Agregación de proteínas

En biología molecular , la agregación de proteínas es un fenómeno en el que las proteínas intrínsecamente desordenadas o mal plegadas se agregan (es decir, se acumulan y se agrupan) ya sea intra o extracelularmente. [1] [2] Los agregados de proteínas se han visto implicados en una amplia variedad de enfermedades conocidas como amiloidosis , incluidas la ELA , el Alzheimer , el Parkinson y la enfermedad priónica . [3] [4]
Después de la síntesis, las proteínas normalmente se pliegan en una conformación tridimensional particular que es la más favorable termodinámicamente : su estado nativo . [5] Este proceso de plegado es impulsado por el efecto hidrofóbico : una tendencia de las partes hidrofóbicas (que temen al agua) de la proteína a protegerse del entorno hidrofílico (que ama el agua) de la célula enterrándose en el interior de la proteína. Por lo tanto, el exterior de una proteína es típicamente hidrofílico, mientras que el interior es típicamente hidrofóbico.
Las estructuras de las proteínas se estabilizan mediante interacciones no covalentes y enlaces disulfuro entre dos residuos de cisteína . Las interacciones no covalentes incluyen interacciones iónicas e interacciones débiles de van der Waals . Las interacciones iónicas se forman entre un anión y un catión y forman puentes salinos que ayudan a estabilizar la proteína. Las interacciones de van der Waals incluyen interacciones no polares (es decir, fuerza de dispersión de London ) e interacciones polares (es decir, enlaces de hidrógeno , enlace dipolo-dipolo ). Estas juegan un papel importante en la estructura secundaria de una proteína , como la formación de una hélice alfa o una lámina beta , y la estructura terciaria. Las interacciones entre los residuos de aminoácidos en una proteína específica son muy importantes en la estructura final de esa proteína.
Cuando hay cambios en las interacciones no covalentes, como puede suceder con un cambio en la secuencia de aminoácidos, la proteína es susceptible a plegarse o desplegarse incorrectamente. En estos casos, si la célula no ayuda a la proteína a plegarse nuevamente, o degrada la proteína desplegada, la proteína desplegada o mal plegada puede agregarse, en la que las porciones hidrofóbicas expuestas de la proteína pueden interactuar con las partes hidrofóbicas expuestas de otras proteínas. [6] [7] Hay tres tipos principales de agregados proteicos que pueden formarse: agregados amorfos, oligómeros y fibrillas amiloides . [8]
Causas
La agregación de proteínas puede producirse por diversas causas. Existen cuatro clases en las que se pueden clasificar estas causas, que se detallan a continuación.
Mutaciones
Las mutaciones que se producen en la secuencia de ADN pueden afectar o no a la secuencia de aminoácidos de la proteína. Cuando la secuencia se ve afectada, un aminoácido diferente puede cambiar las interacciones entre las cadenas laterales que afectan el plegamiento de la proteína. Esto puede dar lugar a que las regiones hidrofóbicas de la proteína queden expuestas y se agrupen con la misma proteína mal plegada o desplegada o con una proteína diferente. [9]
Además de las mutaciones en las propias proteínas afectadas, la agregación de proteínas también podría ser causada indirectamente a través de mutaciones en proteínas en vías reguladoras como la vía de replegamiento ( chaperonas moleculares ) o la vía ubiquitina-proteasoma (ubiquitina ligasas). [10] Las chaperonas ayudan con el replegamiento de proteínas al proporcionar un entorno seguro para que la proteína se pliegue. Las ubiquitina ligasas se dirigen a las proteínas para su degradación a través de la modificación de la ubiquitina. [11]
Problemas con la síntesis de proteínas
La agregación de proteínas puede ser causada por problemas que ocurren durante la transcripción o la traducción . Durante la transcripción, el ADN se copia en ARNm, formando una cadena de pre-ARNm que se somete a un procesamiento de ARN para formar ARNm. [12] Durante la traducción, los ribosomas y el ARNt ayudan a traducir la secuencia de ARNm en una secuencia de aminoácidos. [12] Si surgen problemas durante cualquiera de los pasos, generando una cadena de ARNm incorrecta y/o una secuencia de aminoácidos incorrecta, esto puede hacer que la proteína se pliegue incorrectamente, lo que lleva a la agregación de proteínas. [ cita requerida ]
Estrés ambiental
El estrés ambiental, como las temperaturas y el pH extremos, o el estrés oxidativo , también pueden provocar la agregación de proteínas. [13] Una de estas enfermedades es la crioglobulinemia .
Las temperaturas extremas pueden debilitar y desestabilizar las interacciones no covalentes entre los residuos de aminoácidos. Los valores de pH fuera del rango de pH de la proteína pueden cambiar el estado de protonación de los aminoácidos, lo que puede aumentar o disminuir las interacciones no covalentes. Esto también puede provocar interacciones menos estables y provocar el desdoblamiento de la proteína.
El estrés oxidativo puede ser causado por radicales como las especies reactivas de oxígeno (ROS). Estos radicales inestables pueden atacar los residuos de aminoácidos, lo que lleva a la oxidación de las cadenas laterales (por ejemplo, cadenas laterales aromáticas , cadenas laterales de metionina ) y/o la ruptura de los enlaces polipeptídicos. [14] Esto puede afectar las interacciones no covalentes que mantienen unida correctamente a la proteína, lo que puede causar la desestabilización de la proteína y puede hacer que la proteína se despliegue. [13]
Envejecimiento
Las células tienen mecanismos que pueden replegar o degradar los agregados proteicos. Sin embargo, a medida que las células envejecen, estos mecanismos de control se debilitan y la célula tiene menos capacidad para resolver los agregados. [13]
La hipótesis de que la agregación de proteínas es un proceso causal del envejecimiento es comprobable ahora, ya que se dispone de algunos modelos de envejecimiento retardado. Si el desarrollo de agregados de proteínas fuera un proceso independiente del envejecimiento, ralentizar el envejecimiento no mostraría ningún efecto sobre la tasa de proteotoxicidad a lo largo del tiempo. Sin embargo, si el envejecimiento está asociado con una disminución de la actividad de los mecanismos de protección contra la proteotoxicidad, los modelos de envejecimiento lento mostrarían una agregación y proteotoxicidad reducidas. Para abordar este problema, se han realizado varios ensayos de toxicidad en C. elegans . Estos estudios indicaron que la reducción de la actividad de la señalización de insulina/IGF (IIS), una importante vía reguladora del envejecimiento, protege de la agregación de proteínas tóxicas vinculada a la neurodegeneración. La validez de este enfoque se ha probado y confirmado en mamíferos, ya que la reducción de la actividad de la vía de señalización de IGF-1 protegió a los ratones modelo de Alzheimer de los deterioros conductuales y bioquímicos asociados con la enfermedad. [15]
Localización agregada
Varios estudios han demostrado que las respuestas celulares a la agregación de proteínas están bien reguladas y organizadas. Los agregados de proteínas se localizan en áreas específicas de la célula, y se han realizado investigaciones sobre estas localizaciones en procariotas (E. coli) y eucariotas (levaduras, células de mamíferos). [16] Desde el punto de vista macroscópico, los trazadores de tomografía por emisión de positrones se utilizan para ciertas proteínas mal plegadas. [17] Recientemente, un equipo de investigadores dirigido por el Dr. Alessandro Crimi ha propuesto un método de aprendizaje automático para predecir la deposición futura en el cerebro. [18]
Bacteria
Los agregados de las bacterias terminan asimétricamente en uno de los polos de la célula, el "polo más viejo". Después de que la célula se divide, las células hijas con el polo más viejo obtienen el agregado de proteínas y crecen más lentamente que las células hijas sin el agregado. Esto proporciona un mecanismo de selección natural para reducir los agregados de proteínas en la población bacteriana. [19]
Levadura
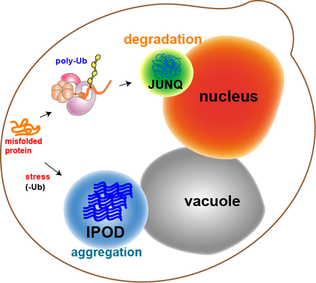
La mayoría de los agregados de proteínas en las células de levadura se repliegan por chaperonas moleculares. Sin embargo, algunos agregados, como las proteínas dañadas por oxidación o las proteínas marcadas para degradación, no se pueden replegar. En cambio, hay dos compartimentos en los que pueden terminar. Los agregados de proteínas pueden localizarse en el compartimento de control de calidad yuxtanuclear ( JUNQ ), que está cerca de la membrana nuclear, o en el depósito de proteína insoluble ( IPOD ), cerca de la vacuola en las células de levadura. [13] Los agregados de proteínas se localizan en JUNQ cuando están ubiquitinados y dirigidos a la degradación. Las proteínas agregadas e insolubles se localizan en IPOD como un depósito más permanente. Hay evidencia de que las proteínas aquí pueden eliminarse por autofagia. [20] Estas dos vías trabajan juntas en el sentido de que las proteínas tienden a llegar al IPOD cuando la vía del proteasoma está sobrecargada. [20]
Células de mamíferos
En las células de los mamíferos, estos agregados de proteínas se denominan "agresomas" y se forman cuando la célula está enferma. Esto se debe a que los agregados tienden a formarse cuando hay proteínas heterólogas presentes en la célula, lo que puede surgir cuando la célula está mutada. Diferentes mutaciones de la misma proteína pueden formar agresomas de diferentes morfologías, que van desde la dispersión difusa de especies solubles hasta grandes puntos, que a su vez tienen diferente patogenicidad. [21] La ligasa de ubiquitina E3 es capaz de reconocer proteínas mal plegadas y ubiquinarlas. HDAC6 puede entonces unirse a la ubiquitina y a la proteína motora dineína para llevar los agregados marcados al centro organizador de microtúbulos ( MTOC ). Allí, se empaquetan juntos en una esfera que rodea el MTOC. Traen chaperonas y proteosomas y activan la autofagia. [22]
Eliminación
Existen dos sistemas principales de control de calidad de proteínas en la célula que son responsables de eliminar los agregados de proteínas. Las proteínas mal plegadas pueden ser plegadas nuevamente por el sistema de bichaperona o degradadas por el sistema de ubiquitina proteosomal o por autofagia. [23]
Replegamiento
El sistema de bi-chaperona utiliza las chaperonas Hsp70 (DnaK-DnaJ-GrpE en E. coli y Ssa1-Ydj1/Sis1-Sse1/Fe1 en levadura) y Hsp100 (ClpB en E. coli y Hsp104 en levadura) para la desagregación y el plegamiento de proteínas. [24]
La Hsp70 interactúa con los agregados proteicos y recluta a la Hsp100. La Hsp70 estabiliza a la Hsp100 activada. Las proteínas Hsp100 tienen bucles de poros aromáticos que se utilizan para la actividad de enhebrado para desenredar polipéptidos individuales. Esta actividad de enhebrado puede iniciarse en el extremo N, el extremo C o en el medio del polipéptido. El polipéptido se transloca a través de la Hsp100 en una serie de pasos, utilizando un ATP en cada paso. [24] El polipéptido se desdobla y luego se le permite volver a plegarse por sí solo o con la ayuda de proteínas de choque térmico. [25]
Degradación
Las proteínas mal plegadas pueden eliminarse a través del sistema ubiquitina-proteasoma ( UPS ). Este consiste en una vía E1-E2-E3 que ubiquina las proteínas para marcarlas para su degradación. En eucariotas, las proteínas son degradadas por el proteasoma 26S. En células de mamíferos, la ligasa E3, proteína carboxi-terminal que interactúa con Hsp70 (CHIP), se dirige a las proteínas unidas a Hsp70. En levadura, las ligasas E3 Doa10 y Hrd1 tienen funciones similares en las proteínas del retículo endoplasmático . [26] A nivel molecular, la tasa de degradación de los agregados varía de proteína a proteína debido a sus diferentes entornos internos y, por lo tanto, a la diferente accesibilidad para las moléculas de proteasa. [27]

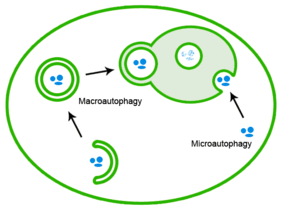
Las proteínas mal plegadas también pueden eliminarse mediante autofagia, en la que los agregados de proteínas se envían al lisosoma. [26]
Toxicidad
Aunque se ha pensado que los agregados proteicos maduros son tóxicos por sí mismos, la evidencia sugiere que, de hecho, son los agregados proteicos inmaduros los más tóxicos. [28] [29] Los parches hidrofóbicos de estos agregados pueden interactuar con otros componentes de la célula y dañarlos. Las hipótesis son que la toxicidad de los agregados proteicos está relacionada con mecanismos de secuestro de componentes celulares, la generación de especies reactivas de oxígeno y la unión a receptores específicos en la membrana o mediante la disrupción de membranas. [30] Se ha utilizado un ensayo cuantitativo para determinar que las especies de mayor peso molecular son responsables de la permeación de la membrana. [31] Se sabe que los agregados proteicos in vitro pueden desestabilizar las bicapas artificiales de fosfolípidos , lo que lleva a la permeabilización de la membrana. [ cita requerida ]
En biofabricación
La agregación de proteínas también es un fenómeno común en el proceso de fabricación biofarmacéutica, que puede suponer riesgos para los pacientes al generar respuestas inmunes adversas. [32]
Véase también
Referencias
- ^ Aguzzi A, O'Connor T (marzo de 2010). "Enfermedades por agregación de proteínas: patogenicidad y perspectivas terapéuticas". Nature Reviews. Drug Discovery . 9 (3): 237–248. doi :10.1038/nrd3050. PMID 20190788. S2CID 5756683.
- ^ Stefani M, Dobson CM (noviembre de 2003). "Agregación de proteínas y toxicidad de los agregados: nuevos conocimientos sobre el plegamiento de proteínas, enfermedades por plegamiento incorrecto y evolución biológica". Journal of Molecular Medicine . 81 (11): 678–699. doi :10.1007/s00109-003-0464-5. PMID 12942175. S2CID 23544974.
- ^ De Felice FG, Vieira MN, Meirelles MN, Morozova-Roche LA, Dobson CM, Ferreira ST (julio de 2004). "Formación de agregados amiloides a partir de lisozima humana y sus variantes asociadas a enfermedades mediante presión hidrostática". FASEB Journal . 18 (10): 1099–1101. doi : 10.1096/fj.03-1072fje . PMID 15155566. S2CID 13647147.
- ^ Tanzi RE, Bertram L (febrero de 2005). "Veinte años de la hipótesis amiloide de la enfermedad de Alzheimer: una perspectiva genética". Cell . 120 (4): 545–555. doi : 10.1016/j.cell.2005.02.008 . PMID 15734686. S2CID 206559875.
- ^ Brüning A, Jückstock J (1 de enero de 2015). "Proteínas mal plegadas: de pequeñas villanas a pequeñas ayudantes en la lucha contra el cáncer". Frontiers in Oncology . 5 : 47. doi : 10.3389/fonc.2015.00047 . PMC 4338749 . PMID 25759792.
- ^ Gething MJ, Sambrook J (enero de 1992). "Plegamiento de proteínas en la célula". Nature . 355 (6355): 33–45. Bibcode :1992Natur.355...33G. doi :10.1038/355033a0. PMID 1731198. S2CID 4330003.
- ^ Roberts CJ (diciembre de 2007). "Cinética de agregación de proteínas no nativas". Biotecnología y bioingeniería . 98 (5): 927–938. doi : 10.1002/bit.21627 . PMID 17705294. S2CID 21787377.
- ^ Cox DL, Nelson MM (2013). Principios de bioquímica de Lehninger . Nueva York: WH Freeman. pág. 143. ISBN 978-1-4292-3414-6.
- ^ Palanikumar L, Karpauskaite L, Al-Sayegh M, Chehade I, Alam M, Hassan S, et al. (junio de 2021). "El inhibidor de amiloide mimético de proteína anula de forma potente la agregación de p53 mutante asociada al cáncer y restaura la función supresora de tumores". Nature Communications . 12 (1): 3962. Bibcode :2021NatCo..12.3962P. doi :10.1038/s41467-021-23985-1. PMC 8233319 . PMID 34172723.
- ^ Berke SJ, Paulson HL (junio de 2003). "Agregación de proteínas y la vía del proteasoma ubiquitina: ganando terreno en la neurodegeneración". Current Opinion in Genetics & Development . 13 (3): 253–261. doi :10.1016/S0959-437X(03)00053-4. PMID 12787787.
- ^ Grillari J, Grillari-Voglauer R, Jansen-Dürr P (2010). Urbani F, Magariños R, Puertas S, Carretero M, Rodriguez V, Liñares R, et al. (eds.). Modificación postraduccional de proteínas celulares por ubiquitina y moléculas similares a la ubiquitina: papel en la senescencia y el envejecimiento celular . Springer Science+Business Media . págs. 172–196. ISBN . 978-1-4419-7002-2.
- ^ ab Weaver RF (2012). Biología molecular . Nueva York: McGraw-Hill. págs. 122-156, 523-600. ISBN 978-0-07-352532-7.
- ^ abcd Tyedmers J, Mogk A, Bukau B (noviembre de 2010). "Estrategias celulares para controlar la agregación de proteínas". Nature Reviews. Molecular Cell Biology . 11 (11): 777–788. doi :10.1038/nrm2993. PMID 20944667. S2CID 22449895.
- ^ Stadtman ER, Levine RL (diciembre de 2003). "Oxidación de aminoácidos libres y residuos de aminoácidos en proteínas mediada por radicales libres". Amino Acids . 25 (3–4): 207–218. doi :10.1007/s00726-003-0011-2. PMID 14661084. S2CID 26844881.
- ^ Morley JF, Brignull HR, Weyers JJ, Morimoto RI (agosto de 2002). "El umbral de agregación de proteínas de expansión de poliglutamina y la toxicidad celular es dinámico y está influenciado por el envejecimiento en Caenorhabditis elegans". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (16): 10417–10422. Bibcode :2002PNAS...9910417M. doi : 10.1073/pnas.152161099 . PMC 124929 . PMID 12122205.
- ^ Coquel AS, Jacob JP, Primet M, Demarez A, Dimiccoli M, Julou T, et al. (abril de 2013). "La localización de la agregación de proteínas en Escherichia coli está regida por la difusión y el efecto de aglomeración macromolecular del nucleoide". PLOS Computational Biology . 9 (4): e1003038. arXiv : 1303.1904 . Bibcode :2013PLSCB...9E3038C. doi : 10.1371/journal.pcbi.1003038 . PMC 3636022 . PMID 23633942.
- ^ Scott JJ (enero de 1987). "La reinervación de los husos musculares del gato por los axones fusimotores esqueléticos". Investigación cerebral . 401 (1): 152–154. doi :10.1016/0006-8993(87)91175-9. PMID 2949798. S2CID 42447184.
- ^ Gherardini L, Zajdel A, Pini L, Crimi A (octubre de 2023). "Predicción de la propagación de proteínas mal plegadas en la enfermedad de Alzheimer mediante aprendizaje automático y modelos de propagación". Corteza cerebral . 33 (24): 11471–11485. doi : 10.1093/cercor/bhad380 . PMC 10724880 . PMID 37833822.
- ^ Bednarska NG, Schymkowitz J, Rousseau F, Van Eldere J (septiembre de 2013). "Agregación de proteínas en bacterias: el delgado límite entre funcionalidad y toxicidad". Microbiología . 159 (Pt 9): 1795–1806. doi : 10.1099/mic.0.069575-0 . PMID 23894132.
- ^ ab Takalo M, Salminen A, Soininen H, Hiltunen M, Haapasalo A (8 de marzo de 2013). "Mecanismos de agregación y degradación de proteínas en enfermedades neurodegenerativas". Revista Estadounidense de Enfermedades Neurodegenerativas . 2 (1): 1–14. PMC 3601466 . PMID 23516262.
- ^ Wan W, Zeng L, Jin W, Chen X, Shen D, Huang Y, et al. (diciembre de 2021). "Una sonda fluorescente solvatocrómica revela heterogeneidad de polaridad tras la agregación de proteínas en las células". Química aplicada . 60 (49): 25865–25871. doi :10.1002/anie.202107943. PMID 34562048. S2CID 237626399.
- ^ Garcia-Mata R, Gao YS, Sztul E (junio de 2002). "Problemas al sacar la basura: problemas agravantes". Traffic . 3 (6): 388–396. doi : 10.1034/j.1600-0854.2002.30602.x . PMID 12010457. S2CID 305786.
- ^ Gregersen N, Bolund L, Bross P (octubre de 2005). "Plegamiento incorrecto de proteínas, agregación y degradación en enfermedades". Biotecnología molecular . 31 (2): 141–150. doi :10.1385/MB:31:2:141. PMID 16170215. S2CID 36403914.
- ^ ab Mogk A, Kummer E, Bukau B (1 de enero de 2015). "Cooperación de las máquinas chaperonas Hsp70 y Hsp100 en la desagregación de proteínas". Frontiers in Molecular Biosciences . 2 : 22. doi : 10.3389/fmolb.2015.00022 . PMC 4436881 . PMID 26042222.
- ^ Liberek K, Lewandowska A, Zietkiewicz S (enero de 2008). "Chaperonas en el control de la desagregación de proteínas". The EMBO Journal . 27 (2): 328–335. doi :10.1038/sj.emboj.7601970. PMC 2234349 . PMID 18216875.
- ^ ab Chen B, Retzlaff M, Roos T, Frydman J (agosto de 2011). "Estrategias celulares de control de calidad de proteínas". Cold Spring Harbor Perspectives in Biology . 3 (8): a004374. doi :10.1101/cshperspect.a004374. PMC 3140689 . PMID 21746797.
- ^ Wan W, Zeng L, Jin W, Chen X, Shen D, Huang Y, et al. (diciembre de 2021). "Una sonda fluorescente solvatocrómica revela heterogeneidad de polaridad tras la agregación de proteínas en las células". Química aplicada . 60 (49): 25865–25871. doi :10.1002/anie.202107943. PMID 34562048. S2CID 237626399.
- ^ Zhu YJ, Lin H, Lal R (junio de 2000). "La proteína beta amiloide (1-40) fresca y no fibrilar induce una rápida degeneración celular en fibroblastos humanos envejecidos: evidencia de toxicidad celular mediada por el canal AbetaP". FASEB Journal . 14 (9): 1244–1254. doi : 10.1096/fasebj.14.9.1244 . PMID 10834946. S2CID 42263619.
- ^ Nilsberth C, Westlind-Danielsson A, Eckman CB, Condron MM, Axelman K, Forsell C, et al. (septiembre de 2001). "La mutación 'ártica' de APP (E693G) causa la enfermedad de Alzheimer al aumentar la formación de protofibrillas Abeta". Nature Neuroscience . 4 (9): 887–893. doi :10.1038/nn0901-887. PMID 11528419. S2CID 13516479.
- ^ Soto C (enero de 2003). "Descubriendo el papel del plegamiento incorrecto de proteínas en enfermedades neurodegenerativas". Nature Reviews. Neuroscience . 4 (1): 49–60. doi :10.1038/nrn1007. PMID 12511861. S2CID 205499427.
- ^ Flagmeier P, De S, Wirthensohn DC, Lee SF, Vincke C, Muyldermans S, et al. (junio de 2017). "Medición ultrasensible del influjo de Ca2+ en vesículas lipídicas inducido por agregados proteicos". Angewandte Chemie . 56 (27): 7750–7754. doi :10.1002/anie.201700966. PMC 5615231 . PMID 28474754.
- ^ Vázquez-Rey M, Lang DA (julio 2011). "Agregados en procesos de fabricación de anticuerpos monoclonales". Biotecnología y Bioingeniería . 108 (7): 1494–1508. doi :10.1002/bit.23155. PMID 21480193. S2CID 33285577.