Virus del Ébola en Zaire
| Virus del Ébola en Zaire | |
|---|---|
 | |
| Micrografía electrónica de barrido coloreada de partículas del virus del Ébola (verde) encontradas tanto como partículas extracelulares como partículas en ciernes de una célula renal de mono verde africano infectada crónicamente (azul); aumento de 20 000x | |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Virus de la ortiga |
| Filo: | Negarnaviricota |
| Clase: | Monjiviricetes |
| Orden: | Mononegavirus |
| Familia: | Filovirus |
| Género: | Virus del Ébola |
| Especies: | Virus del Ébola en Zaire |
El virus del Ébola de Zaire , más comúnmente conocido como virus del Ébola ( / i ˈ b oʊ l ə , ɪ - / ; EBOV ), es una de las seis especies conocidas dentro del género Ebolavirus . [1] Cuatro de los seis virus del Ébola conocidos, incluido el EBOV, causan una fiebre hemorrágica grave y a menudo mortal en humanos y otros mamíferos , conocida como enfermedad del virus del Ébola (EVE). El virus del Ébola ha causado la mayoría de las muertes humanas por EVE y fue la causa de la epidemia de 2013-2016 en África occidental , [2] que resultó en al menos 28.646 casos sospechosos y 11.323 muertes confirmadas. [3] [4]
El virus del Ébola y su género fueron nombrados originalmente por Zaire (ahora la República Democrática del Congo ), el país donde fue descrito por primera vez , [1] y al principio se sospechó que era una nueva "cepa" del virus Marburg estrechamente relacionado . [5] [6] El virus fue renombrado "virus del Ébola" en 2010 para evitar confusiones. El virus del Ébola es el único miembro de la especie Zaire ebolavirus , que está asignada al género Ebolavirus , familia Filoviridae , orden Mononegavirales . Los miembros de la especie se llaman Zaire ebolavirus. [1] [7] Se cree que el reservorio natural del virus del Ébola son los murciélagos , particularmente los murciélagos frugívoros , [8] y se transmite principalmente entre humanos y de animales a humanos a través de fluidos corporales . [9]
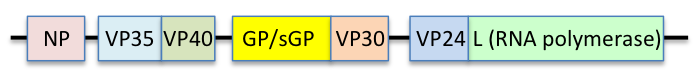
El genoma del virus del Ébola es un ARN monocatenario de aproximadamente 19.000 nucleótidos de longitud. Codifica siete proteínas estructurales : nucleoproteína (NP), cofactor de la polimerasa (VP35), (VP40), GP, activador de la transcripción (VP30), VP24 y ARN polimerasa dependiente de ARN (L). [10]
Debido a su alta tasa de mortalidad (hasta 83 a 90 por ciento), [11] [12] el EBOV también está catalogado como un agente selecto , un patógeno del Grupo de Riesgo 4 de la Organización Mundial de la Salud (que requiere contención equivalente al Nivel 4 de Bioseguridad ), un patógeno prioritario de Categoría A de los Institutos Nacionales de Salud de EE. UU. / Instituto Nacional de Alergias y Enfermedades Infecciosas , un agente de bioterrorismo de Categoría A de los Centros para el Control y la Prevención de Enfermedades de EE. UU. y un agente biológico para el control de exportaciones por el Grupo de Australia . [ cita requerida ]
Estructura

.jpg/440px-Ebola_virus_Makona_from_West_African_Epidemic_(43234028254).jpg)
El virus del Ébola transporta un genoma de ARN de sentido negativo en viriones que son cilíndricos/tubulares y contienen componentes de envoltura, matriz y nucleocápside virales . Los cilindros generales tienen generalmente aproximadamente 80 nm de diámetro y tienen una glicoproteína (GP) codificada por el virus que se proyecta como espigas de 7 a 10 nm de largo desde su superficie de bicapa lipídica. [13] Los cilindros tienen una longitud variable, típicamente 800 nm, pero a veces hasta 1000 nm de largo. La envoltura viral externa del virión se deriva de la gemación de dominios de la membrana de la célula huésped en la que se han insertado las espigas de GP durante su biosíntesis. Las moléculas individuales de GP aparecen con espaciamientos de aproximadamente 10 nm. Las proteínas virales VP40 y VP24 se encuentran entre la envoltura y la nucleocápside (ver a continuación), en el espacio de la matriz . [14] En el centro de la estructura del virión se encuentra la nucleocápside , que está compuesta por una serie de proteínas virales unidas a un ARN lineal de sentido negativo de 18-19 kb sin poliadenilación 3' ni protección 5' (ver a continuación); el ARN está enrollado helicoidalmente y forma un complejo con las proteínas NP, VP35, VP30 y L; esta hélice tiene un diámetro de 80 nm. [15] [16] [17]
La forma general de los viriones después de la purificación y visualización (por ejemplo, por ultracentrifugación y microscopía electrónica , respectivamente) varía considerablemente; los cilindros simples son mucho menos frecuentes que las estructuras que muestran dirección invertida, ramas y bucles (por ejemplo, formas de U, cayado de pastor , 9 o perno de ojo , u otras apariencias circulares/enrolladas), cuyo origen puede estar en las técnicas de laboratorio aplicadas. [18] [19] La característica estructura "filiforme" es, sin embargo, una característica morfológica más general de los filovirus (junto con su envoltura viral decorada con GP, nucleocápside de ARN, etc.). [18]
Genoma
Cada virión contiene una molécula de ARN monocatenario lineal de sentido negativo, de 18.959 a 18.961 nucleótidos de longitud. [20] El extremo 3′ no está poliadenilado y el extremo 5′ no está cubierto. Este genoma viral codifica siete proteínas estructurales y una proteína no estructural. El orden de los genes es 3′ – líder – NP – VP35 – VP40 – GP/sGP – VP30 – VP24 – L – trailer – 5′; siendo el líder y el trailer regiones no transcritas, que llevan señales importantes para controlar la transcripción, replicación y empaquetamiento de los genomas virales en nuevos viriones. Se han identificado secciones de los genes NP, VP35 y L de los filovirus como endógenas en los genomas de varios grupos de pequeños mamíferos. [21] [22] [23]
Se encontró que 472 nucleótidos del extremo 3' y 731 nucleótidos del extremo 5' son suficientes para la replicación de un "minigenoma" viral, aunque no suficientes para la infección. [18] La secuenciación del virus de 78 pacientes con enfermedad confirmada por el virus del Ébola, que representan más del 70% de los casos diagnosticados en Sierra Leona desde fines de mayo hasta mediados de junio de 2014, [24] [25] proporcionó evidencia de que el brote de 2014 ya no estaba siendo alimentado por nuevos contactos con su reservorio natural. Usando tecnología de secuenciación de tercera generación , los investigadores pudieron secuenciar muestras tan rápido como 48 horas. [26] Al igual que otros virus de ARN, [24] el virus del Ébola muta rápidamente, tanto dentro de una persona durante la progresión de la enfermedad como en el reservorio entre la población humana local. [25] La tasa de mutación observada de 2.0 x 10 −3 sustituciones por sitio por año es tan rápida como la de la influenza estacional . [27]
| Símbolo | Nombre | Protección unificada | Función |
|---|---|---|---|
| notario público | Nucleoproteína | P18272 | Envuelve el genoma para protegerlo de las nucleasas y la inmunidad innata. |
| VP35 | Cofactor de la polimerasa VP35 | Q05127 | Cofactor de la polimerasa; suprime la inmunidad innata al unirse al ARN. |
| VP40 | Proteína de matriz VP40 | Q05128 | Matriz. |
| Médico de cabecera | Glicoproteína de envoltura | Q05320 | La furina del huésped la divide en GP1/2 para formar una envoltura con púas. También produce GP desprendido como señuelo. |
| sGP | Glicoproteína prepequeña/secretada | P60170 | Comparte ORF con GP. La furina del huésped lo escinde en sGP (antiinflamatorio) y delta-péptido (viroporina). |
| ssGP | Glicoproteína secretada superpequeña | Q9YMG2 | Comparte ORF con GP; creado mediante edición de ARNm. Función desconocida. |
| VP30 | Proteína hexamérica con dedos de zinc VP30 | Q05323 | Activador transcripcional. |
| VP24 | Proteína asociada a la membrana VP24 | Q05322 | Bloquea la señalización de IFN-alfa/beta y IFN-gamma. |
| yo | ARN polimerasa L dirigida por ARN | Q05318 | Replicasa de ARN . |
Entrada

Hay dos candidatos para las proteínas de entrada a la célula huésped. La primera es una proteína transportadora de colesterol, la Niemann–Pick C1 ( NPC1 ) codificada por el huésped, que parece ser esencial para la entrada de los viriones del Ébola en la célula huésped y para su replicación final. [28] [29] En un estudio, los ratones a los que se les había eliminado una copia del gen NPC1 mostraron una tasa de supervivencia del 80 por ciento quince días después de la exposición al virus del Ébola adaptado al ratón, mientras que solo el 10 por ciento de los ratones no modificados sobrevivieron durante tanto tiempo. [28] En otro estudio, se demostró que pequeñas moléculas inhiben la infección por el virus del Ébola al evitar que la glicoproteína de la envoltura viral (GP) se una a NPC1. [29] [30] Por lo tanto, se demostró que NPC1 es fundamental para la entrada de este filovirus , porque media la infección uniéndose directamente a la GP viral. [29]
Cuando las células de individuos de tipo C de Niemann-Pick que carecían de este transportador se expusieron al virus del Ébola en el laboratorio, las células sobrevivieron y parecieron inmunes al virus, lo que indica además que el Ébola depende del NPC1 para entrar en las células; [28] Se conjeturó que las mutaciones en el gen NPC1 en humanos eran un posible modo de hacer que algunos individuos fueran resistentes a esta enfermedad viral mortal. Los mismos estudios describieron resultados similares con respecto al papel del NPC1 en la entrada del virus de Marburgo , un filovirus relacionado . [28] Un estudio adicional también ha presentado evidencia de que el NPC1 es el receptor crítico que media la infección del Ébola a través de su unión directa al GP viral, y que es el segundo dominio "lisosomal" del NPC1 el que media esta unión. [31]
El segundo candidato es TIM-1 (también conocido como HAVCR1 ). [32] Se ha demostrado que TIM-1 se une al dominio de unión del receptor de la glicoproteína EBOV, para aumentar la receptividad de las células Vero . Silenciar su efecto con ARNi impidió la infección de las células Vero . TIM1 se expresa en tejidos que se sabe que se ven gravemente afectados por la lisis de EBOV (tráquea, córnea y conjuntiva). Un anticuerpo monoclonal contra el dominio IgV de TIM-1, ARD5, bloqueó la unión y la infección de EBOV. En conjunto, estos estudios sugieren que NPC1 y TIM-1 pueden ser objetivos terapéuticos potenciales para un fármaco antiviral contra el ébola y como base para un ensayo de diagnóstico rápido de campo. [ cita requerida ]
Replicación

Al ser acelulares, los virus como el ébola no se replican a través de ningún tipo de división celular; en cambio, utilizan una combinación de enzimas codificadas por el huésped y por el virus, junto con las estructuras de la célula huésped, para producir múltiples copias de sí mismos. Estas luego se autoensamblan en estructuras macromoleculares virales en la célula huésped. [33] El virus completa una serie de pasos cuando infecta cada célula individual. El virus comienza su ataque uniéndose a los receptores del huésped a través del peplómero de superficie de la glicoproteína (GP) y es endocitado en macropinosomas en la célula huésped. [34] Para penetrar en la célula, la membrana viral se fusiona con la membrana de la vesícula y la nucleocápside se libera en el citoplasma . El ssRNA genómico de sentido negativo encapsidado se utiliza como plantilla para la síntesis (3'–5') de ARNm poliadenilados, monocistrónicos y, utilizando los ribosomas de la célula huésped, las moléculas de ARNt, etc., el ARNm se traduce en proteínas virales individuales. [35] [36] [37]
Estas proteínas virales se procesan: un precursor de la glicoproteína (GP0) se escinde en GP1 y GP2, que luego se glucosilan intensamente utilizando enzimas celulares y sustratos. Estas dos moléculas se ensamblan, primero en heterodímeros y luego en trímeros para dar los peplómeros de superficie. El precursor de la glicoproteína secretada (sGP) se escinde en sGP y péptido delta, los cuales se liberan de la célula. A medida que aumentan los niveles de proteína viral, se produce un cambio de traducción a replicación. Utilizando el ARN genómico de sentido negativo como plantilla, se sintetiza un +ssRNA complementario; este se utiliza luego como plantilla para la síntesis de nuevo (-)ssRNA genómico, que se encapsida rápidamente. Las nucleocápsides y las proteínas de la envoltura recién formadas se asocian en la membrana plasmática de la célula huésped; se produce la gemación , destruyendo la célula. [ cita requerida ]
Ecología
El virus del Ébola es un patógeno zoonótico . Se ha informado de que los huéspedes intermediarios son "diversas especies de murciélagos frugívoros... en toda África central y subsahariana". Se ha detectado evidencia de infección en murciélagos a través de medios moleculares y serológicos. Sin embargo, no se han aislado virus del Ébola en murciélagos. [8] [38] Los huéspedes finales son los humanos y los grandes simios, infectados a través del contacto con murciélagos o a través de otros huéspedes finales. Se ha informado de que los cerdos en Filipinas están infectados con el virus Reston , por lo que pueden existir otros huéspedes provisionales o amplificadores. [38] Los brotes del virus del Ébola tienden a ocurrir cuando las temperaturas son más bajas y la humedad es más alta de lo habitual en África. [39] Incluso después de que una persona se recupera de la fase aguda de la enfermedad, el virus del Ébola sobrevive durante meses en ciertos órganos como los ojos y los testículos. [40]
Enfermedad por el virus del Ébola
El virus del Ébola del Zaire es uno de los cuatro virus del Ébola que se sabe que causan enfermedades en los seres humanos. Tiene la tasa de letalidad más alta de estos virus del Ébola, con un promedio del 83 por ciento desde los primeros brotes en 1976, aunque se registró una tasa de letalidad de hasta el 90 por ciento en un brote en la República del Congo entre diciembre de 2002 y abril de 2003. También ha habido más brotes de virus del Ébola del Zaire que de cualquier otro virus del Ébola. El primer brote ocurrió el 26 de agosto de 1976 en Yambuku . [41] El primer caso registrado fue Mabalo Lokela, un maestro de escuela de 44 años. Los síntomas se parecían a la malaria , y los pacientes posteriores recibieron quinina . La transmisión se ha atribuido a la reutilización de agujas no esterilizadas y al contacto personal cercano, fluidos corporales y lugares que la persona ha tocado. Durante el brote de ébola de 1976 en Zaire , Ngoy Mushola viajó de Bumba a Yambuku , donde registró la primera descripción clínica de la enfermedad en su diario:
La enfermedad se caracteriza por una temperatura alta de alrededor de 39°C, hematemesis , diarrea con sangre, dolor abdominal retroesternal, postración con articulaciones "pesadas" y muerte de rápida evolución después de una media de tres días. [42]
Desde la primera descripción clínica registrada de la enfermedad en 1976 en Zaire, el reciente brote de ébola que comenzó en marzo de 2014 alcanzó proporciones epidémicas y, hasta enero de 2015, ha matado a más de 8000 personas. Este brote se centró en África occidental, una zona que no había sido afectada anteriormente por la enfermedad. El saldo fue especialmente grave en tres países: Guinea, Liberia y Sierra Leona. También se notificaron algunos casos en países fuera de África occidental, todos relacionados con viajeros internacionales que estuvieron expuestos en las regiones más afectadas y que luego mostraron síntomas de fiebre del ébola después de llegar a sus destinos. [43]
La gravedad de la enfermedad en los seres humanos varía ampliamente, desde una rápida fatalidad hasta una enfermedad leve o incluso una respuesta asintomática. [44] Los estudios de brotes a finales del siglo XX no lograron encontrar una correlación entre la gravedad de la enfermedad y la naturaleza genética del virus. Por lo tanto, se sospechó que la variabilidad en la gravedad de la enfermedad se correlacionaba con diferencias genéticas en las víctimas. Esto ha sido difícil de estudiar en modelos animales que responden al virus con fiebre hemorrágica de manera similar a los seres humanos, porque los modelos de ratón típicos no responden de esa manera y no se dispone fácilmente de la gran cantidad necesaria de sujetos de prueba adecuados. A finales de octubre de 2014, una publicación informó sobre un estudio de la respuesta a una cepa de ébolavirus de Zaire adaptada al ratón presentada por una población genéticamente diversa de ratones que se crió para tener una gama de respuestas al virus que incluye la fatalidad por fiebre hemorrágica. [45]
Vacuna
En diciembre de 2016, un estudio determinó que la vacuna VSV-EBOV tenía una eficacia del 70 al 100 % contra el virus del ébola de Zaire (no el virus del ébola de Sudán ), lo que la convirtió en la primera vacuna contra la enfermedad. [46] [47] La Administración de Alimentos y Medicamentos de los EE. UU. aprobó la vacuna VSV-EBOV en diciembre de 2019. [48]
Historia y nomenclatura

El virus del Ébola fue identificado por primera vez como una posible nueva "cepa" del virus de Marburgo en 1976. [5] [6] [49] El Comité Internacional de Taxonomía de Virus (ICTV) identifica al virus del Ébola como la especie Zaire ebolavirus , que forma parte del género Ebolavirus , familia Filoviridae , orden Mononegavirales . El nombre "virus del Ébola" se deriva del río Ébola (un río que al principio se pensó que estaba muy cerca del área de la República Democrática del Congo , anteriormente llamada Zaire , donde ocurrió el brote del virus del Ébola de Zaire en 1976 ) y del sufijo taxonómico virus . [1] [5] [6] [50]
En 1998, el nombre del virus fue cambiado a "Zaire Ebola virus" [51] [52] y en 2002 a la especie Zaire ebolavirus . [53] [54] Sin embargo, la mayoría de los artículos científicos continuaron haciendo referencia al "virus del Ébola" o usaron los términos "virus del Ébola" y " Zaire ebolavirus " en paralelo. En consecuencia, en 2010, un grupo de investigadores recomendó que se adoptara el nombre "virus del Ébola" para una subclasificación dentro de la especie Zaire ebolavirus , con la abreviatura correspondiente EBOV. [1] Las abreviaturas anteriores para el virus eran EBOV-Z (para "virus del Ébola Zaire") y ZEBOV (para "virus del Ébola Zaire" o " Zaire ebolavirus "). En 2011, el ICTV rechazó explícitamente una propuesta (2010.010bV) para reconocer este nombre, ya que el ICTV no designa nombres para subtipos, variantes, cepas u otras agrupaciones a nivel de subespecie. [55] En la actualidad, el ICTV no reconoce oficialmente al "virus del Ébola" como un rango taxonómico, sino que continúa utilizando y recomendando solo la designación de especie Zaire ebolavirus . [56] El prototipo del virus del Ébola, variante Mayinga (EBOV/May), recibió su nombre de Mayinga N'Seka, una enfermera que murió durante el brote de Zaire de 1976. [1] [57] [58]
El nombre Zaire ebolavirus se deriva de Zaire y del sufijo taxonómico ebolavirus (que denota una especie de ebolavirus y hace referencia al río Ébola ). [1] De acuerdo con las reglas para la denominación de taxones establecidas por el Comité Internacional de Taxonomía de Virus (ICTV), el nombre Zaire ebolavirus siempre debe escribirse con mayúscula inicial , en cursiva y debe ir precedido de la palabra "especie". Los nombres de sus miembros (Zaire ebolaviruses) deben escribirse con mayúscula inicial, no en cursiva y usarse sin artículos . [1]
Criterios de inclusión del virus
Un virus del género Ebolavirus es miembro de la especie Zaire ebolavirus si: [1]
- Es endémica en la República Democrática del Congo , Gabón o la República del Congo.
- Tiene un genoma con dos o tres superposiciones de genes ( VP35 / VP40 , GP / VP30 , VP24 / L )
- Tiene una secuencia genómica que difiere del virus tipo EBOV/May en menos del 30%.
Evolución
El virus del Ébola de Zaire se separó de sus ancestros entre 1960 y 1976. [59] La diversidad genética del virus del Ébola se mantuvo constante antes de 1900. [59] [60] Luego, alrededor de la década de 1960, probablemente debido al cambio climático o a las actividades humanas, la diversidad genética del virus disminuyó rápidamente y la mayoría de los linajes se extinguieron. [60] A medida que disminuye el número de huéspedes susceptibles, también lo hace el tamaño efectivo de la población y su diversidad genética. Este efecto de cuello de botella genético tiene implicaciones para la capacidad de la especie de causar la enfermedad del virus del Ébola en huéspedes humanos. [ cita requerida ]
Es probable que entre 1996 y 2001 se produjera un evento de recombinación entre linajes del virus del Ébola de Zaire en simios salvajes, lo que dio lugar a virus de progenie recombinantes. [61] Estos virus recombinantes parecen haber sido responsables de una serie de brotes entre humanos en África Central entre 2001 y 2003. [61]
El virus del Ébola de Zaire , variante Makona, causó el brote de 2014 en África occidental. [62] El brote se caracterizó por ser el caso más prolongado de transmisión de persona a persona de la especie viral. [62] En ese momento se observaron presiones para adaptarse al huésped humano, sin embargo, no se observaron cambios fenotípicos en el virus (como mayor transmisión o mayor evasión inmunológica por parte del virus). [ cita requerida ]
En la literatura
- La novela policial de 2008 de Alex Kava , Exposed , se centra en el virus como arma preferida de un asesino en serie. [63]
- Los libros de William Close Ébola: A Documentary Novel of Its First Explosion ( 1995) y Ébola: Through the Eyes of the People ( 2002) se centraron en las reacciones de los individuos al brote de ébola de 1976 en Zaire. [64] [65] [66] [67]
- La zona caliente: una historia real aterradora : un libro superventas de 1994 escrito por Richard Preston sobre el virus del Ébola y otros virus relacionados, que incluye un relato del brote de un virus del Ébola en primates alojados en un centro de cuarentena en Reston, Virginia, EE. UU. [68]
- La novela de Tom Clancy de 1996, Executive Orders , trata sobre un ataque terrorista en Oriente Medio contra los Estados Unidos utilizando una forma aérea de un virus mortal del Ébola llamado "Ébola Mayinga". [69] [70]
Referencias
- ^ abcdefghi Kuhn JH, Becker S, Ebihara H, Geisbert TW, Johnson KM, Kawaoka Y, Lipkin WI, Negredo AI, et al. (2010). "Propuesta para una taxonomía revisada de la familia Filoviridae: clasificación, nombres de taxones y virus, y abreviaturas de virus". Archivos de Virología . 155 (12): 2083–2103. doi :10.1007/s00705-010-0814-x. PMC 3074192 . PMID 21046175.
- ^ Na, Woonsung; Park, Nanuri; Yeom, Minju; Song, Daesub (4 de diciembre de 2016). "Brote de ébola en África occidental 2014: ¿qué está pasando con el virus del ébola?". Clinical and Experimental Vaccine Research . 4 (1): 17–22. doi :10.7774/cevr.2015.4.1.17. ISSN 2287-3651. PMC 4313106 . PMID 25648530.
- ^ Enfermedad por el virus del Ébola (Informe). Organización Mundial de la Salud . Consultado el 6 de junio de 2019 .
- ^ "Brote de enfermedad por el virus del Ébola". Organización Mundial de la Salud . Archivado desde el original el 23 de marzo de 2021. Consultado el 4 de diciembre de 2016 .
- ^ abc Pattyn S, Jacob W, van der Groen G, Piot P, Courteille G (1977). "Aislamiento de virus similar a Marburg de un caso de fiebre hemorrágica en Zaire". Lanceta . 309 (8011): 573–574. doi :10.1016/s0140-6736(77)92002-5. PMID 65663. S2CID 33060636.
- ^ abc Bowen ETW, Lloyd G, Harris WJ, Platt GS, Baskerville A, Vella EE (1977). "Fiebre hemorrágica viral en el sur de Sudán y el norte de Zaire. Estudios preliminares sobre el agente etiológico". Lancet . 309 (8011): 571–573. doi :10.1016/s0140-6736(77)92001-3. PMID 65662. S2CID 3092094.
- ^ OMS. «Enfermedad por el virus del Ébola». Archivado desde el original el 14 de diciembre de 2014. Consultado el 5 de octubre de 2020 .
- ^ ab Quammen, David (30 de diciembre de 2014). «Un nuevo estudio sugiere que un murciélago que se alimenta de insectos podría ser el origen del brote de ébola». news.nationalgeographic.com . Washington, DC: National Geographic Society . Archivado desde el original el 31 de diciembre de 2014 . Consultado el 30 de diciembre de 2014 .
- ^ Angier, Natalie (27 de octubre de 2014). «Asesinos en una celda pero sueltos: el ébola y el vasto universo viral». New York Times . Archivado desde el original el 12 de noviembre de 2020. Consultado el 27 de octubre de 2014 .
- ^ Nanbo, Asuka; Watanabe, Shinji; Halfmann, Peter; Kawaoka, Yoshihiro (4 de febrero de 2013). "La dinámica de distribución espacio-temporal de las proteínas y el ARN del virus del Ébola en células infectadas". Informes científicos . 3 (1): 1206. Código bibliográfico : 2013NatSR...3E1206N. doi :10.1038/srep01206. PMC 3563031 . PMID 23383374.
- ^ "Hoja informativa N°103 sobre la enfermedad por el virus del Ébola". Organización Mundial de la Salud . Marzo de 2014. Archivado desde el original el 14 de diciembre de 2014. Consultado el 12 de abril de 2014 .
- ^ Fauquet CM, Mayo MA, Maniloff J, Desselberger U, Ball LA, eds. (2005). Taxonomía de virus: octavo informe del Comité Internacional de Taxonomía de Virus. Oxford: Elsevier/Academic Press. pág. 648. ISBN 978-0080575483Archivado desde el original el 14 de enero de 2023 . Consultado el 7 de febrero de 2016 .
- ^ Klenk, H.-D.; Feldmann, H., eds. (2004). Virus del Ébola y Marburgo: biología molecular y celular . Wymondham, Norfolk, Reino Unido: Horizon Bioscience. pág. 28. ISBN. 978-0-9545232-3-7.
- ^ Feldmann, HK (1993). "Biología molecular y evolución de los filovirus". Agentes no convencionales y virus no clasificados . Archivos de Virología. Vol. 7. Viena: Springer. págs. 81-100. doi :10.1007/978-3-7091-9300-6_8. ISBN . 978-3211824801. ISSN 0939-1983. PMID 8219816.
{{cite book}}:|work=ignorado ( ayuda ) - ^ Lee, Jeffrey E; Saphire, Erica Ollmann (2009). "Estructura de la glicoproteína del virus del Ébola y mecanismo de entrada". Future Virology . 4 (6): 621–635. doi :10.2217/fvl.09.56. ISSN 1746-0794. PMC 2829775 . PMID 20198110.
- ^ Falasca L, Agrati C, Petrosillo N, Di Caro A, Capobianchi MR, Ippolito G, Piacentini M (4 de diciembre de 2016). "Mecanismos moleculares de la patogénesis del virus del Ébola: enfoque en la muerte celular". Muerte celular y diferenciación . 22 (8): 1250–1259. doi :10.1038/cdd.2015.67. ISSN 1350-9047. PMC 4495366 . PMID 26024394.
- ^ Swetha, Rayapadi G.; Ramaiah, Sudha; Anbarasu, Anand; Sekar, Kanagaraj (2016). "Base de datos sobre el virus del Ébola: recurso de información sobre genes y proteínas para los virus del Ébola". Avances en Bioinformática . 2016 : 1673284. doi : 10.1155/2016/1673284 . ISSN 1687-8027. PMC 4848411 . PMID 27190508.
- ^ abc Klenk, H.-D.; Feldmann, H., eds. (2004). Virus del Ébola y Marburgo: biología molecular y celular . Horizon Bioscience. ISBN 978-1904933496.[ página necesaria ]
- ^ Hillman, H. (1991). El caso de nuevos paradigmas en biología celular y en neurobiología . Edwin Mellen Press.
- ^ Aislado del virus del Ébola de Zaire H.sapiens-wt/GIN/2014/Makona-Kissidougou-C15, genoma completo Archivado el 24 de enero de 2018 en Wayback Machine , GenBank
- ^ Taylor D, Leach R, Bruenn J (2010). "Los filovirus son antiguos y están integrados en los genomas de los mamíferos". BMC Evolutionary Biology . 10 (1): 193. Bibcode :2010BMCEE..10..193T. doi : 10.1186/1471-2148-10-193 . PMC 2906475 . PMID 20569424.
- ^ Belyi, VA; Levine, AJ; Skalka, AM (2010). Buchmeier, Michael J. (ed.). "Herencia inesperada: integraciones múltiples de secuencias antiguas de bornavirus y ébolavirus/marburgvirus en genomas de vertebrados". PLOS Pathogens . 6 (7): e1001030. doi : 10.1371/journal.ppat.1001030 . PMC 2912400 . PMID 20686665.
- ^ Taylor DJ, Ballinger MJ, Zhan JJ, Hanzly LE, Bruenn JA (2014). "Evidencia de que los virus del Ébola y los cuevavirus se han ido diferenciando de los virus de Marburgo desde el Mioceno". PeerJ . 2 : e556. doi : 10.7717/peerj.556 . PMC 4157239 . PMID 25237605.
- ^ de Richard Preston (27 de octubre de 2014). «The Ebola Wars». The New Yorker . Nueva York: Condé Nast . Archivado desde el original el 25 de enero de 2021. Consultado el 20 de octubre de 2014 .
- ^ ab Gire, Stephen K.; et al. (2014). "La vigilancia genómica dilucida el origen y la transmisión del virus del Ébola durante el brote de 2014". Science . 345 (6202): 1369–1372. Bibcode :2014Sci...345.1369G. doi :10.1126/science.1259657. PMC 4431643 . PMID 25214632.
- ^ Check Hayden, Erika (5 de mayo de 2015). "Un secuenciador de ADN de tamaño reducido impresiona a los primeros usuarios". Nature . 521 (7550): 15–16. Bibcode :2015Natur.521...15C. doi : 10.1038/521015a . ISSN 0028-0836. PMID 25951262.
- ^ Jenkins GM, Rambaut A, Pybus OG, Holmes EC (2002). "Tasas de evolución molecular en virus de ARN: un análisis filogenético cuantitativo". Journal of Molecular Evolution . 54 (2): 156–165. Bibcode :2002JMolE..54..156J. doi :10.1007/s00239-001-0064-3. PMID 11821909. S2CID 20759532.
- ^ abcd Carette JE, Raaben M, Wong AC, Herbert AS, Obernosterer G, Mulherkar N, Kuehne AI, Kranzusch PJ, Griffin AM, Ruthel G, Dal Cin P, Dye JM, Whelan SP, Chandran K, Brummelkamp TR (septiembre de 2011). "La entrada del virus del Ébola requiere el transportador de colesterol Niemann-Pick C1". Nature . 477 (7364): 340–343. Bibcode :2011Natur.477..340C. doi :10.1038/nature10348. PMC 3175325 . PMID 21866103.
- Amanda Schaffer (16 de enero de 2012). «Una proteína clave podría abrirle las puertas al virus del Ébola» . The New York Times . Archivado desde el original el 20 de febrero de 2022. Consultado el 26 de febrero de 2017 .
- ^ abc Côté M, Misasi J, Ren T, Bruchez A, Lee K, Filone CM, Hensley L, Li Q, Ory D, Chandran K, Cunningham J (septiembre de 2011). "Los inhibidores de moléculas pequeñas revelan que Niemann-Pick C1 es esencial para la infección por el virus del Ébola". Nature . 477 (7364): 344–348. Bibcode :2011Natur.477..344C. doi :10.1038/nature10380. PMC 3230319 . PMID 21866101.
- Amanda Schaffer (16 de enero de 2012). «Una proteína clave podría abrirle las puertas al virus del Ébola» . The New York Times . Archivado desde el original el 20 de febrero de 2022. Consultado el 26 de febrero de 2017 .
- ^ Flemming A (octubre de 2011). "El talón de Aquiles de la entrada del virus del Ébola". Nat Rev Drug Discov . 10 (10): 731. doi : 10.1038/nrd3568 . PMID 21959282. S2CID 26888076.
- ^ Miller EH, Obernosterer G, Raaben M, Herbert AS, Deffieu MS, Krishnan A, Ndungo E, Sandesara RG, Carette JE, Kuehne AI, Ruthel G, Pfeffer SR, Dye JM, Whelan SP, Brummelkamp TR, Chandran K (marzo) 2012). "La entrada del virus del Ébola requiere el reconocimiento de un receptor intracelular programado por el huésped". Revista EMBO . 31 (8): 1947-1960. doi :10.1038/emboj.2012.53. PMC 3343336 . PMID 22395071.
- ^ Kondratowicz AS, Lennemann NJ, Sinn PL, et al. (mayo de 2011). "El dominio 1 de inmunoglobulina y mucina de células T (TIM-1) es un receptor para el virus del Ébola de Zaire y el virus de Marburgo del Lago Victoria". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (20): 8426–8431. Bibcode :2011PNAS..108.8426K. doi : 10.1073/pnas.1019030108 . PMC 3100998 . PMID 21536871.
- ^ Base de datos de biomarcadores. Virus del Ébola. Instituto Nacional de Salud de Corea. Archivado desde el original el 22 de abril de 2008. Consultado el 31 de mayo de 2009 .
- ^ Saeed MF, Kolokoltsov AA, Albrecht T, Davey RA (2010). Basler CF (ed.). "La entrada celular del virus del Ébola implica la captación por un mecanismo similar a la macropinocitosis y el posterior tráfico a través de endosomas tempranos y tardíos". PLOS Pathogens . 6 (9): e1001110. doi : 10.1371/journal.ppat.1001110 . PMC 2940741 . PMID 20862315.
- ^ Mühlberger, Elke (4 de diciembre de 2016). "Replicación y transcripción de filovirus". Future Virology . 2 (2): 205–215. doi :10.2217/17460794.2.2.205. ISSN 1746-0794. PMC 3787895 . PMID 24093048.
- ^ Feldmann, H.; Klenk, H.-D. (1996). Filovirus. Rama Médica de la Universidad de Texas en Galveston. ISBN 978-0963117212Archivado desde el original el 9 de septiembre de 2018 . Consultado el 4 de diciembre de 2016 .
{{cite book}}:|work=ignorado ( ayuda ) - ^ Lai, Kang Yiu; Ng, Wing Yiu George; Cheng, Fan Fanny (28 de noviembre de 2014). "Infección humana por el virus del Ébola en África occidental: una revisión de los agentes terapéuticos disponibles que se dirigen a diferentes etapas del ciclo de vida del virus del Ébola". Enfermedades infecciosas de la pobreza . 3 (1): 43. doi : 10.1186/2049-9957-3-43 . ISSN 2049-9957. PMC 4334593 . PMID 25699183.
- ^ ab Feldmann H (mayo de 2014). "Ébola: ¿una amenaza creciente?". N. Engl. J. Med . 371 (15): 1375–1378. doi : 10.1056/NEJMp1405314 . PMID 24805988. S2CID 4657264.
- ^ Ng, S.; Cowling, B. (2014). "Asociación entre temperatura, humedad y brotes de enfermedad por virus del Ébola en África, 1976 a 2014". Eurosurveillance . 19 (35): 20892. doi : 10.2807/1560-7917.ES2014.19.35.20892 . PMID 25210981.
- ^ "Atención clínica para sobrevivientes de la enfermedad por el virus del Ébola" (PDF) . Organización Mundial de la Salud. 2016. Archivado (PDF) del original el 31 de agosto de 2016 . Consultado el 4 de diciembre de 2016 .
- ^ Isaacson M, Sureau P, Courteille G, Pattyn, SR. "Aspectos clínicos de la enfermedad del virus del Ébola en el Hospital Ngaliema, Kinshasa, Zaire, 1976". Red Europea para el Diagnóstico de Enfermedades Víricas "Importadas" (ENIVD). Archivado desde el original el 4 de agosto de 2014 . Consultado el 24 de junio de 2014 .
- ^ Bardi, Jason Socrates. "La muerte se llama río". The Scripps Research Institute . Archivado desde el original el 2 de agosto de 2014. Consultado el 9 de octubre de 2014 .
- ^ Nombre: S. Reardan.; N Engl. J Med. (2014) "Los primeros nueve meses de la epidemia y las proyecciones de la enfermedad por el virus del Ébola en África occidental". Archivo del Equipo de Respuesta al Ébola. 511(75.11):520
- ^ Gina Kolata (30 de octubre de 2014). «Los genes influyen en la reacción de los ratones al ébola, según un estudio que supone un «avance significativo»». New York Times . Archivado desde el original el 9 de marzo de 2021 . Consultado el 30 de octubre de 2014 .
- ^ Rasmussen, Angela L.; et al. (30 de octubre de 2014). "La diversidad genética del huésped permite la patogénesis y la resistencia a la fiebre hemorrágica del Ébola". Science . 346 (6212): 987–991. Bibcode :2014Sci...346..987R. doi :10.1126/science.1259595. PMC 4241145 . PMID 25359852.
- ^ Henao-Restrepo, Ana Maria; et al. (22 de diciembre de 2016). "Eficacia y efectividad de una vacuna con vector rVSV en la prevención de la enfermedad por el virus del Ébola: resultados finales del ensayo de vacunación en anillo, abierto y aleatorizado por grupos en Guinea (¡Ébola es suficiente!)". The Lancet . 389 (10068): 505–518. doi :10.1016/S0140-6736(16)32621-6. PMC 5364328 . PMID 28017403.
- ^ Berlinger, Joshua (22 de diciembre de 2016). «Estudio revela que la vacuna contra el ébola ofrece un 100 % de protección». CNN . Archivado desde el original el 27 de diciembre de 2016. Consultado el 27 de diciembre de 2016 .
- ^ "Primera vacuna aprobada por la FDA para la prevención de la enfermedad del virus del Ébola, lo que marca un hito crítico en la preparación y respuesta de la salud pública". Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) . 19 de diciembre de 2019. Archivado desde el original el 20 de diciembre de 2019. Consultado el 19 de diciembre de 2019 .
- ^ Brown, Rob (18 de julio de 2014). «El detective de virus que descubrió el ébola». BBC News . Archivado desde el original el 26 de octubre de 2021. Consultado el 21 de junio de 2018 .
- ^ Johnson KM, Webb PA, Lange JV, Murphy FA (1977). "Aislamiento y caracterización parcial de un nuevo virus causante de fiebre hemorrágica en Zambia". Lancet . 309 (8011): 569–571. doi :10.1016/s0140-6736(77)92000-1. PMID 65661. S2CID 19368457.
- ^ Netesov SV, Feldmann H, Jahrling PB, Klenk HD, Sanchez A (2000). "Familia Filoviridae". En van Regenmortel MHV, Fauquet CM, Bishop DHL, Carstens EB, Estes MK, Lemon SM, Maniloff J, Mayo MA, McGeoch DJ, Pringle CR, Wickner RB (eds.). Taxonomía de virus: séptimo informe del Comité Internacional de Taxonomía de Virus . San Diego: Academic Press. págs. 539–548. ISBN. 978-0123702005.
- ^ Pringle, CR (1998). "Taxonomía de virus – San Diego 1998". Archivos de Virología . 143 (7): 1449–1459. doi :10.1007/s007050050389. PMID 9742051. S2CID 13229117.
- ^ Feldmann H, Geisbert TW, Jahrling PB, Klenk HD, Netesov SV, Peters CJ, Sanchez A, Swanepoel R, Volchkov VE (2005). "Familia Filoviridae". En Fauquet CM, Mayo MA, Maniloff J, Desselberger U, Ball LA (eds.). Taxonomía de virus: octavo informe del Comité Internacional de Taxonomía de Virus . San Diego: Elsevier/Academic Press. págs. 645–653. ISBN. 978-0123702005.
- ^ Mayo, MA (2002). «ICTV en la ICV de París: resultados de la sesión plenaria y de la votación binomial». Archivos de Virología . 147 (11): 2254–2260. doi : 10.1007/s007050200052 . S2CID 43887711.
- ^ "Reemplazar el nombre de la especie Lake Victoria marburgvirus por Marburg marburgvirus en el género Marburgvirus". Archivado desde el original el 5 de marzo de 2016 . Consultado el 31 de octubre de 2014 .
- ^ Comité Internacional de Taxonomía de Virus. «Taxonomía de virus: versión de 2013». Archivado desde el original el 10 de julio de 2015. Consultado el 31 de octubre de 2014 .
- ^ Wahl-Jensen V, Kurz SK, Hazelton PR, Schnittler HJ, Stroher U, Burton DR, Feldmann H (2005). "El papel de las glicoproteínas secretadas por el virus del Ébola y las partículas similares al virus en la activación de los macrófagos humanos". Journal of Virology . 79 (4): 2413–2419. doi :10.1128/JVI.79.4.2413-2419.2005. PMC 546544 . PMID 15681442.
- ^ Kesel AJ, Huang Z, Murray MG, Prichard MN, Caboni L, Nevin DK, Fayne D, Lloyd DG, Detorio MA, Schinazi RF (2014). "Retinazone inhibe ciertos virus humanos transmitidos por la sangre, incluido el virus del Ébola Zaire". Química antiviral y quimioterapia . 23 (5): 197–215. doi :10.3851/IMP2568. PMC 7714485. PMID 23636868. S2CID 34249020 .
- ^ ab Carroll, SA (2012). "Evolución molecular de los virus de la familia Filoviridae basada en 97 secuencias de genoma completo". Journal of Virology . 87 (5): 2608–2616. doi :10.1128/JVI.03118-12. PMC 3571414 . PMID 23255795.
- ^ ab Li, YH (2013). "Historia evolutiva del virus del Ébola". Epidemiología e infección . 142 (6): 1138–1145. doi :10.1017/S0950268813002215. PMC 9151191 . PMID 24040779. S2CID 9873900.
- ^ ab Wittmann TJ, Biek R, Hassanin A, Rouquet P, Reed P, Yaba P, Pourrut X, Real LA, Gonzalez JP, Leroy EM. "Aislamientos de virus del Ébola de Zaire de simios salvajes revelan linaje genético y recombinantes". Proc Natl Acad Sci USA . 23 de octubre de 2007;104(43):17123–17127. Publicación electrónica 17 de octubre de 2007. "Fe de erratas" en: Proc Natl Acad Sci USA . 4 de diciembre de 2007;104(49):19656. PMID 17942693
- ^ ab "Cronología de los brotes: enfermedad por el virus del Ébola". Fiebre hemorrágica del Ébola . CDC. 2 de agosto de 2017. Archivado desde el original el 17 de mayo de 2019. Consultado el 11 de noviembre de 2017 .
- ^ Kava, Alex (octubre de 2008). Reseña de libro de ficción: 'Exposed' de Alex Kava, autor. Mira (332 págs.). PublishersWeekly.com. ISBN 978-0778325574Archivado del original el 7 de noviembre de 2021 . Consultado el 7 de noviembre de 2021 .
- ^ Close, William T. (1995). Ébola: una novela documental sobre su primera explosión . Nueva York: Ivy Books . ISBN 978-0804114325.OCLC 32753758 .En Google Libros .
- ^ Grove, Ryan (2 de junio de 2006). Más sobre la gente que sobre el virus. Archivado desde el original el 21 de octubre de 2014. Consultado el 17 de septiembre de 2014 .
{{cite book}}:|work=ignorado ( ayuda ) - ^ Close, William T. (2002). El ébola a través de los ojos de la gente. Marbleton, Wyoming: Meadowlark Springs Productions. ISBN 978-0970337115. OCLC 49193962. Archivado desde el original el 14 de enero de 2023. Consultado el 7 de febrero de 2016 . En Google Libros .
- ^ Pink, Brenda (24 de junio de 2008). "Una perspectiva fascinante". Reseña de Close, William T., Ebola: Through the Eyes of the People . Archivado desde el original el 21 de octubre de 2014. Consultado el 17 de septiembre de 2014 .
- ^ Preston, Richard (1995). La zona caliente . Nueva York: Anchor. ISBN 0385479565.OCLC 32052009 .
- ^ Clancy, Tom (1996). Órdenes ejecutivas . Nueva York: Putnam. ISBN 978-0399142185.OCLC 34878804 .
- ^ Stone, Oliver (2 de septiembre de 1996). "¿Quién es ese en la Oficina Oval?". Noticias y reseñas de libros. The New York Times Company . Archivado desde el original el 10 de abril de 2009. Consultado el 10 de septiembre de 2014 .
Lectura adicional
- Pacheco, Daniela Alexandra de Meneses Rocha; Rodrigues, Acácio Agostinho Gonçalves; Silva, Carmen Maria Lisboa da (octubre de 2016). "Virus del Ébola: de una amenaza desatendida a un estado de emergencia global". Revista de la Asociación Médica Brasileira . 62 (5): 458–467. doi : 10.1590/1806-9282.62.05.458 . PMID 27656857.
Enlaces externos
- Biología molecular del virus del Ébola
- Proteínas del virus del Ébola (PDB-101)
- Archivos y debates del ICTV: foro de debate y distribución de archivos para el Comité Internacional de Taxonomía de Virus Archivado el 7 de octubre de 2011 en Wayback Machine
- Datos genómicos sobre aislamientos del virus del Ébola y otros miembros de la familia Filoviridae
- ViralZone: Virus similares al ébola – Repositorio virológico del Instituto Suizo de Bioinformática
- Recurso sobre virus patógenos: Portal del ébola: datos genómicos y de otras investigaciones sobre el ébola y otros virus patógenos humanos
- Modelo 3D del virus del Ébola, preparado por Visual Science, Moscú.
- FILOVIR – recursos científicos para la investigación sobre filovirus Archivado el 30 de julio de 2020 en Wayback Machine
- "'Zaire ebolavirus'". Navegador de taxonomía del NCBI . 186538.
- "'Ebola virus sp.'". Navegador de taxonomía del NCBI . 205488.