Modelado de sistemas biológicos
El modelado de sistemas biológicos es una tarea importante de la biología de sistemas y la biología matemática . [a] La biología de sistemas computacionales [b] [1] tiene como objetivo desarrollar y utilizar algoritmos eficientes , estructuras de datos , herramientas de visualización y comunicación con el objetivo de modelar por computadora los sistemas biológicos. Implica el uso de simulaciones por computadora de sistemas biológicos, incluidos los subsistemas celulares (como las redes de metabolitos y enzimas que comprenden el metabolismo , las vías de transducción de señales y las redes reguladoras de genes ), para analizar y visualizar las conexiones complejas de estos procesos celulares. [2]
Una propiedad emergente inesperada de un sistema complejo puede ser el resultado de la interacción de la causa y el efecto entre partes más simples e integradas (véase organización biológica ). Los sistemas biológicos manifiestan muchos ejemplos importantes de propiedades emergentes en la interacción compleja de componentes. El estudio tradicional de los sistemas biológicos requiere métodos reductivos en los que se recopilan cantidades de datos por categoría, como la concentración a lo largo del tiempo en respuesta a un cierto estímulo. Las computadoras son fundamentales para el análisis y el modelado de estos datos. El objetivo es crear modelos precisos en tiempo real de la respuesta de un sistema a estímulos ambientales e internos, como un modelo de una célula cancerosa para encontrar debilidades en sus vías de señalización, o un modelo de mutaciones de canales iónicos para ver los efectos en los cardiomiocitos y, a su vez, la función de un corazón que late.
Normas
El formato estándar más aceptado para almacenar e intercambiar modelos en el campo es el lenguaje de marcado de biología de sistemas (SBML) . [3] El sitio web SBML.org incluye una guía de muchos paquetes de software importantes utilizados en biología de sistemas computacionales. Se puede recuperar una gran cantidad de modelos codificados en SBML desde BioModels . Otros lenguajes de marcado con diferentes énfasis incluyen BioPAX y CellML .
Tareas particulares
Modelo celular

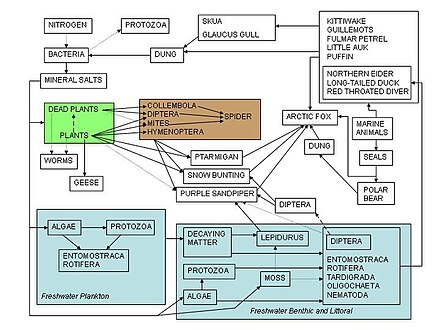

La creación de un modelo celular ha sido una tarea particularmente desafiante para la biología de sistemas y la biología matemática . Implica el uso de simulaciones por computadora de los numerosos subsistemas celulares , como las redes de metabolitos , enzimas que comprenden el metabolismo y la transcripción , la traducción , la regulación y la inducción de redes reguladoras de genes. [4]
La compleja red de procesos de reacción/transporte bioquímico y su organización espacial hacen que el desarrollo de un modelo predictivo de una célula viva sea un gran desafío para el siglo XXI, catalogado como tal por la National Science Foundation (NSF) en 2006. [5]
Un modelo computacional de célula completa para la bacteria Mycoplasma genitalium , incluidos sus 525 genes, productos genéticos y sus interacciones, fue construido por científicos de la Universidad de Stanford y el Instituto J. Craig Venter y publicado el 20 de julio de 2012 en Cell. [6]
Un modelo informático dinámico de señalización intracelular fue la base para que Merrimack Pharmaceuticals descubriera el objetivo de su medicamento contra el cáncer MM-111. [7]
La computación de membrana es la tarea de modelar específicamente una membrana celular .
Simulación de organismos multicelulares
La comunidad OpenWorm está trabajando en una simulación de código abierto de C. elegans a nivel celular . Hasta ahora se ha creado el motor de física Gepetto y se han creado modelos del conectoma neuronal y de una célula muscular en formato NeuroML. [8]
Plegamiento de proteínas
La predicción de la estructura de proteínas es la predicción de la estructura tridimensional de una proteína a partir de su secuencia de aminoácidos , es decir, la predicción de la estructura terciaria de una proteína a partir de su estructura primaria . Es uno de los objetivos más importantes que persiguen la bioinformática y la química teórica . La predicción de la estructura de proteínas es de gran importancia en medicina (por ejemplo, en el diseño de fármacos ) y biotecnología (por ejemplo, en el diseño de nuevas enzimas ). Cada dos años, se evalúa el rendimiento de los métodos actuales en el experimento CASP .
Sistemas biológicos humanos
Modelo de cerebro
El Proyecto Cerebro Azul es un intento de crear un cerebro sintético mediante ingeniería inversa del cerebro de los mamíferos hasta el nivel molecular. El objetivo de este proyecto, fundado en mayo de 2005 por el Instituto del Cerebro y la Mente de la Escuela Politécnica de Lausana , Suiza, es estudiar los principios arquitectónicos y funcionales del cerebro. El proyecto está encabezado por el director del Instituto, Henry Markram. Utilizando una supercomputadora Blue Gene que ejecuta el software NEURON de Michael Hines , la simulación no consiste simplemente en una red neuronal artificial , sino que implica un modelo parcialmente biológicamente realista de neuronas . [9] [10] Sus defensores esperan que eventualmente arroje luz sobre la naturaleza de la conciencia . Hay una serie de subproyectos, incluido el Cerebro Azul de Cajal , coordinado por el Centro de Supercomputación y Visualización de Madrid (CeSViMa), y otros dirigidos por universidades y laboratorios independientes en el Reino Unido, Estados Unidos e Israel. El Proyecto Cerebro Humano se basa en el trabajo del Proyecto Cerebro Azul. [11] [12] Es uno de los seis proyectos piloto del Programa de Investigación de Tecnologías Emergentes Futuras de la Comisión Europea, [13] que compite por una financiación de mil millones de euros.
Modelo del sistema inmune
En la última década ha surgido un número creciente de simulaciones del sistema inmunológico. [14] [15]
Hígado virtual
El proyecto Hígado Virtual es un programa de investigación financiado por el Gobierno alemán con 43 millones de euros y formado por setenta grupos de investigación distribuidos por toda Alemania. El objetivo es producir un hígado virtual, un modelo matemático dinámico que represente la fisiología , la morfología y la función del hígado humano. [16]
Modelo de árbol
Los árboles electrónicos (e-trees) suelen utilizar sistemas L para simular el crecimiento. Los sistemas L son muy importantes en el campo de la ciencia de la complejidad y la vida artificial . Aún no se ha ideado un sistema universalmente aceptado para describir los cambios en la morfología de las plantas a nivel celular o modular. [17] Los algoritmos de generación de árboles más ampliamente implementados se describen en los artículos "Creation and Rendering of Realistic Trees" y Real-Time Tree Rendering.
Modelos ecológicos
Los modelos ecosistémicos son representaciones matemáticas de los ecosistemas . Por lo general, simplifican las redes alimentarias complejas hasta sus componentes principales o niveles tróficos y los cuantifican como cantidad de organismos , biomasa o el inventario / concentración de algún elemento químico pertinente (por ejemplo, carbono o una especie de nutriente como nitrógeno o fósforo ).
Modelos en ecotoxicología
El objetivo de los modelos en ecotoxicología es comprender, simular y predecir los efectos que provocan los tóxicos en el medio ambiente. La mayoría de los modelos actuales describen los efectos en uno de los muchos niveles diferentes de organización biológica (por ejemplo, organismos o poblaciones). Un desafío es el desarrollo de modelos que predigan los efectos en todas las escalas biológicas. Ecotoxicología y modelos analiza algunos tipos de modelos ecotoxicológicos y proporciona vínculos a muchos otros.
Modelización de enfermedades infecciosas
Es posible modelar matemáticamente el progreso de la mayoría de las enfermedades infecciosas para descubrir el resultado probable de una epidemia o ayudar a controlarlas mediante la vacunación . Este campo intenta encontrar parámetros para diversas enfermedades infecciosas y utilizar esos parámetros para realizar cálculos útiles sobre los efectos de un programa de vacunación masiva .
Véase también
- Visualización de datos biológicos
- Biosimulación
- Algoritmo de Gillespie
- Software de modelado molecular
- Simulación estocástica
Notas
- ^ A veces llamada biología teórica, biología seca o incluso biomatemática.
- ^ La biología de sistemas computacionales es una rama que busca generar una comprensión a nivel de sistema mediante el análisis de datos biológicos utilizando técnicas computacionales.
Referencias
- ^ Andres Kriete, Roland Eils, Biología de sistemas computacionales, Elsevier Academic Press, 2006.
- ^ Tavassoly, Iman; Goldfarb, Joseph; Iyengar, Ravi (4 de octubre de 2018). "Introducción a la biología de sistemas: métodos y enfoques básicos". Ensayos en bioquímica . 62 (4): 487–500. doi :10.1042/EBC20180003. ISSN 0071-1365. PMID 30287586. S2CID 52922135.
- ^ Klipp, Liebermeister, Helbig, Kowald y Schaber. (2007). "Estándares de biología de sistemas: la comunidad habla" (2007), Nature Biotechnology 25(4):390–391.
- ^ Carbonell-Ballestero M, Duran-Nebreda S, Montañez R, Solé R, Macía J, Rodríguez-Caso C (diciembre de 2014). "Una caracterización ascendente de funciones de transferencia para diseños de biología sintética: lecciones de la enzimología". Investigación de ácidos nucleicos . 42 (22): 14060–14069. doi : 10.1093/nar/gku964. PMC 4267673 . PMID 25404136.
- ^ Asociación Estadounidense para el Avance de la Ciencia
- ^ Karr, J. (2012) Un modelo computacional de célula completa predice el fenotipo a partir del genotipo celular
- ^ McDonagh, CF (2012) Actividad antitumoral de un nuevo anticuerpo biespecífico que se dirige a la unidad oncogénica ErbB2/ErbB3 e inhibe la activación inducida por la heregulina de ErbB3. Terapéutica molecular contra el cáncer
- ^ Descargas de OpenWorm
- ^ Graham-Rowe, Duncan. "Comienza la misión de construir un cerebro simulado", NewScientist , junio de 2005.
- ^ Palmer, Jason. Cerebro simulado más cerca del pensamiento, BBC News.
- ^ El Proyecto Cerebro Humano. Archivado el 5 de julio de 2012 en Wayback Machine .
- ^ Vídeo de Henry Markram presentando El Proyecto Cerebro Humano el 22 de junio de 2012.
- ^ Página de inicio de la Iniciativa Flagships FET.
- ^ Balicki, Jerzy (2004). "Algoritmo evolutivo multicriterio con modelo del sistema inmunitario para gestionar restricciones en la asignación de tareas". Inteligencia artificial y computación blanda - ICAISC 2004. Apuntes de clase en informática. Vol. 3070. págs. 394–399. doi :10.1007/978-3-540-24844-6_57. ISBN 978-3-540-22123-4.
- ^ "Simulación por computadora captura la respuesta inmunitaria a la gripe" . Consultado el 19 de agosto de 2009 .
- ^ "Red hepática virtual". Archivado desde el original el 30 de septiembre de 2012. Consultado el 14 de octubre de 2016 .
- ^ "Simulación del crecimiento de las plantas". Archivado desde el original el 9 de diciembre de 2009. Consultado el 18 de octubre de 2009 .
Fuentes
- Antmann, SS; Marsden, JE; Sirovich, L., eds. (2009). Fisiología matemática (2.ª ed.). Nueva York, Nueva York: Springer. ISBN 978-0-387-75846-6.
- Barnes, DJ; Chu, D. (2010), Introducción al modelado para biociencias, Springer Verlag
- Introducción al modelado de enfermedades infecciosas, de Emilia Vynnycky y Richard G White. Un libro introductorio al modelado de enfermedades infecciosas y sus aplicaciones.
Lectura adicional
- Barab, A. -L.; Oltvai, Z. (2004). "Biología de redes*: comprensión de la organización funcional de la célula". Nature Reviews Genetics . 5 (2): 101–113. doi :10.1038/nrg1272. PMID 14735121. S2CID 10950726.
- Covert; Schilling, C.; Palsson, B. (2001). "Regulación de la expresión génica en modelos de equilibrio de flujo del metabolismo". Journal of Theoretical Biology . 213 (1): 73–88. Bibcode :2001JThBi.213...73C. CiteSeerX 10.1.1.110.1647 . doi :10.1006/jtbi.2001.2405. PMID 11708855.
- Covert, MW; Palsson, B. . (2002). "Regulación transcripcional en modelos metabólicos basados en restricciones de Escherichia coli". The Journal of Biological Chemistry . 277 (31): 28058–28064. doi : 10.1074/jbc.M201691200 . PMID 12006566.
- Edwards; Palsson, B. (2000). "El genotipo metabólico in silico de Escherichia coli MG1655*: su definición, características y capacidades". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 97 (10): 5528–5533. Bibcode :2000PNAS...97.5528E. doi : 10.1073/pnas.97.10.5528 . PMC 25862 . PMID 10805808.
- Bonneau, R. (2008). "Aprendizaje de redes biológicas* desde módulos hasta dinámicas". Nature Chemical Biology . 4 (11): 658–664. doi :10.1038/nchembio.122. PMID 18936750.
- Edwards, JS; Ibarra, RU; Palsson, BO (2001). "Las predicciones in silico de las capacidades metabólicas de Escherichia coli son consistentes con los datos experimentales". Nature Biotechnology . 19 (2): 125–130. doi :10.1038/84379. PMID 11175725. S2CID 1619105.
- Fell, DA (1998). "Aumento del flujo en las vías metabólicas* Una perspectiva de análisis del control metabólico". Biotecnología y bioingeniería . 58 (2–3): 121–124. doi :10.1002/(SICI)1097-0290(19980420)58:2/3<121::AID-BIT2>3.0.CO;2-N. PMID 10191380.
- Hartwell, LH; Hopfield, JJ; Leibler, S.; Murray, AW (1999). "De la biología celular molecular a la modular". Nature . 402 (6761 Suppl): C47–C52. doi : 10.1038/35011540 . PMID 10591225. S2CID 34290973.
- Ideker; Galitski, T.; Hood, L. (2001). "Un nuevo enfoque para descifrar la biología de sistemas de la vida". Revisión anual de genómica y genética humana . 2 (1): 343–372. doi :10.1146/annurev.genom.2.1.343. PMID 11701654. S2CID 922378.
- Kitano, H. (2002). "Biología de sistemas computacionales". Nature . 420 (6912): 206–210. Bibcode :2002Natur.420..206K. doi :10.1038/nature01254. PMID 12432404. S2CID 4401115.
- Kitano, H. (2002). "Biología de sistemas*: una breve descripción general". Science . 295 (5560): 1662–1664. Bibcode :2002Sci...295.1662K. CiteSeerX 10.1.1.473.8389 . doi :10.1126/science.1069492. PMID 11872829. S2CID 2703843.
- Kitano (2002). "Mirar más allá de los detalles: un aumento de los enfoques orientados a sistemas en genética y biología molecular". Current Genetics . 41 (1): 1–10. doi :10.1007/s00294-002-0285-z. PMID 12073094. S2CID 18976498.
- Gilman, AG; Simon, MI; Bourne, HR; Harris, BA; Long, R.; Ross, EM; Stull, JT; Taussig, R.; Bourne, HR; Arkin, AP; Cobb, MH; Cyster, JG; Devreotes, PN; Ferrell, JE; Fruman, D.; Gold, M.; Weiss, A.; Stull, JT; Berridge, MJ; Cantley, LC; Catterall, WA; Coughlin, SR; Olson, EN; Smith, TF; Brugge, JS; Botstein, D.; Dixon, JE; Hunter, T.; Lefkowitz, RJ; Pawson, AJ (2002). "Descripción general de la Alianza para la señalización celular" (PDF) . Nature . 420 (6916): 703–706. Código Bibliográfico :2002Natur.420..703G. doi : 10.1038/nature01304 . PMID: 12478301. S2CID : 4367083.
- Palsson, Bernhard (2006). Biología de sistemas* Propiedades de redes reconstruidas . Cambridge: Cambridge University Press. ISBN 978-0-521-85903-5.
- Kauffman; Prakash, P.; Edwards, JS (2003). "Avances en el análisis del balance de flujo". Current Opinion in Biotechnology . 14 (5): 491–496. doi :10.1016/j.copbio.2003.08.001. PMID 14580578.
- Segrè, D.; Vitkup, D.; Church, GM (2002). "Análisis de optimalidad en redes metabólicas naturales y perturbadas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (23): 15112–15117. Bibcode :2002PNAS...9915112S. doi : 10.1073/pnas.232349399 . PMC 137552 . PMID 12415116.
- Wildermuth, MC (2000). "Análisis de control metabólico*: aplicaciones biológicas y perspectivas". Genome Biology . 1 (6): REVIEWS1031. doi : 10.1186/gb-2000-1-6-reviews1031 . PMC 138895 . PMID 11178271.
Enlaces externos
- Centro de Modelado de Inmunidad a Patógenos Entéricos (MIEP)