Penalización por brecha
Una penalización por espacios es un método para puntuar alineaciones de dos o más secuencias. Al alinear secuencias, la introducción de espacios en las secuencias puede permitir que un algoritmo de alineación coincida con más términos que una alineación sin espacios. Sin embargo, minimizar los espacios en una alineación es importante para crear una alineación útil. Demasiados espacios pueden hacer que una alineación pierda sentido. Las penalizaciones por espacios se utilizan para ajustar las puntuaciones de alineación en función de la cantidad y la longitud de los espacios. Los cinco tipos principales de penalizaciones por espacios son constantes, lineales, afines, convexas y basadas en perfiles. [1]
Aplicaciones
- Alineamiento de secuencias genéticas - En bioinformática, los huecos se utilizan para dar cuenta de las mutaciones genéticas que se producen a partir de inserciones o deleciones en la secuencia, a veces denominadas indels . Las inserciones o deleciones pueden ocurrir debido a mutaciones individuales, cruce desequilibrado en la meiosis , desemparejamiento de hebras deslizadas y translocación cromosómica . [2] La noción de un hueco en un alineamiento es importante en muchas aplicaciones biológicas, ya que las inserciones o deleciones comprenden una subsecuencia completa y a menudo ocurren a partir de un solo evento mutacional. [3] Además, los eventos mutacionales individuales pueden crear huecos de diferentes tamaños. Por lo tanto, al puntuar, los huecos deben puntuarse como un todo al alinear dos secuencias de ADN. Considerar múltiples huecos en una secuencia como un solo hueco más grande reducirá la asignación de un alto costo a las mutaciones. Por ejemplo, dos secuencias de proteínas pueden ser relativamente similares pero diferir en ciertos intervalos ya que una proteína puede tener una subunidad diferente en comparación con la otra. Representar estas subsecuencias diferentes como espacios nos permitirá tratar estos casos como “buenas coincidencias” incluso aunque haya largas ejecuciones consecutivas con operaciones de indel en la secuencia. Por lo tanto, usar un buen modelo de penalización de espacios evitará puntajes bajos en las alineaciones y mejorará las posibilidades de encontrar una alineación verdadera. [3] En las alineaciones de secuencias genéticas, los espacios se representan como guiones (-) en una alineación de secuencia de proteína/ADN. [1]
- Función diff de Unix : calcula la diferencia mínima entre dos archivos de forma similar a la detección de plagio.
- Revisión ortográfica : las penalizaciones por espacios en blanco pueden ayudar a encontrar palabras escritas correctamente con la menor distancia de edición hasta una palabra mal escrita. Los espacios en blanco pueden indicar una letra faltante en la palabra mal escrita.
- Detección de plagio : las penalizaciones por espacios en blanco permiten que los algoritmos detecten dónde se han plagiado secciones de un documento colocando espacios en blanco en las secciones originales y haciendo coincidir lo que es idéntico. La penalización por espacios en blanco para un documento determinado cuantifica qué parte de un documento determinado es probablemente original o plagiada.
Aplicaciones de la bioinformática
Alineación global
Una alineación global realiza una alineación de extremo a extremo de la secuencia de consulta con la secuencia de referencia. Idealmente, esta técnica de alineación es la más adecuada para secuencias estrechamente relacionadas de longitudes similares. El algoritmo Needleman-Wunsch es una técnica de programación dinámica que se utiliza para realizar una alineación global. Básicamente, el algoritmo divide el problema en un conjunto de subproblemas y luego utiliza los resultados de los subproblemas para reconstruir una solución a la consulta original. [4]
Alineación semiglobal
El alineamiento semiglobal se utiliza para encontrar una coincidencia particular dentro de una secuencia grande. Un ejemplo incluye la búsqueda de promotores dentro de una secuencia de ADN. A diferencia del alineamiento global, este método no implica la existencia de espacios en los extremos de una o ambas secuencias. Si los espacios en los extremos se penalizan en una secuencia 1 pero no en la secuencia 2, se produce un alineamiento que contiene la secuencia 2 dentro de la secuencia 1.
Alineación local
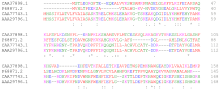
Una alineación de secuencia local hace coincidir una subsección contigua de una secuencia con una subsección contigua de otra. [5] El algoritmo Smith-Waterman está motivado por dar puntuaciones para coincidencias y desajustes. Las coincidencias aumentan la puntuación general de una alineación, mientras que los desajustes la disminuyen. Una buena alineación tiene entonces una puntuación positiva y una mala alineación tiene una puntuación negativa. El algoritmo local encuentra una alineación con la puntuación más alta considerando solo las alineaciones que puntúan positivamente y eligiendo la mejor de ellas. El algoritmo es un algoritmo de programación dinámica . Al comparar proteínas, se utiliza una matriz de similitud que asigna una puntuación a cada par de residuos posible. La puntuación debe ser positiva para residuos similares y negativa para pares de residuos diferentes. Los huecos suelen penalizarse utilizando una función de hueco lineal que asigna una penalización inicial para una apertura de hueco y una penalización adicional para las extensiones de hueco, aumentando la longitud del hueco.
Matriz de puntuación

Las matrices de sustitución como BLOSUM se utilizan para la alineación de secuencias de proteínas. [6] Una matriz de sustitución asigna una puntuación para alinear cualquier par posible de residuos. [6] En general, diferentes matrices de sustitución están diseñadas para detectar similitudes entre secuencias que divergen en diferentes grados. Una sola matriz puede ser razonablemente eficiente en un rango relativamente amplio de cambio evolutivo. [6] La matriz BLOSUM-62 es una de las mejores matrices de sustitución para detectar similitudes débiles en proteínas. [6] Las matrices BLOSUM con números altos están diseñadas para comparar secuencias estrechamente relacionadas, mientras que aquellas con números bajos están diseñadas para comparar secuencias relacionadas distantes. Por ejemplo, BLOSUM-80 se utiliza para alineaciones que son más similares en secuencia, y BLOSUM-45 se utiliza para alineaciones que han divergido entre sí. [6] Para alineaciones particularmente largas y débiles, la matriz BLOSUM-45 puede proporcionar los mejores resultados. Las alineaciones cortas se detectan más fácilmente utilizando una matriz con una "entropía relativa" más alta que la de BLOSUM-62. La serie BLOSUM no incluye ninguna matriz con entropías relativas adecuadas para las consultas más cortas. [6]
Indeles
Durante la replicación del ADN , la maquinaria de replicación celular es propensa a cometer dos tipos de errores al duplicar el ADN. Estos dos errores de replicación son inserciones y deleciones de bases de ADN individuales de la cadena de ADN (indels). [7] Los indels pueden tener graves consecuencias biológicas al causar mutaciones en la cadena de ADN que podrían resultar en la inactivación o sobreactivación de la proteína objetivo. Por ejemplo, si se produce un indel de uno o dos nucleótidos en una secuencia codificante, el resultado será un cambio en el marco de lectura, o una mutación por cambio de marco que puede hacer que la proteína sea inactiva. [7] Las consecuencias biológicas de los indels suelen ser perjudiciales y se asocian con frecuencia con patologías como el cáncer . Sin embargo, no todos los indels son mutaciones por cambio de marco. Si los indels ocurren en trinucleótidos, el resultado es una extensión de la secuencia de la proteína que también puede tener implicaciones en la función de la proteína. [7]
Tipos

Constante
Este es el tipo más simple de penalización por espacio: se otorga una puntuación negativa fija a cada espacio, independientemente de su longitud. [3] [8] Esto alienta al algoritmo a crear menos espacios, pero más grandes, dejando secciones contiguas más grandes.
Ataque de pánico||||||EN---CCTGA
Alineación de dos secuencias cortas de ADN, en la que el signo "-" representa un espacio de un par de bases. Si cada coincidencia valiera 1 punto y el espacio completo -1, la puntuación total sería: 7 − 1 = 6.
Lineal
En comparación con la penalización por espacio constante, la penalización por espacio lineal tiene en cuenta la longitud (L) de cada inserción/eliminación en el espacio. Por lo tanto, si la penalización por cada elemento insertado/eliminado es B y la longitud del espacio L, la penalización por espacio total sería el producto de las dos BL. [9] Este método favorece los espacios más cortos, y la puntuación total disminuye con cada espacio adicional.
Ataque de pánico||||||EN---CCTGA
A diferencia de la penalización por brecha constante, se tiene en cuenta el tamaño de la brecha. En un partido con puntuación 1 y cada brecha -1, la puntuación aquí es (7 − 3 = 4).
Afín
La función de penalización de brecha más utilizada es la penalización de brecha afín. La penalización de brecha afín combina los componentes de la penalización de brecha constante y lineal, tomando la forma . Esto introduce nuevos términos, A se conoce como la penalización de apertura de brecha, B la penalización de extensión de brecha y L la longitud de la brecha. La apertura de brecha se refiere al costo requerido para abrir una brecha de cualquier longitud, y la extensión de brecha el costo de extender la longitud de una brecha existente por 1. [10] A menudo no está claro cuáles deberían ser los valores A y B, ya que difieren según el propósito. En general, si el interés es encontrar coincidencias estrechamente relacionadas (por ejemplo, la eliminación de la secuencia del vector durante la secuenciación del genoma), se debe usar una penalización de brecha más alta para reducir las aperturas de brecha. Por otro lado, la penalización de brecha se debe reducir cuando se está interesado en encontrar una coincidencia más distante. [9] La relación entre A y B también tiene un efecto en el tamaño de la brecha. Si el tamaño de la brecha es importante, se usa una A pequeña y una B grande (más costoso para extender una brecha) y viceversa. Solo la relación A/B es importante, ya que multiplicar ambas por la misma constante positiva aumentará todas las penalizaciones en : lo que no cambia la penalización relativa entre diferentes alineaciones.
Convexo
El uso de la penalización por brecha afín requiere la asignación de valores de penalización fijos tanto para la apertura como para la extensión de una brecha. Esto puede ser demasiado rígido para su uso en un contexto biológico. [11]
El gap logarítmico toma la forma y se propuso como estudios que habían demostrado que la distribución de tamaños de indel obedece a una ley de potencia. [12] Otro problema propuesto con el uso de gaps afines es el favoritismo de alinear secuencias con gaps más cortos. La penalización de gap logarítmico se inventó para modificar el gap afín de modo que los gaps largos sean deseables. [11] Sin embargo, en contraste con esto, se ha encontrado que el uso de modelos logarítmicos había producido alineaciones deficientes en comparación con los modelos afines. [12]
Basado en perfiles
Los algoritmos de alineación perfil-perfil son herramientas poderosas para detectar relaciones de homología de proteínas con una precisión de alineación mejorada. [13] Las alineaciones perfil-perfil se basan en los perfiles de frecuencia estadística de indel de múltiples alineaciones de secuencias generadas por búsquedas PSI-BLAST. [13] En lugar de utilizar matrices de sustitución para medir la similitud de pares de aminoácidos, los métodos de alineación perfil-perfil requieren una función de puntuación basada en perfiles para medir la similitud de pares de vectores de perfil. [13] Las alineaciones perfil-perfil emplean funciones de penalización de brecha. La información de brecha se utiliza generalmente en forma de perfiles de frecuencia de indel, que es más específica para las secuencias que se alinearán. ClustalW y MAFFT adoptaron este tipo de determinación de penalización de brecha para sus alineaciones de secuencias múltiples. [13] La precisión de la alineación se puede mejorar utilizando este modelo, especialmente para proteínas con baja identidad de secuencia. Algunos algoritmos de alineación perfil-perfil también ejecutan la información de estructura secundaria como un término en sus funciones de puntuación, lo que mejora la precisión de la alineación. [13]
Comparando complejidades temporales
El uso de la alineación en biología computacional a menudo implica secuencias de longitudes variables. Es importante elegir un modelo que funcione de manera eficiente con un tamaño de entrada conocido. El tiempo que lleva ejecutar el algoritmo se conoce como complejidad temporal.
| Tipo | Tiempo |
|---|---|
| Penalización por brecha constante | O(mn) |
| Penalización por brecha afín | O(mn) |
| Penalización por brecha convexa | O(mn lg(m+n)) |
Desafíos
Existen algunos desafíos cuando se trata de trabajar con gaps. Cuando se trabaja con algoritmos populares parece haber poca base teórica para la forma de las funciones de penalización de gap. [14] En consecuencia, para cualquier situación de alineación, la ubicación de los gaps debe determinarse empíricamente. [14] Además, las penalizaciones de gap por alineación por pares, como la penalización de gap afín, a menudo se implementan independientemente de los tipos de aminoácidos en el fragmento insertado o eliminado o en los extremos rotos, a pesar de la evidencia de que se prefieren tipos de residuos específicos en las regiones de gap. [14] Finalmente, la alineación de secuencias implica la alineación de las estructuras correspondientes, pero las relaciones entre las características estructurales de los gaps en las proteínas y sus secuencias correspondientes solo se conocen de manera imperfecta. Debido a esto, la incorporación de información estructural en las penalizaciones de gap es difícil de hacer. [14] Algunos algoritmos utilizan información estructural predicha o real para sesgar la ubicación de los gaps. Sin embargo, solo una minoría de secuencias tienen estructuras conocidas, y la mayoría de los problemas de alineación involucran secuencias de estructura secundaria y terciaria desconocidas. [14]
Referencias
- ^ ab "Glosario". Rosalind . Equipo Rosalind . Consultado el 20 de mayo de 2021 .
- ^ Carroll, Ridge, Clement, Snell, Hyrum, Perry, Mark, Quinn (1 de enero de 2007). "Efectos de las penalizaciones por apertura y extensión de huecos". Revista internacional de investigación y aplicaciones de bioinformática . Consultado el 9 de septiembre de 2014 .
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ) - ^ abc "Penalización por brecha" (PDF) . Algoritmos para biología molecular . 1 de enero de 2006. Archivado desde el original (PDF) el 26 de junio de 2013. Consultado el 13 de septiembre de 2014 .
- ^ Lesk, Arthur M (26 de julio de 2013). «bioinformática». Encyclopædia Britannica . Consultado el 12 de septiembre de 2014 .
- ^ Vingron, M.; Waterman, MS (1994). "Alineamiento de secuencias y elección de penalizaciones. Revisión de conceptos, estudios de casos e implicaciones". Journal of Molecular Biology . 235 (1): 1–12. doi :10.1016/S0022-2836(05)80006-3. PMID 8289235.
- ^ abcdef "Matrices de sustitución BLAST". NCBI . Consultado el 27 de noviembre de 2012 .
- ^ abc Garcia-Diaz, Miguel (2006). "Mecanismo de un glissando genético: biología estructural de mutaciones indel". Tendencias en Ciencias Bioquímicas . 31 (4): 206–214. doi :10.1016/j.tibs.2006.02.004. PMID 16545956.
- ^ "Glosario - Penalización por brecha constante". Rosalind . Equipo Rosalind. 12 de agosto de 2014 . Consultado el 12 de agosto de 2014 .
- ^ ab Hodgman C, French A, Westhead D (2009). Notas instantáneas de BIOS en bioinformática . Garland Science. págs. 143-144. ISBN 978-0203967249.
- ^ "Alineación global con la matriz de puntuación y la penalización por brecha afín". Rosalind . Equipo Rosalind. 2012-07-02 . Consultado el 2014-09-12 .
- ^ ab Sung, Wing-Kin (2011). Algoritmos en bioinformática: una introducción práctica . CRC Press. págs. 42–47. ISBN 978-1420070347.
- ^ ab Cartwright, Reed (5 de diciembre de 2006). "Los costos de la brecha logarítmica disminuyen la precisión de la alineación". BMC Bioinformatics . 7 : 527. doi : 10.1186/1471-2105-7-527 . PMC 1770940 . PMID 17147805.
- ^ abcde Wang C, Yan RX, Wang XF, Si JN, Zhang Z (12 de octubre de 2011). "Comparación de penalizaciones por brechas lineales y penalizaciones por brechas variables basadas en perfiles en alineaciones perfil-perfil". Comput Biol Chem . 35 (5): 308–318. doi :10.1016/j.compbiolchem.2011.07.006. PMID 22000802.
- ^ abcde Wrabl JO, Grishin NV (1 de enero de 2004). "Brechas en proteínas estructuralmente similares: hacia la mejora del alineamiento de secuencias múltiples". Proteins . 54 (1): 71–87. doi :10.1002/prot.10508. PMID 14705025. S2CID 20474119.
Lectura adicional
- Taylor WR, Munro RE (1997). "Enhebrado de secuencias múltiples: colocación condicional de espacios". Fold Des . 2 (4): S33-9. doi : 10.1016/S1359-0278(97)00061-8 . PMID 9269566.
- Taylor WR (1996). "Una penalización por brecha no local para la alineación de perfiles". Bull Math Biol . 58 (1): 1–18. doi :10.1007/BF02458279. PMID 8819751. S2CID 189884646.
- Vingron M, Waterman MS (1994). "Alineación de secuencias y elección de penalizaciones. Revisión de conceptos, estudios de casos e implicaciones". J Mol Biol . 235 (1): 1–12. doi :10.1016/S0022-2836(05)80006-3. PMID 8289235.
- Panjukov VV (1993). "Encontrar alineaciones estables: similitud y distancia". Comput Appl Biosci . 9 (3): 285–90. doi :10.1093/bioinformatics/9.3.285. PMID 8324629.
- Alexandrov NN (1992). "Alineamiento local múltiple por matriz de consenso". Comput Appl Biosci . 8 (4): 339–45. doi :10.1093/bioinformatics/8.4.339. PMID 1498689.
- Hein J (1989). "Un nuevo método que alinea y reconstruye simultáneamente secuencias ancestrales para cualquier número de secuencias homólogas, cuando se proporciona la filogenia". Mol Biol Evol . 6 (6): 649–68. doi : 10.1093/oxfordjournals.molbev.a040577 . PMID 2488477.
- Henneke CM (1989). "Un algoritmo de alineamiento de secuencias múltiples para proteínas homólogas que utiliza información de estructura secundaria y opcionalmente alineamientos clave para sitios funcionalmente importantes". Comput Appl Biosci . 5 (2): 141–50. doi :10.1093/bioinformatics/5.2.141. PMID 2751764.
- Reich JG, Drabsch H, Daumler A (1984). "Sobre la evaluación estadística de similitudes en secuencias de ADN". Nucleic Acids Res . 12 (13): 5529–43. doi :10.1093/nar/12.13.5529. PMC 318937 . PMID 6462914.



