Receptor del factor de crecimiento derivado de plaquetas
Este artículo necesita citas adicionales para su verificación . ( noviembre de 2019 ) |
| Receptor del factor de crecimiento derivado de plaquetas | |||||||||
|---|---|---|---|---|---|---|---|---|---|
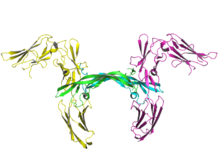 Imagen en cinta de dos moléculas del receptor beta de PDGF humano (amarillo y magenta) en complejo con PDGF-B dimérico (cian y verde). [1] | |||||||||
| Identificadores | |||||||||
| Símbolo | PDGFR | ||||||||
| Pfam | PF04692 | ||||||||
| Interprofesional | IPR006782 | ||||||||
| Membranoma | 1204 | ||||||||
| |||||||||
| receptor del factor de crecimiento derivado de plaquetas, polipéptido alfa | |||||||
|---|---|---|---|---|---|---|---|
| Identificadores | |||||||
| Símbolo | PDGFRA | ||||||
| Gen NCBI | 5156 | ||||||
| HGNC | 8803 | ||||||
| OMI | 173490 | ||||||
| Secuencia de referencia | Número de modelo_006206 | ||||||
| Protección unificada | P16234 | ||||||
| Otros datos | |||||||
| Lugar | Crónica 4 q12 | ||||||
| |||||||
| receptor del factor de crecimiento derivado de plaquetas, polipéptido beta | |||||||
|---|---|---|---|---|---|---|---|
| Identificadores | |||||||
| Símbolo | PDGFRB | ||||||
| Símbolos alternativos | PDGFR | ||||||
| Gen NCBI | 5159 | ||||||
| HGNC | 8804 | ||||||
| OMI | 173410 | ||||||
| Secuencia de referencia | Número de modelo_002609 | ||||||
| Protección unificada | P09619 | ||||||
| Otros datos | |||||||
| Lugar | Crónica 5 q31-q32 | ||||||
| |||||||
Los receptores del factor de crecimiento derivado de plaquetas ( PDGF-R ) son receptores de tirosina quinasa de la superficie celular para miembros de la familia del factor de crecimiento derivado de plaquetas (PDGF). Las subunidades -A y -B del PDGF son factores importantes que regulan la proliferación celular , la diferenciación celular , el crecimiento celular , el desarrollo y muchas enfermedades, incluido el cáncer . [2] Hay dos formas del PDGF-R, alfa y beta , cada una codificada por un gen diferente. [3] Dependiendo de qué factor de crecimiento esté unido, el PDGF-R se homo- o heterodimeriza. [4]
Mecanismo de acción
La familia PDGF consta de PDGF-A, -B, -C y -D, que forman homo- o heterodímeros ( PDGF-AA, -AB, -BB, -CC, -DD). Los cuatro PDGF son inactivos en sus formas monoméricas . Los PDGF se unen a los receptores de la proteína tirosina quinasa PDGF receptor-α y -β. Estas dos isoformas del receptor se dimerizan al unirse al dímero de PDGF, lo que da lugar a tres posibles combinaciones de receptores, a saber, -αα, -ββ y -αβ. La región extracelular del receptor consta de cinco dominios similares a inmunoglobulina , mientras que la parte intracelular es un dominio de tirosina quinasa . Los sitios de unión del ligando de los receptores se encuentran en los tres primeros dominios similares a inmunoglobulina. PDGF-CC interactúa específicamente con PDGFR-αα y -αβ, pero no con -ββ, y por lo tanto se parece a PDGF-AB. El PDGF-DD se une al PDGFR-ββ con alta afinidad, y al PDGFR-αβ en un grado notablemente menor, por lo que se lo considera específico del PDGFR-ββ. El PDGF-AA se une únicamente al PDGFR-αα, mientras que el PDGF-BB es el único PDGF que puede unirse a las tres combinaciones de receptores con alta afinidad. [5]
La dimerización es un prerrequisito para la activación de la quinasa . La activación de la quinasa se visualiza como la fosforilación de tirosina de las moléculas del receptor, que ocurre entre las moléculas del receptor dimerizadas ( transfosforilación ). Junto con la dimerización y la activación de la quinasa, las moléculas del receptor experimentan cambios conformacionales , que permiten que una actividad quinasa basal fosforile un residuo crítico de tirosina , "desbloqueando" así la quinasa, lo que conduce a una actividad enzimática completa dirigida hacia otros residuos de tirosina en las moléculas del receptor, así como otros sustratos para la quinasa. La expresión de ambos receptores y cada uno de los cuatro PDGF está bajo control independiente, lo que le da al sistema PDGF/PDGFR una alta flexibilidad. Diferentes tipos de células varían mucho en la proporción de isoformas de PDGF y PDGFR expresados. Diferentes estímulos externos como la inflamación , el desarrollo embrionario o la diferenciación modulan la expresión del receptor celular permitiendo la unión de algunos PDGF pero no de otros. Además, algunas células muestran sólo una de las isoformas del PDGFR mientras que otras expresan ambas isoformas, simultáneamente o por separado.
Interacción con moléculas de transducción de señales
Los sitios de fosforilación de tirosina en los receptores de factores de crecimiento cumplen dos propósitos principales: controlar el estado de actividad de la quinasa y crear sitios de unión para las moléculas de transducción de señales posteriores , que en muchos casos también son sustratos para la quinasa. La segunda parte del dominio de la tirosina quinasa en el receptor PDGFβ está fosforilada en Tyr-857, y los receptores mutantes que llevan fenilalanina en esta posición tienen una actividad de quinasa reducida. Por lo tanto, a Tyr-857 se le ha asignado un papel en la regulación positiva de la actividad de la quinasa. [6] Se han identificado sitios de fosforilación de tirosina involucrados en la unión de moléculas de transducción de señales en el dominio yuxtamembrana, el inserto de la quinasa y en la cola C-terminal en el receptor PDGFβ. El residuo de tirosina fosforilado y, en general, tres residuos de aminoácidos C-terminales adyacentes forman sitios de unión específicos para moléculas de transducción de señales. La unión a estos sitios implica un tramo conservado común, denominado dominio de homología Src (SH) 2 y/o dominios de unión a fosfotirosina (PTB). La especificidad de estas interacciones parece ser muy alta, ya que los receptores mutantes que llevan residuos de fenilalanina en uno o varios de los diferentes sitios de fosforilación generalmente carecen de la capacidad de unirse a la molécula de transducción de señales objetivo. Las moléculas de transducción de señales están equipadas con diferentes actividades enzimáticas o son moléculas adaptadoras, que en algunos casos, pero no en todos, se encuentran en complejos con subunidades que llevan una actividad catalítica. Tras la interacción con el receptor activado, las actividades catalíticas se regulan al alza, a través de la fosforilación de tirosina u otros mecanismos, generando una señal que puede ser única para cada tipo de molécula de transducción de señales.
El examen de las diferentes cascadas de señalización inducidas por RTK estableció las vías de la proteína quinasa activada por mitógeno/Ras (MAPK), la quinasa PI-3 y la fosfolipasa-γ (PLCγ) como mediadores descendentes clave de la señalización de PDGFR. [7] [8] Además, se ha establecido que la activación de STAT3 dependiente de especies reactivas de oxígeno (ROS) es un mediador descendente clave de la señalización de PDGFR en las células musculares lisas vasculares. [9]
Vía MAPK
La proteína adaptadora Grb2 forma un complejo con Sos mediante el dominio SH3 de Grb2 . Grb2 (o el complejo Grb2/Sos) es reclutado a la membrana por la unión del dominio SH2 de Grb2 a SHP2 unida a PDGFR activado (también conocida como PTPN11 , una PTP citosólica ), lo que permite la interacción con Ras y el intercambio de GDP por GTP en Ras . Mientras que la interacción entre Grb2 y PDGFR ocurre a través de la interacción con la proteína SHP2, Grb2 se une al EGFR activado a través de Shc , otra proteína adaptadora que forma un complejo con muchos receptores a través de su dominio PTB . [10] Una vez activado, Ras interactúa con varias proteínas, a saber, Raf. Raf activado estimula la MAPK-quinasa (MAPKK o MEK) al fosforilar un residuo de serina en su bucle de activación . La MAPKK fosforila entonces a la MAPK (ERK1/2) en los residuos T e Y en el bucle de activación, lo que conduce a su activación. La MAPK activada fosforila una variedad de sustratos citoplasmáticos, así como factores de transcripción, cuando se transloca al núcleo. Se ha descubierto que los miembros de la familia MAPK regulan varias funciones biológicas mediante la fosforilación de moléculas diana particulares (como factores de transcripción, otras quinasas, etc.) ubicadas en la membrana celular, el citoplasma y el núcleo, y contribuyen así a la regulación de diferentes procesos celulares como la proliferación celular, la diferenciación, la apoptosis y las inmunorrespuestas .
Vía PI3K
La fosfolípido quinasa de clase IA, PI-3 quinasa, es activada por la mayoría de las RTK. De manera similar a otras proteínas que contienen el dominio SH2, la PI-3 quinasa forma un complejo con los sitios PY en los receptores activados. La función principal de la activación de PI3K es la generación de PIP3, que funciona como un segundo mensajero para activar las tirosina quinasas Btk e Itk, las Ser/Thr quinasas PDK1 y Akt (PKB). Las principales funciones biológicas de la activación de Akt se pueden clasificar en tres categorías: supervivencia, proliferación y crecimiento celular. También se sabe que Akt está implicada en varios cánceres, particularmente de mama. La PLCγ es reclutada inmediatamente por una RTK activada a través de la unión de sus dominios SH2 a los sitios de fosfotirosina del receptor. Después de la activación, la PLCγ hidroliza su sustrato PtdIns(4,5)P2 y forma dos segundos mensajeros, diacilglicerol e Ins(1,4,5)P3. El Ins(1,4,5)P3 estimula la liberación de Ca 2+ de los suministros intracelulares. Luego, el Ca 2+ se une a la calmodulina, que posteriormente activa una familia de proteínas quinasas independientes de calmodulina (CamK). Además, tanto el diacilglicerol como el Ca 2+ activan a miembros de la familia PKC. Los segundos mensajeros generados por la hidrólisis de PtdIns(4,5)P2 estimulan una variedad de procesos intracelulares como la proliferación, la angiogénesis y la motilidad celular.
Véase también
- Receptor de tirosina quinasa
- PDGF
- Imatinib
- PDGFRA
- PDGFRB
- Crenolanib (CP-868,596-26)
Referencias
- ^ PDB : 3MJG ; Shim AH, Liu H, Focia PJ, Chen X, Lin PC, He X (junio de 2010). "Estructuras de un complejo de factor de crecimiento/propéptido derivado de plaquetas y un complejo de factor de crecimiento/receptor derivado de plaquetas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 107 (25): 11307–12. Bibcode :2010PNAS..10711307H. doi : 10.1073/pnas.1000806107 . PMC 2895058 . PMID 20534510.; renderizado utilizando PyMOL.
- ^ Williams LT (marzo de 1989). "Transducción de señales por el receptor del factor de crecimiento derivado de plaquetas". Science . 243 (4898): 1564–70. Bibcode :1989Sci...243.1564W. doi :10.1126/science.2538922. PMID 2538922.
- ^ Heldin CH, Westermark B (abril de 1989). "Factor de crecimiento derivado de plaquetas: tres isoformas y dos tipos de receptores". Tendencias en genética . 5 (4): 108–11. doi :10.1016/0168-9525(89)90040-1. PMID 2543106.
- ^ Heldin CH, Ostman A, Eriksson A, Siegbahn A, Claesson-Welsh L, Westermark B (marzo de 1992). "Factor de crecimiento derivado de plaquetas: señalización específica de isoformas a través de complejos receptores heterodiméricos u homodiméricos". Kidney International . 41 (3): 571–4. doi : 10.1038/ki.1992.84 . PMID 1315403.
- ^ Cao Y, Cao R, Hedlund EM (julio de 2008). "Regulación de la angiogénesis tumoral y la metástasis por las vías de señalización de FGF y PDGF". Journal of Molecular Medicine . 86 (7): 785–9. doi :10.1007/s00109-008-0337-z. PMID 18392794. S2CID 21872247.
- ^ Kazlauskas A, Cooper JA (septiembre de 1989). "La autofosforilación del receptor PDGF en la región de inserción de la quinasa regula las interacciones con las proteínas celulares". Cell . 58 (6): 1121–33. doi :10.1016/0092-8674(89)90510-2. PMID 2550144. S2CID 25586248.
- ^ Valius M, Kazlauskas A (abril de 1993). "La fosfolipasa C-gamma 1 y la fosfatidilinositol 3 quinasa son los mediadores descendentes de la señal mitogénica del receptor PDGF". Cell . 73 (2): 321–34. doi :10.1016/0092-8674(93)90232-F. PMID 7682895. S2CID 36305139.
- ^ Montmayeur JP, Valius M, Vandenheede J, Kazlauskas A (diciembre de 1997). "El receptor del factor de crecimiento beta derivado de plaquetas desencadena múltiples cascadas de señalización citoplasmática que llegan al núcleo como entradas distinguibles". The Journal of Biological Chemistry . 272 (51): 32670–8. doi : 10.1074/jbc.272.51.32670 . PMID 9405485.
- ^ Blazevic T, Schwaigerer AV, Schreiner CE, Schachner D, Schaible AM, Grojer CS, Atanasov AG, Werz O, Dirsch VM, Heiss EH (diciembre de 2013). "La 12/15-lipoxigenasa contribuye a la activación inducida por el factor de crecimiento derivado de plaquetas del transductor de señal y activador de la transcripción 3". The Journal of Biological Chemistry . 288 (49): 35592–603. doi : 10.1074/jbc.M113.489013 . PMC 3853304 . PMID 24165129.
- ^ Schlessinger, J. Proteínas de señalización SH2/SH3. Curr. Op. Gen. Dev. 1994, 4 (1):25-30.
Enlaces externos
- Receptores de factores de crecimiento derivados de plaquetas en los encabezados de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.

