Haplogrupo T (ADNmt)
| Haplogrupo T | |
|---|---|
| Posible época de origen | 25.149 ± 4.668 años antes del presente |
| Posible lugar de origen | Oriente Próximo y/o Cáucaso |
| Antepasado | JT |
| Descendientes | T1 y T2 |
| Definición de mutaciones | G709A, G1888A, A4917G, G8697A, T10463C, G13368A, G14905A, A15607G, G15928A, C16294T |
El haplogrupo T es un haplogrupo de ADN mitocondrial (ADNmt) humano . Se cree que se originó hace unos 25.100 años en Oriente Próximo . [ cita requerida ]
Orígenes
El clado mitocondrial T deriva del haplogrupo JT , que también dio origen al haplogrupo J de ADNmt . Se cree que el clado materno T emanó del Cercano Oriente . [ cita requerida ]
Distribución

El haplogrupo basal T* se encuentra entre los argelinos de Orán (1,67%) y los saharauis de Reguibate (0,93%). [1] También se distribuye entre los soqotri (1,2%). [2]
El haplogrupo T está presente en frecuencias bajas en Asia occidental y central y Europa, con diversos grados de prevalencia y ciertamente podría haber estado presente en otros grupos de las áreas circundantes. T se encuentra en aproximadamente el 10% de los europeos nativos. [3] [4] También es común entre los iraníes modernos . Con base en una muestra de más de 400 iraníes modernos, [ cita requerida ] el haplogrupo T representa aproximadamente el 8,3% de la población (alrededor de 1 de cada 12 individuos), y el subtipo T1 más específico constituye aproximadamente la mitad de ellos. Además, el subtipo específico T1 tiende a encontrarse más al este y es común en las poblaciones turcas modernas y de Asia central (Lalueza-Fox 2004), que habitan gran parte del mismo territorio que los antiguos saka , sármatas , andronovo y otros supuestos pueblos iraníes del segundo y primer milenio a. C. Lalueza-Fox et al. (2004) también encontraron varias secuencias T y T1 en enterramientos antiguos, incluidos los kurganes , en la estepa kazaja entre los siglos XIV y X a. C., así como más tarde en el primer milenio a. C. Estos coinciden con la última parte del período Andronovo y el período Saka en la región. [5]
La distribución geográfica dentro del subclado T2 varía mucho y se ha informado que la proporción entre el subhaplogrupo T2e y el subhaplogrupo T2b varía 40 veces entre las poblaciones examinadas, desde un mínimo en Gran Bretaña e Irlanda hasta un máximo en Arabia Saudita (Bedford 2012). Dentro del subhaplogrupo T2e, se identifica un motivo muy raro entre los judíos sefardíes de Turquía y Bulgaria y entre los conversos sospechosos del Nuevo Mundo (Bedford 2012).
Se encuentra en la población Svan del Cáucaso (Georgia) T* 10,4% y T1 4,2%. T1a1a1 es particularmente común en países con altos niveles del haplogrupo Y R1a, como Europa central y nororiental. El clado también se encuentra en todas partes de Asia central y en el norte de Asia, tan al este como Mongolia.
Parece que el origen de T2c y T2d se remonta al Cercano Oriente, en torno a la época del Último Máximo Glacial (LGM) y a dispersiones más recientes en Europa. La mayor parte de T2c comprende el haplogrupo T2c1. Aparte de un pico en Chipre, T2c1 es más común en la región del Golfo Pérsico , pero también se encuentra en el Levante y en la Europa mediterránea, con una distribución más extensa en niveles muy bajos. [6]
La T2 también se encuentra entre los Soqotri (7,7%). [2]
Arqueología
Wilde et al. (2014) analizaron muestras de ADNmt de la cultura Yamna , la presunta patria de los hablantes protoindoeuropeos . Encontraron T2a1b en la región del Volga Medio y Bulgaria , y T1a tanto en Ucrania central como en el Volga Medio. La frecuencia de T1a y T2 en las muestras de Yamna fue del 14,5 %, un porcentaje más alto que en cualquier país en la actualidad y que solo se encontró en frecuencias igualmente altas entre los udmurtos de la región del Volga-Ural. [7]
El haplogrupo T también se ha encontrado entre especímenes iberomaurusianos que datan del Epipaleolítico en el sitio prehistórico de Afalou en Argelia . Un individuo antiguo portaba el subclado T2b (1/9; 11%). [8] Además, el haplogrupo T se ha observado entre las momias del antiguo Egipto excavadas en el sitio arqueológico de Abusir el-Meleq en el Medio Egipto, que datan de los períodos preptolemaico / Imperio Nuevo tardío (T1, T2), ptolemaico (T1, T2) y romano (T, T1 indiferenciado). [9] También se ha observado que los fósiles excavados en el sitio neolítico tardío de Kelif el Boroud en Marruecos , que se han datado alrededor de 3000 a. C., llevan el subclado T2. [10] Además, se ha observado el haplogrupo T en fósiles guanches antiguos excavados en Gran Canaria y Tenerife en las Islas Canarias , que han sido datados por radiocarbono entre los siglos VII y XI d.C. Los individuos portadores del clado fueron inhumados en el yacimiento de Tenerife, y se encontró que un espécimen pertenecía al subclado T2c1d2 (1/7; 14%). [11]
África
En África, el haplogrupo T se encuentra principalmente entre las poblaciones de habla afroasiática , incluido el clado T* basal. [1] Algunos clados T no basales también se encuentran comúnmente entre los serer de habla niger-congoleña debido a la difusión desde el Magreb , probablemente con la expansión del Islam. [12]
| Población | Ubicación | Familia de lenguas | norte | Frecuencia | Fuente |
|---|---|---|---|---|---|
| Amhara | Etiopía | Afroasiático > Semítico | 5/120 | 4,17% | Kivisild 2004 |
| Beja | Sudán | Afroasiático > Cushitico | 1/48 | 2,1% | Hassan 2009 |
| Beta Israel | Etiopía | Afroasiático > Cushitico | 0/29 | 0,00% | Behar 2008a |
| copto | Egipto | Afroasiático > egipcio | 29/5 | 17,2% | Hassan 2009 |
| Dawro K. | Etiopía | Afroasiático > Omótico | 2/137 | 1,46% | Castrì 2008 y Boattini 2013 |
| Egipcios (El-Hayez) | Egipto | Afroasiático > Semítico | 10/35 | 28,6% | Kujanova 2009 |
| Etiopía | Etiopía | Indeterminado | 2/77 | 2,60% | Soares 2011 |
| Judío etíope | Etiopía | Afroasiático > Cushitico | 0/41 | 0,00% | No 2011 |
| Guraje | Etiopía | Afroasiático > Semítico | 0/21 | 0,00% | Kivisild 2004 |
| Hamer | Etiopía | Afroasiático > Omótico | 0/11 | 0,00% | Castrì 2008 y Boattini 2013 |
| Ongota | Etiopía | Afroasiático > Cushitico | 0/19 | 0,00% | Castrì 2008 y Boattini 2013 |
| Oromo | Etiopía | Afroasiático > Cushitico | 0/33 | 0,00% | Kivisild 2004 |
| Tigray | Etiopía | Afroasiático > Semítico | 3/44 | 6,82% | Kivisild 2004 |
| Daasanach | Kenia | Afroasiático > Cushitico | 0/49 | 0,00% | Poloni 2009 |
| Elmolo | Kenia | Afroasiático > Cushitico | 0/52 | 0,00% | Castrì 2008 y Boattini 2013 |
| Luo | Kenia | Nilo-Sahariano | 0/49 | 0,00% | Castrì 2008 y Boattini 2013 |
| Masai | Kenia | Nilo-Sahariano | 0/81 | 0,00% | Castrì 2008 y Boattini 2013 |
| Nairobi | Kenia | Níger-Congo | 0/100 | 0,00% | Brandstätter 2004 |
| Nyangatom | Kenia | Nilo-Sahariano | 0/112 | 0,00% | Poloni 2009 |
| Rendille | Kenia | Afroasiático > Cushitico | 0/17 | 0,00% | Castrì 2008 y Boattini 2013 |
| Samburu | Kenia | Nilo-Sahariano | 0/35 | 0,00% | Castrì 2008 y Boattini 2013 |
| Turkana | Kenia | Nilo-Sahariano | 0/51 | 0,00% | Castrì 2008 y Boattini 2013 |
| Hutu | Ruanda | Níger-Congo | 0/42 | 0,00% | Castri 2009 |
| Dinka | Sudán | Nilo-Sahariano | 0/46 | 0,00% | Anillos 1999 |
| Sudán | Sudán | Indeterminado | 3/102 | 2,94% | Soares 2011 |
| Burbuja | Tanzania | Afroasiático > Cushitico | 0/38 | 0,00% | Tishkoff 2007 |
| Datoga | Tanzania | Nilo-Sahariano | 1/57 | 1,75% | Tishkoff 2007 y Knight 2003 |
| Irak | Tanzania | Afroasiático > Cushitico | 0/12 | 0,00% | Caballero 2003 |
| Sukuma | Tanzania | Níger-Congo | 0/32 | 0,00% | Tishkoff 2007 y Knight 2003 |
| Turú | Tanzania | Níger-Congo | 0/29 | 0,00% | Tishkoff 2007 |
| yemenita | Yemen | Afroasiático > Semítico | 1/114 | 0,88% | Kivisild 2004 |
Asia
Europa
Subclados
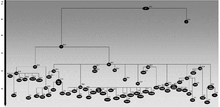
Árbol
Este árbol filogenético de los subclados del haplogrupo I se basa en el artículo (van Oven 2008) y en investigaciones publicadas posteriormente (Behar 2012b). Para abreviar, solo se muestran los tres primeros niveles de subclados (ramas).
- yo
- T1
- T1a
- T1a1
- T1a2
- T1b
- T1a
- T2
- T2a
- T2a1
- T2b
- T2b1
- T2b2
- T2b3
- T2b4
- T2b5
- T2b6
- T2c
- T2c1
- diabetes tipo 2
- T2e
- T2e2
- T2f
- T2f1
- T2g
- T2a
- T1
Problemas de salud
Un estudio ha demostrado que el haplogrupo T está asociado con un mayor riesgo de enfermedad de la arteria coronaria . [ cita requerida ] Sin embargo, algunos estudios también han demostrado que las personas del haplogrupo T son menos propensas a la diabetes (Chinnery 2007 y González 2012).
Algunos estudios médicos preliminares han demostrado que el haplogrupo T puede ofrecer cierta resistencia tanto a la enfermedad de Parkinson como a la enfermedad de Alzheimer . [ cita requerida ]
Un estudio ha descubierto que entre la población española, la miocardiopatía hipertrófica (MCH), también conocida como miocardiopatía hipertrófica obstructiva (MCHO), es más probable que se presente en personas de ascendencia T2 que en otros haplogrupos maternos. [13] Se desconoce si esto es específico de esta subclaudia del haplogrupo T o si es un factor de riesgo compartido por todos los haplogrupos T. Con una diferencia estadísticamente significativa encontrada en una muestra tan pequeña, puede ser recomendable que las personas de ascendencia materna conocida del haplogrupo T sean conscientes de esto y que su médico compruebe si hay evidencia de esta afección cuando se realicen un examen de rutina a una edad temprana. Por lo general, no presenta síntomas y aumenta el riesgo de muerte cardíaca súbita, que a menudo ocurre a personas tan jóvenes como la adolescencia y puede afectar a quienes son activos y no tienen otros factores de riesgo. [14]
Algunos estudios médicos han demostrado que el haplogrupo T mitocondrial está asociado con una menor movilidad de los espermatozoides en los varones, aunque estos resultados han sido cuestionados (Mishmar 2002). Según el Departamento de Bioquímica y Biología Molecular y Celular de la Universidad de Zaragoza, el haplogrupo T puede predisponer a la astenozoospermia (Ruiz-Pesini 2000). Sin embargo, estos hallazgos han sido cuestionados debido al pequeño tamaño de la muestra en el estudio (Mishmar 2002).
Miembros famosos
Durante el documental de BBC One Meet the Izzards , el actor y comediante Eddie Izzard se entera de que su ADN mitocondrial es del haplogrupo T, específicamente el subclade T2f1a1. [15]
Henry Louis Gates Jr. pertenece al haplogrupo T2b2. [16]
Nicolás II de Rusia
Se ha demostrado que el último zar ruso , Nicolás II , pertenece al haplogrupo T, específicamente al subclado T2 (Ivanov 1996). Suponiendo que todos los pedigríes relevantes sean correctos, esto incluye a todos los descendientes de línea femenina de su antepasada de línea femenina Bárbara de Celje (1390-1451), esposa de Segismundo, emperador del Sacro Imperio Romano Germánico. Esto incluye a un gran número de nobles europeos, incluidos Jorge I de Gran Bretaña y Federico Guillermo I de Prusia (a través de la electora Sofía de Hannover ), Carlos I de Inglaterra , Jorge III del Reino Unido , Jorge V del Reino Unido , Carlos X Gustavo de Suecia , Gustavo Adolfo de Suecia , Mauricio de Nassau, príncipe de Orange , Olaf V de Noruega y Jorge I de Grecia .
Véase también
Genética
Árbol de la estructura principal del ADNmt
Referencias
Notas al pie
Citas
- ^ ab Asmahan Bekada; Lara R. Arauna; Tahria Deba; Francisco Calafell; Soraya Benhamamouch; David Comas (24 de septiembre de 2015). "Heterogeneidad genética en poblaciones humanas argelinas". MÁS UNO . 10 (9): e0138453. Código Bib : 2015PLoSO..1038453B. doi : 10.1371/journal.pone.0138453 . PMC 4581715 . PMID 26402429.; Tabla S5
- ^ ab Černý, Viktor; et al. (2009). "Out of Arabia—the settle of island Soqotra as revealed by mitochondrial and Y cromosoma gene diversity" (PDF) . American Journal of Physical Anthropology . 138 (4): 439–447. doi :10.1002/ajpa.20960. PMID 19012329. Archivado desde el original (PDF) el 6 de octubre de 2016 . Consultado el 13 de junio de 2016 .
- ^ Bryan Sykes (2001). Las siete hijas de Eva . Londres; Nueva York: Bantam Press. ISBN 978-0393020182.
- ^ "Ascendencia materna". Oxford Ancestors. Archivado desde el original el 15 de julio de 2017. Consultado el 7 de febrero de 2013 .
- ^ Bennett, Casey; Kaestle, Frederika A. (2010). "Investigación del ADN antiguo de Siberia occidental y la cultura Sargat". Biología Humana . 82 (2): 143-156. arXiv : 1112.2014 . doi :10.3378/027.082.0202. PMID 20649397. S2CID 54566651.
- ^ Pala, M; Olivieri, A; Aquiles, A; Accetturo, M; Metspalu, E; Reidla, M; Tamm, E; Karmín, M; Reisberg, T; Hooshiar Kashani, B; Perego, UA; Carosa, V; Gandini, F; Pereira, JB; Soares, P; Angerhofer, N; Rychkov, S; Al-Zahery, N; Carelli, V; Sanati, MH; Houshmand, M; Hatina, J; Macaulay, V; Pereira, L; Woodward, SR; Davies, W; Apuesta, C; Baird, D; Semino, O; Villems, R; Torroní, A; Richards, MB (4 de mayo de 2012). "Señales de ADN mitocondrial de la recolonización glacial tardía de Europa desde refugios del Cercano Oriente". La Revista Estadounidense de Genética Humana . 90 (5): 915–924. doi :10.1016/j.ajhg.2012.04.003. PMC 3376494 . PMID 22560092. http://haplogroup.org/sources/mitochondrial-dna-signals-of-late-glacial-recolonization-of-europe-from-near-eastern-refugia/ Archivado el 7 de febrero de 2023 en Wayback Machine.
- ^ Wilde, Sandra (2014). "Evidencia directa de selección positiva de la pigmentación de la piel, el cabello y los ojos en los europeos durante los últimos 5000 años". Actas de la Academia Nacional de Ciencias . 111 (13): 4832–4837. Bibcode :2014PNAS..111.4832W. doi : 10.1073/pnas.1316513111 . PMC 3977302 . PMID 24616518.
- ^ Kefi, Rym; et al. (2018). "Sobre el origen de los iberomaurusianos: nuevos datos basados en ADN mitocondrial antiguo y análisis filogenético de las poblaciones de Afalou y Taforalt". ADN mitocondrial parte A . 29 (1): 147–157. doi :10.1080/24701394.2016.1258406. PMID 28034339. S2CID 4490910.
- ^ Schuenemann, Verena J.; et al. (2017). "Los genomas de las momias del Antiguo Egipto sugieren un aumento de la ascendencia africana subsahariana en los períodos postrromanos". Nature Communications . 8 : 15694. Bibcode :2017NatCo...815694S. doi :10.1038/ncomms15694. PMC 5459999 . PMID 28556824.
- ^ Fregel; et al. (2018). "Los genomas antiguos del norte de África evidencian migraciones prehistóricas al Magreb tanto desde el Levante como desde Europa". bioRxiv 10.1101/191569 .
- ^ Rodríguez-Varela; et al. (2017). "Análisis genómicos de restos humanos preeuropeos de las Islas Canarias revelan una estrecha afinidad con los norteafricanos modernos". Current Biology . 27 (1–7): 3396–3402.e5. doi : 10.1016/j.cub.2017.09.059 . hdl : 2164/13526 . PMID 29107554.
- ^ Ball, Edward (2007). La cadena genética: exploración de la historia familiar a través del ADN. Simon and Schuster. pág. 233. ISBN 978-1416554257. Recuperado el 31 de mayo de 2016 .
- ^ Castro, M (2006). "Haplogrupos de ADN mitocondrial en pacientes españoles con miocardiopatía hipertrófica". Int J Cardiol . 112 (2): 202–6. doi :10.1016/j.ijcard.2005.09.008. PMID 16313983.
- ^ Chen, Michael. "Miocardiopatía hipertrófica - Enciclopedia médica". Medline Plus . Biblioteca Nacional de Medicina . Consultado el 3 de octubre de 2015 .
- ^ Conoce a los Izzards: La línea de la mamá . BBC One . 12 de marzo de 2013. 48 minutos.
- ^ Gates Jr., Henry Louis (2010). Rostros de América: cómo 12 personas extraordinarias descubrieron su pasado . New York University Press. pág. 4.
Fuentes
- Pike DA, Barton TJ, Bauer SL, Kipp E (2010). "Filogenia del haplogrupo T de ADNmt basada en secuencias mitocondriales completas". Journal of Genetic Genealogy . 6 (1). Archivado desde el original el 2016-04-22 . Consultado el 2011-10-19 .
- Malyarchuk, BA; Derenko, MV (noviembre de 1999). "Inestabilidad molecular de las secuencias del haplogrupo T mitocondrial en las posiciones de nucleótidos 16292 y 16296". Anales de genética humana . 63 (6): 489–497. doi :10.1017/S0003480099007794. PMID 11246451.
- Abu-Amero, Khaled K; Larruga, José M; Cabrera, Vicente M; González, Ana M (2008). "Estructura del ADN mitocondrial en la Península Arábiga". Biología Evolutiva del BMC . 8 (1): 45. Código bibliográfico : 2008BMCEE...8...45A. doi : 10.1186/1471-2148-8-45 . PMC 2268671 . PMID 18269758.
- Babalini, Carla; Martínez-Labarga, Cristina; Tolk, Helle-Viivi; Kivisild, Toomas; Giampaolo, Rita; Tarsi, Tiziana; Continí, Irene; Barać, Lovorka; et al. (2005). "La historia de la población de la minoría lingüística croata de Molise (sur de Italia): una visión materna". Revista europea de genética humana . 13 (8): 902–12. doi : 10.1038/sj.ejhg.5201439 . PMID 15886710.
- Bedford, Felice L (2012). "Firma sefardí en el ADN mitocondrial del haplogrupo T". Revista Europea de Genética Humana . 20 (4): 441–8. doi :10.1038/ejhg.2011.200. PMC 3306851 . PMID 22108605.
- Behar, DM; Metspalu, E; Kivisild, T; Rosset, S; Tzur, S; Hadid, Y; Yudkovsky, G; Rosengarten, D; et al. (2008). MacAulay, Vincent (ed.). "Contando a los fundadores: La ascendencia genética matrilineal de la diáspora judía". PLOS ONE . 3 (4): e2062. Bibcode :2008PLoSO...3.2062B. doi : 10.1371/journal.pone.0002062 . PMC 2323359 . PMID 18446216.
- Behar, Doron M.; Van Horno, Mannis; Rosset, Saharon; Metspalu, Mait; Loogväli, Eva-Liis; Silva, Nuño M.; Kivisild, Toomas; Torroní, Antonio; Villems, Richard (2012). "Una reevaluación" copernicana "del árbol del ADN mitocondrial humano desde su raíz". La Revista Estadounidense de Genética Humana . 90 (4): 675–84. doi :10.1016/j.ajhg.2012.03.002. PMC 3322232 . PMID 22482806.
- Belyaeva, Olga; Bermisheva, Marina; Khrunin, Andrei; Slominsky, Petr; Bebyakova, Natalia; Khusnutdinova, EK (Elza Kamilevna); Mikulich, Aleksei Ignatevich; Limborskaia, SA (Svetlana Andreevna) (2003). "Variaciones del ADN mitocondrial en poblaciones rusas y bielorrusas". Biología Humana . 75 (5): 647–60. doi :10.1353/hub.2003.0069. PMID 14763602. S2CID 23876546.
- Boattini, Alessio; Castrì, Loredana; Sarno, Stefania; Useli, Antonella; Cioffi, Manuela; Sazzini, Marco; Garagnani, Paolo; De Fanti, Sara; Pettener, Davide; Luiselli, Donata (2013). "La variación del ADNmt en África Oriental desvela la historia de los grupos afroasiáticos". Revista Estadounidense de Antropología Física . 150 (3): 375–385. doi :10.1002/ajpa.22212. PMID 23283748.
- Bosch, E.; Calafell, F.; Gonzalez-Neira, A.; Flaiz, C.; Mateu, E.; Scheil, H.-G.; Huckenbeck, W.; Efremovska, L.; et al. (2006). "Los linajes paternos y maternos en los Balcanes muestran un paisaje homogéneo a pesar de las barreras lingüísticas, excepto en el caso de los aislados Aromuns". Anales de Genética Humana . 70 (4): 459–87. doi :10.1111/j.1469-1809.2005.00251.x. PMID 16759179. S2CID 23156886.
- Brandstatter, Anita; Peterson, Christine T.; Irwin, Jodi A.; Mpoke, Solomon; Koech, Davy K.; Parson, Walther; Parsons, Thomas J. (2004). "Secuencias de la región de control del ADN mitocondrial de Nairobi (Kenia): inferencia de parámetros filogenéticos para el establecimiento de una base de datos forense". Revista Internacional de Medicina Legal . 118 (5): 294–306. doi :10.1007/s00414-004-0466-z. PMID 15248073. S2CID 19703169.
- Castrì, Loredana; Garagnani, P; Useli, A; Pettener, D; Luiselli, D (2008). "Encrucijadas kenianas: migración y flujo genético en seis grupos étnicos de África oriental". Revista de Ciencias Antropológicas . 86 : 189–192. PMID 19934476.
- Castrì, Loredana; Tofanelli, Sergio; Garagnani, Paolo; Bini, Carla; Fosella, Xenia; Pelotti, Susi; Paoli, Giorgio; Pettener, Davide; Luiselli, Donata (2009). "Variabilidad del ADNmt en dos poblaciones de habla bantú (shona y hutu) de África oriental: implicaciones para los patrones de poblamiento y migración en el África subsahariana". American Journal of Physical Anthropology . 140 (2): 302–11. doi :10.1002/ajpa.21070. PMID 19425093.
- Chinnery, PF; Mowbray, C; Patel, SK; Elson, JL; Sampson, M; Hitman, GA; McCarthy, MI; Hattersley, AT; Walker, M (2007). "Haplogrupos de ADN mitocondrial y diabetes tipo 2: un estudio de 897 casos y 1010 controles". Journal of Medical Genetics . 44 (6): e80. doi :10.1136/jmg.2007.048876. PMC 2740896 . PMID 17551080.
- Coble, Michael D.; Loreille, Odile M.; Wadhams, Mark J.; Edson, Suni M.; Maynard, Kerry; Meyer, Carna E.; Niederstätter, Harald; Berger, Cordula; et al. (2009). Hofreiter, Michael (ed.). "Misterio resuelto: la identificación de los dos niños Romanov desaparecidos mediante análisis de ADN". PLOS ONE . 4 (3): e4838. Bibcode :2009PLoSO...4.4838C. doi : 10.1371/journal.pone.0004838 . PMC 2652717 . PMID 19277206.
- Costa, MD; Cherni, L; Fernandes, V; Freitas, F; Ammar El Gaaied, AB; Pereira, L (2009). "Datos de la secuenciación completa de ADNmt de centenarios tunecinos: prueba de asociación de haplogrupos y la "media dorada" de la longevidad". Mecanismos de envejecimiento y desarrollo . 130 (4): 222–6. doi :10.1016/j.mad.2008.12.001. PMID 19133286. S2CID 6102820.
- Cvjetan, S; Tolk, HV; Lauc, LB; Colak, I; Dordević, D; Efremovska, L; Janićijević, B; Kvesić, A; et al. (2004). "Frecuencias de haplogrupos de ADNmt en el sureste de Europa: croatas, bosnios y herzegovinos, serbios, macedonios y romaníes macedonios". Collegium Antropologicum . 28 (1): 193–8. PMID 15636075.
- Derenko, M; Malyarchuk, B; Grzybowski, T; Denisova, G; Dambueva, yo; Perkova, M; Dorzhu, C; Luzina, F; et al. (2007). "Análisis filogeográfico del ADN mitocondrial en poblaciones del norte de Asia". Revista Estadounidense de Genética Humana . 81 (5): 1025–41. doi :10.1086/522933. PMC 2265662 . PMID 17924343.
- Dubut, Vicente; Chollet, Lionel; Murail, Pascal; Cartault, François; Béraud-Colomb, Eliane; Serré, Myriam; Mogentale-Profizi, Nérina (2003). "Polimorfismos del ADNmt en cinco grupos franceses: importancia del muestreo regional". Revista europea de genética humana . 12 (4): 293–300. doi : 10.1038/sj.ejhg.5201145 . PMID 14694359.
- Fernández, Verónica; Alshamali, Farida; Alves, Marco; Costa, Marta D.; Pereira, Joana B.; Silva, Nuño M.; Cherni, Lotfi; Harich, Nourdin; et al. (2012). "La cuna árabe: reliquias mitocondriales de los primeros pasos a lo largo de la ruta sur fuera de África". La Revista Estadounidense de Genética Humana . 90 (2): 347–355. doi :10.1016/j.ajhg.2011.12.010. PMC 3276663 . PMID 22284828.
- Finnila, JS; Finnila, S; Majamaa, K (2001). "Selección específica de linaje en el mtADN humano: Falta de polimorfismos en un segmento del gen MTND5 en el haplogrupo J". Biología molecular y evolución . 20 (12): 2132–42. doi : 10.1093/molbev/msg230 . PMID 12949126.
- Gasparre, G.; Porcelli, AM; Bonora, E.; Pennisi, LF; Toller, M.; Iommarini, L.; Ghelli, A.; Moretti, M.; et al. (2007). "Las mutaciones disruptivas del ADN mitocondrial en las subunidades del complejo I son marcadores del fenotipo oncocítico en los tumores tiroideos". Actas de la Academia Nacional de Ciencias . 104 (21): 9001–9006. Bibcode :2007PNAS..104.9001G. doi : 10.1073/pnas.0703056104 . PMC 1885617 . PMID 17517629.
- Gonder, MK; Mortensen, HM; Reed, FA; De Sousa, A.; Tishkoff, SA (2006). "Análisis de la secuencia genómica de ADNmt completo de linajes africanos antiguos". Biología molecular y evolución . 24 (3): 757–68. doi : 10.1093/molbev/msl209 . PMID 17194802.
- González, Ana M.; MacEira, Benito M.; Pérez, Estefanía; Cabrera, Vicente M.; López, Alfonso J.; Larruga, José M. (2012). "Genética, medio ambiente y enfermedad renal terminal relacionada con la diabetes en Canarias". Pruebas genéticas y biomarcadores moleculares . 16 (8): 859–64. doi :10.1089/gtmb.2011.0207. PMC 3422557 . PMID 22480375.
- Hartmann, A; Thieme, M; Nanduri, LK; Stempfl, T; Moehle, C; Kivisild, T; Oefner, PJ (2009). "Validación de la resecuenciación basada en microarrays de 93 genomas mitocondriales de todo el mundo". Human Mutation . 30 (1): 115–22. doi :10.1002/humu.20816. PMID 18623076. S2CID 205918494.
- Hassan, Hisham Yousif (2009). "Patrones genéticos de la variación del ADN mitocondrial y del cromosoma Y, con implicaciones para el poblamiento del Sudán". Universidad de Jartum. Archivado desde el original (PDF) el 2020-11-10 . Consultado el 2016-04-18 .
- Helgason, Agnar; Hickey, Eileen; Goodacre, Sara; Bosnes, Vidar; Stefánsson, Kári; Ward, Ryk; Sykes, Bryan (2001). "ADNmt y las islas del Atlántico Norte: estimación de las proporciones de ascendencia nórdica y gaélica". The American Journal of Human Genetics . 68 (3): 206–15. doi :10.1086/318785. PMC 1274484 . PMID 11179019.
- Hofreiter, Linea; Lynnerup, Niels; Siegismund, Hans R.; Kivisild, Toomas; Dissing, Jørgen (2010). Hofreiter, Michael (ed.). "Diversidad genética entre las poblaciones nórdicas antiguas". PLOS ONE . 5 (7): e11898. Bibcode :2010PLoSO...511898M. doi : 10.1371/journal.pone.0011898 . PMC 2912848 . PMID 20689597.
La aparición general de haplogrupos no se desvió de los escandinavos actuales, sin embargo, el haplogrupo I fue significativamente más frecuente entre los daneses antiguos (promedio 13%) que entre los daneses y escandinavos actuales (~2,5%), así como entre otras muestras de población antigua informadas. Por lo tanto, el haplogrupo I podría haber sido un antiguo tipo escandinavo del sur "diluido" por eventos de inmigración posteriores.
- Ivanov, Pavel L.; Wadhams, Mark J.; Roby, Rhonda K.; Holland, Mitchell M.; Weedn, Victor W.; Parsons, Thomas J. (1996). "La heteroplasmia de la secuencia de ADN mitocondrial en el Gran Duque de Rusia Georgij Romanov establece la autenticidad de los restos del Zar Nicolás II". Nature Genetics . 12 (4): 417–20. doi :10.1038/ng0496-417. PMID 8630496. S2CID 287478.
- Janssen, GM; Neu, A; 't Hart, LM; Van De Sande, CM; Antonie Maassen, J (2006). "Nuevas variantes de longitud del ADN mitocondrial e inestabilidad genética en una familia con diabetes y sordera". Experimental and Clinical Endocrinology & Diabetes . 114 (4): 168–74. doi :10.1055/s-2006-924066. PMID 16705548.
- Keyser, Christine; Bouakaze, Caroline; Crubézy, Eric; Nikolaev, Valery G.; Montagnon, Daniel; Reis, Tatiana; Ludes, Bertrand (2009). "El ADN antiguo proporciona nuevos conocimientos sobre la historia de los pueblos kurganos del sur de Siberia". Genética humana . 126 (3): 395–410. doi :10.1007/s00439-009-0683-0. PMID 19449030. S2CID 21347353.
- Kivisild, T; Reidla, M; Metspalu, E; Rosa, A; Brehm, A; Pennarun, E; Parik, J; Geberhiwot, T; et al. (2004). "Herencia del ADN mitocondrial etíope: seguimiento del flujo de genes a través y alrededor de la puerta de las lágrimas". Revista Estadounidense de Genética Humana . 75 (5): 752–70. doi :10.1086/425161. PMC 1182106 . PMID 15457403.
- Knight, A; Underhill, PA; Mortensen, HM; Zhivotovsky, LA; Lin, AA; Henn, BM; Louis, D; Ruhlen, M; Mountain, JL (2003). "La divergencia del cromosoma Y africano y el ADNmt proporciona información sobre la historia de los lenguajes de clic". Current Biology . 13 (6): 464–73. doi : 10.1016/S0960-9822(03)00130-1 . PMID 12646128. S2CID 52862939.
- Krings, M; Salem, AE; Bauer, K; Geisert, H; Malek, AK; Chaix, L; Simon, C; Welsby, D; et al. (1999). "Análisis de ADNmt de las poblaciones del valle del río Nilo: ¿un corredor genético o una barrera para la migración?". American Journal of Human Genetics . 64 (4): 1166–1176. doi :10.1086/302314. PMC 1377841 . PMID 10090902.
- Martina Kujanova; Luisa Pereira; Verónica Fernandes; Joana B. Pereira; Viktor Cerny (2009). "Insumo genético neolítico del Cercano Oriente en un pequeño oasis del desierto occidental egipcio". American Journal of Physical Anthropology . 140 (2): 336–346. doi :10.1002/ajpa.21078. PMID 19425100.
- Lalueza-Fox, C; Sampietro, ML; Gilbert, MT; Castri, L; Facchini, F; Pettener, D; Bertranpetit, J (2004). "Descifrando las migraciones en la estepa: secuencias de ADN mitocondrial de antiguos asiáticos centrales". Actas: Ciencias Biológicas . 271 (1542): 941–7. doi :10.1098/rspb.2004.2698. PMC 1691686 . PMID 15255049.
- Maca-Meyer, N; González, AM; Larruga, JM; Flores, C; Cabrera, VM (2001). "Los principales linajes mitocondriales genómicos delinean las expansiones humanas tempranas". BMC Genetics . 2 : 13. doi : 10.1186/1471-2156-2-13 . PMC 55343 . PMID 11553319.
- MacAulay, Vicente; Richards, Martín; Hickey, Eileen; Vega, Emilce; Cruciani, Fulvio; Guida, Valentina; Scozzari, Rosaria; Bonné-Tamir, Batsheva; et al. (1999). "El árbol emergente de los ADNmt de Eurasia occidental: una síntesis de secuencias de regiones de control y RFLP". La Revista Estadounidense de Genética Humana . 64 (1): 232–49. doi :10.1086/302204. PMC 1377722 . PMID 9915963.
- Malyarchuk, BA; Derenko, MV (1999). "Inestabilidad molecular de las secuencias del haplogrupo T mitocondrial en las posiciones de nucleótidos 16292 y 16296". Anales de genética humana . 63 (6): 489–497. doi : 10.1046/j.1469-1809.1999.6360489.x . PMID 11246451. S2CID 45007072.
- Malyarchuk, BA; Derenko, MV (2001). "Variabilidad del ADN mitocondrial en rusos y ucranianos: implicaciones para el origen de los eslavos orientales". Anales de genética humana . 65 (parte 1): 63–78. doi : 10.1046/j.1469-1809.2001.6510063.x . PMID 11415523. S2CID 9392520.
- Malyarchuk, BA; Grzybowski, T; Derenko, MV; Czarny, J; Drobnic, K; Miścicka-Sliwka, D (2003). "Variabilidad del ADN mitocondrial en bosnios y eslovenos". Anales de genética humana . 67 (parte 5): 412–25. doi :10.1046/j.1469-1809.2003.00042.x. PMID 12940915. S2CID 2105448.
- Malyarchuk, B; Derenko, M; Denisova, G; Kravtsova, O (2010). "Diversidad mitogenómica en tártaros de la región Volga-Ural de Rusia". Biología molecular y evolución . 27 (10): 2220–6. doi : 10.1093/molbev/msq065 . PMID 20457583.
- Martínez-Cruz, B.; Harmant, C.; Platt, DE; Haak, W.; Manry, J.; Ramos-Luis, E.; Soria-Hernanz, DF; Bauduer, F.; et al. (2012). "Evidencia de estructura genética tribal prerromana en vascos a partir de marcadores heredados uniparentalmente". Biología Molecular y Evolución . 29 (9): 2211–22. doi : 10.1093/molbev/mss091 . hdl : 10261/112478 . PMID 22411853.
- Melchior, Linea; Kivisild, Toomas; Lynnerup, Niels; Dissing, Jørgen (2008). Ahmed, Niyaz (ed.). "Evidencia de ADN auténtico en esqueletos de la era vikinga danesa que no han sido tocados por humanos durante 1000 años". PLOS ONE . 3 (5): e2214. Bibcode :2008PLoSO...3.2214M. doi : 10.1371/journal.pone.0002214 . PMC 2386972 . PMID 18509537.
- Metspalu, Mait; Kivisild, Toomas; Metspalu, Ene; Parik, Jüri; Hudjashov, Georgi; Kaldma, Katrin; Serk, Piia; Karmin, Monika; Behar, Doron M; Gilbert, M Thomas P; Endicott, Phillip; Mastana, Sarabjit; Papiha, Surinder S; Skorecki, Karl; Torroni, Antonio; Villems, Richard (2004). "La mayoría de los límites de ADNmt existentes en el sur y suroeste de Asia probablemente se formaron durante el asentamiento inicial de Eurasia por humanos anatómicamente modernos". BMC Genetics . 5 : 26. doi : 10.1186/1471-2156-5-26 . PMC 516768 . PMID 15339343.
- Mikkelsen, Martín; Sorensen, Erik; Rasmussen, Erik Michael; Morling, Niels (2010). "Variación del ADN mitocondrial HV1 y HV2 en daneses". Ciencia Forense Internacional: Genética . 4 (4): e87–8. doi :10.1016/j.fsigen.2009.07.007. PMID 20457038.
- Mishmar, D; Ruiz-Pesini, E; Golik, P; MacAulay, V; Clark, AG; Hosseini, S; Brandon, M; Easley, K; et al. (2003). "La selección natural moldeó la variación regional del mtADN en humanos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 100 (1): 171–6. Bibcode :2003PNAS..100..171M. doi : 10.1073/pnas.0136972100 . PMC 140917 . PMID 12509511.
- Musilová, Eliška; Fernández, Verónica; Silva, Nuño M.; Soares, Pedro; Alshamali, Farida; Harich, Nourdin; Cherni, Lotfi; Gaaied, Amel Ben Ammar El; et al. (2011). "La historia de la población de los intercambios genéticos del Mar Rojo entre la Península Arábiga y África Oriental señalados en el haplogrupo HV1 del ADN mitocondrial". Revista Estadounidense de Antropología Física . 145 (4): 592–8. doi :10.1002/ajpa.21522. PMID 21660931.
- Nikitin, Alexey G.; Kochkin, Igor T.; junio, Cynthia M.; Willis, Catalina M.; McBain, Ian; Videiko, Mykhailo Y. (2009). "Variación de la secuencia de ADN mitocondrial en las poblaciones Boyko, Hutsul y Lemko de las tierras altas de los Cárpatos". Biología Humana . 81 (1): 43–58. doi :10.3378/027.081.0104. PMID 19589018. S2CID 45791162.
- No, Amy L.; Al-Meeri, Ali; Raaum, Ryan L.; Sánchez, Luisa F.; Mulligan, Connie J. (2011). "El ADN mitocondrial revela distintas historias evolutivas de las poblaciones judías en Yemen y Etiopía". Revista Estadounidense de Antropología Física . 144 (1): 1–10. doi :10.1002/ajpa.21360. PMID 20623605.
- Palanichamy, MG; Sun, C; Agrawal, S; Bandelt, HJ; Kong, QP; Khan, F; Wang, CY; Chaudhuri, TK; et al. (2004). "Filogenia del macrohaplogrupo N del ADN mitocondrial en la India, basada en la secuenciación completa: implicaciones para el poblamiento del sur de Asia". American Journal of Human Genetics . 75 (6): 966–78. doi :10.1086/425871. PMC 1182158 . PMID 15467980.
- Pereira, L; Gonçalves, J; Franco-Duarte, R; Silva, J; Rocha, T; Arnold, C; Richards, M; MacAulay, V (2007). "No hay evidencia de un papel del mtADN en la motilidad de los espermatozoides: datos de la secuenciación completa de machos astenozoospérmicos". Biología molecular y evolución . 24 (3): 868–74. doi : 10.1093/molbev/msm004 . PMID 17218641.
- Pericić, M; Barać Lauc, L; Martinović Klarić, yo; Janićijević, B; Rudan, P (2005). "Revisión de la herencia genética croata revelada por el ADN mitocondrial y los linajes del cromosoma Y". Revista médica croata . 46 (4): 502–13. PMID 16100752.
- Pichler, Irene; Fuchsberger, Christian; Platzer, Christa; Çalişkan, Minal; Marroni, Fabio; Pramstaller, Peter P; Ober, Carole (2010). "Dibujando la historia de la población huterita en un paisaje genético: inferencia a partir de genotipos del cromosoma Y y del ADNmt". Revista Europea de Genética Humana . 18 (4): 463–70. doi :10.1038/ejhg.2009.172. PMC 2987252 . PMID 19844259.
- Poloni, Estella S.; Naciri, Yamama; Bucho, Rute; Niba, Régine; Kervaire, Bárbara; Excoffier, Laurent; Langaney, André; Sánchez-Mazas, Alicia (2009). "Evidencia genética de la complejidad de la diferenciación étnica y la historia en África oriental". Anales de genética humana . 73 (6): 582–600. doi :10.1111/j.1469-1809.2009.00541.x. PMID 19706029. S2CID 2488794.
- Pope, AM; Carr, SM; Smith, KN; Marshall, HD; Marshall, HD (2011). "Variación mitogenómica y microsatélite en los descendientes de la población fundadora de Terranova: Alta diversidad genética en una población históricamente aislada". Genoma . 54 (2): 110–9. doi :10.1139/G10-102. PMID 21326367.
- Quintana-Murci, Lluís; Chaix, Raphaëlle; Wells, R. Spencer; Behar, Doron M.; Sayar, Hamid; Scozzari, Rosaria; Rengo, Chiara; Al-Zahery, Nadia; et al. (2004). "Donde Occidente se encuentra con Oriente: el complejo paisaje del ADNmt en el corredor del suroeste y centro asiático". The American Journal of Human Genetics . 74 (5): 827–45. doi :10.1086/383236. PMC 1181978 . PMID 15077202.
- Richards, Martin; MacAulay, Vincent; Hickey, Eileen; Vega, Emilce; Sykes, Bryan; Guida, Valentina; Rengo, Chiara; Sellitto, Daniele; et al. (2000). "Rastreo de linajes fundadores europeos en el acervo de ADNmt del Cercano Oriente". The American Journal of Human Genetics . 67 (5): 1251–76. doi :10.1016/S0002-9297(07)62954-1. PMC 1288566 . PMID 11032788.
- Richards, Martin; Rengo, Chiara; Cruciani, Fulvio; Gratrix, Fiona; Wilson, James F.; Scozzari, Rosaria; MacAulay, Vincent; Torroni, Antonio (2003). "Extenso flujo genético mediado por mujeres desde el África subsahariana hacia las poblaciones árabes del Cercano Oriente". The American Journal of Human Genetics . 72 (4): 1058–64. doi :10.1086/374384. PMC 1180338 . PMID 12629598.
- Rogaev, EI; Grigorenko, AP; Moliaka, YK; Faskhutdinova, G.; Goltsov, A.; Lahti, A.; Hildebrandt, C.; Kittler, ELW; Morozova, I. (2009). "Identificación genómica en el caso histórico de la familia real de Nicolás II". Actas de la Academia Nacional de Ciencias . 106 (13): 5258–63. Bibcode :2009PNAS..106.5258R. doi : 10.1073/pnas.0811190106 . PMC 2664067 . PMID 19251637.
- Ruiz-Pesini, Eduardo; Lapeña, Ana Cristina; Díez-Sánchez, Carmen; Pérez-Martos, Acisclo; Montoya, Julio; Álvarez, Enrique; Díaz, Miguel; Urriés, Antonio; et al. (2000). "Haplogrupos de ADNmt humano asociados con una motilidad de espermatozoides alta o reducida". La Revista Estadounidense de Genética Humana . 67 (3): 682–96. doi :10.1086/303040. PMC 1287528 . PMID 10936107.
- Shlush, Liran I.; Behar, Doron M.; Yudkovsky, Guennady; Templeton, Alan; Hadid, Yarin; Basis, Fuad; Hammer, Michael; Itzkovitz, Shalev; Skorecki, Karl (2008). Gemmell, Neil John (ed.). "Los drusos: un refugio genético poblacional del Cercano Oriente". PLOS ONE . 3 (5): e2105. Bibcode :2008PLoSO...3.2105S. doi : 10.1371/journal.pone.0002105 . PMC 2324201 . PMID 18461126.
- Soares, P.; Alshamali, F.; Pereira, JB; Fernández, V.; Silva, Nuevo México; Alfonso, C.; Costa, MD; Musílova, E.; et al. (2011). "La expansión del haplogrupo L3 del ADNmt dentro y fuera de África". Biología Molecular y Evolución . 29 (3): 915–27. doi : 10.1093/molbev/msr245 . PMID 22096215.
- Stanger, Olaf; Müller, Edith; Zimmermann, Franz; Wiesbauer, Martina; Mayr, Johannes A.; Paulweber, Bernhard; Iglseder, Bernhard; Renner, Wilfried; et al. (2007). "30 El haplogrupo mitocondrial T está asociado con la enfermedad de la arteria coronaria". Mitocondria . 7 (6): 412. doi :10.1016/j.mito.2007.08.034.
- Stone, AC; Starrs, JE; Stoneking, M (2001). "Análisis de ADN mitocondrial de los presuntos restos de Jesse James". Revista de Ciencias Forenses . 46 (1): 173–6. doi :10.1520/JFS14932J. PMID 11210907.
- Terreros, Maria C; Rowold, Diane J; Mirabal, Sheyla; Herrera, Rene J (2011). "ADN mitocondrial y estratificación del cromosoma Y en Irán: relación entre Irán y la Península Arábiga". Journal of Human Genetics . 56 (3): 235–46. doi : 10.1038/jhg.2010.174 . PMID 21326310.
- Thomas, Mark G; Barnes, Ian; Weale, Michael E; Jones, Abigail L; Forster, Peter; Bradman, Neil; Pramstaller, Peter P (2008). "Nueva evidencia genética apoya el aislamiento y la deriva en las comunidades ladinas de los Alpes del Tirol del Sur, pero no un origen antiguo en Oriente Medio". Revista Europea de Genética Humana . 16 (1): 124–34. doi : 10.1038/sj.ejhg.5201906 . PMID 17712356.
- Tishkoff, SA; Gonder, MK; Henn, BM; Mortensen, H.; Knight, A.; Gignoux, C.; Fernandopulle, N.; Lema, G.; et al. (2007). "Historia de las poblaciones de habla chasquida de África inferida a partir de la variación genética del ADNmt y del cromosoma Y". Biología molecular y evolución . 24 (10): 2180–95. doi : 10.1093/molbev/msm155 . PMID 17656633.
- Topf, AL; Gilbert, MT; Dumbacher, JP; Hoelzel, AR (2005). "Rastreo de la filogeografía de las poblaciones humanas en Gran Bretaña basada en genotipos de ADNmt de los siglos IV al XI". Biología molecular y evolución . 23 (1): 152–61. doi : 10.1093/molbev/msj013 . PMID 16151183.
- Torroni, A; Huoponen, K; Francalacci, P; Petrozzi, M; Morelli, L; Scozzari, R; Obinu, D; Savontaus, ML; Wallace, DC (1996). "Clasificación de los mtADN europeos a partir de un análisis de tres poblaciones europeas". Genética . 144 (4): 1835–50. doi :10.1093/genetics/144.4.1835. PMC 1207732 . PMID 8978068.
- van Oven, Mannis; Kayser, Manfred (2009). "Árbol filogenético actualizado y completo de la variación global del ADN mitocondrial humano". Human Mutation . 30 (2): E386–94. doi : 10.1002/humu.20921 . PMID 18853457. S2CID 27566749.
Sitios web
- Behar; Family Tree DNA (2012). «Comunidad de ADNmt». Archivado desde el original el 2018-01-02 . Consultado el 2013-01-12 .
Lectura adicional
- Černý, Viktor; Pereira, Luisa; Kujanová, Martina; VašÍková, Alžběta; Hájek, Martín; Morris, Miranda; Mulligan, Connie J. (2009). "Fuera de Arabia: el asentamiento de la isla Soqotra según lo revelado por la diversidad genética mitocondrial y del cromosoma Y". Revista Estadounidense de Antropología Física . 138 (4): 439–47. doi :10.1002/ajpa.20960. PMID 19012329.
- Kitchen, A.; Ehret, C.; Assefa, S.; Mulligan, CJ (2009). "El análisis filogenético bayesiano de las lenguas semíticas identifica un origen semítico en la Edad del Bronce Temprano en Oriente Próximo". Actas de la Royal Society B: Biological Sciences . 276 (1668): 2703–10. doi :10.1098/rspb.2009.0408. PMC 2839953 . PMID 19403539.
- Petit-Maire, Nicole; Bouysse, Philippe (2000). "Registros geológicos del pasado reciente, una clave para los entornos mundiales del futuro cercano" (PDF) . Episodios . 23 (4): 230–246. doi : 10.18814/epiiugs/2000/v23i4/001 .
Enlaces externos
- General
- Sitio de ADN mitocondrial de Ian Logan
- El árbol filogenético de Mannis van Oven
- Base de datos de ADN mitocondrial de participación pública del Proyecto Genográfico
- Haplogrupo T
- Haplogrupo T de YFull MTree
- Haplogrupo T de MITOMAP
- Árbol de haplos del ADNmt de FamilyTreeDNA : haplogrupo T
- Propagación del haplogrupo T, de National Geographic
- Genealogía genética: una perspectiva personal sobre Tara, Karelia y Kent, Inglaterra
- Análisis de una secuencia del haplogrupo T (T5/T2)
- Redes filogenéticas para el haplogrupo T del ADNmt humano Archivado el 31 de mayo de 2008 en Wayback Machine
- Redes filogenéticas para el haplogrupo T del ADNmt humano Archivado el 9 de mayo de 2008 en Wayback Machine
- Proyecto de investigación de secuencia genómica completa del haplogrupo T del ADNmt
