Mapa de SP1
| Mapa de SP1 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificadores | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Alias | SPMAP1 , marco de lectura abierto 98 del cromosoma 17, proteína 1 asociada a los microtúbulos del esperma | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificaciones externas | MGI : 1919465; HomoloGene : 19140; GeneCards : SPMAP1; OMA : SPMAP1 - ortólogos | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidatos | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
La proteína 1 asociada a los microtúbulos espermáticos es una proteína que en los humanos está codificada por el gen SPMAP1 . La proteína se deriva del cromosoma 17 del Homo sapiens . [5] El gen SPMAP1 consta de una secuencia de 6.302 bases. Su ARNm tiene tres exones y ningún sitio de empalme alternativo. La proteína tiene 154 aminoácidos, sin niveles anormales de aminoácidos. [6] SPMAP1 tiene un dominio de función desconocida (DUF4542) y tiene un peso de 17,6 kDa. [7] [8] SPMAP1 no pertenece a ninguna otra familia ni tiene isoformas. [9] La proteína tiene ortólogos con un alto porcentaje de similitud en mamíferos y reptiles. La proteína tiene ortólogos adicionales distantemente relacionados en todo el reino de los metazoos , que culminan con la familia de las esponjas. [10]
Al igual que la mayoría de las proteínas, se sabe que SPMAP1 se expresa en gran medida en los testículos. [11] También se sabe que la proteína tiene niveles elevados en el cáncer. [11] Se ha demostrado que la proteína se expresa cerca o dentro de los filamentos intermedios y el nucléolo . [11] Además, SPMAP1 tiene factores de transcripción que también son activos en las células madre hematopoyéticas , el sistema inmunológico y el sistema cardiovascular , entre otros. [12] El gen se sobreexpresa en muchos tipos de cáncer, incluido el carcinoma de células claras renal y el carcinoma de células escamosas pulmonar. [13] El análisis de motivos y factores de transcripción apunta a que SPMAP1 desempeña un papel en la proliferación, especialmente en la proliferación de células inmunes.
Gene
Fondo
El gen SPMAP1 consta de 6.303 bases. Tiene tres exones y dos intrones grandes. El gen no tiene sitios de empalme alternativos. [14] La secuencia 5' UTR de SPMAP1 está altamente conservada en primates. No se pudo determinar ninguna coincidencia 5' UTR en animales no mamíferos. [15] [16] SPMAP1 tiene 11 repeticiones Alu . [17]
Potenciadores
GeneCards determinó que SPMAP1 tiene cinco secuencias potenciadoras . El papel de las secuencias puede proporcionar información sobre la función de SPMAP1. Cuatro de los cinco potenciadores están activos en el timo. Los cinco potenciadores están activos en las células madre embrionarias humanas H1. Además, los cinco potenciadores están activos en iPS DF 19.11 derivada de fibroblastos del prepucio. [18]
Factores de transcripción
El promotor SPMAP1 tiene muchos sitios de unión de factores de transcripción. [19] Los factores de transcripción de SPMAP1 se encuentran comúnmente en células hematopoyéticas, tejido conectivo, tejido cardiovascular y el sistema inmunológico. La presencia de factores de transcripción similares a Krueppel sugiere un papel para SPMAP1 en la proliferación o apoptosis. La presencia de SMAD indica una participación en la vía TGF-β, mientras que la presencia de factores de transcripción relacionados con Myc indica una posible función de proliferación de la proteína. Además, otros factores de transcripción de SPMAP1 , como RBPJ-Kappa, están involucrados en la proliferación y la señalización.
Variantes
Se encontraron numerosos SNP en el 5' UTR, 3' UTR y la región codificante de SPMAP1 . [20] Se encontraron pocos SNP en regiones altamente conservadas. En total, se encontraron cuatro SNP en los aminoácidos altamente conservados. Se encontró un SNP en la secuencia del codón de inicio. De estos cinco, tres tenían un SNP en la tercera posición del codón. Debido a la hipótesis del bamboleo , tres de los cinco SNP no tendrían efecto en la estructura general de la proteína.
ARNm
SPMAP1 no tiene ningún sitio de unión de miRNA. [21] Su ARNm tiene una abundancia baja (0,44%). [22] La secuencia de ARNm tiene tres hexaloops, ninguno de los cuales es significativo. [23]
Proteína
Estructura primaria
SPMAP1 es una proteína de 17,6 kDa. [8] Los ortólogos distantes son de 5 a 6 kDa más grandes, pero algunas de las discrepancias provienen de una secuencia NLS agregada, que el Homo sapiens no tiene No hay grupos de carga positiva o negativa. No hay componentes transmembrana. El punto isoeléctrico es 9,80 / 17564,67 pI/Mw. [24] SPMAP1 es hidrófobo y soluble.

Estructura secundaria y terciaria
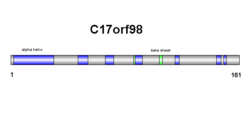
La estructura secundaria de SPMAP1 consta de láminas beta y hélices alfa (ver diagrama a la derecha). Los resultados se confirman en la estructura terciaria, sin embargo, la cantidad de hélices alfa y láminas beta difiere ligeramente (ver diagrama a la derecha).
Motivos y sitios de unión
No hay péptidos señal N-terminales. No se encontraron motivos de escisión. No hay señales de retención de membrana de RE, ni señal de orientación peroxisomal. SKL2 no está presente, por lo tanto, no hay una señal de peroxisoma secundaria. No hay señales de orientación vacuolar. No hay motivos de unión de ARN o motivos de unión de actina de tipo actina. No hay patrón de N-miristoilación ni patrones de prenilación. [25]

El buscador de quinasas de Cuckoo determinó los sitios de unión de quinasas para SPMAP1. Hay muchos sitios de fosforilación de quinasas de serina/treonina y tirosina. [26] Los sitios de unión de quinasas de serina y treonina son los más frecuentes por encima del umbral estadísticamente significativo. No hay sitios de SUMOilación. [27] El gen SPMAP1 tiene seis sitios en la secuencia de posibles sitios de O-GlcNAc . [28] Los sitios de aminoácidos de O-GlcNAc altamente conservados son 24, 32, 117 y 142. La modificación postraduccional de O-GlcNAc ocurre en residuos Ser/Thr, específicamente en oncogenes, supresores de tumores y proteínas involucradas en la señalización del factor de crecimiento. [29]
SPMAP1 tiene un motivo Caspasa3/7, donde la Caspasa 3 o 7 se escindirían. [30] Esto respalda la idea de que SPMAP1 está involucrado en la proliferación, ya que una caspasa proapoptótica querría destruir cualquier proteína que impulse la proliferación. La proteína también tiene un motivo donde se une la peptidil-prolil cis-trans isomerasa NIMA interactuando 1 (Pin1). [30] La regulación positiva de Pin1 está involucrada en el cáncer y los trastornos inmunológicos. [31] Esto respalda la afirmación de que SPMAP1 está involucrado en el cáncer, las células inmunes y quizás los cánceres del sistema inmunológico. Además, la proteína SPMAP1 tiene un sitio IBM, donde se unen los inhibidores de la apoptosis (IAP). [30] Esto nuevamente respalda la idea de que SPMAP1 está involucrado en la inhibición de la apoptosis y, lógicamente, en el impulso del cáncer. Además, SPMAP1 tiene motivos donde se une el dominio SH2 de GRB2 . GRB2 es una proteína adaptadora involucrada en la vía de señalización RAS, una vía que cuando se desregula impulsa una proliferación descontrolada.
Secuencia de aminoácidos
Es posible que se haya producido una duplicación en las posiciones 59-71.
Homo sapiens
MAYLSECRLRLEKGFILDGVAVSTAARAYGRSRPKLWSAIPPYNAQQDYHARSYFQ SHVVPPLLRVVPPLLRKTDQDHGGTGRDGWIVDYIHIFGQGQRYLNRRRNWAGTGHS LQQVTGHDHYNADLKPIDGFNGRFGYRRNTPALRQSTSVFGEVTHFPLF
Proteínas asociadas
No se conocen proteínas asociadas. [32] [33] [34] [35]
Expresión
La abundancia de proteínas en el organismo entero del Homo sapiens es bastante baja. No hay datos disponibles para otras especies. [36] El Atlas cerebral de Allen no ofrece ningún atlas cerebral para SPMAP1. [37]
Localización subcelular
Se ha descubierto que la proteína SPMAP1 se expresa en los filamentos intermedios y los nucléolos. [38] Sigma-Aldrich ofrece un anticuerpo SPMAP1. [39] Además, SPMAP1 se localiza en el citoplasma. Los ortólogos de SPMAP1 distantemente relacionados en organismos como Macrostomum lignano y Amphimedon queenslandica exhiben expresión nuclear. [40] Las señales de localización nuclear están presentes en organismos distantemente relacionados en sitios no conservados. Los resultados de la predicción k-NN son la localización citoplasmática. [41] SPMAP1 no es un péptido señal. [42] La proteína es soluble. [43]
Tejido
Al igual que la mayoría de las proteínas, la proteína SPMAP1 se expresa en gran medida en los testículos. [44] La proteína se expresa en tejidos adultos, así como en tejido fetal. Se ha descubierto que la proteína se expresa levemente en el tejido conectivo. [45] Además, se ha observado expresión en el esperma, las células epiteliales mamarias y varias células del sistema inmunológico. [46]
Importancia clínica
Cáncer
La expresión de proteínas está elevada en muchos pacientes con cáncer. Específicamente, se ha demostrado que la expresión de proteínas es alta en el cáncer colorrectal, de mama, de próstata y de pulmón. [47] SPMAP1 también se expresa en el cáncer papilar de tiroides. [48] Además, se encontraron mutaciones en SPMAP1 en cáncer de endometrio, estómago, coloratura y riñón. [49] La expresión de SPMAP1 está elevada en pacientes con cáncer con BRCA. En pacientes con carcinoma renal de células claras, la expresión de SPMAP1 disminuyó drásticamente en comparación con el estado no canceroso. [13] En el 80% de los pacientes con carcinoma de células renales cromófobo, estaba presente al menos una duplicación del gen SPMAP1. [13]
Otras condiciones
La expresión de proteínas es menor en los machos con teratozoospermia en comparación con los que no la padecen. [50] Se han realizado muchos experimentos de Geo Profile con SPMAP1, sin embargo, ninguno arrojó datos que mostraran un cambio significativo en la expresión. [51]
Evolución
La SPMAP1 es una proteína de mutación lenta. Se parece al citocromo c en su tasa de divergencia, determinada por las ecuaciones del reloj molecular. [52]

Parálogos
No se conocen parálogos del Homo sapiens para SPMAP1. [53]
Ortólogos
La proteína SPMAP1 tiene ortólogos distantes adicionales en todo el reino de los metazoos. Su pariente más lejano está en la familia de las esponjas. No se conoce ningún ortólogo en ctenóforos, nematodos, bacterias, hongos, plantas o pez cebra. [10] Solo hay dos peces con el gen SPMAP1 . Los organismos modelo como Caenorhabditis elegans y Drosophila melanogaster no tienen el gen.
Ortólogos de SPMAP1 [10]
| Secuencia # | Género y especie | Nombre común | Número de acceso | Longitud de la proteína | División MYA | Id. de secuencia | Confianza |
|---|---|---|---|---|---|---|---|
| 1 | Homo sapiens | Humano | Número de serie 001073934 | 154 | 0 | 100% | n / A |
| 2 | Camello ferus | Camello bactriano salvaje | XP_006176436 | 154 | 96 | 83% | 2.00E-94 |
| 3 | Pteropus alecto | Zorro volador negro | XP_006924784 | 154 | 96 | 81% | 1.00E-92 |
| 4 | Lipotes vexilifer | Delfín del río Yangtze | XP_007465208 | 154 | 96 | 81% | 6.00E-89 |
| 5 | Condilura cristata | Topo de nariz estrellada | XP_004684322 | 154 | 96 | 75% | 5.00E-78 |
| 6 | Myotis brandtii | El bate de Brandt | EPQ05064 | 171 | 96 | 78% | 6.00E-78 |
| 7 | Marmata marmata marmata | Marmota alpina | XP_015362150.1 | 154 | 90 | 81% | 3.00E-94 |
| 8 | Octodon degus | Roedor chileno | XP_004633931 | 153 | 90 | 73% | 1.00E-76 |
| 9 | Caimán sinensis | Caimán chino | XP_006022630 | 154 | 312 | 63% | 8.00E-68 |
| 10 | Anolis carolinensis | Lagarto | XP_003222553 | 154 | 312 | 62% | 6.00E-67 |
| 11 | Xenopus laevis | Rana africana con garras | XP_018090228 | 244 | 352 | 51% | 4.00E-38 |
| 12 | Tipo de rinoceronte | Tiburón ballena | XP_020388051.1 | 164 | 476 | 53% | 5.00E-52 |
| 13 | Acanthaster planci | Estrella de mar | XP_022086463 | 209 | 684 | 48% | 1.00E-37 |
| 14 | Mizuhopecten yessoensis | Vieira | XP_021340301 | 275 | 797 | 45% | 5.00E-06 |
| 15 | Lotia gigantea | Caracol de mar | XP_009063876 | 173 | 797 | 45% | 2.00E-37 |
| 16 | Língula anatina | Carcasa de lámpara | XP_013388744.1 | 211 | 797 | 43% | 2.00E-35 |
| 17 | Biomphalaria glabrata | Caracol de agua dulce | XP_013088317 | 198 | 797 | 41% | 6.00E-15 |
| 18 | Nematostella vectensis | Anémona de mar | XP_001629616 | 173 | 824 | 48% | 2.00E-35 |
| 19 | Stylophora pistillata | Coral | XP_022795125 | 226 | 824 | 46% | 3.00E-38 |
| 20 | Macrostonum lignano | Gusano plano | PAA73615 | 235 | 824 | 36% | 4.00E-25 |
| 21 | Amphimedon queenslandica | Esponja | XP_003389909 | 275 | 951.8 | 32% | 2.00E-12 |
Referencias
- ^ abc ENSG00000276913 GRCh38: Versión 89 de Ensembl: ENSG00000275489, ENSG00000276913 – Ensembl , mayo de 2017
- ^ abc GRCm38: Lanzamiento de Ensembl 89: ENSMUSG00000018543 – Ensembl , mayo de 2017
- ^ "Referencia de PubMed humana:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ "Referencia de PubMed sobre ratón". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ Zody MC, Garber M, Adams DJ, Sharpe T, Harrow J, Lupski JR, et al. (abril de 2006). "Secuencia de ADN del cromosoma humano 17 y análisis de reordenamiento en el linaje humano". Nature . 440 (7087): 1045–9. Bibcode :2006Natur.440.1045Z. doi :10.1038/nature04689. PMC 2610434 . PMID 16625196.
- ^ Entrada de PSORT II sobre c17orf98 https://psort.hgc.jp/form2.html
- ^ Entrada C17orf98 de dominios conservados del NCBI
- ^ ab ENMBL-EBI SAPS entrada en c17orf98
- ^ "BLAST: herramienta básica de búsqueda de alineaciones locales". blast.ncbi.nlm.nih.gov . Consultado el 2 de mayo de 2018 .
- ^ abc "Protein BLAST: búsqueda de bases de datos de proteínas mediante una consulta de proteínas". blast.ncbi.nlm.nih.gov . Consultado el 2 de mayo de 2018 .
- ^ abc Entrada del atlas de proteínas humanas en c17orf98
- ^ Genomatix El Derado etnry en c17orf98
- ^ Entrada abc TissGDB en c17orf98
- ^ Entrada de Acieview sobre c17orf98
- ^ Entrada de ClustalW en c17orf98 5' UTR
- ^ Entrada de NCBI Blast sobre c17orf98 5' UTR https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blastho me
- ^ Genomatix El Derado etnry en c17orf98 [ enlace muerto permanente ]
- ^ Base de datos GeneCards Human Gene. «C17orf98 Gene - GeneCards - Proteína CQ098 - Anticuerpo CQ098». www.genecards.org . Consultado el 2 de mayo de 2018 .
- ^ "Genomatix El Derado etnry en c17orf98".[ enlace muerto permanente ]
- ^ Visor de datos genómicos del NCBI
- ^ Entrada de Target Scan sobre c17orf98 http://www.targetscan.org/cgibin/targetscan/vert_71/view_gene.cgi?rs=ENST00000398575.4&taxid=9606&showcnc=0&shownc=0&shownc_nc=&showncf1=&showncf2=&subset=1 [ enlace muerto permanente ]
- ^ Entrada de Pax-db sobre c17orf98
- ^ "Entrada mFold en c17orf98 5' UTR".[ enlace muerto permanente ]
- ^ Entrada de pI/mW de ExPASy sobre c17orf98 https://web.expasy.org/cgi-bin/compute_pi/pi_tool [ enlace muerto permanente ]
- ^ Entrada de PSort II sobre C17orf98 [ enlace muerto permanente ]
- ^ Entrada de GPS de Bio Cockoo en C17orf98 http://gps.biocu [ enlace muerto permanente ]
- ^ Entrada de GPS Sumo en c17orf98
- ^ Entrada de YinOyang en c17orf98 http://www.cbs.dtu.dk/services/YinOYang/
- ^ Hanover, John A.; Krause, Michael W.; Love, Dona C. (2010). "La vía de señalización de la hexosamina: ciclo de O-GlcNAc en abundancia o en hambruna". Biochimica et Biophysica Acta (BBA) - Temas generales . 1800 (2): 80–95. doi :10.1016/j.bbagen.2009.07.017. PMC 2815088 . PMID 19647043.
- ^ Búsqueda de motivos lineales eucariotas en la secuencia de aminoácidos c17orf98
- ^ Esnault S, Braun RK, Shen ZJ, Xiang Z, Heninger E, Love RB, Sandor M, Malter JS (febrero de 2007). "Pin1 modula la respuesta inmunitaria de tipo 1". PLOS ONE . 2 (2): e226. Bibcode :2007PLoSO...2..226E. doi : 10.1371/journal.pone.0000226 . PMC 1790862 . PMID 17311089.
- ^ Entrada de BioGrid sobre c17orf98
- ^ Entrada MINT en c17orf98
- ^ Entrada STRING en C17orf98
- ^ PSICQUIC Ver entrada en c17orf98
- ^ Entrada de pax-db sobre c17orf98 https://pax-db.org/protein/1858623#
- ^ "Datos de microarrays:: Atlas cerebral de Allen: cerebro humano". human.brain-map.org . Consultado el 6 de mayo de 2018 .
- ^ Atlas de proteínas humanas (sigma) entrada sobre c17orf98 https://www.proteinatlas.org/ENSG00000275489-C17orf98/cell [ enlace muerto permanente ]
- ^ Entrada de Sigma Aldrich sobre c17orf98 https://www.sigmaaldrich.com/catalog/product/sigma/hpa051696?lang=en®ion=US
- ^ Entrada de PSORT II sobre la secuencia de aminoácidos c17orf98 https://psort.hgc.jp/form2.html
- ^ Entrada de PSort II sobre C17orf98 https://psort.hgc.jp/cgi-bin/runpsort.pl [ enlace muerto permanente ]
- ^ Entrada de Bioinformática de la DTU sobre c17orf98
- ^ Entrada de Expasy Sosui en C17orf98
- ^ Entrada del Atlas de Proteínas sobre c17orf98
- ^ Entrada de Unigene del NCBI sobre c17orf98 www.ncbi.nlm.nih.gov/UniGene/clust.cgi?UGID=169593&TAXID=9606&SEARCH=c17orf98
- ^ "Entrada de Bio GPS en c17orf98".
- ^ Atlas de proteínas humanas (sigma) entrada sobre c17orf98 https://www.proteinatlas.org/ENSG00000275489-C17orf98/cell
- ^ Entrada de NCBI GeoProfiles sobre c17orf98 https://www.ncbi.nlm.nih.gov/geoprofiles
- ^ Entrada de fosfosito en c17orf98 https://www.phosphosite.org/proteinAction.action?id=5156341&showAllSites=true
- ^ "C17orf98 - Teratozoospermia (HG-U133 2.0 )".
- ^ "Entrada de NCBI GeoProfiles sobre c17orf98".
- ^ "El reloj molecular y la estimación de la divergencia de las especies - Aprenda ciencias en Scitable" www.nature.com . Consultado el 2 de mayo de 2018 .
- ^ Entrada de Blast sobre c17orf98 https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE=Proteins





