Respuesta SOS

La respuesta SOS es una respuesta global al daño del ADN en la que se detiene el ciclo celular y se induce la reparación y mutagénesis del ADN. El sistema involucra a la proteína RecA ( Rad51 en eucariotas). La proteína RecA, estimulada por ADN monocatenario, está involucrada en la inactivación del represor ( LexA ) de los genes de respuesta SOS, induciendo así la respuesta. Es un sistema de reparación propenso a errores que contribuye significativamente a los cambios del ADN observados en una amplia gama de especies.
Descubrimiento
La respuesta SOS fue articulada por Evelyn Witkin . [3] [4] Más tarde, al caracterizar los fenotipos de E. coli mutagenizada , ella y el estudiante de postdoctorado Miroslav Radman detallaron la respuesta SOS a la radiación UV en bacterias. [3] [5] La respuesta SOS al daño del ADN fue un descubrimiento fundamental porque fue la primera respuesta de estrés coordinada que se elucidara. [6]
Mecanismo
Durante el crecimiento normal, los genes SOS están regulados negativamente por los dímeros de la proteína represora LexA . En condiciones normales, LexA se une a una secuencia de consenso de 20 pb (la caja SOS ) en la región operadora de esos genes. Algunos de estos genes SOS se expresan en ciertos niveles incluso en el estado reprimido, de acuerdo con la afinidad de LexA por su caja SOS. La activación de los genes SOS ocurre después del daño del ADN por la acumulación de regiones monocatenarias (ssDNA) generadas en las horquillas de replicación, donde la ADN polimerasa está bloqueada. RecA forma un filamento alrededor de estas regiones ssDNA de una manera dependiente de ATP y se activa. [7] La forma activada de RecA interactúa con el represor LexA para facilitar la autoescisión del represor LexA del operador. [7] [8]
Una vez que el grupo de LexA disminuye, la represión de los genes SOS disminuye de acuerdo con el nivel de afinidad de LexA por las cajas SOS. [7] Los operadores que se unen débilmente a LexA son los primeros en expresarse completamente. De esta manera, LexA puede activar secuencialmente diferentes mecanismos de reparación. Los genes que tienen una caja SOS débil (como lexA , recA , uvrA , uvrB y uvrD ) se inducen completamente en respuesta a tratamientos inductores de SOS incluso débiles. Por lo tanto, el primer mecanismo de reparación de SOS que se induce es la reparación por escisión de nucleótidos (NER), cuyo objetivo es reparar el daño del ADN sin comprometerse con una respuesta SOS completa. Sin embargo, si NER no es suficiente para reparar el daño, la concentración de LexA se reduce aún más, por lo que se induce la expresión de genes con cajas LexA más fuertes (como sulA , umuD , umuC , que se expresan más tarde). [7] SulA detiene la división celular [7] al unirse a FtsZ , la proteína iniciadora de este proceso. Esto provoca la filamentación y la inducción de la reparación mutagénica dependiente de UmuDC. Como resultado de estas propiedades, algunos genes pueden ser inducidos parcialmente en respuesta a niveles incluso endógenos de daño del ADN, mientras que otros genes parecen ser inducidos solo cuando hay un daño alto o persistente del ADN en la célula.
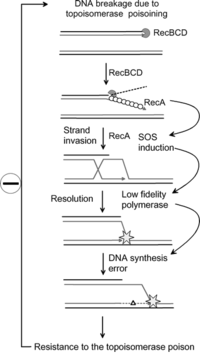
Resistencia a los antibióticos
Las investigaciones han demostrado que el sistema de respuesta SOS puede provocar mutaciones que pueden generar resistencia a los antibióticos. [9] La mayor tasa de mutación durante la respuesta SOS es causada por tres polimerasas de ADN de baja fidelidad : Pol II , Pol IV y Pol V. [ 10] [9] Los investigadores están apuntando ahora a estas proteínas con el objetivo de crear medicamentos que impidan la reparación SOS. Al hacerlo, se podría extender el tiempo necesario para que las bacterias patógenas desarrollen resistencia a los antibióticos, mejorando así la viabilidad a largo plazo de algunos medicamentos antibióticos. [11]
Además de la resistencia genética, la respuesta SOS también puede promover la resistencia fenotípica. En este caso, se preserva el genoma mientras que otros factores no genéticos se modifican para permitir que las bacterias sobrevivan. Por ejemplo, el sistema toxina-antitoxina tisB-istR dependiente de SOS se ha relacionado con la inducción de células persistentes dependiente del daño del ADN . [12]
Prueba de genotoxicidad

En Escherichia coli , diferentes clases de agentes que dañan el ADN pueden iniciar la respuesta SOS, como se describió anteriormente. Aprovechando una fusión de operones que coloca el operón lac (responsable de producir beta-galactosidasa, una proteína que degrada la lactosa) bajo el control de una proteína relacionada con SOS, es posible un ensayo colorimétrico simple para genotoxicidad . Se agrega un análogo de la lactosa a la bacteria, que luego es degradada por beta-galactosidasa, produciendo así un compuesto coloreado que puede medirse cuantitativamente a través de espectrofotometría . El grado de desarrollo del color es una medida indirecta de la beta-galactosidasa producida, que a su vez está directamente relacionada con la cantidad de daño al ADN.
Las bacterias E. coli se modifican aún más para tener una serie de mutaciones, incluida una mutación uvrA que hace que la cepa sea deficiente en la reparación por escisión, lo que aumenta la respuesta a ciertos agentes que dañan el ADN, así como una mutación rfa , que hace que las bacterias sean deficientes en lipopolisacáridos, lo que permite una mejor difusión de ciertos químicos en la célula para inducir la respuesta SOS. [13] Hay kits comerciales disponibles que miden la respuesta primaria de la célula E. coli al daño genético y pueden estar altamente correlacionados con la prueba de Ames para ciertos materiales. [14]
Cianobacterias
Las cianobacterias , las únicas procariotas capaces de desarrollar oxígeno mediante la fotosíntesis , son las principales productoras de la atmósfera oxigenada de la Tierra. [15] Las cianobacterias marinas Prochlorococcus y Synechococcus parecen tener un sistema SOS similar al de E. coli para la reparación del ADN, ya que codifican genes homólogos a los genes SOS clave de E. coli, como lexA y sulA . [16]
Imágenes adicionales
- La respuesta SOS inhibe la formación del septo hasta que se pueda reparar el ADN bacteriano y se observa como filamentación cuando las células se examinan mediante microscopía (arriba a la derecha de la imagen).
Véase también
Referencias
- ^ Little, John W.; Mount, David W. (mayo de 1982). "El sistema regulador SOS de Escherichia coli". Cell . 29 (1): 11–22. doi :10.1016/0092-8674(82)90085-X. PMID 7049397. S2CID 12476812.
- ^ Michel, Bénédicte (12 de julio de 2005). "Tras 30 años de estudio, la respuesta SOS bacteriana aún nos sorprende". PLOS Biology . 3 (7): e255. doi : 10.1371/journal.pbio.0030255 . PMC 1174825 . PMID 16000023.
- ^ ab Fitzgerald, Devon M.; Hastings, PJ; Rosenberg, Susan M. (6 de marzo de 2017). "Mutagénesis inducida por estrés: implicaciones en el cáncer y la resistencia a los fármacos". Revisión anual de la biología del cáncer . 1 (1): 119–140. doi :10.1146/annurev-cancerbio-050216-121919. PMC 5794033 . PMID 29399660 . Consultado el 6 de marzo de 2023 .
- ^ Witkin, EM (mayo de 1967). "La sensibilidad a la radiación de Escherichia coli B: una hipótesis que relaciona la formación de filamentos y la inducción de profagos". Actas de la Academia Nacional de Ciencias . 57 (5): 1275–9. Bibcode :1967PNAS...57.1275W. doi : 10.1073/pnas.57.5.1275 . PMC 224468 . PMID 5341236.
- ^ Witkin, EM (1976). "Mutagénesis ultravioleta y reparación inducible del ADN en Escherichia coli". Bacteriological Reviews . 40 (4): 869–907. doi :10.1128/MMBR.40.4.869-907.1976. PMC 413988 . PMID 795416.
- ^ Radman, M (1975). "Fenomenología de una vía de reparación de ADN mutagénica inducible en Escherichia coli : hipótesis de reparación SOS". Ciencias Básicas de la Vida . 5A : 355–367. doi :10.1007/978-1-4684-2895-7_48. PMID 1103845.
- ^ abcde Maslowska, KH; Makiela-Dzbenska, K.; Fijalkowska, IJ (mayo de 2019). "El sistema SOS: una respuesta compleja y estrechamente regulada al daño del ADN". Mutagénesis ambiental y molecular . 60 (4): 368–384. Bibcode :2019EnvMM..60..368M. doi :10.1002/em.22267. PMC 6590174 . PMID 30447030.
- ^ Lehninger, Albert L.; Nelson, David Lee; Cox, Michael M. (2005). Principios de bioquímica de Lehninger (4.ª ed.). Nueva York: WH Freeman. pág. 1098. ISBN 978-0-7167-4339-2.OCLC 55476414 .
- ^ ab Cirz, RT; Chin, JK; Andes, DR; De Crécy-Lagard, V; Craig, WA; Romesberg, FE (junio de 2005). "Inhibición de la mutación y lucha contra la evolución de la resistencia a los antibióticos". PLOS Biology . 3 (6): e176. doi : 10.1371/journal.pbio.0030176 . PMC 1088971 . PMID 15869329.

- ^ Jaszczur, M; Bertram, JG; Robinson, A; van Oijen, AM; Woodgate, R; Cox, MM; Goodman, MF (abril de 2016). "Mutaciones para peor o mejor: la síntesis de ADN de baja fidelidad por la ADN polimerasa V de SOS es un arma de doble filo estrictamente regulada". Bioquímica . 55 (16): 2309–18. doi :10.1021/acs.biochem.6b00117. PMC 4846499 . PMID 27043933.
- ^ Lee, AM; Ross, CT; Zeng, BB; Singleton, SF (julio de 2005). "Un objetivo molecular para la supresión de la evolución de la resistencia a los antibióticos: inhibición de la proteína RecA de Escherichia coli por N6-(1-naftil)-ADP". Journal of Medicinal Chemistry . 48 (17): 5408–5411. doi :10.1021/jm050113z. PMID 16107138.
- ^ Dörr, T; Vulić, M; Lewis, K (febrero de 2010). "La ciprofloxacina provoca la formación de peroxidasa al inducir la toxina TisB en Escherichia coli". PLOS Biology . 8 (2): e1000317. doi : 10.1371/journal.pbio.1000317 . PMC 2826370 . PMID 20186264.
- ^ Quillardet, Philippe; Hofnung, Maurice (octubre de 1993). "El cromotest SOS: una revisión". Investigación sobre mutaciones/Revisiones en toxicología genética . 297 (3): 235–279. doi :10.1016/0165-1110(93)90019-J. PMID 7692273.
- ^ Quillardet, Philippe; de Bellecombe, Christine; Hofnung, Maurice (junio de 1985). "El SOS Chromotest, un ensayo bacteriano colorimétrico para genotoxinas: estudio de validación con 83 compuestos". Investigación sobre mutaciones/Mutagénesis ambiental y temas relacionados . 147 (3): 79–95. doi :10.1016/0165-1161(85)90021-4. PMID 3923333.
- ^ Hamilton, Trinity L.; Bryant, Donald A.; Macalady, Jennifer L. (febrero de 2016). "El papel de la biología en la evolución planetaria: producción primaria de cianobacterias en océanos proterozoicos con bajo contenido de oxígeno". Microbiología ambiental . 18 (2): 325–340. doi :10.1111/1462-2920.13118. PMC 5019231 . PMID 26549614.
- ^ Cassier-Chauvat, Corinne; Veaudor, Théo; Chauvat, Franck (9 de noviembre de 2016). "Genómica comparativa de la recombinación y reparación del ADN en cianobacterias: implicaciones biotecnológicas". Frontiers in Microbiology . 7 : 1809. doi : 10.3389/fmicb.2016.01809 . PMC 5101192 . PMID 27881980.

