Mecanismo de acción
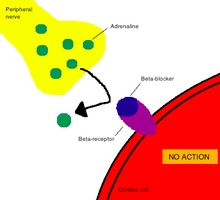
En farmacología , el término mecanismo de acción ( MOA ) se refiere a la interacción bioquímica específica a través de la cual una sustancia farmacológica produce su efecto farmacológico . [2] Un mecanismo de acción generalmente incluye la mención de los objetivos moleculares específicos a los que se une el fármaco, como una enzima o un receptor . [3] Los sitios receptores tienen afinidades específicas para los fármacos en función de la estructura química del fármaco, así como de la acción específica que ocurre allí.
Los fármacos que no se unen a los receptores producen su efecto terapéutico correspondiente simplemente interactuando con las propiedades químicas o físicas del organismo. Algunos ejemplos comunes de fármacos que actúan de esta manera son los antiácidos y los laxantes . [2]
Por el contrario, un modo de acción (MoA) describe cambios funcionales o anatómicos, a nivel celular, resultantes de la exposición de un organismo vivo a una sustancia.
Importancia
Esclarecer el mecanismo de acción de nuevos fármacos y medicamentos es importante por varias razones:
- En el caso del desarrollo de fármacos antiinfecciosos , la información permite anticipar problemas relacionados con la seguridad clínica. Los fármacos que alteran la membrana citoplasmática o la cadena de transporte de electrones , por ejemplo, tienen más probabilidades de causar problemas de toxicidad que los que actúan sobre componentes de la pared celular ( peptidoglicano o β-glucanos ) o el ribosoma 70S , estructuras que están ausentes en las células humanas. [4] [5]
- Al conocer la interacción entre un sitio determinado de un fármaco y un receptor, se pueden formular otros fármacos de forma que repliquen esa interacción y produzcan así los mismos efectos terapéuticos. De hecho, este método se utiliza para crear nuevos fármacos.
- Puede ayudar a identificar qué pacientes tienen más probabilidades de responder al tratamiento. Debido a que se sabe que el medicamento contra el cáncer de mama trastuzumab tiene como blanco la proteína HER2 , por ejemplo, se pueden analizar los tumores para detectar la presencia de esta molécula y así determinar si el paciente se beneficiará o no de la terapia con trastuzumab. [6] [7]
- Esto puede permitir una mejor dosificación porque los efectos del fármaco sobre la vía diana pueden ser monitoreados en el paciente. La dosis de estatinas , por ejemplo, generalmente se determina midiendo los niveles de colesterol en sangre del paciente . [6]
- Permite combinar fármacos de forma que se reduzca la probabilidad de aparición de resistencias. Al conocer sobre qué estructura celular actúa un fármaco antiinfeccioso o anticancerígeno , es posible administrar un cóctel que inhibe múltiples dianas simultáneamente, reduciendo así el riesgo de que una única mutación en el ADN microbiano o tumoral provoque resistencia a los fármacos y el fracaso del tratamiento. [4] [8] [9] [10]
- Esto podría permitir la identificación de otras indicaciones para el fármaco. El descubrimiento de que el sildenafil inhibe las proteínas de la fosfodiesterasa-5 (PDE-5) , por ejemplo, permitió que este fármaco se reutilizara para el tratamiento de la hipertensión arterial pulmonar , ya que la PDE-5 se expresa en los pulmones hipertensos pulmonares. [11] [12]
Determinación
Métodos basados en microscopía

Los compuestos bioactivos inducen cambios fenotípicos en las células objetivo, cambios que son observables mediante microscopía y que pueden dar información sobre el mecanismo de acción del compuesto. [13]
Con agentes antibacterianos , la conversión de células diana en esferoplastos puede ser una indicación de que se está inhibiendo la síntesis de peptidoglicano , y la filamentación de células diana puede ser una indicación de que se está inhibiendo la síntesis de PBP 3, FtsZ o ADN . Otros cambios inducidos por agentes antibacterianos incluyen la formación de células ovoides, formas pseudomulticelulares, hinchazón localizada, formación de protuberancias, formación de ampollas y engrosamiento del peptidoglicano. [4] En el caso de agentes anticancerígenos , la formación de ampollas puede ser una indicación de que el compuesto está alterando la membrana plasmática . [14]
Una limitación actual de este enfoque es el tiempo necesario para generar e interpretar manualmente los datos, pero los avances en el software de microscopía automatizada y análisis de imágenes pueden ayudar a resolverlo. [4] [13]
Métodos bioquímicos directos
Los métodos bioquímicos directos incluyen métodos en los que se marca una proteína o una molécula pequeña, como un fármaco candidato, y se rastrea a través del cuerpo. [15] Este resulta ser el enfoque más directo para encontrar la proteína objetivo que se unirá a pequeños objetivos de interés, como una representación básica del contorno de un fármaco, con el fin de identificar el farmacóforo del fármaco. Debido a las interacciones físicas entre la molécula marcada y una proteína, se pueden utilizar métodos bioquímicos para determinar la toxicidad, la eficacia y el mecanismo de acción del fármaco.
Métodos de inferencia computacional
Por lo general, los métodos de inferencia computacional se utilizan principalmente para predecir dianas proteicas para fármacos de moléculas pequeñas basados en el reconocimiento de patrones basado en computadora. [15] Sin embargo, este método también podría utilizarse para encontrar nuevas dianas para fármacos existentes o recientemente desarrollados. Al identificar el farmacóforo de la molécula del fármaco, se puede llevar a cabo el método de perfilado de reconocimiento de patrones donde se identifica una nueva diana. [15] Esto proporciona una idea de un posible mecanismo de acción, ya que se sabe de qué son responsables ciertos componentes funcionales del fármaco cuando interactúan con un área determinada de una proteína, lo que conduce a un efecto terapéutico.
Métodos basados en la ómica
Los métodos basados en ómicas utilizan tecnologías ómicas, como la quimioproteómica , la genética inversa y la genómica , la transcriptómica y la proteómica , para identificar los objetivos potenciales del compuesto de interés. [16] Los enfoques de genética inversa y genómica, por ejemplo, utilizan perturbación genética (por ejemplo, CRISPR - Cas9 o ARNi ) en combinación con el compuesto para identificar genes cuyo knockdown o knockout elimina el efecto farmacológico del compuesto. Por otro lado, los perfiles transcriptómicos y proteómicos del compuesto se pueden utilizar para comparar con perfiles de compuestos con objetivos conocidos. Gracias a la inferencia computacional, es posible entonces hacer hipótesis sobre el mecanismo de acción del compuesto, que posteriormente se puede probar. [16]
Medicamentos con MOA conocido
Existen muchos fármacos cuyo mecanismo de acción es conocido. Un ejemplo es la aspirina.
Aspirina
El mecanismo de acción de la aspirina implica la inhibición irreversible de la enzima ciclooxigenasa [17] , suprimiendo así la producción de prostaglandinas y tromboxanos , reduciendo así el dolor y la inflamación. Este mecanismo de acción es específico de la aspirina y no es constante para todos los fármacos antiinflamatorios no esteroideos (AINE). Más bien, la aspirina es el único AINE que inhibe irreversiblemente la COX-1. [18]
Medicamentos con MOA desconocido
Algunos mecanismos de acción de los fármacos son aún desconocidos. Sin embargo, aunque se desconozca el mecanismo de acción de un determinado fármaco, éste sigue funcionando; simplemente se desconoce o no está claro cómo interactúa con los receptores y produce su efecto terapéutico.
Modo de acción
En algunos artículos de la literatura, los términos "mecanismo de acción" y " modo de acción " se utilizan indistintamente, refiriéndose típicamente a la forma en que el fármaco interactúa y produce un efecto médico. Sin embargo, en realidad, un modo de acción describe cambios funcionales o anatómicos, a nivel celular, resultantes de la exposición de un organismo vivo a una sustancia. [19] Esto difiere de un mecanismo de acción ya que es un término más específico que se centra en la interacción entre el fármaco en sí y una enzima o receptor y su forma particular de interacción, ya sea a través de inhibición , activación , agonismo o antagonismo . Además, el término "mecanismo de acción" es el término principal que se utiliza principalmente en farmacología, mientras que "modo de acción" aparecerá con más frecuencia en el campo de la microbiología o ciertos aspectos de la biología.
Véase también
Referencias
- ^ Ogrodowczyk, M.; Dettlaff, K.; Jelinska, A. (2016). "Betabloqueantes: estado actual del conocimiento y perspectivas". Mini Reseñas en Química Medicinal . 16 (1): 40–54. doi :10.2174/1389557515666151016125948. PMID 26471965.
- ^ de Spratto, GR; Woods, AL (2010). Manual de medicamentos para enfermeras de Delmar . Cengage Learning. ISBN 978-1-4390-5616-5.
- ^ Grant, RL; Combs, AB; Acosta, D. (2010) "Modelos experimentales para la investigación de mecanismos toxicológicos". En McQueen, CA Comprehensive Toxicology (2.ª ed.). Oxford: Elsevier. pág. 204. ISBN 978-0-08-046884-6 .
- ^ abcde Cushnie, TP; O'Driscoll, NH; Lamb, AJ (2016). "Cambios morfológicos y ultraestructurales en células bacterianas como indicador del mecanismo de acción antibacteriano". Cellular and Molecular Life Sciences . 73 (23): 4471–4492. doi :10.1007/s00018-016-2302-2. hdl : 10059/2129 . PMC 11108400 . PMID 27392605. S2CID 2065821. Archivado desde el original el 2017-10-07 . Consultado el 2017-10-07 .
- ^ Chang, CC; Slavin, MA; Chen, SC (2017). "Nuevos desarrollos y direcciones en la aplicación clínica de las equinocandinas". Archivos de toxicología . 91 (4): 1613–1621. Bibcode :2017ArTox..91.1613C. doi :10.1007/s00204-016-1916-3. PMID 28180946. S2CID 31029386.
- ^ ab No hay autores listados (2010). "El mecanismo importa". Nature Medicine . 16 (4): 347. doi : 10.1038/nm0410-347 . PMID 20376007.
- ^ Joensuu, H. (2017). "Escalating and de-escalating treatment in HER2-positive early breast cancer" (Escalar y desescalar el tratamiento en el cáncer de mama temprano con HER2 positivo). Cancer Treatment Reviews (Reseñas de tratamiento del cáncer ) . 52 : 1–11. doi : 10.1016/j.ctrv.2016.11.002 . PMID 27866067. Archivado desde el original el 5 de agosto de 2019. Consultado el 7 de octubre de 2017 .
- ^ Cihlar, T.; Fordyce, M. (2016). "Estado actual y perspectivas del tratamiento del VIH". Current Opinion in Virology . 18 : 50–56. doi : 10.1016/j.coviro.2016.03.004 . PMID 27023283.
- ^ Antony, HA; Parija, SC (2016). "Resistencia a los fármacos antimaláricos: una descripción general". Parasitología tropical . 6 (1): 30–41. doi : 10.4103/2229-5070.175081 . PMC 4778180 . PMID 26998432.
- ^ Bozic, I.; Reiter, JG; Allen, B.; Antal, T.; Chatterjee, K.; Shah, P.; Moon, YS; Yaqubie, A.; Kelly, N.; Le, DT; Lipson, EJ; Chapman, PB; Diaz, LA; Vogelstein, B.; Nowak, MA (2013). "Dinámica evolutiva del cáncer en respuesta a la terapia de combinación dirigida". eLife . 2 : ID de artículo e00747. doi : 10.7554/eLife.00747 . PMC 3691570 . PMID 23805382.
- ^ Tari, L.; Vo, N.; Liang, S.; Patel, J.; Baral, C.; Cai, J. (2012). "Identificación de nuevas indicaciones de fármacos mediante razonamiento automatizado". PLOS ONE . 7 (7): Artículo e40946. Bibcode :2012PLoSO...740946T. doi : 10.1371/journal.pone.0040946 . PMC 3402456 . PMID 22911721.
- ^ Hayardeny, L. (2014). ¿Por qué es importante conocer el modo de acción de los fármacos? (Presentación en conferencia) . Taller Nuevas fronteras en neurociencia y métodos de educación transdisciplinaria, Universidad de Tel Aviv, Israel: Universidad de Tel Aviv. Archivado desde el original el 18 de marzo de 2017. Consultado el 18 de marzo de 2017 .
- ^ ab Fetz, V.; Prochnow, H.; Brönstrup, M.; Sasse, F. (2016). "Identificación de objetivos mediante análisis de imágenes" (PDF) . Natural Product Reports . 33 (5): 655–667. doi :10.1039/c5np00113g. hdl :10033/621283. PMID 26777141. Archivado (PDF) desde el original el 2020-06-02 . Consultado el 2019-09-26 .
- ^ Dubovskii, PV; Vassilevski, AA; Kozlov, SA; Feofanov, AV; Grishin, EV; Efremov, RG (2015). "Latarcinas: péptidos versátiles del veneno de araña". Ciencias de la vida celulares y moleculares . 72 (23): 4501–4522. doi :10.1007/s00018-015-2016-x. PMC 11113828 . PMID 26286896. S2CID 14177431.
- ^ abc Schenone, M.; Dančík, V.; Wagner, BK; Clemons, PA (2013). "Identificación de objetivos y mecanismo de acción en biología química y descubrimiento de fármacos". Nature Chemical Biology . 9 (4): 232–240. doi :10.1038/nchembio.1199. ISSN 1552-4450. PMC 5543995 . PMID 23508189.
- ^ ab Wecke, T.; Mascher, T. (2011). "Investigación sobre antibióticos en la era de la ciencia ómica: desde los perfiles de expresión hasta la comunicación entre especies". Journal of Antimicrobial Chemotherapy . 66 (12): 2689–2704. doi :10.1093/jac/dkr373. PMID 21930574.
- ^ Tóth, L.; Muszbek, L.; Komaromi, I. (2013). "Mecanismo de la inhibición irreversible de la ciclooxigenasa-1 humana por aspirina según lo predicho por los cálculos de QM/MM". Journal of Molecular Graphics and Modelling . 40 : 99–109. doi :10.1016/j.jmgm.2012.12.013. PMID 23384979.
- ^ Sharma, S.; Sharma, SC (1997). "Una actualización sobre eicosanoides e inhibidores de los sistemas enzimáticos de la ciclooxigenasa". Indian Journal of Experimental Biology . 35 (10): 1025–1031. ISSN 0019-5189. PMID 9475035.
- ^ "Mecanismos y modo de acción de las dioxinas" (PDF) . Agencia de Protección Ambiental de Estados Unidos. Archivado (PDF) del original el 28 de diciembre de 2016. Consultado el 11 de junio de 2012 .