Secuenciación de ARN

RNA-Seq (denominado así por su sigla en inglés como RNA sequencing ) es una técnica que utiliza la secuenciación de próxima generación para revelar la presencia y cantidad de moléculas de ARN en una muestra biológica, proporcionando una instantánea de la expresión genética en la muestra, también conocida como transcriptoma . [2] [3]
Específicamente, RNA-Seq facilita la capacidad de observar transcripciones alternativas de genes empalmados , modificaciones postranscripcionales , fusión de genes , mutaciones/ SNP y cambios en la expresión génica a lo largo del tiempo, o diferencias en la expresión génica en diferentes grupos o tratamientos. [4] Además de las transcripciones de ARNm, RNA-Seq puede observar diferentes poblaciones de ARN para incluir ARN total, ARN pequeño, como miRNA , ARNt y perfiles ribosómicos . [5] RNA-Seq también se puede utilizar para determinar los límites de exón / intrón y verificar o modificar los límites de genes 5' y 3' anotados previamente . Los avances recientes en RNA-Seq incluyen secuenciación de células individuales , secuenciación de ARN en masa , [6] secuenciación de ARNm 3' , secuenciación in situ de tejido fijo y secuenciación de moléculas de ARN nativas con secuenciación de moléculas individuales en tiempo real. [7] Otros ejemplos de aplicaciones emergentes de RNA-Seq debido al avance de los algoritmos bioinformáticos son la alteración del número de copias, la contaminación microbiana, los elementos transponibles, el tipo de célula (deconvolución) y la presencia de neoantígenos. [8]
Antes de RNA-Seq, los estudios de expresión génica se realizaban con microarreglos basados en hibridación . Los problemas con los microarreglos incluyen artefactos de hibridación cruzada, mala cuantificación de genes poco y altamente expresados y la necesidad de conocer la secuencia a priori . [9] Debido a estos problemas técnicos, la transcriptómica hizo la transición a métodos basados en secuenciación. Estos progresaron desde la secuenciación de Sanger de bibliotecas de etiquetas de secuencia expresada , a métodos basados en etiquetas químicas (por ejemplo, análisis en serie de la expresión génica ) y finalmente a la tecnología actual, la secuenciación de próxima generación de ADN complementario (ADNc), en particular RNA-Seq.

Métodos
Preparación de la biblioteca

Los pasos generales para preparar una biblioteca de ADN complementario (ADNc) para secuenciación se describen a continuación, pero a menudo varían entre plataformas. [10] [3] [11]
- Aislamiento de ARN: el ARN se aísla del tejido y se mezcla con desoxirribonucleasa (DNasa). La DNasa reduce la cantidad de ADN genómico. La cantidad de degradación del ARN se verifica con electroforesis capilar y en gel y se utiliza para asignar un número de integridad del ARN a la muestra. Esta calidad del ARN y la cantidad total de ARN inicial se tienen en cuenta durante los pasos posteriores de preparación de la biblioteca, secuenciación y análisis.
- Selección/agotamiento de ARN: para analizar señales de interés, el ARN aislado puede conservarse tal cual, enriquecerse con ARN con colas poliadeniladas 3' (poli(A)) para incluir solo ARNm eucariota , despojarse de ARN ribosómico (ARNr) y/o filtrarse para ARN que se une a secuencias específicas ( tabla de métodos de selección y agotamiento de ARN , a continuación). Las moléculas de ARN que tienen colas poli(A) 3' en eucariotas se componen principalmente de secuencias codificantes maduras y procesadas. La selección poli(A) se realiza mezclando ARN con oligómeros poli(T) unidos covalentemente a un sustrato, típicamente perlas magnéticas. [12] [13] La selección poli(A) tiene limitaciones importantes en la detección del biotipo de ARN. Muchos biotipos de ARN no están poliadenilados, incluidos muchos ARN no codificantes y transcripciones de proteínas del núcleo de histonas, o están regulados a través de la longitud de su cola de poli(A) (p. ej., citocinas) y, por lo tanto, es posible que no se detecten después de la selección de poli(A). [14] Además, la selección de poli(A) puede mostrar un mayor sesgo 3', especialmente con ARN de menor calidad. [15] [16] Estas limitaciones se pueden evitar con el agotamiento ribosómico, eliminando el ARNr que normalmente representa más del 90% del ARN en una célula. Tanto el enriquecimiento de poli(A) como los pasos de agotamiento ribosómico requieren mucha mano de obra y podrían introducir sesgos, por lo que se han desarrollado enfoques más simples para omitir estos pasos. [17] Los objetivos de ARN pequeños, como el miRNA , se pueden aislar aún más mediante la selección de tamaño con geles de exclusión, perlas magnéticas o kits comerciales.
- Síntesis de ADNc: el ARN se transcribe de forma inversa a ADNc porque el ADN es más estable y para permitir la amplificación (que utiliza ADN polimerasas ) y aprovechar una tecnología de secuenciación de ADN más madura. La amplificación posterior a la transcripción inversa da como resultado la pérdida de la cadena , que se puede evitar con el etiquetado químico o la secuenciación de moléculas individuales. La fragmentación y la selección de tamaño se realizan para purificar secuencias que tienen la longitud adecuada para la máquina de secuenciación. El ARN, el ADNc o ambos se fragmentan con enzimas, sonicación , iones divalentes o nebulizadores. La fragmentación del ARN reduce el sesgo 5' de la transcripción inversa cebada aleatoriamente y la influencia de los sitios de unión del cebador , [13] con la desventaja de que los extremos 5' y 3' se convierten en ADN de forma menos eficiente. La fragmentación es seguida por la selección de tamaño, donde se eliminan secuencias pequeñas o se selecciona un rango estrecho de longitudes de secuencia. Debido a que los ARN pequeños como los miARN se pierden, estos se analizan de forma independiente. El ADNc de cada experimento se puede indexar con un código de barras de hexámero u octámero, de modo que estos experimentos se puedan agrupar en un solo carril para la secuenciación multiplexada.
| Estrategia | Tipo predominante de ARN | Contenido de ARN ribosómico | Contenido de ARN sin procesar | Método de aislamiento |
|---|---|---|---|---|
| ARN total | Todo | Alto | Alto | Ninguno |
| Selección de PolyA | Codificación | Bajo | Bajo | Hibridación con oligómeros poli(dT) |
| Agotamiento del ARNr | Codificación, no codificación | Bajo | Alto | Eliminación de oligómeros complementarios al ARNr |
| Captura de ARN | Dirigido | Bajo | Moderado | Hibridación con sondas complementarias a las transcripciones deseadas |
Secuenciación complementaria de ADN (cDNA-Seq)
La biblioteca de ADNc derivada de los biotipos de ARN se secuencia luego en un formato legible por computadora. Existen muchas tecnologías de secuenciación de alto rendimiento para la secuenciación de ADNc, incluidas las plataformas desarrolladas por Illumina , Thermo Fisher , BGI/MGI , PacBio y Oxford Nanopore Technologies . [18] Para la secuenciación de lectura corta de Illumina, una tecnología común para la secuenciación de ADNc, los adaptadores se ligan al ADNc, el ADN se une a una celda de flujo, los grupos se generan a través de ciclos de amplificación de puente y desnaturalización, y la secuencia por síntesis se realiza en ciclos de síntesis de cadena complementaria y excitación láser de bases con terminadores reversibles. La elección de la plataforma de secuenciación y los parámetros están guiados por el diseño experimental y el costo. Las consideraciones de diseño experimental comunes incluyen decidir sobre la longitud de secuenciación, la profundidad de secuenciación, el uso de secuenciación de extremo único versus secuenciación de extremos emparejados, número de réplicas, multiplexación, aleatorización y agregaciones. [19]
Secuenciación de ARN pequeño/ARN no codificante
Al secuenciar ARN que no sea ARNm, se modifica la preparación de la biblioteca. El ARN celular se selecciona en función del rango de tamaño deseado. Para dianas de ARN pequeñas, como miRNA , el ARN se aísla mediante selección de tamaño. Esto se puede realizar con un gel de exclusión por tamaño, a través de perlas magnéticas de selección de tamaño o con un kit desarrollado comercialmente. Una vez aislado, se agregan enlaces a los extremos 3' y 5' y luego se purifica. El paso final es la generación de ADNc mediante transcripción inversa.
Secuenciación directa de ARN

Dado que se ha demostrado que la conversión de ARN en ADNc , la ligadura, la amplificación y otras manipulaciones de muestras introducen sesgos y artefactos que pueden interferir tanto con la caracterización como con la cuantificación adecuadas de las transcripciones, [20] empresas como Helicos (en quiebra), Oxford Nanopore Technologies [21] y otras han explorado la secuenciación directa de ARN de una sola molécula . Esta tecnología secuencia moléculas de ARN directamente de una manera masivamente paralela.
Secuenciación de ARN de una sola molécula en tiempo real
Se ha explorado la secuenciación directa de ARN de una sola molécula en paralelo masivo como una alternativa a la secuenciación de ARN tradicional, en la que la conversión de ARN a ADNc , la ligadura, la amplificación y otros pasos de manipulación de muestras pueden introducir sesgos y artefactos. [22] Las plataformas tecnológicas que realizan secuenciación de ARN de una sola molécula en tiempo real incluyen la secuenciación Nanopore de Oxford Nanopore Technologies (ONT) , [21] PacBio IsoSeq y Helicos (en quiebra). La secuenciación de ARN en su forma nativa preserva modificaciones como la metilación, lo que permite investigarlas de manera directa y simultánea. [21] Otro beneficio de la secuenciación de ARN de una sola molécula es que las transcripciones se pueden cubrir en su longitud completa, lo que permite una detección y cuantificación de isoformas de mayor confianza en comparación con la secuenciación de lectura corta. Tradicionalmente, los métodos de secuenciación de ARN de una sola molécula tienen tasas de error más altas en comparación con la secuenciación de lectura corta, pero los métodos más nuevos como la secuenciación de ARN directa ONT limitan los errores al evitar la fragmentación y la conversión de ADNc. Los usos recientes de la secuenciación de ARN directa ONT para la expresión diferencial en poblaciones de células humanas han demostrado que esta tecnología puede superar muchas limitaciones de la secuenciación de ADNc corta y larga. [23]
Secuenciación de ARN de una sola célula (scRNA-Seq)
Los métodos estándar, como los microarrays y el análisis estándar de secuenciación de ARN en masa, analizan la expresión de ARN de grandes poblaciones de células. En poblaciones de células mixtas, estas mediciones pueden ocultar diferencias críticas entre células individuales dentro de estas poblaciones. [24] [25]
La secuenciación de ARN de una sola célula (scRNA-Seq) proporciona los perfiles de expresión de células individuales. Aunque no es posible obtener información completa sobre cada ARN expresado por cada célula, debido a la pequeña cantidad de material disponible, se pueden identificar patrones de expresión génica a través de análisis de agrupamiento de genes . Esto puede descubrir la existencia de tipos de células raras dentro de una población celular que tal vez nunca se hayan visto antes. Por ejemplo, en 2018, dos grupos que realizaron scRNA-Seq en epitelios de las vías respiratorias pulmonares identificaron células especializadas raras en el pulmón llamadas ionocitos pulmonares que expresan el regulador de conductancia transmembrana de la fibrosis quística . [26] [27]
Procedimientos experimentales
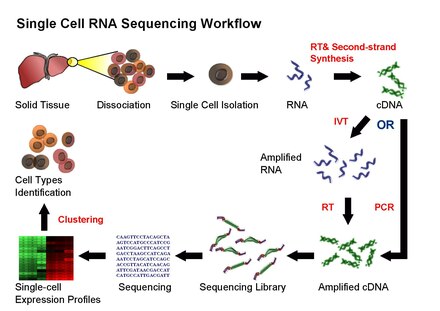
Los protocolos actuales de scRNA-Seq implican los siguientes pasos: aislamiento de células individuales y ARN, transcripción inversa (RT), amplificación, generación de bibliotecas y secuenciación. Las células individuales se separan mecánicamente en micropocillos (p. ej., BD Rhapsody, Takara ICELL8, Vycap Puncher Platform o CellMicrosystems CellRaft) o se encapsulan en gotitas (p. ej., 10x Genomics Chromium, Illumina Bio-Rad ddSEQ, 1CellBio InDrop, Dolomite Bio Nadia). [28] Las células individuales se marcan añadiendo perlas con oligonucleótidos con código de barras; tanto las células como las perlas se suministran en cantidades limitadas de modo que la co-ocupación con múltiples células y perlas es un evento muy raro. Una vez que se completa la transcripción inversa, los ADNc de muchas células se pueden mezclar para la secuenciación; las transcripciones de una célula en particular se identifican por el código de barras único de cada célula. [29] [30] Se pueden adjuntar identificadores moleculares únicos (UMI) a secuencias objetivo de ARNm/ADNc para ayudar a identificar artefactos durante la preparación de la biblioteca. [31]
Los desafíos para scRNA-Seq incluyen preservar la abundancia relativa inicial de ARNm en una célula e identificar transcripciones raras. [32] El paso de transcripción inversa es crítico ya que la eficiencia de la reacción de RT determina qué cantidad de la población de ARN de la célula será analizada finalmente por el secuenciador. La procesividad de las transcriptasas inversas y las estrategias de cebado utilizadas pueden afectar la producción de ADNc de longitud completa y la generación de bibliotecas sesgadas hacia el extremo 3' o 5' de los genes.
En la etapa de amplificación, actualmente se utiliza PCR o transcripción in vitro (IVT) para amplificar el ADNc. Una de las ventajas de los métodos basados en PCR es la capacidad de generar ADNc de longitud completa. Sin embargo, la diferente eficiencia de la PCR en secuencias particulares (por ejemplo, contenido de GC y estructura de snapback) también puede amplificarse exponencialmente, produciendo bibliotecas con cobertura desigual. Por otro lado, mientras que las bibliotecas generadas por IVT pueden evitar el sesgo de secuencia inducido por PCR, secuencias específicas pueden transcribirse de manera ineficiente, causando así la pérdida de secuencia o generando secuencias incompletas. [33] [24] Se han publicado varios protocolos scRNA-Seq: Tang et al., [34] STRT, [35] SMART-seq, [36] CEL-seq, [37] RAGE-seq, [38] Quartz-seq [39] y C1-CAGE. [40] Estos protocolos difieren en términos de estrategias para la transcripción inversa, la síntesis y amplificación de ADNc y la posibilidad de acomodar códigos de barras específicos de secuencia (es decir, UMI ) o la capacidad de procesar muestras agrupadas. [41]
En 2017, se introdujeron dos enfoques para medir simultáneamente la expresión de ARNm y proteínas de una sola célula a través de anticuerpos marcados con oligonucleótidos conocidos como REAP-seq, [42] y CITE-seq. [43]
Aplicaciones
El scRNA-Seq se está utilizando ampliamente en disciplinas biológicas, incluido el desarrollo, la neurología , [44] la oncología , [45] [46] [47] las enfermedades autoinmunes , [48] y las enfermedades infecciosas . [49]
La secuenciación de ARNm ha proporcionado una perspectiva considerable sobre el desarrollo de embriones y organismos, incluido el gusano Caenorhabditis elegans [50] y la planaria regenerativa Schmidtea mediterranea [51] [52] Los primeros animales vertebrados en ser mapeados de esta manera fueron el pez cebra [53] [54] y el Xenopus laevis [55] En cada caso se estudiaron múltiples etapas del embrión, lo que permitió mapear todo el proceso de desarrollo célula por célula [10] Science reconoció estos avances como el Avance del Año 2018 [ 56]
Consideraciones experimentales
Al diseñar y realizar experimentos de RNA-Seq se tienen en cuenta diversos parámetros :
- Especificidad tisular: la expresión génica varía dentro y entre los tejidos, y la secuenciación de ARN mide esta mezcla de tipos de células. Esto puede dificultar el aislamiento del mecanismo biológico de interés. La secuenciación de células individuales se puede utilizar para estudiar cada célula individualmente, lo que mitiga este problema.
- Dependencia del tiempo: la expresión génica cambia con el tiempo y la secuenciación de ARN solo toma una instantánea. Se pueden realizar experimentos de evolución temporal para observar cambios en el transcriptoma.
- Cobertura (también conocida como profundidad): el ARN contiene las mismas mutaciones observadas en el ADN, y su detección requiere una cobertura más profunda. Con una cobertura lo suficientemente alta, la secuenciación de ARN se puede utilizar para estimar la expresión de cada alelo. Esto puede proporcionar información sobre fenómenos como la impronta o los efectos reguladores cis . La profundidad de secuenciación necesaria para aplicaciones específicas se puede extrapolar a partir de un experimento piloto. [57]
- Artefactos de generación de datos (también conocidos como variación técnica): los reactivos (por ejemplo, el kit de preparación de la biblioteca), el personal involucrado y el tipo de secuenciador (por ejemplo, Illumina , Pacific Biosciences ) pueden generar artefactos técnicos que podrían malinterpretarse como resultados significativos. Como con cualquier experimento científico, es prudente realizar RNA-Seq en un entorno bien controlado. Si esto no es posible o el estudio es un metanálisis , otra solución es detectar artefactos técnicos infiriendo variables latentes (normalmente análisis de componentes principales o análisis factorial ) y corrigiendo posteriormente estas variables. [58]
- Gestión de datos: Un único experimento de RNA-Seq en humanos suele ocupar entre 1 y 5 Gb (comprimido), o más si se incluyen archivos intermedios. [59] Este gran volumen de datos puede plantear problemas de almacenamiento. Una solución es comprimir los datos utilizando esquemas computacionales multipropósito (p. ej., gzip ) o esquemas específicos de genómica. Estos últimos pueden basarse en secuencias de referencia o de novo. Otra solución es realizar experimentos de microarrays, que pueden ser suficientes para trabajos basados en hipótesis o estudios de replicación (a diferencia de la investigación exploratoria).
Análisis

Ensamblaje del transcriptoma
Se utilizan dos métodos para asignar lecturas de secuencias sin procesar a características genómicas (es decir, ensamblar el transcriptoma):
- De novo: este enfoque no requiere un genoma de referencia para reconstruir el transcriptoma y se utiliza normalmente si el genoma es desconocido, incompleto o sustancialmente alterado en comparación con la referencia. [60] Los desafíos al utilizar lecturas cortas para el ensamblaje de novo incluyen 1) determinar qué lecturas deben unirse en secuencias contiguas ( contigs ), 2) robustez ante errores de secuenciación y otros artefactos, y 3) eficiencia computacional. El algoritmo principal utilizado para el ensamblaje de novo pasó de los gráficos de superposición, que identifican todas las superposiciones por pares entre lecturas, a los gráficos de Bruijn , que dividen las lecturas en secuencias de longitud k y colapsan todos los k-meros en una tabla hash. [61] Los gráficos de superposición se utilizaron con la secuenciación de Sanger, pero no se escalan bien a los millones de lecturas generadas con RNA-Seq. Ejemplos de ensambladores que utilizan grafos de Bruijn son Trinity, [60] Oases [62] (derivado del ensamblador genómico Velvet [63] ), Bridger [64] y rnaSPAdes. [65] La secuenciación de extremos emparejados y de lectura larga de la misma muestra puede mitigar los déficits en la secuenciación de lectura corta al servir como plantilla o esqueleto. Las métricas para evaluar la calidad de un ensamblaje de novo incluyen la longitud media de contig, el número de contig y N50 . [66]

- Guiado por el genoma: este enfoque se basa en los mismos métodos utilizados para la alineación del ADN, con la complejidad adicional de alinear lecturas que cubren porciones no continuas del genoma de referencia. [67] Estas lecturas no continuas son el resultado de la secuenciación de transcripciones empalmadas (ver figura). Normalmente, los algoritmos de alineación tienen dos pasos: 1) alinear porciones cortas de la lectura (es decir, sembrar el genoma) y 2) usar programación dinámica para encontrar una alineación óptima, a veces en combinación con anotaciones conocidas. Las herramientas de software que utilizan la alineación guiada por el genoma incluyen Bowtie , [68] TopHat (que se basa en los resultados de BowTie para alinear las uniones de empalme), [69] [70] Subread, [71] STAR, [67] HISAT2, [72] y GMAP. [73] La salida de las herramientas de alineación (mapeo) guiada por genoma puede ser utilizada además por herramientas como Cufflinks [70] o StringTie [74] para reconstruir secuencias de transcripción contiguas ( es decir , un archivo FASTA). La calidad de un ensamblaje guiado por genoma puede medirse con 1) métricas de ensamblaje de novo (por ejemplo, N50) y 2) comparaciones con secuencias de transcripción, unión de empalme, genoma y proteína conocidas usando precisión, recuperación o su combinación (por ejemplo, puntuación F1). [66] Además, la evaluación in silico podría realizarse usando lecturas simuladas. [75] [76]
Una nota sobre la calidad del ensamblaje: el consenso actual es que 1) la calidad del ensamblaje puede variar según la métrica utilizada, 2) las herramientas de ensamblaje que obtuvieron buenos resultados en una especie no necesariamente funcionan bien en la otra especie, y 3) la combinación de diferentes enfoques podría ser la más confiable. [77] [78] [79]
Cuantificación de la expresión génica
La expresión se cuantifica para estudiar los cambios celulares en respuesta a estímulos externos, las diferencias entre estados sanos y enfermos y otras cuestiones de investigación. Los niveles de transcripción se utilizan a menudo como un indicador de la abundancia de proteínas, pero a menudo no son equivalentes debido a eventos postranscripcionales como la interferencia del ARN y la descomposición mediada por sinsentidos . [80]
La expresión se cuantifica contando el número de lecturas que se asignaron a cada locus en el paso de ensamblaje del transcriptoma. La expresión se puede cuantificar para exones o genes utilizando contigs o anotaciones de transcripción de referencia. [10] Estos recuentos de lecturas de RNA-Seq observados se han validado de manera sólida frente a tecnologías más antiguas, incluidas las micromatrices de expresión y la qPCR . [57] [81] Las herramientas que cuantifican los recuentos son HTSeq, [82] FeatureCounts, [83] Rcount, [84] maxcounts, [85] FIXSEQ, [86] y Cuffquant. Estas herramientas determinan los recuentos de lecturas a partir de datos de RNA-Seq alineados, pero también se pueden obtener recuentos sin alineación con Sailfish [87] y Kallisto. [88] Luego, los recuentos de lecturas se convierten en métricas apropiadas para pruebas de hipótesis, regresiones y otros análisis. Los parámetros para esta conversión son:
- Profundidad/cobertura de secuenciación : aunque la profundidad se especifica previamente al realizar múltiples experimentos de RNA-Seq, aún variará ampliamente entre experimentos. [89] Por lo tanto, el número total de lecturas generadas en un solo experimento generalmente se normaliza convirtiendo los recuentos en fragmentos, lecturas o recuentos por millón de lecturas mapeadas (FPM, RPM o CPM). La diferencia entre RPM y FPM se derivó históricamente durante la evolución de la secuenciación de fragmentos de un solo extremo a la secuenciación de extremos emparejados. En la secuenciación de un solo extremo, solo hay una lectura por fragmento ( es decir , RPM = FPM). En la secuenciación de extremos emparejados, hay dos lecturas por fragmento ( es decir , RPM = 2 x FPM). La profundidad de secuenciación a veces se denomina tamaño de la biblioteca , la cantidad de moléculas de ADNc intermediarias en el experimento.
- Longitud del gen: los genes más largos tendrán más fragmentos/lecturas/conteos que los genes más cortos si la expresión de la transcripción es la misma. Esto se ajusta dividiendo el FPM por la longitud de una característica (que puede ser un gen, una transcripción o un exón), lo que da como resultado la métrica fragmentos por kilobase de característica por millón de lecturas mapeadas (FPKM). [90] Al observar grupos de características en muestras, FPKM se convierte en transcripciones por millón (TPM) dividiendo cada FPKM por la suma de FPKM dentro de una muestra. [91] [92] [93]
- Salida total de ARN de la muestra: debido a que se extrae la misma cantidad de ARN de cada muestra, las muestras con más ARN total tendrán menos ARN por gen. Estos genes parecen tener una expresión reducida, lo que da como resultado falsos positivos en los análisis posteriores. [89] Las estrategias de normalización, que incluyen cuantiles, DESeq2, TMM y proporción mediana, intentan explicar esta diferencia comparando un conjunto de genes expresados de manera no diferencial entre muestras y escalando en consecuencia. [94]
- Varianza para la expresión de cada gen: se modela para tener en cuenta el error de muestreo (importante para genes con recuentos de lectura bajos), aumentar la potencia y disminuir los falsos positivos. La varianza se puede estimar como unadistribución normal , de Poisson o binomial negativa [95] [96] [97] y con frecuencia se descompone en varianza técnica y biológica.
Spike-ins para cuantificación absoluta y detección de efectos en todo el genoma
Los agregados de ARN son muestras de ARN en concentraciones conocidas que pueden usarse como estándares de oro en el diseño experimental y durante los análisis posteriores para la cuantificación absoluta y la detección de efectos en todo el genoma.
- Cuantificación absoluta: la cuantificación absoluta de la expresión génica no es posible con la mayoría de los experimentos de secuenciación de ARN, que cuantifican la expresión relativa a todas las transcripciones. Es posible realizar la secuenciación de ARN con secuencias de adición de secuencias, muestras de ARN en concentraciones conocidas. Después de la secuenciación, se utilizan los recuentos de lecturas de las secuencias de adición de secuencias para determinar la relación entre los recuentos de lecturas de cada gen y las cantidades absolutas de fragmentos biológicos. [13] [98] En un ejemplo, esta técnica se utilizó en embriones de Xenopus tropicalis para determinar la cinética de la transcripción. [99]
- Detección de efectos en todo el genoma: los cambios en los reguladores globales, incluidos los remodeladores de cromatina , los factores de transcripción (por ejemplo, MYC ), los complejos de acetiltransferasa y el posicionamiento de los nucleosomas no son congruentes con los supuestos de normalización y los controles de adición pueden ofrecer una interpretación precisa. [100] [101]
Expresión diferencial
El uso más simple pero a menudo más poderoso de RNA-Seq es encontrar diferencias en la expresión genética entre dos o más condiciones ( por ejemplo , tratado vs no tratado); este proceso se llama expresión diferencial. Los resultados se denominan con frecuencia genes expresados diferencialmente (DEG) y estos genes pueden ser regulados al alza o a la baja ( es decir , más altos o más bajos en la condición de interés). Hay muchas herramientas que realizan la expresión diferencial . La mayoría se ejecutan en R , Python o la línea de comandos de Unix . Las herramientas comúnmente utilizadas incluyen DESeq, [96] edgeR, [97] y voom+limma, [95] [102] todas las cuales están disponibles a través de R/ Bioconductor . [103] [104] Estas son las consideraciones comunes al realizar la expresión diferencial:
- Entradas: Las entradas de expresión diferencial incluyen (1) una matriz de expresión de RNA-Seq (M genes x N muestras) y (2) una matriz de diseño que contiene condiciones experimentales para N muestras. La matriz de diseño más simple contiene una columna, correspondiente a las etiquetas para la condición que se está probando. Otras covariables (también denominadas factores, características, etiquetas o parámetros) pueden incluir efectos de lote , artefactos conocidos y cualquier metadato que pueda confundir o mediar la expresión genética. Además de las covariables conocidas, las covariables desconocidas también se pueden estimar a través de enfoques de aprendizaje automático no supervisado que incluyen análisis de componentes principales , variables sustitutas [105] y PEER [58] . Los análisis de variables ocultas a menudo se emplean para datos de RNA-Seq de tejido humano, que normalmente tienen artefactos adicionales no capturados en los metadatos ( p. ej. , tiempo de isquemia, obtención de múltiples instituciones, rasgos clínicos subyacentes, recopilación de datos a lo largo de muchos años con mucho personal).
- Métodos: La mayoría de las herramientas utilizan regresión o estadísticas no paramétricas para identificar genes expresados diferencialmente, y se basan en recuentos de lecturas mapeados a un genoma de referencia (DESeq2, limma, edgeR) o en recuentos de lecturas derivados de cuantificación sin alineamiento (sleuth, [106] Cuffdiff, [107] Ballgown [108] ). [109] Después de la regresión, la mayoría de las herramientas emplean ajustes de valor p de tasa de error familiar (FWER) o tasa de falsos descubrimientos (FDR) para tener en cuenta múltiples hipótesis (en estudios humanos, ~20 000 genes codificadores de proteínas o ~50 000 biotipos).
- Resultados: Un resultado típico consiste en filas correspondientes al número de genes y al menos tres columnas, el cambio de log fold de cada gen ( transformación de log de la relación en la expresión entre condiciones, una medida del tamaño del efecto ), valor p y valor p ajustado para comparaciones múltiples . Los genes se definen como biológicamente significativos si pasan los puntos de corte para el tamaño del efecto (cambio de log fold) y la significación estadística . Estos puntos de corte idealmente deberían especificarse a priori , pero la naturaleza de los experimentos de RNA-Seq es a menudo exploratoria, por lo que es difícil predecir los tamaños del efecto y los puntos de corte pertinentes con anticipación.
- Dificultades: La razón de ser de estos métodos complejos es evitar la gran cantidad de dificultades que pueden llevar a errores estadísticos e interpretaciones engañosas. Las dificultades incluyen mayores tasas de falsos positivos (debido a comparaciones múltiples), artefactos en la preparación de muestras, heterogeneidad de las muestras (como antecedentes genéticos mixtos), muestras altamente correlacionadas, diseños experimentales de múltiples niveles no tenidos en cuenta y un diseño experimental deficiente . Una dificultad notable es ver los resultados en Microsoft Excel sin usar la función de importación para garantizar que los nombres de los genes sigan siendo texto. [110] Aunque es conveniente, Excel convierte automáticamente algunos nombres de genes ( SEPT1 , DEC1 , MARCH2 ) en fechas o números de punto flotante.
- Elección de herramientas y evaluación comparativa: existen numerosos esfuerzos que comparan los resultados de estas herramientas, y DESeq2 tiende a superar moderadamente a otros métodos. [111] [112] [113] [114] [19] [109] [115] [116] Al igual que con otros métodos, la evaluación comparativa consiste en comparar los resultados de las herramientas entre sí y con estándares de oro conocidos .
Los análisis posteriores para una lista de genes expresados diferencialmente vienen en dos sabores, validar observaciones y hacer inferencias biológicas. Debido a las trampas de la expresión diferencial y RNA-Seq, las observaciones importantes se replican con (1) un método ortogonal en las mismas muestras (como PCR en tiempo real ) o (2) otro experimento, a veces pre-registrado , en una nueva cohorte. Este último ayuda a asegurar la generalización y típicamente puede ser seguido con un metanálisis de todas las cohortes agrupadas. El método más común para obtener una comprensión biológica de alto nivel de los resultados es el análisis de enriquecimiento del conjunto de genes , aunque a veces se emplean enfoques de genes candidatos. El enriquecimiento del conjunto de genes determina si la superposición entre dos conjuntos de genes es estadísticamente significativa, en este caso la superposición entre genes expresados diferencialmente y conjuntos de genes de vías/bases de datos conocidas ( por ejemplo , Gene Ontology , KEGG , Human Phenotype Ontology ) o de análisis complementarios en los mismos datos (como redes de coexpresión). Las herramientas comunes para el enriquecimiento de conjuntos de genes incluyen interfaces web ( por ejemplo , ENRICHR, g:profiler, WEBGESTALT) [117] y paquetes de software. Al evaluar los resultados del enriquecimiento, una heurística es buscar primero el enriquecimiento de la biología conocida como una comprobación de cordura y luego ampliar el alcance para buscar biología nueva.

Empalme alternativo
El empalme de ARN es parte integral de los eucariotas y contribuye significativamente a la regulación y diversidad de proteínas, y ocurre en >90% de los genes humanos. [118] Existen múltiples modos de empalme alternativo : omisión de exón (modo de empalme más común en humanos y eucariotas superiores), exones mutuamente excluyentes, sitios donantes o aceptores alternativos, retención de intrones (modo de empalme más común en plantas, hongos y protozoos), sitio de inicio de transcripción alternativo (promotor) y poliadenilación alternativa. [118] Un objetivo de RNA-Seq es identificar eventos de empalme alternativo y probar si difieren entre condiciones. La secuenciación de lectura larga captura la transcripción completa y, por lo tanto, minimiza muchos de los problemas en la estimación de la abundancia de isoformas, como el mapeo de lectura ambigua. Para RNA-Seq de lectura corta, existen múltiples métodos para detectar empalme alternativo que se pueden clasificar en tres grupos principales: [119] [91] [120]
- Basado en recuentos (también basado en eventos, empalme diferencial): estima la retención de exones. Algunos ejemplos son DEXSeq, [121] MATS, [122] y SeqGSEA. [123]
- Basado en isoformas (también módulos de lectura múltiple, expresión diferencial de isoformas) : primero se estima la abundancia de isoformas y luego la abundancia relativa entre condiciones. Algunos ejemplos son Cufflinks 2 [124] y DiffSplice. [125]
- Basado en la escisión de intrones: calcule el empalme alternativo utilizando lecturas divididas. Algunos ejemplos son MAJIQ [126] y Leafcutter. [120]
Las herramientas de expresión genética diferencial también se pueden utilizar para la expresión de isoformas diferenciales si las isoformas se cuantifican con anticipación con otras herramientas como RSEM. [127]
Redes de coexpresión
Las redes de coexpresión son representaciones derivadas de datos de genes que se comportan de manera similar en diferentes tejidos y condiciones experimentales. [128] Su propósito principal radica en la generación de hipótesis y en enfoques de culpabilidad por asociación para inferir funciones de genes previamente desconocidos. [128] Los datos de RNA-Seq se han utilizado para inferir genes involucrados en vías específicas basadas en la correlación de Pearson , tanto en plantas [129] como en mamíferos. [130] La principal ventaja de los datos de RNA-Seq en este tipo de análisis sobre las plataformas de microarrays es la capacidad de cubrir todo el transcriptoma, lo que permite la posibilidad de desentrañar representaciones más completas de las redes reguladoras de genes. La regulación diferencial de las isoformas de empalme del mismo gen se puede detectar y utilizar para predecir sus funciones biológicas. [131] [132] El análisis de redes de coexpresión de genes ponderados se ha utilizado con éxito para identificar módulos de coexpresión y genes centrales intramodulares basados en datos de RNA seq. Los módulos de coexpresión pueden corresponder a tipos de células o vías. Los centros intramodulares altamente conectados pueden interpretarse como representantes de su respectivo módulo. Un gen propio es una suma ponderada de la expresión de todos los genes en un módulo. Los genes propios son biomarcadores útiles (características) para el diagnóstico y el pronóstico. [133] Se han propuesto enfoques de transformación estabilizadora de varianza para estimar coeficientes de correlación basados en datos de secuenciación de ARN. [129]
Descubrimiento de variantes
RNA-Seq captura la variación del ADN, incluyendo variantes de un solo nucleótido , pequeñas inserciones/deleciones y variación estructural . La llamada de variantes en RNA-Seq es similar a la llamada de variantes de ADN y a menudo emplea las mismas herramientas (incluyendo SAMtools mpileup [134] y GATK HaplotypeCaller [135] ) con ajustes para tener en cuenta el empalme. Una dimensión única para las variantes de ARN es la expresión específica de alelos (ASE) : las variantes de un solo haplotipo podrían expresarse preferentemente debido a efectos reguladores que incluyen impronta y expresión de loci de rasgos cuantitativos y variantes raras no codificantes . [136] [137] Las limitaciones de la identificación de variantes de ARN incluyen que solo refleja regiones expresadas (en humanos, <5% del genoma), podría estar sujeta a sesgos introducidos por el procesamiento de datos (p. ej., los ensamblajes de transcriptomas de novo subestiman la heterocigosidad [138] ) y tiene menor calidad en comparación con la secuenciación directa de ADN.
Edición de ARN (alteraciones postranscripcionales)
Disponer de las secuencias genómicas y transcriptómicas coincidentes de un individuo puede ayudar a detectar ediciones postranscripcionales ( edición de ARN ). [3] Un evento de modificación postranscripcional se identifica si la transcripción del gen tiene un alelo/variante que no se observa en los datos genómicos.

Detección de genes de fusión
Los genes de fusión, que son consecuencia de diferentes modificaciones estructurales en el genoma, han ganado atención debido a su relación con el cáncer. [139] La capacidad de RNA-Seq para analizar el transcriptoma completo de una muestra de manera imparcial lo convierte en una herramienta atractiva para encontrar este tipo de eventos comunes en el cáncer. [4]
La idea surge del proceso de alinear las lecturas transcriptómicas cortas con un genoma de referencia. La mayoría de las lecturas cortas caerán dentro de un exón completo, y se esperaría que un conjunto más pequeño, pero aún grande, se mapeara en uniones exón-exón conocidas. Las lecturas cortas restantes sin mapear se analizarían más a fondo para determinar si coinciden con una unión exón-exón donde los exones provienen de diferentes genes. Esto sería evidencia de un posible evento de fusión, sin embargo, debido a la longitud de las lecturas, esto podría resultar muy ruidoso. Un enfoque alternativo es usar lecturas de extremos emparejados, cuando un número potencialmente grande de lecturas emparejadas mapearía cada extremo a un exón diferente, brindando una mejor cobertura de estos eventos (ver figura). No obstante, el resultado final consiste en múltiples y potencialmente novedosas combinaciones de genes que proporcionan un punto de partida ideal para una mayor validación.
Alteración del número de copias
Los análisis de alteración del número de copias (CNA) se utilizan comúnmente en los estudios sobre el cáncer. La ganancia y pérdida de genes tiene implicaciones en las vías de señalización y son un biomarcador clave de disfunción molecular en oncología. La obtención de la información de la CNA a partir de los datos de RNA-Seq no es sencilla debido a las diferencias en la expresión genética, que conducen a una variación de la profundidad de lectura de diferentes magnitudes entre los genes. Debido a estas dificultades, la mayoría de estos análisis se realizan habitualmente mediante secuenciación del genoma completo/secuenciación del exoma completo (WGS/WES). Pero las herramientas bioinformáticas avanzadas pueden obtener la CNA a partir de RNA-Seq. [140]
Otros análisis y aplicaciones emergentes
Las aplicaciones de RNA-Seq están creciendo día a día. Otras nuevas aplicaciones de RNA-Seq incluyen la detección de contaminantes microbianos, [141] la determinación de la abundancia de tipos celulares (desconvolución de tipos celulares), [8] la medición de la expresión de TE y la predicción de neoantígenos, etc. [8]
Historia

El ARN-Seq se desarrolló por primera vez a mediados de la década de 2000 con la llegada de la tecnología de secuenciación de próxima generación. [144] Los primeros manuscritos que utilizaron ARN-Seq incluso sin usar el término incluyen los de líneas celulares de cáncer de próstata [145] (fechado en 2006), Medicago truncatula [146] (2006), maíz [147] (2007) y Arabidopsis thaliana [148] (2007), mientras que el término "ARN-Seq" en sí se mencionó por primera vez en 2008. [13] [149] La cantidad de manuscritos que hacen referencia al ARN-Seq en el título o resumen (Figura, línea azul) aumenta continuamente con 6754 manuscritos publicados en 2018. La intersección del ARN-Seq y la medicina (Figura, línea dorada) tiene una celeridad similar. [150]
Aplicaciones a la medicina
La secuenciación de ARN tiene el potencial de identificar nuevas enfermedades biológicas, crear perfiles de biomarcadores para indicaciones clínicas, inferir vías farmacológicas y realizar diagnósticos genéticos. [151] [152] Estos resultados podrían personalizarse aún más para subgrupos o incluso para pacientes individuales, lo que podría poner de relieve una prevención, un diagnóstico y una terapia más eficaces. La viabilidad de este enfoque está determinada en parte por los costes en dinero y tiempo; una limitación relacionada es el equipo de especialistas necesario (bioinformáticos, médicos/clínicos, investigadores básicos, técnicos) para interpretar por completo la enorme cantidad de datos generados por este análisis. [153]
Esfuerzos de secuenciación a gran escala
Se ha dado mucho énfasis a los datos de RNA-Seq después de que los proyectos Encyclopedia of DNA Elements (ENCODE) y The Cancer Genome Atlas (TCGA) hayan utilizado este enfoque para caracterizar docenas de líneas celulares [154] y miles de muestras de tumores primarios, [155] respectivamente. ENCODE tenía como objetivo identificar regiones reguladoras de todo el genoma en diferentes cohortes de líneas celulares y los datos transcriptómicos son fundamentales para comprender el efecto descendente de esas capas reguladoras epigenéticas y genéticas. TCGA, en cambio, tenía como objetivo recopilar y analizar miles de muestras de pacientes de 30 tipos de tumores diferentes para comprender los mecanismos subyacentes de la transformación y progresión malignas. En este contexto, los datos de RNA-Seq brindan una instantánea única del estado transcriptómico de la enfermedad y analizan una población imparcial de transcripciones que permite la identificación de nuevas transcripciones, transcripciones de fusión y ARN no codificantes que podrían pasar desapercibidos con diferentes tecnologías.
Véase también
Referencias
 Este artículo fue enviado a WikiJournal of Science para su revisión académica externa por pares en 2019 (informes de los revisores). El contenido actualizado fue reintegrado a la página de Wikipedia bajo una licencia CC-BY-SA-3.0 ( 2021 ). La versión de registro revisada es: Felix Richter, et al. (17 de mayo de 2021). "Una amplia introducción a RNA-Seq" (PDF) . WikiJournal of Science . 4 (2): 4. doi : 10.15347/WJS/2021.004 . ISSN 2470-6345. Wikidata Q100146647.
Este artículo fue enviado a WikiJournal of Science para su revisión académica externa por pares en 2019 (informes de los revisores). El contenido actualizado fue reintegrado a la página de Wikipedia bajo una licencia CC-BY-SA-3.0 ( 2021 ). La versión de registro revisada es: Felix Richter, et al. (17 de mayo de 2021). "Una amplia introducción a RNA-Seq" (PDF) . WikiJournal of Science . 4 (2): 4. doi : 10.15347/WJS/2021.004 . ISSN 2470-6345. Wikidata Q100146647.
- ^ Lowe R, Shirley N, Bleackley M, Dolan S, Shafee T (mayo de 2017). "Tecnologías transcriptómicas". PLOS Computational Biology . 13 (5): e1005457. Bibcode :2017PLSCB..13E5457L. doi : 10.1371/journal.pcbi.1005457 . PMC 5436640 . PMID 28545146.
- ^ Chu Y, Corey DR (agosto de 2012). "Secuenciación de ARN: selección de plataforma, diseño experimental e interpretación de datos". Terapéutica de ácidos nucleicos . 22 (4): 271–4. doi :10.1089/nat.2012.0367. PMC 3426205 . PMID 22830413.
- ^ abc Wang Z, Gerstein M, Snyder M (enero de 2009). "RNA-Seq: una herramienta revolucionaria para la transcriptómica". Nature Reviews. Genetics . 10 (1): 57–63. doi :10.1038/nrg2484. PMC 2949280 . PMID 19015660.
- ^ ab Maher CA, Kumar-Sinha C, Cao X, Kalyana-Sundaram S, Han B, Jing X, et al. (marzo de 2009). "Secuenciación del transcriptoma para detectar fusiones genéticas en el cáncer". Nature . 458 (7234): 97–101. Bibcode :2009Natur.458...97M. doi :10.1038/nature07638. PMC 2725402 . PMID 19136943.
- ^ Ingolia NT, Brar GA, Rouskin S, McGeachy AM, Weissman JS (julio de 2012). "La estrategia de perfilado de ribosomas para monitorear la traducción in vivo mediante secuenciación profunda de fragmentos de ARNm protegidos por ribosomas". Nature Protocols . 7 (8): 1534–50. doi :10.1038/nprot.2012.086. PMC 3535016 . PMID 22836135.
- ^ Alpern D, Gardeux V, Russeil J, Mangeat B, Meireles-Filho AC, Breysse R, et al. (abril de 2019). "BRB-seq: transcriptómica de alto rendimiento y ultra asequible posibilitada por secuenciación y codificación de ARN en masa". Genome Biology . 20 (1): 71. doi : 10.1186/s13059-019-1671-x . PMC 6474054 . PMID 30999927.
- ^ Lee JH, Daugharthy ER, Scheiman J, Kalhor R, Yang JL, Ferrante TC, et al. (marzo de 2014). "Secuenciación de ARN subcelular altamente multiplexada in situ". Science . 343 (6177): 1360–3. Bibcode :2014Sci...343.1360L. doi :10.1126/science.1250212. PMC 4140943 . PMID 24578530.
- ^ abc Thind AS, Monga I, Thakur PK, Kumari P, Dindhoria K, Krzak M, et al. (noviembre de 2021). "Desmitificando las aplicaciones emergentes de secuenciación masiva de ARN: la aplicación y la utilidad de la metodología bioinformática". Briefings in Bioinformatics . 22 (6). doi :10.1093/bib/bbab259. PMID 34329375.
- ^ Kukurba KR, Montgomery SB (abril de 2015). "Secuenciación y análisis de ARN". Protocolos de Cold Spring Harbor . 2015 (11): 951–69. doi :10.1101/pdb.top084970. PMC 4863231. PMID 25870306 .
- ^ abcde Griffith M, Walker JR, Spies NC, Ainscough BJ, Griffith OL (agosto de 2015). "Informática para la secuenciación de ARN: un recurso web para el análisis en la nube". PLOS Computational Biology . 11 (8): e1004393. Bibcode :2015PLSCB..11E4393G. doi : 10.1371/journal.pcbi.1004393 . PMC 4527835 . PMID 26248053.
- ^ "RNA-seqlopedia". rnaseq.uoregon.edu . Consultado el 8 de febrero de 2017 .
- ^ Morin R, Bainbridge M, Fejes A, Hirst M, Krzywinski M, Pugh T, et al. (julio de 2008). "Perfilado del transcriptoma HeLa S3 utilizando ADNc cebado aleatoriamente y secuenciación masiva paralela de lectura corta". BioTechniques . 45 (1): 81–94. doi : 10.2144/000112900 . PMID 18611170.
- ^ abcd Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (julio de 2008). "Mapeo y cuantificación de transcriptomas de mamíferos mediante RNA-Seq". Nature Methods . 5 (7): 621–8. doi :10.1038/nmeth.1226. PMID 18516045. S2CID 205418589.
- ^ Sun Q, Hao Q, Prasanth KV (febrero de 2018). "ARN nucleares largos no codificantes: reguladores clave de la expresión génica". Tendencias en genética . 34 (2): 142–157. doi :10.1016/j.tig.2017.11.005. PMC 6002860 . PMID 29249332.
- ^ Sigurgeirsson B, Emanuelsson O, Lundeberg J (2014). "Secuenciación de ARN degradado mediante recuento de etiquetas 3'". PLOS ONE . 9 (3): e91851. Bibcode :2014PLoSO...991851S. doi : 10.1371/journal.pone.0091851 . PMC 3954844 . PMID 24632678.
- ^ Chen EA, Souaiaia T, Herstein JS, Evgrafov OV, Spitsyna VN, Rebolini DF, et al. (octubre de 2014). "Efecto de la integridad del ARN en lecturas mapeadas de forma única en RNA-Seq". BMC Research Notes . 7 : 753. doi : 10.1186/1756-0500-7-753 . PMC 4213542 . PMID 25339126.
- ^ Moll P, Ante M, Seitz A, Reda T (diciembre de 2014). "Secuenciación de ARNm QuantSeq 3′ para cuantificación de ARN". Nature Methods . 11 (12): i–iii. doi :10.1038/nmeth.f.376. ISSN 1548-7105. S2CID 83424788.
- ^ Oikonomopoulos S, Bayega A, Fahiminiya S, Djambazian H, Berube P, Ragoussis J (2020). "Metodologías para la elaboración de perfiles de transcripción utilizando tecnologías de lectura larga". Frontiers in Genetics . 11 : 606. doi : 10.3389/fgene.2020.00606 . PMC 7358353 . PMID 32733532.
- ^ ab Conesa A, Madrigal P, Tarazona S, Gomez-Cabrero D, Cervera A, McPherson A, et al. (enero de 2016). "Una encuesta sobre las mejores prácticas para el análisis de datos de ARN-seq". Genome Biology . 17 (1): 13. doi : 10.1186/s13059-016-0881-8 . PMC 4728800 . PMID 26813401.
- ^ Liu D, Graber JH (febrero de 2006). "La comparación cuantitativa de bibliotecas EST requiere compensación por sesgos sistemáticos en la generación de ADNc". BMC Bioinformatics . 7 : 77. doi : 10.1186/1471-2105-7-77 . PMC 1431573 . PMID 16503995.
- ^ abc Garalde DR, Snell EA, Jachimowicz D, Sipos B, Lloyd JH, Bruce M, et al. (Marzo de 2018). "Secuenciación directa de ARN altamente paralela en una serie de nanoporos". Métodos de la naturaleza . 15 (3): 201–206. doi :10.1038/nmeth.4577. PMID 29334379. S2CID 3589823.
- ^ Liu D, Graber JH (febrero de 2006). "La comparación cuantitativa de bibliotecas EST requiere compensación por sesgos sistemáticos en la generación de ADNc". BMC Bioinformatics . 7 : 77. doi : 10.1186/1471-2105-7-77 . PMC 1431573 . PMID 16503995.
- ^ Gleeson J, Lane TA, Harrison PJ, Haerty W, Clark MB (3 de agosto de 2020). "La secuenciación directa de ARN con nanoporos detecta la expresión diferencial entre poblaciones de células humanas". bioRxiv : 2020.08.02.232785. doi : 10.1101/2020.08.02.232785 . S2CID 220975367.
- ^ ab " Shapiro E, Biezuner T, Linnarsson S (septiembre de 2013). "Las tecnologías basadas en secuenciación de células individuales revolucionarán la ciencia de organismos completos". Nature Reviews. Genetics . 14 (9): 618–30. doi :10.1038/nrg3542. PMID 23897237. S2CID 500845."
- ^ Kolodziejczyk AA, Kim JK, Svensson V, Marioni JC, Teichmann SA (mayo de 2015). "La tecnología y la biología de la secuenciación de ARN de células individuales". Molecular Cell . 58 (4): 610–20. doi : 10.1016/j.molcel.2015.04.005 . PMID 26000846.
- ^ Montoro DT, Haber AL, Biton M, Vinarsky V, Lin B, Birket SE, et al. (Agosto de 2018). "Una jerarquía epitelial de las vías respiratorias revisada incluye ionocitos que expresan CFTR". Naturaleza . 560 (7718): 319–324. Código Bib :2018Natur.560..319M. doi :10.1038/s41586-018-0393-7. PMC 6295155 . PMID 30069044.
- ^ Plasschaert LW, Žilionis R, Choo-Wing R, Savova V, Knehr J, Roma G, et al. (agosto de 2018). "Un atlas unicelular del epitelio de las vías respiratorias revela el ionocito pulmonar rico en CFTR". Nature . 560 (7718): 377–381. Bibcode :2018Natur.560..377P. doi :10.1038/s41586-018-0394-6. PMC 6108322 . PMID 30069046.
- ^ Valihrach L, Androvic P, Kubista M (marzo de 2018). "Plataformas para la recolección y análisis de células individuales". Revista internacional de ciencias moleculares . 19 (3): 807. doi : 10.3390/ijms19030807 . PMC 5877668 . PMID 29534489.
- ^ Klein AM, Mazutis L, Akartuna I, Tallapragada N, Veres A, Li V, et al. (mayo de 2015). "Código de barras de gotitas para la transcriptómica de células individuales aplicada a células madre embrionarias". Cell . 161 (5): 1187–1201. doi :10.1016/j.cell.2015.04.044. PMC 4441768 . PMID 26000487.
- ^ Macosko EZ, Basu A, Satija R, Nemesh J, Shekhar K, Goldman M, et al. (mayo de 2015). "Perfiles de expresión de todo el genoma altamente paralelos de células individuales utilizando gotas de nanolitros". Cell . 161 (5): 1202–1214. doi :10.1016/j.cell.2015.05.002. PMC 4481139 . PMID 26000488.
- ^ Islam S, Zeisel A, Joost S, La Manno G, Zajac P, Kasper M, et al. (febrero de 2014). "Secuenciación cuantitativa de ARN unicelular con identificadores moleculares únicos". Nature Methods . 11 (2): 163–6. doi :10.1038/nmeth.2772. PMID 24363023. S2CID 6765530.
- ^ " Hebenstreit D (noviembre de 2012). "Métodos, desafíos y potenciales de la secuenciación de ARN de una sola célula". Biology . 1 (3): 658–67. doi : 10.3390/biology1030658 . PMC 4009822 . PMID 24832513. "
- ^ Eberwine J, Sul JY, Bartfai T, Kim J (enero de 2014). "La promesa de la secuenciación unicelular". Nature Methods . 11 (1): 25–7. doi :10.1038/nmeth.2769. PMID 24524134. S2CID 11575439.
- ^ Tang F, Barbacioru C, Wang Y, Nordman E, Lee C, Xu N, et al. (mayo de 2009). "Análisis del transcriptoma completo de ARNm-Seq de una sola célula". Nature Methods . 6 (5): 377–82. doi :10.1038/NMETH.1315. PMID 19349980. S2CID 16570747.
- ^ Islam S, Kjällquist U, Moliner A, Zajac P, Fan JB, Lönnerberg P, et al. (julio de 2011). "Caracterización del panorama transcripcional de células individuales mediante secuenciación de ARN altamente multiplexada". Genome Research . 21 (7): 1160–7. doi :10.1101/gr.110882.110. PMC 3129258 . PMID 21543516.
- ^ Ramsköld D, Luo S, Wang YC, Li R, Deng Q, Faridani OR, et al. (agosto de 2012). "Secuenciación completa de ARNm a partir de niveles de ARN de células individuales y células tumorales circulantes individuales". Nature Biotechnology . 30 (8): 777–82. doi :10.1038/nbt.2282. PMC 3467340 . PMID 22820318.
- ^ Hashimshony T, Wagner F, Sher N, Yanai I (septiembre de 2012). "CEL-Seq: secuenciación de ARN de una sola célula mediante amplificación lineal multiplexada". Cell Reports . 2 (3): 666–73. doi : 10.1016/j.celrep.2012.08.003 . PMID 22939981.
- ^ Singh M, Al-Eryani G, Carswell S, Ferguson JM, Blackburn J, Barton K, et al. (2018). "La secuenciación de células individuales de lectura larga dirigida y de alto rendimiento revela el panorama clonal y transcripcional de los linfocitos". bioRxiv . 10 (1): 3120. doi : 10.1101/424945 . PMC 6635368 . PMID 31311926.
- ^ Sasagawa Y, Nikaido I, Hayashi T, Danno H, Uno KD, Imai T, et al. (abril de 2013). "Quartz-Seq: un método de secuenciación de ARN unicelular altamente reproducible y sensible, revela heterogeneidad no genética en la expresión génica". Genome Biology . 14 (4): R31. doi : 10.1186/gb-2013-14-4-r31 . PMC 4054835 . PMID 23594475.
- ^ Kouno T, Moody J, Kwon AT, Shibayama Y, Kato S, Huang Y, et al. (enero de 2019). "C1 CAGE detecta sitios de inicio de transcripción y actividad potenciadora con resolución de célula única". Nature Communications . 10 (1): 360. Bibcode :2019NatCo..10..360K. doi :10.1038/s41467-018-08126-5. PMC 6341120 . PMID 30664627.
- ^ Dal Molin A, Di Camillo B (2019). "Cómo diseñar un experimento de secuenciación de ARN de una sola célula: dificultades, desafíos y perspectivas". Briefings in Bioinformatics . 20 (4): 1384–1394. doi :10.1093/bib/bby007. PMID 29394315.
- ^ Peterson VM, Zhang KX, Kumar N, Wong J, Li L, Wilson DC, et al. (octubre de 2017). "Cuantificación multiplexada de proteínas y transcripciones en células individuales". Nature Biotechnology . 35 (10): 936–939. doi :10.1038/nbt.3973. PMID 28854175. S2CID 205285357.
- ^ Stoeckius M, Hafemeister C, Stephenson W, Houck-Loomis B, Chattopadhyay PK, Swerdlow H, et al. (septiembre de 2017). "Medición simultánea de epítopos y transcriptomas en células individuales". Nature Methods . 14 (9): 865–868. doi :10.1038/nmeth.4380. PMC 5669064 . PMID 28759029.
- ^ Raj B, Wagner DE, McKenna A, Pandey S, Klein AM, Shendure J, et al. (junio de 2018). "Perfiles simultáneos de células individuales de linajes y tipos de células en el cerebro de vertebrados". Nature Biotechnology . 36 (5): 442–450. doi :10.1038/nbt.4103. PMC 5938111 . PMID 29608178.
- ^ Olmos D, Arkenau HT, Ang JE, Ledaki I, Attard G, Carden CP, et al. (enero de 2009). "Las células tumorales circulantes (CTC) se consideran puntos finales intermedios en el cáncer de próstata resistente a la castración (CRPC): una experiencia en un solo centro". Anales de Oncología . 20 (1): 27–33. doi : 10.1093/annonc/mdn544 . PMID 18695026.
- ^ Levitin HM, Yuan J, Sims PA (abril de 2018). "Análisis transcriptómico de células individuales de la heterogeneidad tumoral". Tendencias en el cáncer . 4 (4): 264–268. doi :10.1016/j.trecan.2018.02.003. PMC 5993208 . PMID 29606308.
- ^ Jerby-Arnon L, Shah P, Cuoco MS, Rodman C, Su MJ, Melms JC, et al. (noviembre de 2018). "Un programa de células cancerosas promueve la exclusión de células T y la resistencia al bloqueo de puntos de control". Cell . 175 (4): 984–997.e24. doi :10.1016/j.cell.2018.09.006. PMC 6410377 . PMID 30388455.
- ^ Stephenson W, Donlin LT, Butler A, Rozo C, Bracken B, Rashidfarrokhi A, et al. (febrero de 2018). "Secuenciación de ARN de células individuales de tejido sinovial de artritis reumatoide mediante instrumentación microfluídica de bajo costo". Nature Communications . 9 (1): 791. Bibcode :2018NatCo...9..791S. doi :10.1038/s41467-017-02659-x. PMC 5824814 . PMID 29476078.
- ^ Avraham R, Haseley N, Brown D, Penaranda C, Jijon HB, Trombetta JJ, et al. (septiembre de 2015). "La variabilidad entre células de los patógenos impulsa la heterogeneidad en las respuestas inmunitarias del huésped". Cell . 162 (6): 1309–21. doi :10.1016/j.cell.2015.08.027. PMC 4578813 . PMID 26343579.
- ^ Cao J, Packer JS, Ramani V, Cusanovich DA, Huynh C, Daza R, et al. (agosto de 2017). "Perfil transcripcional unicelular integral de un organismo multicelular". Science . 357 (6352): 661–667. Bibcode :2017Sci...357..661C. doi :10.1126/science.aam8940. PMC 5894354 . PMID 28818938.
- ^ Plass M, Solana J, Wolf FA, Ayoub S, Misios A, Glažar P, et al. (mayo de 2018). "Atlas de tipos celulares y árbol de linaje de un animal complejo completo mediante transcriptómica de células individuales". Science . 360 (6391): eaaq1723. doi : 10.1126/science.aaq1723 . PMID 29674432.
- ^ Fincher CT, Wurtzel O, de Hoog T, Kravarik KM, Reddien PW (mayo de 2018). "Schmidtea mediterránea". Ciencia . 360 (6391): eaaq1736. doi : 10.1126/ciencia.aaq1736. PMC 6563842 . PMID 29674431.
- ^ Wagner DE, Weinreb C, Collins ZM, Briggs JA, Megason SG, Klein AM (junio de 2018). "Mapeo unicelular de paisajes de expresión génica y linaje en el embrión de pez cebra". Science . 360 (6392): 981–987. Bibcode :2018Sci...360..981W. doi :10.1126/science.aar4362. PMC 6083445 . PMID 29700229.
- ^ Farrell JA, Wang Y, Riesenfeld SJ, Shekhar K, Regev A, Schier AF (junio de 2018). "Reconstrucción unicelular de trayectorias de desarrollo durante la embriogénesis del pez cebra". Science . 360 (6392): eaar3131. doi :10.1126/science.aar3131. PMC 6247916 . PMID 29700225.
- ^ Briggs JA, Weinreb C, Wagner DE, Megason S, Peshkin L, Kirschner MW, et al. (junio de 2018). "La dinámica de la expresión génica en la embriogénesis de vertebrados con resolución unicelular". Science . 360 (6392): eaar5780. doi :10.1126/science.aar5780. PMC 6038144 . PMID 29700227.
- ^ You J. "El avance científico del año 2018: seguimiento del desarrollo célula por célula". Revista Science . Asociación Estadounidense para el Avance de la Ciencia.
- ^ ab Li H, Lovci MT, Kwon YS, Rosenfeld MG, Fu XD, Yeo GW (diciembre de 2008). "Determinación de la densidad de etiquetas requerida para el análisis del transcriptoma digital: aplicación a un modelo de cáncer de próstata sensible a los andrógenos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (51): 20179–84. Bibcode :2008PNAS..10520179L. doi : 10.1073/pnas.0807121105 . PMC 2603435 . PMID 19088194.
- ^ ab Stegle O, Parts L, Piipari M, Winn J, Durbin R (febrero de 2012). "Uso de la estimación probabilística de residuos de expresión (PEER) para obtener mayor potencia e interpretabilidad de los análisis de expresión génica". Nature Protocols . 7 (3): 500–7. doi :10.1038/nprot.2011.457. PMC 3398141 . PMID 22343431.
- ^ Kingsford C, Patro R (junio de 2015). "Compresión basada en referencia de secuencias de lectura corta utilizando codificación de ruta". Bioinformática . 31 (12): 1920–8. doi :10.1093/bioinformatics/btv071. PMC 4481695 . PMID 25649622.
- ^ ab Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, et al. (mayo de 2011). "Ensamblaje del transcriptoma de longitud completa a partir de datos de ARN-Seq sin un genoma de referencia". Nature Biotechnology . 29 (7): 644–52. doi :10.1038/nbt.1883. PMC 3571712 . PMID 21572440.
- ^ "Ensamblaje de novo con lecturas de Illumina" (PDF) . Consultado el 22 de octubre de 2016 .
- ^ Oasis: un ensamblador de transcriptomas para lecturas muy cortas
- ^ Zerbino DR, Birney E (mayo de 2008). "Velvet: algoritmos para el ensamblaje de lecturas cortas de novo utilizando grafos de Bruijn". Genome Research . 18 (5): 821–9. doi :10.1101/gr.074492.107. PMC 2336801 . PMID 18349386.
- ^ Chang Z, Li G, Liu J, Zhang Y, Ashby C, Liu D, et al. (febrero de 2015). "Bridger: un nuevo marco para el ensamblaje de novo del transcriptoma utilizando datos de ARN-seq". Genome Biology . 16 (1): 30. doi : 10.1186/s13059-015-0596-2 . PMC 4342890 . PMID 25723335.
- ^ Bushmanova E, Antipov D, Lapidus A, Prjibelski AD (septiembre de 2019). "rnaSPAdes: un ensamblador de transcriptoma de novo y su aplicación a datos de ARN-Seq". GigaScience . 8 (9). doi :10.1093/gigascience/giz100. PMC 6736328 . PMID 31494669.
- ^ ab Li B, Fillmore N, Bai Y, Collins M, Thomson JA, Stewart R, et al. (diciembre de 2014). "Evaluación de ensamblajes de transcriptomas de novo a partir de datos de ARN-Seq". Genome Biology . 15 (12): 553. doi : 10.1186/s13059-014-0553-5 . PMC 4298084 . PMID 25608678.
- ^ ab Dobin A, Davis CA, Schlesinger F, Drenkow J, Zaleski C, Jha S, et al. (enero de 2013). "STAR: alineador universal ultrarrápido de ARN-seq". Bioinformática . 29 (1): 15–21. doi :10.1093/bioinformatics/bts635. PMC 3530905 . PMID 23104886.
- ^ Langmead B , Trapnell C, Pop M, Salzberg SL (2009). "Alineamiento ultrarrápido y con memoria eficiente de secuencias cortas de ADN con el genoma humano". Genome Biology . 10 (3): R25. doi : 10.1186/gb-2009-10-3-r25 . PMC 2690996 . PMID 19261174.
- ^ Trapnell C, Pachter L , Salzberg SL (mayo de 2009). "TopHat: descubrimiento de uniones de empalme con RNA-Seq". Bioinformática . 25 (9): 1105–11. doi :10.1093/bioinformatics/btp120. PMC 2672628. PMID 19289445 .
- ^ ab Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley DR, et al. (marzo de 2012). "Análisis diferencial de la expresión génica y de transcripción de experimentos de secuenciación de ARN con TopHat y Cufflinks". Nature Protocols . 7 (3): 562–78. doi :10.1038/nprot.2012.016. PMC 3334321 . PMID 22383036.
- ^ Liao Y, Smyth GK, Shi W (mayo de 2013). "El alineador Subread: mapeo de lectura rápido, preciso y escalable mediante semilla y votación". Investigación de ácidos nucleicos . 41 (10): e108. doi :10.1093/nar/gkt214. PMC 3664803 . PMID 23558742.
- ^ Kim D, Langmead B, Salzberg SL (abril de 2015). "HISAT: un alineador de empalme rápido con bajos requisitos de memoria". Nature Methods . 12 (4): 357–60. doi :10.1038/nmeth.3317. PMC 4655817 . PMID 25751142.
- ^ Wu TD, Watanabe CK (mayo de 2005). "GMAP: un programa de mapeo y alineamiento genómico para secuencias de ARNm y EST". Bioinformática . 21 (9): 1859–75. doi : 10.1093/bioinformatics/bti310 . PMID 15728110.
- ^ Pertea M, Pertea GM, Antonescu CM, Chang TC, Mendell JT, Salzberg SL (marzo de 2015). "StringTie permite una reconstrucción mejorada de un transcriptoma a partir de lecturas de secuenciación de ARN". Nature Biotechnology . 33 (3): 290–5. doi :10.1038/nbt.3122. PMC 4643835 . PMID 25690850.
- ^ Baruzzo G, Hayer KE, Kim EJ, Di Camillo B, FitzGerald GA, Grant GR (febrero de 2017). "Evaluación comparativa integral basada en simulación de alineadores de secuenciación de ARN". Nature Methods . 14 (2): 135–139. doi :10.1038/nmeth.4106. PMC 5792058 . PMID 27941783.
- ^ Engström PG, Steijger T, Sipos B, Grant GR, Kahles A, Rätsch G, et al. (diciembre de 2013). "Evaluación sistemática de programas de alineamiento empalmado para datos de ARN-seq". Nature Methods . 10 (12): 1185–91. doi :10.1038/nmeth.2722. PMC 4018468 . PMID 24185836.
- ^ Lu B, Zeng Z, Shi T (febrero de 2013). "Estudio comparativo de las estrategias de ensamblaje de novo y de ensamblaje guiado por genoma para la reconstrucción del transcriptoma basada en RNA-Seq". Science China Life Sciences . 56 (2): 143–55. doi : 10.1007/s11427-013-4442-z . PMID 23393030.
- ^ Bradnam KR, Fass JN, Alexandrov A, Baranay P, Bechner M, Birol I, et al. (julio de 2013). "Assemblathon 2: evaluación de métodos de novo de ensamblaje del genoma en tres especies de vertebrados". GigaScience . 2 (1): 10. arXiv : 1301.5406 . Bibcode :2013arXiv1301.5406B. doi : 10.1186/2047-217X-2-10 . PMC 3844414 . PMID 23870653.
- ^ Hölzer M, Marz M (mayo de 2019). "Ensamblaje de transcriptomas de novo: una comparación integral entre especies de ensambladores de ARN-Seq de lectura corta". GigaScience . 8 (5). doi :10.1093/gigascience/giz039. PMC 6511074 . PMID 31077315.
- ^ Greenbaum D, Colangelo C, Williams K, Gerstein M (2003). "Comparación de la abundancia de proteínas y los niveles de expresión de ARNm a escala genómica". Genome Biology . 4 (9): 117. doi : 10.1186/gb-2003-4-9-117 . PMC 193646 . PMID 12952525.
- ^ Zhang ZH, Jhaveri DJ, Marshall VM, Bauer DC, Edson J, Narayanan RK, et al. (agosto de 2014). "Un estudio comparativo de técnicas para el análisis de expresión diferencial en datos de ARN-Seq". PLOS ONE . 9 (8): e103207. Bibcode :2014PLoSO...9j3207Z. doi : 10.1371/journal.pone.0103207 . PMC 4132098 . PMID 25119138.
- ^ Anders S, Pyl PT, Huber W (enero de 2015). "HTSeq: un marco de Python para trabajar con datos de secuenciación de alto rendimiento". Bioinformática . 31 (2): 166–9. doi :10.1093/bioinformatics/btu638. PMC 4287950 . PMID 25260700.
- ^ Liao Y, Smyth GK, Shi W (abril de 2014). "featureCounts: un programa eficiente de propósito general para asignar lecturas de secuencias a características genómicas". Bioinformática . 30 (7): 923–30. arXiv : 1305.3347 . doi :10.1093/bioinformatics/btt656. PMID 24227677.
- ^ Schmid MW, Grossniklaus U (febrero de 2015). "Rcount: conteo de lecturas de ARN-Seq simple y flexible". Bioinformática . 31 (3): 436–7. doi : 10.1093/bioinformatics/btu680 . PMID 25322836.
- ^ Finotello F, Lavezzo E, Bianco L, Barzon L, Mazzon P, Fontana P, et al. (2014). "Reducir el sesgo en los datos de secuenciación de ARN: un enfoque novedoso para calcular recuentos". Bioinformática BMC . 15 (Suplemento 1): T7. doi : 10.1186/1471-2105-15-s1-s7 . PMC 4016203 . PMID 24564404.
- ^ Hashimoto TB, Edwards MD, Gifford DK (marzo de 2014). "Corrección de recuento universal para secuenciación de alto rendimiento". PLOS Computational Biology . 10 (3): e1003494. Bibcode :2014PLSCB..10E3494H. doi : 10.1371/journal.pcbi.1003494 . PMC 3945112 . PMID 24603409.
- ^ Patro R, Mount SM, Kingsford C (mayo de 2014). "Sailfish permite la cuantificación de isoformas sin alineamiento a partir de lecturas de secuenciación de ARN utilizando algoritmos livianos". Nature Biotechnology . 32 (5): 462–4. arXiv : 1308.3700 . doi :10.1038/nbt.2862. PMC 4077321 . PMID 24752080.
- ^ Bray NL, Pimentel H, Melsted P, Pachter L (mayo de 2016). "Cuantificación probabilística de secuenciación de ARN casi óptima". Nature Biotechnology . 34 (5): 525–7. doi :10.1038/nbt.3519. PMID 27043002. S2CID 205282743.
- ^ ab Robinson MD, Oshlack A (2010). "Un método de normalización de escala para el análisis de expresión diferencial de datos de ARN-seq". Genome Biology . 11 (3): R25. doi : 10.1186/gb-2010-11-3-r25 . PMC 2864565 . PMID 20196867.
- ^ Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, et al. (mayo de 2010). "El ensamblaje y cuantificación de transcripciones mediante RNA-Seq revela transcripciones no anotadas y cambio de isoformas durante la diferenciación celular". Nature Biotechnology . 28 (5): 511–5. doi :10.1038/nbt.1621. PMC 3146043 . PMID 20436464.
- ^ ab Pachter L (19 de abril de 2011). "Modelos para cuantificación de transcripción a partir de ARN-Seq". arXiv : 1104.3889 [q-bio.GN].
- ^ "¿Qué es el FPKM? Una revisión de las unidades de expresión de RNA-Seq". El fárrago . 8 de mayo de 2014 . Consultado el 28 de marzo de 2018 .
- ^ Wagner GP, Kin K, Lynch VJ (diciembre de 2012). "Medición de la abundancia de ARNm utilizando datos de secuenciación de ARN: la medida de RPKM es inconsistente entre muestras". Teoría en biociencias . 131 (4): 281–5. doi :10.1007/s12064-012-0162-3. PMID 22872506. S2CID 16752581.
- ^ Evans C, Hardin J, Stoebel DM (septiembre de 2018). "Selección de métodos de normalización de secuencias de ARN entre muestras desde la perspectiva de sus suposiciones". Briefings in Bioinformatics . 19 (5): 776–792. doi :10.1093/bib/bbx008. PMC 6171491 . PMID 28334202.
- ^ ab Law CW, Chen Y, Shi W, Smyth GK (febrero de 2014). "voom: Los pesos de precisión desbloquean herramientas de análisis de modelos lineales para recuentos de lecturas de ARN-seq". Genome Biology . 15 (2): R29. doi : 10.1186/gb-2014-15-2-r29 . PMC 4053721 . PMID 24485249.
- ^ ab Anders S, Huber W (2010). "Análisis de expresión diferencial para datos de recuento de secuencias". Genome Biology . 11 (10): R106. doi : 10.1186/gb-2010-11-10-r106 . PMC 3218662 . PMID 20979621.
- ^ ab Robinson MD, McCarthy DJ, Smyth GK (enero de 2010). "edgeR: un paquete Bioconductor para el análisis de expresión diferencial de datos de expresión génica digital". Bioinformática . 26 (1): 139–40. doi :10.1093/bioinformatics/btp616. PMC 2796818 . PMID 19910308.
- ^ Marguerat S, Schmidt A, Codlin S, Chen W, Aebersold R, Bähler J (octubre de 2012). "Análisis cuantitativo de los transcriptomas y proteomas de la levadura de fisión en células proliferantes y quiescentes". Cell . 151 (3): 671–83. doi :10.1016/j.cell.2012.09.019. PMC 3482660 . PMID 23101633.
- ^ Owens ND, Blitz IL, Lane MA, Patrushev I, Overton JD, Gilchrist MJ, et al. (enero de 2016). "La medición del número absoluto de copias de ARN con alta resolución temporal revela la cinética del transcriptoma en el desarrollo". Cell Reports . 14 (3): 632–647. doi :10.1016/j.celrep.2015.12.050. PMC 4731879 . PMID 26774488.
- ^ Chen K, Hu Z, Xia Z, Zhao D, Li W, Tyler JK (diciembre de 2015). "El hecho pasado por alto: necesidad fundamental de control de la adición de la proteína en prácticamente todos los análisis de todo el genoma". Biología molecular y celular . 36 (5): 662–7. doi :10.1128/MCB.00970-14. PMC 4760223 . PMID 26711261.
- ^ Lovén J, Orlando DA, Sigova AA, Lin CY, Rahl PB, Burge CB, et al. (octubre de 2012). "Revisitando el análisis de expresión génica global". Cell . 151 (3): 476–82. doi :10.1016/j.cell.2012.10.012. PMC 3505597 . PMID 23101621.
- ^ Ritchie ME, Phipson B, Wu D, Hu Y, Law CW, Shi W, et al. (abril de 2015). "Limma potencia los análisis de expresión diferencial para estudios de secuenciación de ARN y microarrays". Nucleic Acids Research . 43 (7): e47. doi :10.1093/nar/gkv007. PMC 4402510 . PMID 25605792.
- ^ "Bioconductor - Software de código abierto para bioinformática".
- ^ Huber W, Carey VJ, Gentleman R, Anders S, Carlson M, Carvalho BS, et al. (febrero de 2015). "Orquestación de análisis genómico de alto rendimiento con Bioconductor". Nature Methods . 12 (2): 115–21. doi :10.1038/nmeth.3252. PMC 4509590 . PMID 25633503.
- ^ Leek JT, Storey JD (septiembre de 2007). "Captura de la heterogeneidad en los estudios de expresión génica mediante análisis de variables sustitutas". PLOS Genetics . 3 (9): 1724–35. doi : 10.1371/journal.pgen.0030161 . PMC 1994707 . PMID 17907809.
- ^ Pimentel H, Bray NL, Puente S, Melsted P, Pachter L (julio de 2017). "Análisis diferencial de secuencias de ARN que incorporan incertidumbre de cuantificación". Nature Methods . 14 (7): 687–690. doi :10.1038/nmeth.4324. PMID 28581496. S2CID 15063247.
- ^ Trapnell C, Hendrickson DG, Sauvageau M, Goff L, Rinn JL, Pachter L (enero de 2013). "Análisis diferencial de la regulación génica en la resolución de la transcripción con RNA-seq". Nature Biotechnology . 31 (1): 46–53. doi :10.1038/nbt.2450. PMC 3869392 . PMID 23222703.
- ^ Frazee AC, Pertea G, Jaffe AE, Langmead B, Salzberg SL, Leek JT (marzo de 2015). "Ballgown cierra la brecha entre el ensamblaje del transcriptoma y el análisis de expresión". Nature Biotechnology . 33 (3): 243–6. doi :10.1038/nbt.3172. PMC 4792117 . PMID 25748911.
- ^ ab Sahraeian SM, Mohiyuddin M, Sebra R, Tilgner H, Afshar PT, Au KF, et al. (julio de 2017). "Obtención de una visión biológica completa del transcriptoma mediante la realización de un análisis de secuenciación de ARN de amplio espectro". Nature Communications . 8 (1): 59. Bibcode :2017NatCo...8...59S. doi :10.1038/s41467-017-00050-4. PMC 5498581 . PMID 28680106.
- ^ Ziemann M, Eren Y, El-Osta A (agosto de 2016). "Los errores en los nombres de los genes están muy extendidos en la literatura científica". Genome Biology . 17 (1): 177. doi : 10.1186/s13059-016-1044-7 . PMC 4994289 . PMID 27552985.
- ^ Soneson C, Delorenzi M (marzo de 2013). "Una comparación de métodos para el análisis de expresión diferencial de datos de secuenciación de ARN". BMC Bioinformatics . 14 : 91. doi : 10.1186/1471-2105-14-91 . PMC 3608160 . PMID 23497356.
- ^ Fonseca NA, Marioni J, Brazma A (30 de septiembre de 2014). "Perfiles genéticos de ARN-Seq: una comparación empírica sistemática". PLOS ONE . 9 (9): e107026. Bibcode :2014PLoSO...9j7026F. doi : 10.1371/journal.pone.0107026 . PMC 4182317 . PMID 25268973.
- ^ Seyednasrollah F, Laiho A, Elo LL (enero de 2015). "Comparación de paquetes de software para detectar la expresión diferencial en estudios de secuenciación de ARN". Briefings in Bioinformatics . 16 (1): 59–70. doi :10.1093/bib/bbt086. PMC 4293378 . PMID 24300110.
- ^ Rapaport F, Khanin R, Liang Y, Pirun M, Krek A, Zumbo P, et al. (2013). "Evaluación integral de métodos de análisis de expresión génica diferencial para datos de RNA-seq". Biología del genoma . 14 (9): R95. doi : 10.1186/gb-2013-14-9-r95 . PMC 4054597 . PMID 24020486.
- ^ Costa-Silva J, Domingues D, Lopes FM (21 de diciembre de 2017). "Análisis de expresión diferencial de ARN-Seq: una revisión ampliada y una herramienta de software". PLOS ONE . 12 (12): e0190152. Bibcode :2017PLoSO..1290152C. doi : 10.1371/journal.pone.0190152 . PMC 5739479 . PMID 29267363.
- ^ Corchete LA, Rojas EA, Alonso-López D, De Las Rivas J, Gutiérrez NC, Burguillo FJ (12 de noviembre de 2020). "Comparación y evaluación sistemática de procedimientos de RNA-seq para el análisis cuantitativo de expresión génica". Informes científicos . 12 (10): 19737. Código bibliográfico : 2020NatSR..1019737C. doi : 10.1038/s41598-020-76881-x . PMC 7665074 . PMID 33184454.
- ^ Liao Y, Wang J, Jaehnig EJ, Shi Z, Zhang B (julio de 2019). "WebGestalt 2019: kit de herramientas de análisis de conjuntos de genes con interfaces de usuario y API renovadas". Nucleic Acids Research . 47 (W1): W199–W205. doi :10.1093/nar/gkz401. PMC 6602449 . PMID 31114916.
- ^ ab Keren H, Lev-Maor G, Ast G (mayo de 2010). "Empalme alternativo y evolución: diversificación, definición y función del exón". Nature Reviews. Genética . 11 (5): 345–55. doi :10.1038/nrg2776. PMID 20376054. S2CID 5184582.
- ^ Liu R, Loraine AE, Dickerson JA (diciembre de 2014). "Comparaciones de métodos computacionales para la detección de splicing alternativo diferencial utilizando RNA-seq en sistemas vegetales". BMC Bioinformatics . 15 (1): 364. doi : 10.1186/s12859-014-0364-4 . PMC 4271460 . PMID 25511303.
- ^ ab Li YI, Knowles DA, Humphrey J, Barbeira AN, Dickinson SP, Im HK, et al. (enero de 2018). "Cuantificación sin anotaciones del empalme de ARN utilizando LeafCutter". Nature Genetics . 50 (1): 151–158. doi :10.1038/s41588-017-0004-9. PMC 5742080 . PMID 29229983.
- ^ Anders S, Reyes A, Huber W (octubre de 2012). "Detección del uso diferencial de exones a partir de datos de secuenciación de ARN". Genome Research . 22 (10): 2008–17. doi :10.1101/gr.133744.111. PMC 3460195 . PMID 22722343.
- ^ Shen S, Park JW, Huang J, Dittmar KA, Lu ZX, Zhou Q, et al. (abril de 2012). "MATS: un marco bayesiano para la detección flexible de empalmes alternativos diferenciales a partir de datos de ARN-Seq". Investigación de ácidos nucleicos . 40 (8): e61. doi :10.1093/nar/gkr1291. PMC 3333886 . PMID 22266656.
- ^ Wang X, Cairns MJ (junio de 2014). "SeqGSEA: un paquete Bioconductor para el análisis de enriquecimiento de conjuntos de genes de datos de ARN-Seq que integran expresión diferencial y empalme". Bioinformática . 30 (12): 1777–9. doi : 10.1093/bioinformatics/btu090 . PMID 24535097.
- ^ Trapnell C, Hendrickson DG, Sauvageau M, Goff L, Rinn JL, Pachter L (enero de 2013). "Análisis diferencial de la regulación génica en la resolución de la transcripción con RNA-seq". Nature Biotechnology . 31 (1): 46–53. doi :10.1038/nbt.2450. PMC 3869392 . PMID 23222703.
- ^ Hu Y, Huang Y, Du Y, Orellana CF, Singh D, Johnson AR, et al. (enero de 2013). "DiffSplice: la detección de eventos de splicing diferencial en todo el genoma con RNA-seq". Nucleic Acids Research . 41 (2): e39. doi :10.1093/nar/gks1026. PMC 3553996 . PMID 23155066.
- ^ Vaquero-Garcia J, Barrera A, Gazzara MR, González-Vallinas J, Lahens NF, Hogenesch JB, et al. (febrero de 2016). "Una nueva visión de la complejidad y regulación del transcriptoma a través de la lente de las variaciones de empalme local". eLife . 5 : e11752. doi : 10.7554/eLife.11752 . PMC 4801060 . PMID 26829591.
- ^ Merino GA, Conesa A, Fernández EA (marzo de 2019). "Una evaluación comparativa de los flujos de trabajo para detectar el splicing diferencial y la expresión diferencial a nivel de isoforma en estudios de ARN-seq humanos". Briefings in Bioinformatics . 20 (2): 471–481. doi :10.1093/bib/bbx122. hdl : 11336/41247 . PMID 29040385. S2CID 22706028.
- ^ ab Marcotte EM, Pellegrini M, Thompson MJ, Yeates TO, Eisenberg D (noviembre de 1999). "Un algoritmo combinado para la predicción de la función de las proteínas en todo el genoma". Nature . 402 (6757): 83–6. Bibcode :1999Natur.402...83M. doi :10.1038/47048. PMID 10573421. S2CID 144447.
- ^ ab Giorgi FM, Del Fabbro C, Licausi F (marzo de 2013). "Estudio comparativo de redes de coexpresión derivadas de ARN-seq y microarrays en Arabidopsis thaliana". Bioinformática . 29 (6): 717–24. doi : 10.1093/bioinformatics/btt053 . hdl : 11390/990155 . PMID 23376351.
- ^ Iancu OD, Kawane S, Bottomly D, Searles R, Hitzemann R, McWeeney S (junio de 2012). "Utilización de datos de RNA-Seq para la inferencia de redes de coexpresión de novo". Bioinformática . 28 (12): 1592–7. doi :10.1093/bioinformatics/bts245. PMC 3493127 . PMID 22556371.
- ^ Eksi R, Li HD, Menon R, Wen Y, Omenn GS, Kretzler M, et al. (noviembre de 2013). "Diferenciación sistemática de funciones para isoformas con empalme alternativo mediante la integración de datos de secuenciación de ARN". PLOS Computational Biology . 9 (11): e1003314. Bibcode :2013PLSCB...9E3314E. doi : 10.1371/journal.pcbi.1003314 . PMC 3820534 . PMID 24244129.
- ^ Li HD, Menon R, Omenn GS, Guan Y (agosto de 2014). "La era emergente de la integración de datos genómicos para analizar la función de las isoformas de empalme". Tendencias en genética . 30 (8): 340–7. doi :10.1016/j.tig.2014.05.005. PMC 4112133 . PMID 24951248.
- ^ Foroushani A, Agrahari R, Docking R, Chang L, Duns G, Hudoba M, et al. (marzo de 2017). "El análisis de la red genética a gran escala revela la importancia de la vía de la matriz extracelular y los genes homeobox en la leucemia mieloide aguda: una introducción al paquete Pigengene y sus aplicaciones". BMC Medical Genomics . 10 (1): 16. doi : 10.1186/s12920-017-0253-6 . PMC 5353782 . PMID 28298217.
- ^ Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, et al. (agosto de 2009). "El formato de alineación/mapa de secuencias y SAMtools". Bioinformática . 25 (16): 2078–9. doi :10.1093/bioinformatics/btp352. PMC 2723002 . PMID 19505943.
- ^ DePristo MA, Banks E, Poplin R, Garimella KV, Maguire JR, Hartl C, et al. (mayo de 2011). "Un marco para el descubrimiento de variaciones y el genotipado utilizando datos de secuenciación de ADN de próxima generación". Genética de la Naturaleza . 43 (5): 491–8. doi :10.1038/ng.806. PMC 3083463 . PMID 21478889.
- ^ Battle A, Brown CD, Engelhardt BE, Montgomery SB (octubre de 2017). "Efectos genéticos en la expresión génica en los tejidos humanos". Nature . 550 (7675): 204–213. Bibcode :2017Natur.550..204A. doi :10.1038/nature24277. hdl : 10230/34202 . PMC 5776756 . PMID 29022597.
- ^ Richter F, Hoffman GE, Manheimer KB, Patel N, Sharp AJ, McKean D, et al. (octubre de 2019). "ORE identifica efectos de expresión extremos enriquecidos para variantes raras". Bioinformática . 35 (20): 3906–3912. doi :10.1093/bioinformatics/btz202. PMC 6792115 . PMID 30903145.
- ^ Freedman AH, Clamp M, Sackton TB (enero de 2021). "Error, ruido y sesgo en ensamblajes de transcriptomas de novo". Recursos de ecología molecular . 21 (1): 18–29. doi :10.1111/1755-0998.13156. PMID 32180366. S2CID 212739959.
- ^ Teixeira MR (diciembre de 2006). "Oncogenes de fusión recurrentes en carcinomas". Critical Reviews in Oncogenesis . 12 (3–4): 257–71. doi :10.1615/critrevoncog.v12.i3-4.40. PMID 17425505. S2CID 40770452.
- ^ Thind AS, Monga I, Thakur PK, Kumari P, Dindhoria K, Krzak M, et al. (noviembre de 2021). "Desmitificando las aplicaciones emergentes de secuenciación masiva de ARN: la aplicación y la utilidad de la metodología bioinformática". Briefings in Bioinformatics . 22 (6). doi :10.1093/bib/bbab259. PMID 34329375.
- ^ Sangiovanni M, Granata I, Thind AS, Guarracino MR (abril de 2019). "De la basura al tesoro: detección de contaminación inesperada en datos NGS no mapeados". BMC Bioinformatics . 20 (Supl 4): 168. doi : 10.1186/s12859-019-2684-x . PMC 6472186 . PMID 30999839.
- ^ "Búsqueda en PubMed: "RNA Seq" O "RNA-Seq" O "RNA sequencing" O "RNASeq"". PubMed . Consultado el 20 de junio de 2021 .
- ^ "Búsqueda en PubMed: ("RNA Seq" O "RNA-Seq" O "RNA sequencing" O "RNASeq") Y "Medicine"". PubMed . Consultado el 20 de junio de 2021 .
- ^ Weber AP (noviembre de 2015). "Descubrimiento de nueva biología a través de la secuenciación del ARN". Fisiología vegetal . 169 (3): 1524–31. doi :10.1104/pp.15.01081. PMC 4634082 . PMID 26353759.
- ^ Bainbridge MN, Warren RL, Hirst M, Romanuik T, Zeng T, Go A, et al. (septiembre de 2006). "Análisis del transcriptoma de la línea celular de cáncer de próstata LNCaP utilizando un enfoque de secuenciación por síntesis". BMC Genomics . 7 : 246. doi : 10.1186/1471-2164-7-246 . PMC 1592491 . PMID 17010196.
- ^ Cheung F, Haas BJ, Goldberg SM, May GD, Xiao Y, Town CD (octubre de 2006). "Secuenciación de etiquetas secuenciadas expresadas en Medicago truncatula utilizando la tecnología 454 Life Sciences". BMC Genomics . 7 : 272. doi : 10.1186/1471-2164-7-272 . PMC 1635983 . PMID 17062153.
- ^ Emrich SJ, Barbazuk WB, Li L, Schnable PS (enero de 2007). "Descubrimiento y anotación de genes mediante la secuenciación del transcriptoma LCM-454". Genome Research . 17 (1): 69–73. doi :10.1101/gr.5145806. PMC 1716268 . PMID 17095711.
- ^ Weber AP, Weber KL, Carr K, Wilkerson C, Ohlrogge JB (mayo de 2007). "Muestreo del transcriptoma de Arabidopsis con pirosecuenciación masivamente paralela". Fisiología vegetal . 144 (1): 32–42. doi :10.1104/pp.107.096677. PMC 1913805 . PMID 17351049.
- ^ Nagalakshmi U, Wang Z, Waern K, Shou C, Raha D, Gerstein M, et al. (junio de 2008). "El panorama transcripcional del genoma de la levadura definido por la secuenciación del ARN". Science . 320 (5881): 1344–9. Bibcode :2008Sci...320.1344N. doi :10.1126/science.1158441. PMC 2951732 . PMID 18451266.
- ^ Richter F (2021). "Una amplia introducción a RNA-Seq". WikiRevista científica . 4 (1): 4. doi : 10.15347/WJS/2021.004 .
- ^ Cummings BB, Marshall JL, Tukiainen T, Lek M, Donkervoort S, Foley AR, et al. (19 de abril de 2017). "Mejora del diagnóstico genético en enfermedades mendelianas con secuenciación del transcriptoma". Science Translational Medicine . 9 (386). doi :10.1126/scitranslmed.aal5209. hdl : 10230/35912 .
- ^ Kremer LS, Bader DM, Mertes C, Kopajtich R, Pichler G, Iuso A, et al. (12 de junio de 2017). "Diagnóstico genético de trastornos mendelianos mediante secuenciación de ARN". Nature Communications . 8 (1). doi :10.1038/ncomms15824.
- ^ Sandberg R (enero de 2014). "Entrando en la era de la transcriptómica unicelular en biología y medicina". Nature Methods . 11 (1): 22–4. doi :10.1038/nmeth.2764. PMID 24524133. S2CID 27632439.
- ^ "Matriz de datos ENCODE" . Consultado el 28 de julio de 2013 .
- ^ "El Atlas del Genoma del Cáncer – Portal de Datos" . Consultado el 28 de julio de 2013 .
Lectura adicional
- Taguchi Y (2019). "Análisis transcriptómico comparativo". Enciclopedia de bioinformática y biología computacional . págs. 814–818. doi :10.1016/B978-0-12-809633-8.20163-5. ISBN . 978-0-12-811432-2.S2CID65302519 .
Enlaces externos
- Cresko B, Voelker R, Small C (2001). Bassham S, Catchen J (eds.). "RNA-seqlopedia". Universidad de Oregon.:una guía de alto nivel para diseñar e implementar un experimento de ARN-Seq.
