Repetición rica en leucina
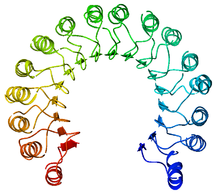 Un ejemplo de una proteína repetida rica en leucina, un inhibidor de la ribonucleasa porcina | |||||||||
| Identificadores | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Símbolo | LRR_1 | ||||||||
| Pfam | PF00560 | ||||||||
| Clan Pfam | CL0022 | ||||||||
| ECOD | 207.1.1 | ||||||||
| Interprofesional | IPR001611 | ||||||||
| SCOP2 | 2 mil millones de habitantes / ALCANCE / SUPFAM | ||||||||
| Membranoma | 605 | ||||||||
| |||||||||
| Variante repetida rica en leucina | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Una variante repetida rica en leucina con un nuevo motivo estructural proteico repetitivo | |||||||||
| Identificadores | |||||||||
| Símbolo | Vehículo de pasajeros ligero (LRV) | ||||||||
| Pfam | PF01816 | ||||||||
| Clan Pfam | CL0020 | ||||||||
| Interprofesional | IPR004830 | ||||||||
| SCOP2 | 1lrv / ALCANCE / SUPFAM | ||||||||
| Membranoma | 737 | ||||||||
| |||||||||
| LRR adyacente | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 internalina h: estructura cristalina de los dominios n-terminales fusionados. | |||||||||
| Identificadores | |||||||||
| Símbolo | LRR_adyacente | ||||||||
| Pfam | PF08191 | ||||||||
| Interprofesional | IPR012569 | ||||||||
| Membranoma | 341 | ||||||||
| |||||||||
| Dominio N-terminal de repetición rica en leucina | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Decorina dimérica extraída de tejido bovino, forma cristalina 2 | |||||||||
| Identificadores | |||||||||
| Símbolo | LRRNT | ||||||||
| Pfam | PF01462 | ||||||||
| Interprofesional | IPR000372 | ||||||||
| ELEGANTE | LRRNT | ||||||||
| SCOP2 | 1m10 / ALCANCE / SUPFAM | ||||||||
| Membranoma | 127 | ||||||||
| |||||||||
| Dominio N-terminal de repetición rica en leucina | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 La estructura cristalina de pgip ( proteína inhibidora de poligalacturonasa ), una proteína repetida rica en leucina involucrada en la defensa de las plantas. | |||||||||
| Identificadores | |||||||||
| Símbolo | LRRNT_2 | ||||||||
| Pfam | PF08263 | ||||||||
| Interprofesional | IPR013210 | ||||||||
| ELEGANTE | LRRNT | ||||||||
| SCOP2 | 1m10 / ALCANCE / SUPFAM | ||||||||
| |||||||||
| Dominio C-terminal de repetición rica en leucina | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Tercer dominio lrr de la hendidura de la drosophila | |||||||||
| Identificadores | |||||||||
| Símbolo | LRRCT | ||||||||
| Pfam | PF01463 | ||||||||
| Interprofesional | IPR000483 | ||||||||
| ELEGANTE | LRRCT | ||||||||
| SCOP2 | 1m10 / ALCANCE / SUPFAM | ||||||||
| |||||||||
| Grupo FeS4 de proteínas LRV | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Una variante repetida rica en leucina con un nuevo motivo estructural proteico repetitivo | |||||||||
| Identificadores | |||||||||
| Símbolo | LRV_FeS | ||||||||
| Pfam | PF05484 | ||||||||
| Clan Pfam | CL0020 | ||||||||
| Interprofesional | IPR008665 | ||||||||
| SCOP2 | 1lrv / ALCANCE / SUPFAM | ||||||||
| |||||||||
Una repetición rica en leucina ( LRR ) es un motivo estructural de proteína que forma un pliegue de herradura α/β . [1] [2] Está compuesta por tramos repetidos de 20 a 30 aminoácidos que son inusualmente ricos en el aminoácido hidrofóbico leucina . Estas repeticiones en tándem comúnmente se pliegan juntas para formar un dominio de proteína solenoide , denominado dominio de repetición rico en leucina . Típicamente, cada unidad de repetición tiene una estructura de hélice alfa de cadena beta - giro , y el dominio ensamblado , compuesto de muchas de estas repeticiones, tiene una forma de herradura con una lámina beta paralela interior y una matriz exterior de hélices. Una cara de la lámina beta y un lado de la matriz de hélices están expuestas al solvente y, por lo tanto, están dominadas por residuos hidrofílicos . La región entre las hélices y las láminas es el núcleo hidrofóbico de la proteína y está firmemente empaquetada estéricamente con residuos de leucina.
Las repeticiones ricas en leucina participan con frecuencia en la formación de interacciones proteína-proteína . [3] [4]
Ejemplos
Se han identificado motivos repetidos ricos en leucina en una gran cantidad de proteínas funcionalmente no relacionadas. [5] El ejemplo más conocido es el inhibidor de la ribonucleasa , pero otras proteínas como el regulador de la tropomiosina, la tropomodulina , y el receptor tipo Toll también comparten el motivo. De hecho, el receptor tipo Toll posee 10 motivos LRR sucesivos que sirven para unir patrones moleculares asociados a patógenos y peligros.
Aunque la proteína LRR canónica contiene aproximadamente una hélice por cada cadena beta, las variantes que forman pliegues de superhélice beta-alfa a veces tienen bucles largos en lugar de hélices que unen cadenas beta sucesivas.
Un dominio variante de repetición rico en leucina (LRV) tiene un nuevo motivo estructural repetitivo que consiste en hélices alfa y 3 10 alternas dispuestas en una superhélice dextrógira, con la ausencia de las láminas beta presentes en otras repeticiones ricas en leucina. [6]
Dominios asociados
Las repeticiones ricas en leucina suelen estar flanqueadas por dominios ricos en cisteína N-terminal y C-terminal , pero no siempre como es el caso de C5orf36.
También coexisten con los dominios adyacentes de LRR. Se trata de dominios pequeños, todos de cadena beta , que se han descrito estructuralmente para la proteína Internalina (InlA) y las proteínas relacionadas InlB, InlE, InlH de la bacteria patógena Listeria monocytogenes . Su función parece ser principalmente estructural: se fusionan al extremo C-terminal de repeticiones ricas en leucina, estabilizando significativamente el LRR y formando una entidad rígida común con el LRR. No participan en las interacciones proteína-proteína , pero ayudan a presentar el dominio LRR adyacente para este propósito. Estos dominios pertenecen a la familia de dominios similares a Ig , ya que consisten en dos láminas beta intercaladas que siguen la conectividad clásica de los dominios Ig. Sin embargo, las cadenas beta en una de las láminas son mucho más pequeñas que en la mayoría de los dominios similares a Ig estándar, lo que la convierte en algo atípico. [7] [8] [9]
En el extremo N de algunas proteínas que contienen el dominio de variante de repetición rico en leucina (LRV) se encuentra un grupo de azufre de hierro . Estas proteínas tienen una estructura de dos dominios, compuesta por un pequeño dominio N-terminal que contiene un grupo de cuatro residuos de cisteína que alberga el grupo 4Fe:4S y un dominio C-terminal más grande que contiene las repeticiones LRV. [6] Los estudios bioquímicos revelaron que el grupo 4Fe:4S es sensible al oxígeno , pero no parece tener actividad redox reversible .
Véase también
Referencias
- ^ Kobe B, Deisenhofer J (octubre de 1994). "La repetición rica en leucina: un motivo de unión versátil". Trends Biochem. Sci . 19 (10): 415–21. doi :10.1016/0968-0004(94)90090-6. PMID 7817399.
- ^ Enkhbayar P, Kamiya M, Osaki M, Matsumoto T, Matsushima N (febrero de 2004). "Principios estructurales de las proteínas con repeticiones ricas en leucina (LRR)". Proteins . 54 (3): 394–403. doi :10.1002/prot.10605. PMID 14747988. S2CID 19951452.
- ^ Kobe B, Kajava AV (diciembre de 2001). "La repetición rica en leucina como motivo de reconocimiento de proteínas". Curr. Opin. Struct. Biol . 11 (6): 725–32. doi :10.1016/S0959-440X(01)00266-4. PMID 11751054.
- ^ Gay NJ, Packman LC, Weldon MA, Barna JC (octubre de 1991). "Un péptido repetido rico en leucina derivado del receptor Toll de Drosophila forma filamentos extendidos con una estructura de lámina beta". FEBS Lett . 291 (1): 87–91. doi : 10.1016/0014-5793(91)81110-T . PMID 1657640. S2CID 84294221.
- ^ Rothberg JM, Jacobs JR, Goodman CS, Artavanis-Tsakonas S (diciembre de 1990). "slit: una proteína extracelular necesaria para el desarrollo de las vías de la glía de la línea media y del axón comisural contiene dominios EGF y LRR". Genes Dev . 4 (12A): 2169–87. doi : 10.1101/gad.4.12a.2169 . PMID 2176636.
- ^ ab Peters JW, Stowell MH, Rees DC (diciembre de 1996). "Una variante repetida rica en leucina con un nuevo motivo estructural repetitivo de proteína". Nat. Struct. Biol . 3 (12): 991–4. doi :10.1038/nsb1296-991. PMID 8946850. S2CID 36535731.
- ^ Schubert WD, Gobel G, Diepholz M, Darji A, Kloer D, Hain T, Chakraborty T, Wehland J, Domann E, Heinz DW (septiembre de 2001). "Las internalinas del patógeno humano Listeria monocytogenes combinan tres pliegues distintos en un dominio de internalina contiguo". J. Mol. Biol . 312 (4): 783–94. doi :10.1006/jmbi.2001.4989. PMID 11575932.
- ^ Schubert WD, Urbanke C, Ziehm T, Beier V, Machner MP, Domann E, Wehland J, Chakraborty T, Heinz DW (diciembre de 2002). "Estructura de la internalina, una proteína de invasión importante de Listeria monocytogenes, en complejo con su receptor humano E-cadherina". Cell . 111 (6): 825–36. doi : 10.1016/S0092-8674(02)01136-4 . PMID 12526809. S2CID 17232767.
- ^ Freiberg A, Machner MP, Pfeil W, Schubert WD, Heinz DW, Seckler R (marzo de 2004). "Plegamiento y estabilidad del dominio de repetición rico en leucina de la internalina B de Listeri monocytogenes". J. Mol. Biol . 337 (2): 453–61. doi :10.1016/j.jmb.2004.01.044. PMID 15003459.
Lectura adicional
- Tooze, John; Brändén, Carl-Ivar (1999). Introducción a la estructura de las proteínas (2.ª ed.). Nueva York: Garland Publishing. ISBN 0-8153-2305-0.
- Wei T, Gong J, Jamitzky F, Heckl WM, Stark RW, Roessle SC (noviembre de 2008). "LRRML: una base de datos conformacional y una descripción XML de repeticiones ricas en leucina (LRR)". BMC Struct. Biol . 8 (1): 47. doi : 10.1186/1472-6807-8-47 . PMC 2645405. PMID 18986514 .
Enlaces externos
- Recurso de motivo lineal eucariota de clase LIG_SCF_Skp2-Cks1_1
- Pliegue SCOP LRR
- Arquitectura de herradura alfa-beta de CATH
- LRRML: una base de datos conformacional de repeticiones ricas en leucina