Receptor de superficie celular
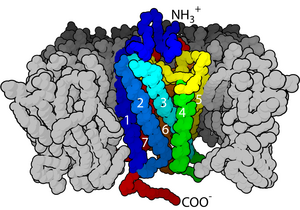
Los receptores de superficie celular ( receptores de membrana , receptores transmembrana ) son receptores que están incrustados en la membrana plasmática de las células . [1] Actúan en la señalización celular al recibir (unirse a) moléculas extracelulares . Son proteínas de membrana integrales especializadas que permiten la comunicación entre la célula y el espacio extracelular . Las moléculas extracelulares pueden ser hormonas , neurotransmisores , citocinas , factores de crecimiento , moléculas de adhesión celular o nutrientes ; reaccionan con el receptor para inducir cambios en el metabolismo y la actividad de una célula. En el proceso de transducción de señales , la unión del ligando afecta un cambio químico en cascada a través de la membrana celular.
Estructura y mecanismo
Muchos receptores de membrana son proteínas transmembrana . Hay varios tipos, incluidas las glicoproteínas y las lipoproteínas . [2] Se conocen cientos de receptores diferentes y aún quedan muchos más por estudiar. [3] [4] Los receptores transmembrana se clasifican típicamente según su estructura terciaria (tridimensional). Si se desconoce la estructura tridimensional, se pueden clasificar según la topología de la membrana . En los receptores más simples, las cadenas polipeptídicas cruzan la bicapa lipídica una vez, mientras que otros, como los receptores acoplados a proteína G , lo hacen hasta siete veces. Cada membrana celular puede tener varios tipos de receptores de membrana, con distribuciones de superficie variables. Un solo receptor también puede estar distribuido de manera diferente en diferentes posiciones de la membrana, dependiendo del tipo de membrana y la función celular. Los receptores a menudo se agrupan en la superficie de la membrana, en lugar de distribuirse uniformemente. [5] [6]
Mecanismo
Se han propuesto dos modelos para explicar el mecanismo de acción de los receptores transmembrana.
- Dimerización : el modelo de dimerización sugiere que antes de la unión del ligando, los receptores existen en forma monomérica . Cuando se produce la unión del agonista, los monómeros se combinan para formar un dímero activo .
- Rotación : la unión del ligando a la parte extracelular del receptor induce una rotación (cambio conformacional) de parte de las hélices transmembrana del receptor. La rotación altera qué partes del receptor están expuestas en el lado intracelular de la membrana, lo que altera la forma en que el receptor puede interactuar con otras proteínas dentro de la célula. [7]
Dominios

Los receptores transmembrana en la membrana plasmática generalmente se pueden dividir en tres partes.
Dominios extracelulares
El dominio extracelular se encuentra justo en el exterior de la célula u orgánulo . Si la cadena polipeptídica cruza la bicapa varias veces, el dominio externo comprende bucles entrelazados a través de la membrana. Por definición, la función principal de un receptor es reconocer y responder a un tipo de ligando. Por ejemplo, un neurotransmisor , una hormona o iones atómicos pueden unirse al dominio extracelular como un ligando acoplado al receptor. Klotho es una enzima que hace que un receptor reconozca el ligando ( FGF23 ).
Dominios transmembrana
Las dos clases más abundantes de receptores transmembrana son los GPCR y las proteínas transmembrana de un solo paso . [8] [9] En algunos receptores, como el receptor nicotínico de acetilcolina , el dominio transmembrana forma un poro proteico a través de la membrana o alrededor del canal iónico . Tras la activación de un dominio extracelular mediante la unión del ligando apropiado, el poro se vuelve accesible a los iones, que luego se difunden. En otros receptores, los dominios transmembrana experimentan un cambio conformacional tras la unión, que afecta a las condiciones intracelulares. En algunos receptores, como los miembros de la superfamilia 7TM , el dominio transmembrana incluye un bolsillo de unión de ligando.
Dominios intracelulares
El dominio intracelular (o citoplasmático ) del receptor interactúa con el interior de la célula u orgánulo, retransmitiendo la señal. Existen dos vías fundamentales para esta interacción:
- El dominio intracelular se comunica a través de interacciones proteína-proteína contra proteínas efectoras , que a su vez pasan una señal al destino.
- En el caso de los receptores ligados a enzimas , el dominio intracelular tiene actividad enzimática . A menudo, se trata de la actividad de la tirosina quinasa . La actividad enzimática también puede deberse a una enzima asociada al dominio intracelular.
Transducción de señales

Los procesos de transducción de señales a través de receptores de membrana involucran las reacciones externas, en las que el ligando se une a un receptor de membrana, y las reacciones internas, en las que se desencadena la respuesta intracelular. [10] [11]
La transducción de señales a través de receptores de membrana requiere cuatro partes:
- Molécula de señalización extracelular: una molécula de señalización extracelular es producida por una célula y es al menos capaz de viajar a las células vecinas.
- Proteína receptora: las células deben tener proteínas receptoras en la superficie celular que se unan a la molécula de señalización y se comuniquen hacia el interior de la célula.
- Proteínas de señalización intracelular: transmiten la señal a los orgánulos de la célula. La unión de la molécula de señal a la proteína receptora activará las proteínas de señalización intracelular que inician una cascada de señalización.
- Proteínas diana: las conformaciones u otras propiedades de las proteínas diana se alteran cuando una vía de señalización está activa y cambia el comportamiento de la célula. [11]

Los receptores de membrana se dividen principalmente por estructura y función en tres clases: el receptor vinculado al canal iónico , el receptor vinculado a enzima y el receptor acoplado a proteína G.
- Los receptores unidos a canales iónicos tienen canales iónicos para aniones y cationes, y constituyen una gran familia de proteínas transmembrana de múltiples pasos. Participan en eventos de señalización rápida que suelen encontrarse en células eléctricamente activas, como las neuronas . También se denominan canales iónicos regulados por ligando . La apertura y el cierre de los canales iónicos están controlados por neurotransmisores .
- Los receptores ligados a enzimas son enzimas en sí mismas o activan directamente enzimas asociadas. Por lo general, son receptores transmembrana de un solo paso, en los que el componente enzimático del receptor se mantiene intracelular. La mayoría de los receptores ligados a enzimas son, o están asociados con, proteínas quinasas.
- Los receptores acoplados a proteína G son proteínas integrales de membrana que poseen siete hélices transmembrana. Estos receptores activan una proteína G al unirse a un agonista , y la proteína G media los efectos del receptor en las vías de señalización intracelular.
Receptor ligado a canal iónico
Durante el proceso de transducción de señales en una neurona, el neurotransmisor se une al receptor y altera la conformación de la proteína. Esto abre el canal iónico, lo que permite el ingreso de iones extracelulares a la célula. Se altera la permeabilidad iónica de la membrana plasmática, lo que transforma la señal química extracelular en una señal eléctrica intracelular que altera la excitabilidad celular . [12]
El receptor de acetilcolina es un receptor ligado a un canal catiónico. La proteína consta de cuatro subunidades: subunidades alfa (α), beta (β), gamma (γ) y delta (δ). Hay dos subunidades α, con un sitio de unión de acetilcolina cada una. Este receptor puede existir en tres conformaciones. El estado cerrado y desocupado es la conformación de la proteína nativa. Como dos moléculas de acetilcolina se unen a los sitios de unión de las subunidades α, la conformación del receptor se altera y la compuerta se abre, lo que permite la entrada de muchos iones y moléculas pequeñas. Sin embargo, este estado abierto y ocupado solo dura un período breve y luego la compuerta se cierra, convirtiéndose en el estado cerrado y ocupado. Las dos moléculas de acetilcolina pronto se disociarán del receptor, devolviéndolo al estado nativo cerrado y desocupado. [13] [14]
Receptores ligados a enzimas

A partir de 2009, existen 6 tipos conocidos de receptores ligados a enzimas : receptores de tirosina quinasas ; receptores asociados a tirosina quinasas; fosfatasas de tirosina similares a receptores ; receptores de serina / treonina quinasas ; receptores de guanilil ciclasas y receptores asociados a histidina quinasa . Los receptores de tirosina quinasas tienen la población más grande y la aplicación más amplia. La mayoría de estas moléculas son receptores para factores de crecimiento como el factor de crecimiento epidérmico (EGF), el factor de crecimiento derivado de plaquetas (PDGF), el factor de crecimiento de fibroblastos (FGF), el factor de crecimiento de hepatocitos (HGF), el factor de crecimiento nervioso (NGF) y hormonas como la insulina . La mayoría de estos receptores se dimerizarán después de unirse con sus ligandos, para activar más transducciones de señales. Por ejemplo, después de que el receptor del factor de crecimiento epidérmico (EGF) se une a su ligando EGF, los dos receptores se dimerizan y luego experimentan una fosforilación de los residuos de tirosina en la porción enzimática de cada molécula de receptor. Esto activará la tirosina quinasa y catalizará otras reacciones intracelulares.
Receptores acoplados a proteína G
Los receptores acoplados a proteína G comprenden una gran familia de proteínas de receptores transmembrana. Se encuentran solo en eucariotas . [15] Los ligandos que se unen y activan estos receptores incluyen: compuestos fotosensibles, olores , feromonas , hormonas y neurotransmisores . Estos varían en tamaño desde pequeñas moléculas hasta péptidos y proteínas grandes . Los receptores acoplados a proteína G están involucrados en muchas enfermedades y, por lo tanto, son el objetivo de muchos medicamentos modernos. [16]
Existen dos vías principales de transducción de señales que involucran a los receptores acoplados a la proteína G: la vía de señalización del AMPc y la vía de señalización del fosfatidilinositol . [17] Ambas están mediadas por la activación de la proteína G. La proteína G es una proteína trimérica, con tres subunidades designadas como α, β y γ. En respuesta a la activación del receptor, la subunidad α libera difosfato de guanosina (GDP) unido, que es desplazado por el trifosfato de guanosina (GTP), activando así la subunidad α, que luego se disocia de las subunidades β y γ. La subunidad α activada puede afectar además a las proteínas de señalización intracelular o dirigirse directamente a las proteínas funcionales.
Enfermedad relacionada con el receptor de membrana
Si los receptores de membrana están desnaturalizados o deficientes, la transducción de señales puede verse obstaculizada y causar enfermedades. Algunas enfermedades son causadas por trastornos de la función del receptor de membrana. Esto se debe a la deficiencia o degradación del receptor a través de cambios en los genes que codifican y regulan la proteína del receptor. El receptor de membrana TM4SF5 influye en la migración de células hepáticas y hepatoma . [18] Además, el receptor NMDA cortical influye en la fluidez de la membrana, y se altera en la enfermedad de Alzheimer. [19] Cuando la célula es infectada por un virus sin envoltura, el virus primero se une a receptores de membrana específicos y luego pasa a sí mismo o un componente subviral al lado citoplasmático de la membrana celular. En el caso del poliovirus , se sabe in vitro que las interacciones con los receptores causan reordenamientos conformacionales que liberan una proteína virión llamada VP4. El extremo N de VP4 está miristilado y, por lo tanto, es hidrófobo [ ácido mirístico =CH 3 (CH 2 ) 12 COOH]. Se propone que los cambios conformacionales inducidos por la unión al receptor dan como resultado la unión del ácido mirístico en VP4 y la formación de un canal para el ARN.
Diseño de fármacos basado en la estructura

A través de métodos como la cristalografía de rayos X y la espectroscopia de RMN , la información sobre las estructuras 3D de las moléculas objetivo ha aumentado drásticamente, y también lo ha hecho la información estructural sobre los ligandos. Esto impulsa el rápido desarrollo del diseño de fármacos basado en la estructura . Algunos de estos nuevos fármacos se dirigen a los receptores de membrana. Los enfoques actuales para el diseño de fármacos basado en la estructura se pueden dividir en dos categorías. La primera categoría trata sobre la determinación de ligandos para un receptor dado. Esto generalmente se logra a través de consultas de bases de datos, simulaciones biofísicas y la construcción de bibliotecas químicas. En cada caso, se examina una gran cantidad de moléculas de ligando potenciales para encontrar aquellas que se ajusten al bolsillo de unión del receptor. Este enfoque generalmente se conoce como diseño de fármacos basado en ligando. La ventaja clave de buscar en una base de datos es que ahorra tiempo y energía para obtener nuevos compuestos efectivos. Otro enfoque del diseño de fármacos basado en la estructura trata sobre el mapeo combinatorio de ligandos, lo que se conoce como diseño de fármacos basado en receptores. En este caso, las moléculas de ligando se diseñan dentro de las limitaciones de un bolsillo de unión ensamblando pequeñas piezas de manera escalonada. Estos fragmentos pueden ser átomos o moléculas. La principal ventaja de este método es que se pueden descubrir nuevas estructuras. [20] [21] [22]
Otros ejemplos
- Receptor adrenérgico
- Receptores olfativos
- Receptores de tirosina quinasas
- Receptor del factor de crecimiento epidérmico
- Receptor de insulina
- Receptores del factor de crecimiento de fibroblastos ,
- Receptores de neurotrofina de alta afinidad
- Receptores de efrina
- Integrinas
- Receptor del factor de crecimiento nervioso de baja afinidad
- Receptor NMDA
- Varios receptores inmunes
Véase también
Referencias
- ^ "9.3: Moléculas de señalización y receptores celulares - Tipos de receptores". Biology LibreTexts . 12 de julio de 2018 . Consultado el 24 de julio de 2023 .
- ^ Cuatrecasas P. (1974). "Receptores de membrana". Revista Anual de Bioquímica . 43 : 169–214. doi :10.1146/annurev.bi.43.070174.001125. PMID 4368906. S2CID 44727052.
- ^ Dautzenberg FM, Hauger RL (febrero de 2002). "La familia de péptidos CRF y sus receptores: se han descubierto más socios". Trends Pharmacol. Sci. 23 (2): 71–7. doi :10.1016/S0165-6147(02)01946-6. PMID 11830263.
- ^ Rivière S, Challet L, Fluegge D, Spehr M, Rodriguez I (mayo de 2009). "Las proteínas similares a los receptores de péptidos formilo son una nueva familia de quimiosensores vomeronasales". Nature . 459 (7246): 574–7. doi :10.1038/nature08029. PMID 19387439.
- ^ Rothberg KG; Ying YS; Kamen BA; Anderson RG (1990). "El colesterol controla la agrupación del receptor de membrana anclado a glicofosfolípidos para el 5-metiltetrahidrofolato". The Journal of Cell Biology . 111 (6): 2931–2938. doi :10.1083/jcb.111.6.2931. PMC 2116385 . PMID 2148564.
- ^ Jacobson C.; Côté PD; Rossi SG; Rotundo RL; Carbonetto S. (2001). "El complejo de distroglicano es necesario para la estabilización de los grupos de receptores de acetilcolina en las uniones neuromusculares y la formación de la membrana basal sináptica". The Journal of Cell Biology . 152 (3): 435–450. doi :10.1083/jcb.152.3.435. PMC 2195998 . PMID 11157973.
- ^ Maruyama, Ichiro N. (1 de septiembre de 2015). "¿Activación de receptores transmembrana de superficie celular a través de un mecanismo común? El "modelo de rotación"". BioEssays . 37 (9): 959–967. doi :10.1002/bies.201500041. ISSN 1521-1878. PMC 5054922 . PMID 26241732.
- ^ Superfamilias de receptores transmembrana de un solo paso en la base de datos Membranome
- ^ Superfamilias de ligandos de proteínas transmembrana de un solo paso y reguladores de receptores en la base de datos Membranome
- ^ Ullricha A., Schlessingerb J.; Schlessinger, J (1990). "Transducción de señales por receptores con actividad de tirosina quinasa". Cell . 61 (2): 203–212. doi :10.1016/0092-8674(90)90801-K. PMID 2158859.
- ^ de Kenneth B. Storey (1990). Metabolismo funcional . Wiley-IEEE. págs. 87-94. ISBN 978-0-471-41090-4.
- ^ Hille B. (2001). Canales iónicos de membranas excitables . Sunderland, Mass. ISBN 978-0-87893-321-1.
- ^ Miyazawa A.; Fujiyoshi Y.; Unwin N. (2003). "Estructura y mecanismo de activación del poro del receptor de acetilcolina". Nature . 423 (6943): 949–955. doi :10.1038/nature01748. PMID 12827192.
- ^ Akabas MH; Stauffer DA; Xu M.; Karlin A. (1992). "Estructura del canal del receptor de acetilcolina investigada en mutantes de sustitución de cisteína". Science . 258 (5080): 307–310. doi :10.1126/science.1384130. PMID 1384130.
- ^ King N, Hittinger CT, Carroll SB (2003). "La evolución de las principales familias de proteínas de adhesión y señalización celular es anterior a los orígenes animales". Science . 301 (5631): 361–3. doi :10.1126/science.1083853. PMID 12869759.
- ^ Filmore, David (2004). "Es un mundo GPCR". Modern Drug Discovery . 2004 (noviembre): 24–28.
- ^ Gilman AG (1987). "Proteínas G: transductores de señales generadas por receptores". Revista anual de bioquímica . 56 : 615–649. doi :10.1146/annurev.bi.56.070187.003151. PMID 3113327. S2CID 33992382.
- ^ Müller-Pillascha F.; Wallrappa C.; Lachera U.; Friessb H.; Büchlerb M.; Adlera G.; GressTM (1998). "Identificación de un nuevo antígeno asociado a tumores TM4SF5 y su expresión en cáncer humano". Gen. 208 (1): 25–30. doi :10.1016/S0378-1119(97)00633-1. PMID 9479038.
- ^ Scheuer K.; Marasb A.; Gattazb WF; Cairnsc N.; Förstlb H.; Müller WE (1996). "Las propiedades del receptor NMDA cortical y la fluidez de la membrana se alteran en la enfermedad de Alzheimer". Demencia . 7 (4): 210–214. doi :10.1159/000106881. PMID 8835885.
- ^ Wang R.; Gao Y.; Lai L. (2000). "LigBuilder: un programa multipropósito para el diseño de fármacos basado en la estructura". Journal of Molecular Modeling . 6 (7–8): 498–516. doi :10.1007/s0089400060498.
- ^ Schneider G.; Fechner U. (2005). "Diseño de novo basado en computadora de moléculas similares a fármacos". Nature Reviews Drug Discovery . 4 (8): 649–663. doi :10.1038/nrd1799. PMID 16056391.
- ^ Jorgensen WL (2004). "Los múltiples roles de la computación en el descubrimiento de fármacos". Science . 303 (5665): 1813–1818. doi :10.1126/science.1096361. PMID 15031495. S2CID 1307935.
Enlaces externos
- Base de datos GPCR de la IUPHAR Archivado el 23 de marzo de 2019 en Wayback Machine
- Receptores de superficie celular en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.