Plegamiento de proteínas


El plegamiento de proteínas es el proceso físico por el cual una proteína , después de ser sintetizada por un ribosoma como una cadena lineal de aminoácidos , cambia de una espiral aleatoria inestable a una estructura tridimensional más ordenada . Esta estructura permite que la proteína se vuelva biológicamente funcional. [1]
El plegamiento de muchas proteínas comienza incluso durante la traducción de la cadena polipeptídica. Los aminoácidos interactúan entre sí para producir una estructura tridimensional bien definida, conocida como estado nativo de la proteína . Esta estructura está determinada por la secuencia de aminoácidos o estructura primaria . [2]
La estructura tridimensional correcta es esencial para el funcionamiento, aunque algunas partes de las proteínas funcionales pueden permanecer sin plegar , [3] lo que indica que la dinámica de las proteínas es importante. La falta de plegamiento en una estructura nativa generalmente produce proteínas inactivas, pero en algunos casos, las proteínas mal plegadas tienen una funcionalidad modificada o tóxica. Se cree que varias enfermedades neurodegenerativas y de otro tipo son resultado de la acumulación de fibrillas amiloides formadas por proteínas mal plegadas, cuyas variedades infecciosas se conocen como priones . [4] Muchas alergias son causadas por el plegamiento incorrecto de algunas proteínas porque el sistema inmunológico no produce los anticuerpos para ciertas estructuras proteínicas. [5]
La desnaturalización de las proteínas es un proceso de transición de un estado plegado a uno desplegado . Ocurre en la cocción , quemaduras , proteinopatías y otros contextos. La estructura residual presente, si la hay, en el estado supuestamente desplegado puede formar un sitio de inicio del plegamiento y guiar las reacciones de plegamiento posteriores. [6]
La duración del proceso de plegamiento varía drásticamente dependiendo de la proteína de interés. Cuando se estudia fuera de la célula , las proteínas de plegamiento más lento requieren muchos minutos u horas para plegarse, principalmente debido a la isomerización de la prolina , y deben pasar por una serie de estados intermedios, como puntos de control, antes de que el proceso se complete. [7] Por otro lado, las proteínas muy pequeñas de un solo dominio con longitudes de hasta cien aminoácidos generalmente se pliegan en un solo paso. [8] Las escalas de tiempo de milisegundos son la norma, y las reacciones de plegamiento de proteínas más rápidas conocidas se completan en unos pocos microsegundos. [9] La escala de tiempo de plegamiento de una proteína depende de su tamaño, orden de contacto y topología del circuito . [10]
Comprender y simular el proceso de plegamiento de proteínas ha sido un desafío importante para la biología computacional desde finales de la década de 1960.
Proceso de plegamiento de proteínas
Estructura primaria
La estructura primaria de una proteína, su secuencia lineal de aminoácidos, determina su conformación nativa. [11] Los residuos de aminoácidos específicos y su posición en la cadena polipeptídica son los factores determinantes para que las porciones de la proteína se plieguen juntas y formen su conformación tridimensional. La composición de aminoácidos no es tan importante como la secuencia. [12] Sin embargo, el hecho esencial del plegamiento sigue siendo que la secuencia de aminoácidos de cada proteína contiene la información que especifica tanto la estructura nativa como la vía para alcanzar ese estado. Esto no quiere decir que las secuencias de aminoácidos casi idénticas siempre se plieguen de manera similar. [13] Las conformaciones también difieren en función de factores ambientales; proteínas similares se pliegan de manera diferente según el lugar donde se encuentren.
Estructura secundaria
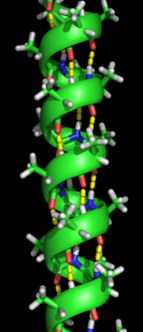

La formación de una estructura secundaria es el primer paso en el proceso de plegamiento que una proteína realiza para asumir su estructura nativa. Las estructuras conocidas como hélices alfa y láminas beta son características de la estructura secundaria , ya que se pliegan rápidamente porque están estabilizadas por enlaces de hidrógeno intramoleculares , como fue caracterizado por primera vez por Linus Pauling . La formación de enlaces de hidrógeno intramoleculares proporciona otra contribución importante a la estabilidad de la proteína. [14] Las hélices α se forman mediante enlaces de hidrógeno de la cadena principal para formar una forma espiral (consulte la figura de la derecha). [12] La lámina β plegada es una estructura que se forma con la cadena principal doblándose sobre sí misma para formar los enlaces de hidrógeno (como se muestra en la figura de la izquierda). Los enlaces de hidrógeno están entre el hidrógeno de la amida y el oxígeno del carbonilo del enlace peptídico . Existen láminas β plegadas antiparalelas y láminas β plegadas paralelas donde la estabilidad de los enlaces de hidrógeno es más fuerte en la lámina β antiparalela ya que forma enlaces de hidrógeno con el ángulo ideal de 180 grados en comparación con los enlaces de hidrógeno inclinados formados por láminas paralelas. [12]
Estructura terciaria
Las hélices α y las láminas β son comúnmente anfipáticas, lo que significa que tienen una porción hidrófila y otra hidrófoba. Esta capacidad ayuda a formar la estructura terciaria de una proteína en la que el plegamiento se produce de modo que los lados hidrófilos estén orientados hacia el entorno acuoso que rodea a la proteína y los lados hidrófobos estén orientados hacia el núcleo hidrófobo de la proteína. [15] La estructura secundaria da paso jerárquicamente a la formación de la estructura terciaria. Una vez que la estructura terciaria de la proteína se forma y se estabiliza mediante las interacciones hidrófobas, también puede haber enlaces covalentes en forma de puentes disulfuro formados entre dos residuos de cisteína . Estos contactos no covalentes y covalentes adoptan una disposición topológica específica en una estructura nativa de una proteína. La estructura terciaria de una proteína implica una sola cadena polipeptídica; sin embargo, interacciones adicionales de cadenas polipeptídicas plegadas dan lugar a la formación de una estructura cuaternaria. [16]
Estructura cuaternaria
La estructura terciaria puede dar lugar a la formación de una estructura cuaternaria en algunas proteínas, lo que generalmente implica el "ensamblaje" o "coensamblaje" de subunidades que ya se han plegado; en otras palabras, múltiples cadenas polipeptídicas podrían interactuar para formar una proteína cuaternaria completamente funcional. [12]
Fuerzas impulsoras del plegamiento de proteínas

El plegamiento es un proceso espontáneo que está guiado principalmente por interacciones hidrofóbicas, formación de enlaces de hidrógeno intramoleculares , fuerzas de van der Waals , y se opone a la entropía conformacional . [17] El proceso de plegamiento a menudo comienza co-traduccionalmente , de modo que el extremo N de la proteína comienza a plegarse mientras que la porción C-terminal de la proteína todavía está siendo sintetizada por el ribosoma ; sin embargo, una molécula de proteína puede plegarse espontáneamente durante o después de la biosíntesis . [18] Si bien estas macromoléculas pueden considerarse como " plegándose a sí mismas ", el proceso también depende del solvente ( agua o bicapa lipídica ), [19] la concentración de sales , el pH , la temperatura , la posible presencia de cofactores y de chaperonas moleculares .
Las proteínas tendrán limitaciones en sus capacidades de plegamiento debido a los ángulos de flexión restringidos o las conformaciones que son posibles. Estos ángulos permisibles de plegamiento de proteínas se describen con un gráfico bidimensional conocido como el gráfico de Ramachandran , representado con ángulos psi y phi de rotación permisible. [20]
Efecto hidrofóbico

El plegamiento de proteínas debe ser termodinámicamente favorable dentro de una célula para que sea una reacción espontánea. Dado que se sabe que el plegamiento de proteínas es una reacción espontánea, entonces debe asumir un valor de energía libre de Gibbs negativo . La energía libre de Gibbs en el plegamiento de proteínas está directamente relacionada con la entalpía y la entropía . [12] Para que surja un delta G negativo y para que el plegamiento de proteínas se vuelva termodinámicamente favorable, entonces la entalpía, la entropía o ambos términos deben ser favorables.
Minimizar el número de cadenas laterales hidrofóbicas expuestas al agua es una fuerza impulsora importante detrás del proceso de plegamiento. [21] El efecto hidrofóbico es el fenómeno en el que las cadenas hidrofóbicas de una proteína colapsan en el núcleo de la proteína (lejos del entorno hidrofílico). [12] En un entorno acuoso, las moléculas de agua tienden a agregarse alrededor de las regiones hidrofóbicas o cadenas laterales de la proteína, creando capas de agua de moléculas de agua ordenadas. [22] Un ordenamiento de las moléculas de agua alrededor de una región hidrofóbica aumenta el orden en un sistema y, por lo tanto, contribuye a un cambio negativo en la entropía (menos entropía en el sistema). Las moléculas de agua se fijan en estas jaulas de agua, lo que impulsa el colapso hidrofóbico o el plegamiento hacia adentro de los grupos hidrofóbicos. El colapso hidrofóbico introduce entropía nuevamente en el sistema a través de la ruptura de las jaulas de agua que liberan las moléculas de agua ordenadas. [12] La multitud de grupos hidrófobos que interactúan dentro del núcleo de la proteína plegada globular contribuye en gran medida a la estabilidad de la proteína después del plegado, debido a las fuerzas de van der Waals enormemente acumuladas (específicamente las fuerzas de dispersión de London ). [12] El efecto hidrófobo existe como una fuerza impulsora en la termodinámica solo si existe la presencia de un medio acuoso con una molécula anfifílica que contiene una gran región hidrófoba. [23] La fuerza de los enlaces de hidrógeno depende de su entorno; por lo tanto, los enlaces de H envueltos en un núcleo hidrófobo contribuyen más que los enlaces de H expuestos al entorno acuoso a la estabilidad del estado nativo. [24]
En las proteínas con pliegues globulares, los aminoácidos hidrófobos tienden a estar intercalados a lo largo de la secuencia primaria, en lugar de estar distribuidos aleatoriamente o agrupados. [25] [26] Sin embargo, las proteínas que han nacido recientemente de novo , que tienden a estar intrínsecamente desordenadas , [27] [28] muestran el patrón opuesto de agrupamiento de aminoácidos hidrófobos a lo largo de la secuencia primaria. [29]
Acompañantes

Las chaperonas moleculares son una clase de proteínas que ayudan en el plegamiento correcto de otras proteínas in vivo . Las chaperonas existen en todos los compartimentos celulares e interactúan con la cadena polipeptídica para permitir que se forme la conformación tridimensional nativa de la proteína; sin embargo, las chaperonas en sí mismas no están incluidas en la estructura final de la proteína en la que están ayudando. [30] Las chaperonas pueden ayudar en el plegamiento incluso cuando el polipéptido naciente está siendo sintetizado por el ribosoma. [31] Las chaperonas moleculares operan uniéndose para estabilizar una estructura de otra manera inestable de una proteína en su vía de plegamiento, pero las chaperonas no contienen la información necesaria para conocer la estructura nativa correcta de la proteína a la que están ayudando; más bien, las chaperonas funcionan previniendo conformaciones de plegamiento incorrectas. [31] De esta manera, las chaperonas en realidad no aumentan la tasa de pasos individuales involucrados en la vía de plegamiento hacia la estructura nativa; en cambio, funcionan reduciendo posibles agregaciones no deseadas de la cadena polipeptídica que de otro modo podrían ralentizar la búsqueda del intermedio adecuado y proporcionan una vía más eficiente para que la cadena polipeptídica asuma las conformaciones correctas. [30] Las chaperonas no deben confundirse con las proteínas catalizadoras de plegamiento , que catalizan las reacciones químicas responsables de los pasos lentos en las vías de plegamiento. Ejemplos de catalizadores de plegamiento son las isomerasas de disulfuro de proteínas y las isomerasas de peptidil-prolil que pueden estar involucradas en la formación de enlaces disulfuro o la interconversión entre estereoisómeros cis y trans del grupo peptídico. [31] Se ha demostrado que las chaperonas son fundamentales en el proceso de plegamiento de proteínas in vivo porque proporcionan a la proteína la ayuda necesaria para asumir sus alineaciones y conformaciones adecuadas con la suficiente eficiencia para volverse "biológicamente relevante". [32] Esto significa que la cadena polipeptídica podría, teóricamente, plegarse en su estructura nativa sin la ayuda de chaperonas, como lo demuestran los experimentos de plegamiento de proteínas realizados in vitro ; [32] Sin embargo, este proceso resulta demasiado ineficiente o demasiado lento para existir en sistemas biológicos; por lo tanto, las chaperonas son necesarias para el plegamiento de proteínas in vivo. Junto con su papel en la ayuda a la formación de la estructura nativa, se ha demostrado que las chaperonas están involucradas en varias funciones, como el transporte de proteínas, la degradación e incluso permiten que las proteínas desnaturalizadas expuestas a ciertos factores desnaturalizantes externos tengan la oportunidad de volver a plegarse en sus estructuras nativas correctas. [33]
Una proteína completamente desnaturalizada carece tanto de estructura terciaria como secundaria, y existe como una denominada bobina aleatoria . Bajo ciertas condiciones, algunas proteínas pueden replegarse; sin embargo, en muchos casos, la desnaturalización es irreversible. [34] Las células a veces protegen sus proteínas contra la influencia desnaturalizante del calor con enzimas conocidas como proteínas de choque térmico (un tipo de chaperona), que ayudan a otras proteínas tanto a plegarse como a permanecer plegadas. Se han encontrado proteínas de choque térmico en todas las especies examinadas, desde bacterias hasta humanos, lo que sugiere que evolucionaron muy temprano y tienen una función importante. Algunas proteínas nunca se pliegan en las células excepto con la ayuda de chaperonas que aíslan proteínas individuales para que su plegamiento no se interrumpa por interacciones con otras proteínas o ayudan a desplegar proteínas mal plegadas, lo que les permite replegarse en la estructura nativa correcta. [35] Esta función es crucial para prevenir el riesgo de precipitación en agregados amorfos insolubles . Los factores externos implicados en la desnaturalización de proteínas o la alteración de su estado nativo incluyen la temperatura, los campos externos (eléctricos, magnéticos), [36] el hacinamiento molecular, [37] e incluso la limitación del espacio (es decir, el confinamiento), que pueden tener una gran influencia en el plegamiento de las proteínas. [38] Las altas concentraciones de solutos , los extremos de pH , las fuerzas mecánicas y la presencia de desnaturalizantes químicos también pueden contribuir a la desnaturalización de proteínas. Estos factores individuales se clasifican juntos como estreses. Se ha demostrado que las chaperonas existen en concentraciones crecientes durante los momentos de estrés celular y ayudan al plegamiento adecuado de las proteínas emergentes, así como de las desnaturalizadas o mal plegadas. [30]
En determinadas condiciones, las proteínas no se plegarán hasta adoptar sus formas bioquímicamente funcionales. Las temperaturas superiores o inferiores al rango en el que las células tienden a vivir harán que las proteínas térmicamente inestables se desplieguen o desnaturalicen (por eso, al hervirlas , la clara de un huevo se vuelve opaca). Sin embargo, la estabilidad térmica de las proteínas dista mucho de ser constante; por ejemplo, se han descubierto bacterias hipertermófilas que crecen a temperaturas de hasta 122 °C [39] , lo que, por supuesto, requiere que su dotación completa de proteínas vitales y conjuntos proteínicos sea estable a esa temperatura o a una temperatura superior.
La bacteria E. coli es el huésped del bacteriófago T4 , y la proteína gp31 codificada por el fago ( P17313 ) parece ser estructural y funcionalmente homóloga a la proteína chaperona GroES de E. coli y capaz de sustituirla en el ensamblaje de partículas del virus del bacteriófago T4 durante la infección. [40] Al igual que GroES, gp31 forma un complejo estable con la chaperonina GroEL que es absolutamente necesario para el plegamiento y ensamblaje in vivo de la proteína gp23 de la cápside principal del bacteriófago T4. [40]
Cambio de pliegues
Algunas proteínas tienen múltiples estructuras nativas y cambian su plegamiento en función de algunos factores externos. Por ejemplo, la proteína KaiB cambia de plegamiento a lo largo del día , actuando como un reloj para las cianobacterias. Se ha estimado que alrededor del 0,5 al 4 % de las proteínas del PDB ( Protein Data Bank ) cambian de plegamiento. [41]
Mal plegamiento de proteínas y enfermedades neurodegenerativas
Se considera que una proteína está mal plegada si no puede alcanzar su estado nativo normal. Esto puede deberse a mutaciones en la secuencia de aminoácidos o a una interrupción del proceso de plegamiento normal por factores externos. [42] La proteína mal plegada normalmente contiene láminas β que están organizadas en una disposición supramolecular conocida como estructura β cruzada. Estos ensamblajes ricos en láminas β son muy estables, muy insolubles y generalmente resistentes a la proteólisis. [43] La estabilidad estructural de estos ensamblajes fibrilares es causada por interacciones extensas entre los monómeros de la proteína, formados por enlaces de hidrógeno de la cadena principal entre sus cadenas β. [43] El plegamiento incorrecto de las proteínas puede desencadenar un mayor plegamiento incorrecto y la acumulación de otras proteínas en agregados u oligómeros. El aumento de los niveles de proteínas agregadas en la célula conduce a la formación de estructuras similares a amiloide que pueden causar trastornos degenerativos y muerte celular. [42] Los amiloides son estructuras fibrilares que contienen enlaces de hidrógeno intermoleculares que son altamente insolubles y están hechos de agregados de proteínas transformadas. [42] Por lo tanto, la vía del proteasoma puede no ser lo suficientemente eficiente para degradar las proteínas mal plegadas antes de la agregación. Las proteínas mal plegadas pueden interactuar entre sí y formar agregados estructurados y adquirir toxicidad a través de interacciones intermoleculares. [42]
Las proteínas agregadas se asocian con enfermedades relacionadas con priones como la enfermedad de Creutzfeldt-Jakob , la encefalopatía espongiforme bovina (enfermedad de las vacas locas), enfermedades relacionadas con amiloide como la enfermedad de Alzheimer y la miocardiopatía o polineuropatía amiloide familiar , [44] así como enfermedades de agregación intracelular como la enfermedad de Huntington y la enfermedad de Parkinson . [4] [45] Estas enfermedades degenerativas de inicio en la edad están asociadas con la agregación de proteínas mal plegadas en agregados extracelulares insolubles y/o inclusiones intracelulares incluyendo fibrillas de amiloide β cruzadas . No está completamente claro si los agregados son la causa o simplemente un reflejo de la pérdida de la homeostasis proteica, el equilibrio entre síntesis, plegamiento, agregación y recambio proteico. Recientemente la Agencia Europea de Medicamentos aprobó el uso de Tafamidis o Vyndaqel (un estabilizador cinético de la transtiretina tetramérica) para el tratamiento de enfermedades amiloides por transtiretina. Esto sugiere que el proceso de formación de fibrillas amiloides (y no las fibrillas en sí) causa la degeneración del tejido postmitótico en las enfermedades amiloides humanas. [46] El plegamiento incorrecto y la degradación excesiva en lugar del plegamiento y la función conducen a una serie de enfermedades proteopáticos como el enfisema asociado a la antitripsina , la fibrosis quística y las enfermedades de almacenamiento lisosomal , donde la pérdida de función es el origen del trastorno. Si bien la terapia de reemplazo de proteínas se ha utilizado históricamente para corregir estos últimos trastornos, un enfoque emergente es utilizar chaperonas farmacéuticas para plegar proteínas mutadas para hacerlas funcionales.
Técnicas experimentales para estudiar el plegamiento de proteínas
Si bien se pueden hacer inferencias sobre el plegamiento de proteínas a través de estudios de mutación , normalmente las técnicas experimentales para estudiar el plegamiento de proteínas se basan en el desdoblamiento o plegamiento gradual de las proteínas y en la observación de cambios conformacionales utilizando técnicas estándar no cristalográficas.
Cristalografía de rayos X

La cristalografía de rayos X es uno de los métodos más eficientes e importantes para intentar descifrar la configuración tridimensional de una proteína plegada. [47] Para poder realizar la cristalografía de rayos X, la proteína bajo investigación debe estar ubicada dentro de una red cristalina. Para colocar una proteína dentro de una red cristalina, uno debe tener un solvente adecuado para la cristalización, obtener una proteína pura a niveles sobresaturados en solución y precipitar los cristales en solución. [48] Una vez que una proteína se cristaliza, los rayos X se pueden concentrar a través de la red cristalina que difractaría los rayos o los dispararía hacia afuera en varias direcciones. Estos rayos salientes están correlacionados con la configuración tridimensional específica de la proteína encerrada en su interior. Los rayos X interactúan específicamente con las nubes de electrones que rodean los átomos individuales dentro de la red cristalina de la proteína y producen un patrón de difracción discernible. [15] Solamente relacionando las nubes de densidad electrónica con la amplitud de los rayos X se puede leer este patrón y conducir a suposiciones de las fases o ángulos de fase involucrados que complican este método. [49] Sin la relación establecida a través de una base matemática conocida como transformada de Fourier , el " problema de fase " haría muy difícil predecir los patrones de difracción. [15] Los métodos emergentes como el reemplazo isomorfo múltiple utilizan la presencia de un ion de metal pesado para difractar los rayos X de una manera más predecible, reduciendo el número de variables involucradas y resolviendo el problema de fase. [47]
Espectroscopia de fluorescencia
La espectroscopia de fluorescencia es un método muy sensible para estudiar el estado de plegamiento de las proteínas. Tres aminoácidos, la fenilalanina (Phe), la tirosina (Tyr) y el triptófano (Trp), tienen propiedades intrínsecas de fluorescencia, pero solo Tyr y Trp se utilizan experimentalmente porque sus rendimientos cuánticos son lo suficientemente altos como para dar buenas señales de fluorescencia. Tanto Trp como Tyr se excitan con una longitud de onda de 280 nm, mientras que solo Trp se excita con una longitud de onda de 295 nm. Debido a su carácter aromático, los residuos de Trp y Tyr a menudo se encuentran enterrados total o parcialmente en el núcleo hidrofóbico de las proteínas, en la interfaz entre dos dominios proteicos o en la interfaz entre subunidades de proteínas oligoméricas. En este entorno apolar, tienen altos rendimientos cuánticos y, por lo tanto, altas intensidades de fluorescencia. Al interrumpirse la estructura terciaria o cuaternaria de la proteína, estas cadenas laterales quedan más expuestas al entorno hidrofílico del disolvente y sus rendimientos cuánticos disminuyen, lo que conduce a bajas intensidades de fluorescencia. Para los residuos de Trp, la longitud de onda de su emisión máxima de fluorescencia también depende de su entorno.
La espectroscopia de fluorescencia se puede utilizar para caracterizar el desdoblamiento en equilibrio de las proteínas midiendo la variación en la intensidad de la emisión de fluorescencia o en la longitud de onda de emisión máxima como funciones de un valor desnaturalizante. [50] [51] El desnaturalizante puede ser una molécula química (urea, clorhidrato de guanidinio), temperatura, pH, presión, etc. El equilibrio entre los diferentes pero discretos estados de la proteína, es decir, estado nativo, estados intermedios, estado desdoblado, depende del valor desnaturalizante; por lo tanto, la señal de fluorescencia global de su mezcla de equilibrio también depende de este valor. De este modo, se obtiene un perfil que relaciona la señal global de la proteína con el valor desnaturalizante. El perfil de desdoblamiento en equilibrio puede permitir detectar e identificar intermediarios de desdoblamiento. [52] [53] Hugues Bedouelle ha desarrollado ecuaciones generales para obtener los parámetros termodinámicos que caracterizan los equilibrios de desdoblamiento para proteínas homoméricas o heteroméricas, hasta trímeros y potencialmente tetrámeros, a partir de dichos perfiles. [50] La espectroscopia de fluorescencia se puede combinar con dispositivos de mezcla rápida, como el flujo detenido , para medir la cinética de plegamiento de proteínas, [54] generar un gráfico de chevron y derivar un análisis del valor Phi .
Dicroísmo circular
El dicroísmo circular es una de las herramientas más generales y básicas para estudiar el plegamiento de proteínas. La espectroscopia de dicroísmo circular mide la absorción de luz polarizada circularmente . En las proteínas, las estructuras como las hélices alfa y las láminas beta son quirales y, por lo tanto, absorben dicha luz. La absorción de esta luz actúa como un marcador del grado de plegamiento del conjunto de proteínas. Esta técnica se ha utilizado para medir el desdoblamiento en equilibrio de la proteína midiendo el cambio en esta absorción como una función de la concentración de desnaturalizante o la temperatura . Una fusión de desnaturalizante mide la energía libre de desdoblamiento, así como el valor m de la proteína, o la dependencia del desnaturalizante. Una fusión de temperatura mide la temperatura de desnaturalización (Tm) de la proteína. [50] En cuanto a la espectroscopia de fluorescencia, la espectroscopia de dicroísmo circular se puede combinar con dispositivos de mezcla rápida como el flujo detenido para medir la cinética de plegamiento de proteínas y generar gráficos de chevron .
Dicroísmo circular vibracional de proteínas
Los desarrollos más recientes de las técnicas de dicroísmo circular vibracional (VCD) para proteínas, que actualmente involucran instrumentos de transformada de Fourier (FT), proporcionan medios poderosos para determinar conformaciones de proteínas en solución incluso para moléculas de proteínas muy grandes. Estos estudios de VCD de proteínas se pueden combinar con datos de difracción de rayos X para cristales de proteínas, datos de FT-IR para soluciones de proteínas en agua pesada (D 2 O) o cálculos cuánticos .
Espectroscopia de resonancia magnética nuclear de proteínas
La resonancia magnética nuclear (RMN) de proteínas es capaz de recolectar datos estructurales de proteínas induciendo un campo magnético a través de muestras de proteína concentrada. En RMN, dependiendo del entorno químico, ciertos núcleos absorberán radiofrecuencias específicas. [55] [56] Debido a que los cambios estructurales de las proteínas operan en una escala de tiempo de ns a ms, la RMN está especialmente equipada para estudiar estructuras intermedias en escalas de tiempo de ps a s. [57] Algunas de las principales técnicas para estudiar la estructura de las proteínas y los cambios estructurales de las proteínas sin plegamiento incluyen COSY , TOCSY , HSQC , relajación temporal (T1 y T2) y NOE . [55] La NOE es especialmente útil porque se pueden observar transferencias de magnetización entre hidrógenos espacialmente proximales. [55] Diferentes experimentos de RMN tienen diferentes grados de sensibilidad a la escala de tiempo que son apropiados para diferentes cambios estructurales de las proteínas. La NOE puede captar vibraciones de enlaces o rotaciones de cadenas laterales, sin embargo, la NOE es demasiado sensible para captar el plegamiento de proteínas porque ocurre en una escala de tiempo mayor. [57]

Debido a que el plegamiento de proteínas ocurre en aproximadamente 50 a 3000 s −1, la dispersión de relajación CPMG y la transferencia de saturación por intercambio químico se han convertido en algunas de las técnicas principales para el análisis de RMN del plegamiento. [56] Además, ambas técnicas se utilizan para descubrir estados intermedios excitados en el paisaje de plegamiento de proteínas. [58] Para hacer esto, la dispersión de relajación CPMG aprovecha el fenómeno del eco de espín . Esta técnica expone los núcleos objetivo a un pulso de 90 seguido de uno o más pulsos de 180. [59] A medida que los núcleos se reorientan, una distribución amplia indica que los núcleos objetivo están involucrados en un estado excitado intermedio. Al observar los gráficos de dispersión de relajación, los datos recopilan información sobre la termodinámica y la cinética entre el estado excitado y el fundamental. [59] [58] La transferencia de saturación mide los cambios en la señal del estado fundamental a medida que los estados excitados se perturban. Utiliza una irradiación de radiofrecuencia débil para saturar el estado excitado de un núcleo particular que transfiere su saturación al estado fundamental. [56] Esta señal se amplifica disminuyendo la magnetización (y la señal) del estado fundamental. [56] [58]
Las principales limitaciones de la RMN es que su resolución disminuye con proteínas mayores de 25 kDa y no es tan detallada como la cristalografía de rayos X. [56] Además, el análisis de RMN de proteínas es bastante difícil y puede proponer múltiples soluciones a partir del mismo espectro de RMN. [55]
En un estudio centrado en el plegamiento de una proteína involucrada en la esclerosis lateral amiotrófica , SOD1 , se estudiaron los intermediarios excitados con dispersión de relajación y transferencia de saturación. [60] La SOD1 se había vinculado previamente a muchos mutantes causantes de enfermedades que se suponía que estaban involucrados en la agregación de proteínas, sin embargo, el mecanismo aún era desconocido. Mediante el uso de experimentos de dispersión de relajación y transferencia de saturación, se descubrió que muchos estados intermedios excitados estaban mal plegados en los mutantes SOD1. [60]
Interferometría de polarización dual
La interferometría de polarización dual es una técnica basada en la superficie para medir las propiedades ópticas de las capas moleculares. Cuando se utiliza para caracterizar el plegamiento de proteínas, mide la conformación determinando el tamaño total de una monocapa de la proteína y su densidad en tiempo real con una resolución sub-Angstrom, [61] aunque la medición en tiempo real de la cinética del plegamiento de proteínas se limita a procesos que ocurren a una velocidad inferior a ~10 Hz. De manera similar al dicroísmo circular , el estímulo para el plegamiento puede ser un desnaturalizante o la temperatura .
Estudios de plegamiento con alta resolución temporal
El estudio del plegamiento de proteínas ha avanzado mucho en los últimos años gracias al desarrollo de técnicas rápidas y resueltas en el tiempo. Los experimentadores desencadenan rápidamente el plegamiento de una muestra de proteína desplegada y observan la dinámica resultante . Las técnicas rápidas en uso incluyen dispersión de neutrones , [62] mezcla ultrarrápida de soluciones, métodos fotoquímicos y espectroscopia de salto de temperatura láser . Entre los muchos científicos que han contribuido al desarrollo de estas técnicas se encuentran Jeremy Cook, Heinrich Roder, Terry Oas, Harry Gray , Martin Gruebele , Brian Dyer, William Eaton, Sheena Radford , Chris Dobson , Alan Fersht , Bengt Nölting y Lars Konermann.
Proteólisis
La proteólisis se utiliza rutinariamente para investigar la fracción desplegada en una amplia gama de condiciones de solución (por ejemplo, proteólisis paralela rápida (FASTpp) . [63] [64]
Espectroscopia de fuerza de una sola molécula
Las técnicas de moléculas individuales, como las pinzas ópticas y el AFM, se han utilizado para comprender los mecanismos de plegamiento de proteínas de proteínas aisladas, así como de proteínas con chaperonas. [65] Las pinzas ópticas se han utilizado para estirar moléculas de proteínas individuales desde sus extremos C y N y desplegarlas para permitir el estudio del replegamiento posterior. [66] La técnica permite medir las tasas de plegamiento a nivel de una sola molécula; por ejemplo, las pinzas ópticas se han aplicado recientemente para estudiar el plegamiento y desplegamiento de proteínas implicadas en la coagulación sanguínea. El factor de von Willebrand (vWF) es una proteína con un papel esencial en el proceso de formación de coágulos sanguíneos. Se descubrió, utilizando la medición con pinzas ópticas de una sola molécula, que el vWF unido al calcio actúa como un sensor de fuerza de corte en la sangre. La fuerza de corte conduce al desplegamiento del dominio A2 del vWF, cuya tasa de replegamiento aumenta drásticamente en presencia de calcio. [67] Recientemente, también se demostró que el simple dominio src SH3 accede a múltiples vías de desplegamiento bajo fuerza. [68]
Pintura de biotina
La pintura con biotina permite obtener instantáneas celulares específicas de la condición de las proteínas (no) plegadas. La "pintura" con biotina muestra un sesgo hacia las proteínas intrínsecamente desordenadas predichas . [69]
Estudios computacionales del plegamiento de proteínas
Los estudios computacionales del plegamiento de proteínas incluyen tres aspectos principales relacionados con la predicción de la estabilidad, la cinética y la estructura de las proteínas. Una revisión de 2013 resume los métodos computacionales disponibles para el plegamiento de proteínas. [70]
La paradoja de Levinthal
En 1969, Cyrus Levinthal observó que, debido a la gran cantidad de grados de libertad en una cadena polipeptídica desplegada, la molécula tiene una cantidad astronómica de conformaciones posibles. En uno de sus artículos, realizó una estimación de 3 300 o 10 143. [71] La paradoja de Levinthal es un experimento mental basado en la observación de que si una proteína se plegara mediante un muestreo secuencial de todas las conformaciones posibles, tomaría una cantidad astronómica de tiempo hacerlo, incluso si las conformaciones se muestrearan a una velocidad rápida (en la escala de nanosegundos o picosegundos ). [72] Basándose en la observación de que las proteínas se pliegan mucho más rápido que esto, Levinthal propuso entonces que no se produce una búsqueda conformacional aleatoria y que, por lo tanto, la proteína debe plegarse a través de una serie de estados intermedios metaestables .
Paisaje energético del plegamiento de proteínas
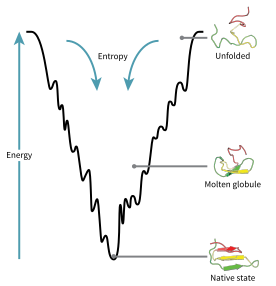
El espacio de configuración de una proteína durante el plegamiento se puede visualizar como un paisaje energético . Según Joseph Bryngelson y Peter Wolynes , las proteínas siguen el principio de frustración mínima , lo que significa que las proteínas evolucionadas naturalmente han optimizado sus paisajes energéticos de plegamiento, [73] y que la naturaleza ha elegido secuencias de aminoácidos de modo que el estado plegado de la proteína sea suficientemente estable. Además, la adquisición del estado plegado tuvo que convertirse en un proceso suficientemente rápido. Aunque la naturaleza ha reducido el nivel de frustración en las proteínas, hasta ahora persiste cierto grado de ella, como se puede observar en la presencia de mínimos locales en el paisaje energético de las proteínas.
Una consecuencia de estas secuencias seleccionadas evolutivamente es que generalmente se piensa que las proteínas tienen "paisajes de energía en embudo" globalmente (un término acuñado por José Onuchic ) [74] que están dirigidos en gran medida hacia el estado nativo. Este paisaje de " embudo de plegamiento " permite que la proteína se pliegue al estado nativo a través de cualquiera de un gran número de vías e intermediarios, en lugar de estar restringida a un solo mecanismo. La teoría está respaldada tanto por simulaciones computacionales de proteínas modelo como por estudios experimentales [73] , y se ha utilizado para mejorar los métodos de predicción y diseño de la estructura de proteínas [73] . La descripción del plegamiento de proteínas por el paisaje de energía libre nivelador también es consistente con la segunda ley de la termodinámica [ 75] . Físicamente, pensar en paisajes en términos de superficies de energía potencial o total visualizables simplemente con máximos, puntos de silla, mínimos y embudos, más bien como paisajes geográficos, es quizás un poco engañoso. La descripción relevante es realmente un espacio de fase de alta dimensión en el que las variedades pueden tomar una variedad de formas topológicas más complicadas [76] .
La cadena polipeptídica desplegada comienza en la parte superior del embudo, donde puede asumir la mayor cantidad de variaciones desplegadas y se encuentra en su estado de energía más alto. Los paisajes energéticos como estos indican que hay una gran cantidad de posibilidades iniciales, pero solo es posible un único estado nativo; sin embargo, no revelan las numerosas vías de plegamiento que son posibles. Una molécula diferente de la misma proteína exacta puede ser capaz de seguir vías de plegamiento marginalmente diferentes, buscando diferentes intermediarios de menor energía, siempre que se alcance la misma estructura nativa. [77] Diferentes vías pueden tener diferentes frecuencias de utilización dependiendo de la favorabilidad termodinámica de cada vía. Esto significa que si se descubre que una vía es termodinámicamente más favorable que otra, es probable que se use con más frecuencia en la búsqueda de la estructura nativa. [77] A medida que la proteína comienza a plegarse y asumir sus diversas conformaciones, siempre busca una estructura termodinámicamente más favorable que antes y, por lo tanto, continúa a través del embudo de energía. La formación de estructuras secundarias es un fuerte indicio de una mayor estabilidad dentro de la proteína, y solo una combinación de estructuras secundarias asumidas por la estructura principal del polipéptido tendrá la energía más baja y, por lo tanto, estará presente en el estado nativo de la proteína. [77] Entre las primeras estructuras que se forman una vez que el polipéptido comienza a plegarse se encuentran las hélices alfa y los giros beta, donde las hélices alfa pueden formarse en tan solo 100 nanosegundos y los giros beta en 1 microsegundo. [30]
Existe un punto de silla en el diagrama del embudo de energía donde se encuentra el estado de transición para una proteína en particular. [30] El estado de transición en el diagrama del embudo de energía es la conformación que debe adoptar cada molécula de esa proteína si la proteína desea finalmente asumir la estructura nativa. Ninguna proteína puede asumir la estructura nativa sin pasar primero por el estado de transición. [30] El estado de transición puede considerarse una variante o forma prematura del estado nativo en lugar de simplemente otro paso intermedio. [78] Se ha demostrado que el plegamiento del estado de transición determina la velocidad y, aunque existe en un estado de energía más alto que el plegamiento nativo, se parece mucho a la estructura nativa. Dentro del estado de transición, existe un núcleo alrededor del cual la proteína puede plegarse, formado por un proceso conocido como "condensación de nucleación", donde la estructura comienza a colapsar sobre el núcleo. [78]
Modelado del plegamiento de proteínas

Las técnicas de novo o ab initio para la predicción computacional de la estructura de proteínas se pueden utilizar para simular varios aspectos del plegamiento de proteínas. La dinámica molecular (MD) se utilizó en simulaciones de plegamiento de proteínas y dinámica in silico . [79] Las primeras simulaciones de plegamiento de equilibrio se realizaron utilizando un modelo de solvente implícito y muestreo de paraguas . [80] Debido al costo computacional, las simulaciones de plegamiento MD ab initio con agua explícita están limitadas a péptidos y proteínas pequeñas. [81] [82] Las simulaciones MD de proteínas más grandes permanecen restringidas a la dinámica de la estructura experimental o su desdoblamiento a alta temperatura. Se puede acceder a los procesos de plegamiento de largo plazo (más allá de aproximadamente 1 milisegundo), como el plegamiento de proteínas más grandes (>150 residuos) utilizando modelos de grano grueso . [83] [84] [85]
Varios proyectos computacionales a gran escala, como Rosetta@home , [86] Folding@home [87] y Foldit , [88], se centran en el plegamiento de proteínas.
Se han realizado simulaciones de trayectoria continua de largo alcance en Anton , una supercomputadora masivamente paralela diseñada y construida alrededor de interconexiones y circuitos integrados de aplicación específica (ASIC) personalizados por DE Shaw Research . El resultado más extenso publicado de una simulación realizada con Anton hasta 2011 fue una simulación de 2,936 milisegundos de NTL9 a 355 K. [89] Dichas simulaciones actualmente pueden desplegar y replegar proteínas pequeñas (<150 residuos de aminoácidos) en equilibrio y predecir cómo las mutaciones afectan la cinética y la estabilidad del plegamiento. [90]
En 2020, un equipo de investigadores que utilizó AlphaFold , un programa de predicción de la estructura de proteínas de inteligencia artificial (IA) desarrollado por DeepMind, obtuvo el primer puesto en CASP , un concurso de predicción de estructuras de larga data. [91] El equipo logró un nivel de precisión mucho mayor que cualquier otro grupo. [92] Obtuvo una puntuación superior al 90 % para alrededor de dos tercios de las proteínas en la prueba de distancia global (GDT) de CASP , una prueba que mide el grado de similitud entre la estructura predicha por un programa computacional y la estructura empírica determinada experimentalmente en un laboratorio. Una puntuación de 100 se considera una coincidencia completa, dentro del límite de distancia utilizado para calcular la GDT. [93]
Los resultados de la predicción de la estructura de las proteínas de AlphaFold en CASP fueron descritos como "transformacionales" y "asombrosos". [94] [95] Algunos investigadores notaron que la precisión no es lo suficientemente alta para un tercio de sus predicciones, y que no revela el mecanismo físico del plegamiento de proteínas para que el problema del plegamiento de proteínas se considere resuelto. [96] Sin embargo, se considera un logro significativo en biología computacional [93] y un gran avance hacia un gran desafío de la biología que lleva décadas en marcha, predecir la estructura de las proteínas. [94]
Véase también
- El dogma de Anfinsen
- Trama de Chevron
- Punto medio de desnaturalización
- Plegado en bajada
- Plegado (química)
- Análisis del valor phi
- Energía potencial de la proteína
- Dinámica de proteínas
- Amplificación cíclica por plegamiento incorrecto de proteínas
- Software de predicción de la estructura de proteínas
- Proteopatía
- Espectrometría de masas resuelta en el tiempo
Referencias
- ^ Alberts B , Johnson A, Lewis J, Raff M, Roberts K, Walters P (2002). "La forma y la estructura de las proteínas". Biología molecular de la célula; cuarta edición . Nueva York y Londres: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Anfinsen CB (julio de 1972). "La formación y estabilización de la estructura de las proteínas". The Biochemical Journal . 128 (4): 737–49. doi :10.1042/bj1280737. PMC 1173893 . PMID 4565129.
- ^ Berg JM, Tymoczko JL, Stryer L (2002). "3. Estructura y función de las proteínas". Bioquímica . San Francisco: WH Freeman. ISBN 978-0-7167-4684-3.
- ^ ab Selkoe DJ (diciembre de 2003). "Plegamiento de proteínas de forma fatal". Nature . 426 (6968): 900–4. Bibcode :2003Natur.426..900S. doi :10.1038/nature02264. PMID 14685251. S2CID 6451881.
- ^ Alberts B, Bray D, Hopkin K, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2010). "Estructura y función de las proteínas". Biología celular esencial (tercera edición). Nueva York, NY: Garland Science. págs. 120–70. ISBN 978-0-8153-4454-4.
- ^ Yagi-Utsumi M, Chandak MS, Yanaka S, Hiranyakorn M, Nakamura T, Kato K, Kuwajima K (noviembre de 2020). "Estructura residual de la ubiquitina desplegada revelada por RMN 2D de intercambio de hidrógeno/deuterio". Revista Biofísica . 119 (10): 2029–38. doi :10.1016/j.bpj.2020.10.003. PMC 7732725 . PMID 33142107.
- ^ Kim PS, Baldwin RL (1990). "Intermediarios en las reacciones de plegamiento de proteínas pequeñas". Revisión anual de bioquímica . 59 : 631–60. doi :10.1146/annurev.bi.59.070190.003215. PMID 2197986.
- ^ Jackson SE (1998). "¿Cómo se pliegan las proteínas pequeñas de dominio único?". Folding & Design . 3 (4): R81-91. doi :10.1016/S1359-0278(98)00033-9. PMID 9710577.
- ^ Kubelka J, Hofrichter J, Eaton WA (febrero de 2004). "El 'límite de velocidad' del plegamiento de proteínas". Current Opinion in Structural Biology . 14 (1): 76–88. doi :10.1016/j.sbi.2004.01.013. PMID 15102453.
- ^ Scalvini, Barbara; Sheikhhassani, Vahid; Mashaghi, Alireza (2021). "Principios topológicos del plegamiento de proteínas". Química física Química Física . 23 (37): 21316–21328. Bibcode :2021PCCP...2321316S. doi :10.1039/D1CP03390E. hdl : 1887/3277889 . PMID 34545868. S2CID 237583577.
- ^ Anfinsen CB (julio de 1973). "Principios que gobiernan el plegamiento de las cadenas proteínicas". Science . 181 (4096): 223–30. Bibcode :1973Sci...181..223A. doi :10.1126/science.181.4096.223. PMID 4124164.
- ^ abcdefgh Voet D, Voet JG, Pratt CW (2016). Principios de bioquímica (Quinta ed.). Wiley. ISBN 978-1-118-91840-1.
- ^ Alexander PA, He Y, Chen Y, Orban J, Bryan PN (julio de 2007). "El diseño y caracterización de dos proteínas con 88% de identidad de secuencia pero diferente estructura y función". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (29): 11963–8. Bibcode :2007PNAS..10411963A. doi : 10.1073/pnas.0700922104 . PMC 1906725 . PMID 17609385.
- ^ Rose GD, Fleming PJ, Banavar JR, Maritan A (noviembre de 2006). "Una teoría del plegamiento de proteínas basada en la estructura básica". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (45): 16623–33. Bibcode :2006PNAS..10316623R. CiteSeerX 10.1.1.630.5487 . doi : 10.1073/pnas.0606843103 . PMC 1636505 . PMID 17075053.
- ^ abc Fersht A (1999). Estructura y mecanismo en la ciencia de las proteínas: una guía para la catálisis enzimática y el plegamiento de proteínas. Macmillan. ISBN 978-0-7167-3268-6.
- ^ "Estructura de la proteína". Scitable . Nature Education . Consultado el 26 de noviembre de 2016 .
- ^ Pratt C, Cornely K (2004). "Termodinámica". Bioquímica esencial . Wiley. ISBN 978-0-471-39387-0. Recuperado el 26 de noviembre de 2016 .
- ^ Zhang G, Ignatova Z (febrero de 2011). "Plegamiento en el nacimiento de la cadena naciente: coordinación de la traducción con plegamiento co-traduccional". Current Opinion in Structural Biology . 21 (1): 25–31. doi :10.1016/j.sbi.2010.10.008. PMID 21111607.
- ^ van den Berg B, Wain R, Dobson CM, Ellis RJ (agosto de 2000). "El hacinamiento macromolecular perturba la cinética de replegamiento de proteínas: implicaciones para el plegamiento dentro de la célula". The EMBO Journal . 19 (15): 3870–5. doi :10.1093/emboj/19.15.3870. PMC 306593 . PMID 10921869.
- ^ Al-Karadaghi S. "Ángulos de torsión y diagrama de Ramachnadran en estructuras proteínicas". www.proteinstructures.com . Consultado el 26 de noviembre de 2016 .
- ^ Pace CN, Shirley BA, McNutt M, Gajiwala K (enero de 1996). "Fuerzas que contribuyen a la estabilidad conformacional de las proteínas". FASEB Journal . 10 (1): 75–83. doi : 10.1096/fasebj.10.1.8566551 . PMID 8566551. S2CID 20021399.
- ^ Cui D, Ou S, Patel S (diciembre de 2014). "Redes de agua que abarcan proteínas e implicaciones para la predicción de interacciones proteína-proteína mediadas a través de efectos hidrofóbicos". Proteins . 82 (12): 3312–26. doi :10.1002/prot.24683. PMID 25204743. S2CID 27113763.
- ^ Tanford C (junio de 1978). "El efecto hidrofóbico y la organización de la materia viva". Science . 200 (4345): 1012–8. Bibcode :1978Sci...200.1012T. doi :10.1126/science.653353. PMID 653353.
- ^ Deechongkit S, Nguyen H, Powers ET, Dawson PE, Gruebele M, Kelly JW (julio de 2004). "Contribuciones dependientes del contexto de los enlaces de hidrógeno de la cadena principal a la energía del plegamiento de la hoja beta". Nature . 430 (6995): 101–5. Bibcode :2004Natur.430..101D. doi :10.1038/nature02611. PMID 15229605. S2CID 4315026.
- ^ Irbäck A, Sandelin E (noviembre de 2000). "Sobre las correlaciones de hidrofobicidad en las cadenas proteínicas". Biophysical Journal . 79 (5): 2252–8. arXiv : cond-mat/0010390 . Bibcode :2000BpJ....79.2252I. doi :10.1016/S0006-3495(00)76472-1. PMC 1301114 . PMID 11053106.
- ^ Irbäck A, Peterson C, Potthast F (septiembre de 1996). "Evidencia de estructuras de hidrofobicidad no aleatorias en cadenas proteínicas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 93 (18): 9533–8. arXiv : chem-ph/9512004 . Bibcode :1996PNAS...93.9533I. doi : 10.1073/pnas.93.18.9533 . PMC 38463 . PMID 8790365.
- ^ Wilson BA, Foy SG, Neme R, Masel J (junio de 2017). "Nacimiento del gen De Novo". Ecología y evolución de la naturaleza . 1 (6): 0146–146. doi :10.1038/s41559-017-0146. PMC 5476217 . PMID 28642936.
- ^ Willis S, Masel J (septiembre de 2018). "El nacimiento de genes contribuye al desorden estructural codificado por genes superpuestos". Genética . 210 (1): 303–313. doi :10.1534/genetics.118.301249. PMC 6116962 . PMID 30026186.
- ^ Foy SG, Wilson BA, Bertram J, Cordes MH, Masel J (abril de 2019). "Un cambio en la estrategia de evitación de la agregación marca una dirección a largo plazo para la evolución de las proteínas". Genética . 211 (4): 1345–1355. doi :10.1534/genetics.118.301719. PMC 6456324 . PMID 30692195.
- ^ abcdef Dobson CM (diciembre de 2003). "Plegamiento y mal plegamiento de proteínas". Nature . 426 (6968): 884–90. Bibcode :2003Natur.426..884D. doi :10.1038/nature02261. PMID 14685248. S2CID 1036192.
- ^ abc Hartl FU (junio de 1996). "Chaperonas moleculares en el plegamiento de proteínas celulares". Nature . 381 (6583): 571–9. Bibcode :1996Natur.381..571H. doi :10.1038/381571a0. PMID 8637592. S2CID 4347271.
- ^ ab Hartl FU, Bracher A, Hayer-Hartl M (julio de 2011). "Chaperonas moleculares en el plegamiento de proteínas y la proteostasis". Nature . 475 (7356): 324–32. doi :10.1038/nature10317. PMID 21776078. S2CID 4337671.
- ^ Kim YE, Hipp MS, Bracher A, Hayer-Hartl M, Hartl FU (2013). "Funciones de la chaperona molecular en el plegamiento de proteínas y la proteostasis". Revisión anual de bioquímica . 82 : 323–55. doi :10.1146/annurev-biochem-060208-092442. PMID 23746257.
- ^ Shortle D (enero de 1996). "El estado desnaturalizado (la otra mitad de la ecuación de plegamiento) y su papel en la estabilidad de las proteínas". FASEB Journal . 10 (1): 27–34. doi : 10.1096/fasebj.10.1.8566543 . PMID 8566543. S2CID 24066207.
- ^ Lee S, Tsai FT (2005). "Chaperonas moleculares en el control de calidad de proteínas". Revista de bioquímica y biología molecular . 38 (3): 259–65. doi : 10.5483/BMBRep.2005.38.3.259 . PMID 15943899.
- ^ Ojeda-May P, Garcia ME (julio de 2010). "Disrupción impulsada por campo eléctrico de la conformación de una proteína de lámina beta nativa y generación de una estructura helicoidal". Revista Biofísica . 99 (2): 595–9. Bibcode :2010BpJ....99..595O. doi :10.1016/j.bpj.2010.04.040. PMC 2905109 . PMID 20643079.
- ^ van den Berg B, Ellis RJ, Dobson CM (diciembre de 1999). "Efectos del hacinamiento macromolecular en el plegamiento y agregación de proteínas". The EMBO Journal . 18 (24): 6927–33. doi :10.1093/emboj/18.24.6927. PMC 1171756 . PMID 10601015.
- ^ Ellis RJ (julio de 2006). "Chaperonas moleculares: ayudan al ensamblaje además del plegamiento". Tendencias en ciencias bioquímicas . 31 (7): 395–401. doi :10.1016/j.tibs.2006.05.001. PMID 16716593.
- ^ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K (agosto de 2008). "Proliferación celular a 122 grados C y producción de CH4 isotópicamente pesado por un metanógeno hipertermofílico bajo cultivo a alta presión". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (31): 10949–54. Bibcode :2008PNAS..10510949T. doi : 10.1073/pnas.0712334105 . PMC 2490668 . PMID 18664583.
- ^ ab Marusich, IE; Kurochkina, LP; Mesyanzhinov, VV (abril de 1998). "Chaperonas en el ensamblaje del bacteriófago T4". Bioquímica. Biokhimia . 63 (4): 399–406. PMID 9556522.
- ^ Porter, Lauren L.; Looger, Loren L. (5 de junio de 2018). "Las proteínas con cambio de plegamiento existentes están muy extendidas". Actas de la Academia Nacional de Ciencias . 115 (23): 5968–5973. Bibcode :2018PNAS..115.5968P. doi : 10.1073/pnas.1800168115 . PMC 6003340 . PMID 29784778.
- ^ abcd Chaudhuri TK, Paul S (abril de 2006). "Enfermedades por plegamiento incorrecto de proteínas y enfoques terapéuticos basados en chaperonas". The FEBS Journal . 273 (7): 1331–49. doi : 10.1111/j.1742-4658.2006.05181.x . PMID 16689923. S2CID 23370420.
- ^ ab Soto C, Estrada L, Castilla J (marzo de 2006). "Amiloides, priones y la naturaleza infecciosa inherente de los agregados de proteínas mal plegadas". Tendencias en Ciencias Bioquímicas . 31 (3): 150–5. doi :10.1016/j.tibs.2006.01.002. PMID 16473510.
- ^ Hammarström P, Wiseman RL, Powers ET, Kelly JW (enero de 2003). "Prevención de la enfermedad amiloide por transtiretina modificando la energía del plegamiento incorrecto de las proteínas". Science . 299 (5607): 713–6. Bibcode :2003Sci...299..713H. doi :10.1126/science.1079589. PMID 12560553. S2CID 30829998.
- ^ Chiti F, Dobson CM (2006). "Plegamiento incorrecto de proteínas, amiloide funcional y enfermedad humana". Revisión anual de bioquímica . 75 : 333–66. doi :10.1146/annurev.biochem.75.101304.123901. PMID 16756495. S2CID 23797549.
- ^ Johnson SM, Wiseman RL, Sekijima Y, Green NS, Adamski-Werner SL, Kelly JW (diciembre de 2005). "Estabilización cinética del estado nativo como estrategia para mejorar las enfermedades por plegamiento incorrecto de proteínas: un enfoque en las amiloidosis por transtiretina". Accounts of Chemical Research . 38 (12): 911–21. doi :10.1021/ar020073i. PMID 16359163.
- ^ ab Cowtan K (2001). "Problema de fase en cristalografía de rayos X y su solución" (PDF) . Enciclopedia de ciencias de la vida . Macmillan Publishers Ltd, Nature Publishing Group . Consultado el 3 de noviembre de 2016 .
- ^ Drenth J (5 de abril de 2007). Principios de la cristalografía de rayos X de proteínas. Springer Science & Business Media. ISBN 978-0-387-33746-3.
- ^ Taylor G (2003). "El problema de las fases". Acta Crystallographica Sección D . 59 (11): 1881–90. Bibcode :2003AcCrD..59.1881T. doi : 10.1107/S0907444903017815 . PMID 14573942.
- ^ abc Bedouelle H (febrero de 2016). "Principios y ecuaciones para medir e interpretar la estabilidad de las proteínas: del monómero al tetrámero". Biochimie . 121 : 29–37. doi :10.1016/j.biochi.2015.11.013. PMID 26607240.
- ^ Monsellier E, Bedouelle H (septiembre de 2005). "Medición cuantitativa de la estabilidad de proteínas a partir de equilibrios de desdoblamiento monitoreados con la longitud de onda máxima de fluorescencia". Ingeniería, diseño y selección de proteínas . 18 (9): 445–56. doi : 10.1093/protein/gzi046 . PMID 16087653.
- ^ Park YC, Bedouelle H (julio de 1998). "La tirosil-ARNt sintetasa dimérica de Bacillus stearothermophilus se despliega a través de un intermediario monomérico. Un análisis cuantitativo en condiciones de equilibrio". The Journal of Biological Chemistry . 273 (29): 18052–9. doi : 10.1074/jbc.273.29.18052 . PMID 9660761.
- ^ Ould-Abeih MB, Petit-Topin I, Zidane N, Baron B, Bedouelle H (junio de 2012). "Múltiples estados de plegamiento y desorden de la proteína ribosomal SA, un receptor de membrana para laminina, anticancerígenos y patógenos". Bioquímica . 51 (24): 4807–21. doi :10.1021/bi300335r. PMID 22640394.
- ^ Royer CA (mayo de 2006). "Estudio del plegamiento de proteínas y las transiciones conformacionales con fluorescencia". Chemical Reviews . 106 (5): 1769–84. doi :10.1021/cr0404390. PMID 16683754.
- ^ abcd Wüthrich K (diciembre de 1990). "Determinación de la estructura de proteínas en solución mediante espectroscopia de RMN". The Journal of Biological Chemistry . 265 (36): 22059–62. doi : 10.1016/S0021-9258(18)45665-7 . PMID 2266107.
- ^ abcde Zhuravleva A, Korzhnev DM (mayo de 2017). "Plegamiento de proteínas por RMN". Progreso en espectroscopia de resonancia magnética nuclear . 100 : 52–77. doi :10.1016/j.pnmrs.2016.10.002. PMID 28552172.
- ^ ab Ortega, Gabriel; Pons, Miquel; Millet, Oscar (2013). "Dinámica funcional de proteínas en múltiples escalas de tiempo estudiada por espectroscopia de RMN". Dinámica de proteínas y ácidos nucleicos . Avances en química de proteínas y biología estructural. Vol. 92. págs. 219–251. doi :10.1016/b978-0-12-411636-8.00006-7. ISBN 9780124116368. Número de identificación personal 23954103.
- ^ abc Vallurupalli P, Bouvignies G, Kay LE (mayo de 2012). "Estudio de estados de proteínas excitados "invisibles" en intercambio lento con una conformación de estado mayor". Revista de la Sociedad Química Americana . 134 (19): 8148–61. doi :10.1021/ja3001419. PMID 22554188.
- ^ ab Neudecker P, Lundström P, Kay LE (marzo de 2009). "Espectroscopia de RMN de dispersión por relajación como herramienta para estudios detallados del plegamiento de proteínas". Revista biofísica . 96 (6): 2045–54. Bibcode :2009BpJ....96.2045N. doi :10.1016/j.bpj.2008.12.3907. PMC 2717354 . PMID 19289032.
- ^ ab Sekhar A, Rumfeldt JA, Broom HR, Doyle CM, Sobering RE, Meiering EM, Kay LE (noviembre de 2016). "Probing the free energy landscapes of ALS disease mutants of SOD1 by NMR spectroscopy" (Investigación de los paisajes de energía libre de mutantes de SOD1 de la enfermedad de ELA mediante espectroscopia de RMN). Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 113 (45): E6939–E6945. Bibcode :2016PNAS..113E6939S. doi : 10.1073/pnas.1611418113 . PMC 5111666 . PMID 27791136.
- ^ Cross GH, Freeman NJ, Swann MJ (2008). "Interferometría de polarización dual: una técnica óptica en tiempo real para medir la orientación, la estructura y la función (bio)moleculares en la interfaz sólido/líquido". Manual de biosensores y biochips . doi :10.1002/9780470061565.hbb055. ISBN 978-0-470-01905-4.
- ^ Bu Z, Cook J, Callaway DJ (septiembre de 2001). "Regímenes dinámicos y dinámica estructural correlacionada en alfa-lactalbúmina nativa y desnaturalizada". Journal of Molecular Biology . 312 (4): 865–73. doi :10.1006/jmbi.2001.5006. PMID 11575938.
- ^ Minde DP, Maurice MM, Rüdiger SG (2012). "Determinación de la estabilidad biofísica de las proteínas en lisados mediante un ensayo de proteólisis rápida, FASTpp". PLOS ONE . 7 (10): e46147. Bibcode :2012PLoSO...746147M. doi : 10.1371/journal.pone.0046147 . PMC 3463568 . PMID 23056252.
- ^ Park C, Marqusee S (marzo de 2005). "Proteólisis pulsátil: un método simple para la determinación cuantitativa de la estabilidad de las proteínas y la unión de ligandos". Nature Methods . 2 (3): 207–12. doi :10.1038/nmeth740. PMID 15782190. S2CID 21364478.
- ^ Mashaghi A, Kramer G, Lamb DC, Mayer MP, Tans SJ (enero de 2014). "Acción de chaperona a nivel de molécula única". Chemical Reviews . 114 (1): 660–76. doi :10.1021/cr400326k. PMID 24001118.
- ^ Jagannathan B, Marqusee S (noviembre de 2013). "Plegado y desplegado de proteínas bajo fuerza". Biopolímeros . 99 (11): 860–9. doi :10.1002/bip.22321. PMC 4065244 . PMID 23784721.
- ^ Jakobi AJ, Mashaghi A, Tans SJ, Huizinga EG (julio de 2011). "El calcio modula la detección de fuerza mediante el dominio A2 del factor von Willebrand". Comunicaciones de la naturaleza . 2 : 385. Código Bib : 2011NatCo...2..385J. doi : 10.1038/ncomms1385. PMC 3144584 . PMID 21750539.
- ^ Jagannathan B, Elms PJ, Bustamante C, Marqusee S (octubre de 2012). "Observación directa de un interruptor inducido por fuerza en la vía de desdoblamiento mecánico anisotrópico de una proteína". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (44): 17820–5. Bibcode :2012PNAS..10917820J. doi : 10.1073/pnas.1201800109 . PMC 3497811 . PMID 22949695.
- ^ Minde DP, Ramakrishna M, Lilley KS (2018). "La biotinilación mediante etiquetado de proximidad favorece a las proteínas desplegadas" (PDF) . bioRxiv . doi : 10.1101/274761 .
- ^ Compiani M, Capriotti E (diciembre de 2013). "Métodos computacionales y teóricos para el plegamiento de proteínas". Bioquímica . 52 (48): 8601–24. doi :10.1021/bi4001529. PMID 24187909.
- ^ "Bioquímica estructural/Proteínas/Plegamiento de proteínas - Wikilibros, libros abiertos para un mundo abierto". es.wikibooks.org . Consultado el 5 de noviembre de 2016 .
- ^ Levinthal C (1968). "¿Existen vías para el plegamiento de proteínas?" (PDF) . Journal de Chimie Physique et de Physico-Chimie Biologique . 65 : 44–45. Bibcode :1968JCP....65...44L. doi :10.1051/jcp/1968650044. Archivado desde el original (PDF) el 2 de septiembre de 2009.
- ^ abc Bryngelson JD, Onuchic JN, Socci ND, Wolynes PG (marzo de 1995). "Embudos, vías y el paisaje energético del plegamiento de proteínas: una síntesis". Proteins . 21 (3): 167–95. arXiv : chem-ph/9411008 . doi :10.1002/prot.340210302. PMID 7784423. S2CID 13838095.
- ^ Leopold PE, Montal M, Onuchic JN (septiembre de 1992). "Embudos de plegamiento de proteínas: un enfoque cinético de la relación secuencia-estructura". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 89 (18): 8721–5. Bibcode :1992PNAS...89.8721L. doi : 10.1073/pnas.89.18.8721 . PMC 49992 . PMID 1528885.
- ^ Sharma V, Kaila VR, Annila A (2009). "El plegamiento de proteínas como un proceso evolutivo". Physica A: Mecánica estadística y sus aplicaciones . 388 (6): 851–62. Bibcode :2009PhyA..388..851S. doi :10.1016/j.physa.2008.12.004.
- ^ Robson B, Vaithilingam A (2008). "Revisitando el plegamiento de proteínas". Biología molecular del plegamiento de proteínas, parte B. Progreso en biología molecular y ciencia traslacional. Vol. 84. págs. 161–202. doi :10.1016/S0079-6603(08)00405-4. ISBN 978-0-12-374595-8. Número de identificación personal 19121702.
- ^ abc Dill KA, MacCallum JL (noviembre de 2012). "El problema del plegamiento de proteínas, 50 años después". Science . 338 (6110): 1042–6. Bibcode :2012Sci...338.1042D. doi :10.1126/science.1219021. PMID 23180855. S2CID 5756068.
- ^ ab Fersht AR (febrero de 2000). "Estructura del estado de transición como base unificadora en los mecanismos de plegamiento de proteínas: orden de contacto, topología de la cadena, estabilidad y el mecanismo del núcleo extendido". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 97 (4): 1525–9. Bibcode :2000PNAS...97.1525F. doi : 10.1073/pnas.97.4.1525 . PMC 26468 . PMID 10677494.
- ^ Rizzuti B, Daggett V (marzo de 2013). "Uso de simulaciones para proporcionar el marco para estudios experimentales de plegamiento de proteínas". Archivos de bioquímica y biofísica . 531 (1–2): 128–35. doi :10.1016/j.abb.2012.12.015. PMC 4084838 . PMID 23266569.
- ^ Schaefer M, Bartels C, Karplus M (diciembre de 1998). "Conformaciones de solución y termodinámica de péptidos estructurados: simulación de dinámica molecular con un modelo de solvatación implícito". Journal of Molecular Biology . 284 (3): 835–48. doi :10.1006/jmbi.1998.2172. PMID 9826519.
- ^ Jones D. "Simulaciones de plegamiento de proteínas basadas en fragmentos". University College London.
- ^ "Plegamiento de proteínas" (por Molecular Dynamics) .
- ^ Kmiecik S, Gront D, Kolinski M, Wieteska L, Dawid AE, Kolinski A (julio de 2016). "Modelos de proteínas de grano grueso y sus aplicaciones". Reseñas químicas . 116 (14): 7898–936. doi : 10.1021/acs.chemrev.6b00163 . PMID 27333362.
- ^ Kmiecik S, Kolinski A (julio de 2007). "Caracterización de las vías de plegamiento de proteínas mediante modelado en espacio reducido". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (30): 12330–5. Bibcode :2007PNAS..10412330K. doi : 10.1073/pnas.0702265104 . PMC 1941469 . PMID 17636132.
- ^ Adhikari AN, Freed KF, Sosnick TR (octubre de 2012). "Predicción de novo de las vías de plegamiento de proteínas y su estructura utilizando el principio de estabilización secuencial". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (43): 17442–7. Bibcode :2012PNAS..10917442A. doi : 10.1073/pnas.1209000109 . PMC 3491489 . PMID 23045636.
- ^ "Rosetta@home". boinc.bakerlab.org . Consultado el 14 de marzo de 2023 .
- ^ "El Consorcio Folding@home (FAHC) – Folding@home" . Consultado el 14 de marzo de 2023 .
- ^ "Foldit". fold.it . Consultado el 14 de marzo de 2023 .
- ^ Lindorff-Larsen K, Piana S, Dror RO, Shaw DE (octubre de 2011). "Cómo se pliegan las proteínas de plegado rápido". Science . 334 (6055): 517–20. Bibcode :2011Sci...334..517L. doi :10.1126/science.1208351. PMID 22034434. S2CID 27988268.
- ^ Piana S, Piana S, Sarkar K, Lindorff-Larsen K, Guo M, Gruebele M, Shaw DE (2010). "Diseño computacional y prueba experimental de la proteína de lámina β de plegamiento más rápido". J. Mol. Biol . 405 (1): 43–48. doi :10.1016/j.jmb.2010.10.023. PMID 20974152.
- ^ Shead, Sam (30 de noviembre de 2020). "DeepMind resuelve un 'gran desafío' de 50 años con inteligencia artificial que pliega proteínas" CNBC . Consultado el 30 de noviembre de 2020 .
- ^ Stoddart, Charlotte (1 de marzo de 2022). "Biología estructural: cómo las proteínas consiguieron su primer plano". Revista Knowable . doi : 10.1146/knowable-022822-1 . S2CID : 247206999. Consultado el 25 de marzo de 2022 .
- ^ ab Robert F. Service, 'El juego ha cambiado'. La IA triunfa en la resolución de estructuras proteínicas, Science , 30 de noviembre de 2020
- ^ ab Callaway, Ewen (30 de noviembre de 2020). ""Cambiará todo": la IA de DeepMind da un salto gigantesco en la resolución de estructuras proteínicas". Nature . 588 (7837): 203–204. Bibcode :2020Natur.588..203C. doi :10.1038/d41586-020-03348-4. PMID 33257889. S2CID 227243204.
- ^ @MoAlQuraishi (30 de noviembre de 2020). "Acaban de salir los números de CASP14 y son asombrosos" ( Tweet ) – vía Twitter .
- ^ Balls, Phillip (9 de diciembre de 2020). "Detrás de las pantallas de AlphaFold". Chemistry World .
Enlaces externos
- Proyecto de plegamiento del proteoma humano