Poliploidía

La poliploidía es una condición en la que las células de un organismo tienen más de dos pares de cromosomas ( homólogos ) . La mayoría de las especies cuyas células tienen núcleo ( eucariotas ) son diploides , lo que significa que tienen dos juegos completos de cromosomas, uno de cada uno de los dos progenitores; cada juego contiene el mismo número de cromosomas, y los cromosomas están unidos en pares de cromosomas homólogos. Sin embargo, algunos organismos son poliploides . La poliploidía es especialmente común en las plantas. La mayoría de los eucariotas tienen células somáticas diploides , pero producen gametos haploides (óvulos y espermatozoides) por meiosis . Un monoploide tiene solo un juego de cromosomas, y el término generalmente solo se aplica a células u organismos que normalmente son diploides. Los machos de las abejas y otros himenópteros , por ejemplo, son monoploides. A diferencia de los animales, las plantas y las algas multicelulares tienen ciclos de vida con dos generaciones multicelulares alternas . La generación gametofítica es haploide y produce gametos por mitosis ; la generación esporofita es diploide y produce esporas por meiosis .
La poliploidía es el resultado de la duplicación de todo el genoma durante la evolución de las especies. Puede ocurrir debido a una división celular anormal , ya sea durante la mitosis, o más comúnmente por la falla de los cromosomas al separarse durante la meiosis o por la fertilización de un óvulo por más de un espermatozoide. [1] Además, puede ser inducida en plantas y cultivos celulares por algunas sustancias químicas: la más conocida es la colchicina , que puede resultar en la duplicación de cromosomas, aunque su uso puede tener también otras consecuencias menos obvias. La orizalina también duplicará el contenido cromosómico existente.
Entre los mamíferos , se encuentra una alta frecuencia de células poliploides en órganos como el cerebro, el hígado, el corazón y la médula ósea. [2] También se presenta en las células somáticas de otros animales , como los peces de colores , [3] el salmón y las salamandras . Es común entre los helechos y las plantas con flores (véase Hibiscus rosa-sinensis ), incluidas tanto las especies silvestres como las cultivadas . El trigo , por ejemplo, después de milenios de hibridación y modificación por parte de los humanos, tiene cepas que son diploides (dos juegos de cromosomas), tetraploides (cuatro juegos de cromosomas) con el nombre común de trigo duro o macarrones, y hexaploides (seis juegos de cromosomas) con el nombre común de trigo panificable. Muchas plantas de importancia agrícola del género Brassica también son tetraploides. La caña de azúcar puede tener niveles de ploidía superiores a los octaploides . [4]
La poliploidización puede ser un mecanismo de especiación simpátrica porque los poliploides normalmente no pueden cruzarse con sus ancestros diploides. Un ejemplo es la planta Erythranthe peregrina . La secuenciación confirmó que esta especie se originó a partir de E. × robertsii , un híbrido triploide estéril entre E. guttata y E. lutea, ambas introducidas y naturalizadas en el Reino Unido. Nuevas poblaciones de E. peregrina surgieron en el continente escocés y en las Islas Orcadas a través de la duplicación del genoma de poblaciones locales de E. × robertsii . [5] Debido a una rara mutación genética, E. peregrina no es estéril. [6]
Por otra parte, la poliploidización también puede ser un mecanismo para una especie de "especiación inversa", [7] por la cual se permite el flujo genético después del evento de poliploidía, incluso entre linajes que previamente no experimentaron flujo genético como diploides. Esto se ha detallado a nivel genómico en Arabidopsis arenosa y Arabidopsis lyrata . [8] Cada una de estas especies experimentó eventos de autopoliploidía independientes (poliploidía dentro de la especie, descrita más adelante), que luego permitieron el flujo genético interespecie posterior de alelos adaptativos, en este caso estabilizando cada linaje poliploide joven. [9] Tal introgresión adaptativa habilitada por la poliploidía puede permitir que los poliploides actúen como "esponjas alélicas", por lo que acumulan variación genómica críptica que puede ser reclutada al encontrar desafíos ambientales posteriores. [10]
Terminología
Tipos

Los tipos poliploides se clasifican según la cantidad de conjuntos de cromosomas en el núcleo . La letra x se utiliza para representar la cantidad de cromosomas en un solo conjunto:
- haploide (un conjunto; 1 x ), por ejemplo, las hormigas de fuego europeas macho
- diploide (dos conjuntos; 2 x ), por ejemplo los humanos
- triploide (tres conjuntos; 3 x ), por ejemplo, el azafrán estéril o las sandías sin semillas , también comunes en el filo Tardigrada [11]
- tetraploide (cuatro conjuntos; 4 x ), por ejemplo, la rata vizcacha de las llanuras , el pez salmónido [12] , el algodonero Gossypium hirsutum [13]
- pentaploide (cinco series; 5 x ), por ejemplo abedul de Kenai ( Betula kenaica )
- hexaploide (seis conjuntos; 6 x ), por ejemplo algunas especies de trigo , [14] kiwi [15]
- heptaploide o septaploide (siete series; 7 x ), por ejemplo, algunos esturiones siberianos cultivados [16]
- octaploide u octoploide , (ocho conjuntos; 8 x ), por ejemplo Acipenser (género de peces esturión ), dalias
- decaploide (diez series; 10 x ), por ejemplo ciertas fresas
- dodecaploide o duodecaploide (doce conjuntos; 12 x ), por ejemplo las plantas Celosia argentea y Spartina anglica [17] o el anfibio Xenopus ruwenzoriensis .
- tetratetracontaploide (cuarenta y cuatro conjuntos; 44 x ), por ejemplo, la morera negra [18]
Clasificación
Autopoliploidía
Los autopoliploides son poliploides con múltiples conjuntos de cromosomas derivados de un solo taxón .
Dos ejemplos de autopoliploides naturales son la planta piggyback, Tolmiea menzisii [19] y el esturión blanco, Acipenser transmontanum . [20] La mayoría de los casos de autopoliploidía resultan de la fusión de gametos no reducidos (2 n ), lo que da como resultado una descendencia triploide ( n + 2 n = 3 n ) o tetraploide (2 n + 2 n = 4 n ). [21] La descendencia triploide es típicamente estéril (como en el fenómeno del bloqueo triploide ), pero en algunos casos pueden producir altas proporciones de gametos no reducidos y así ayudar a la formación de tetraploides. Esta vía hacia la tetraploidía se conoce como el puente triploide . [21] Los triploides también pueden persistir a través de la reproducción asexual . De hecho, la autotriploidía estable en plantas a menudo se asocia con sistemas de apareamiento apomícticos . [22] En los sistemas agrícolas, la autotriploidía puede provocar la ausencia de semillas, como en las sandías y los plátanos . [23] La triploidía también se utiliza en la cría de salmón y trucha para inducir la esterilidad. [24] [25]
En raras ocasiones, los autopoliploides surgen de la duplicación espontánea y somática del genoma, lo que se ha observado en los brotes de manzano ( Malus domesticus ) . [26] Esta es también la vía más común de poliploidía inducida artificialmente, donde se utilizan métodos como la fusión de protoplastos o el tratamiento con colchicina , orizalina o inhibidores mitóticos para interrumpir la división mitótica normal , lo que da como resultado la producción de células poliploides. Este proceso puede ser útil en el fitomejoramiento, especialmente cuando se intenta introgresar germoplasma a través de niveles ploidales. [27]
Los autopoliploides poseen al menos tres conjuntos de cromosomas homólogos , lo que puede conducir a altas tasas de emparejamiento multivalente durante la meiosis (particularmente en autopoliploides recientemente formados, también conocidos como neopoliploides) y una disminución asociada en la fertilidad debido a la producción de gametos aneuploides . [28] La selección natural o artificial para la fertilidad puede estabilizar rápidamente la meiosis en autopoliploides al restaurar el emparejamiento bivalente durante la meiosis. La rápida evolución adaptativa de la maquinaria meiótica, que resulta en niveles reducidos de multivalentes (y por lo tanto una meiosis autopoliploide estable) se ha documentado en Arabidopsis arenosa [29] y Arabidopsis lyrata [30] , con alelos adaptativos específicos de estas especies compartidos solo entre los poliploides evolucionados. [31] [32]
El alto grado de homología entre los cromosomas duplicados hace que los autopoliploides muestren una herencia polisómica . [33] Este rasgo se utiliza a menudo como criterio de diagnóstico para distinguir los autopoliploides de los alopoliploides, que comúnmente muestran una herencia disómica después de progresar más allá de la etapa neopoliploide. [34] Si bien la mayoría de las especies poliploides se caracterizan inequívocamente como autopoliploides o alopoliploides, estas categorías representan los extremos de un espectro de divergencia entre los subgenomas parentales. Los poliploides que se encuentran entre estos dos extremos, que a menudo se denominan alopoliploides segmentarios, pueden mostrar niveles intermedios de herencia polisómica que varían según el locus. [35] [36]
Se cree que aproximadamente la mitad de todos los poliploides son el resultado de la autopoliploidía, [37] [38] aunque muchos factores hacen que esta proporción sea difícil de estimar. [39]
Alopoliploidía
Los alopoliploides , anfipoliploides o heteropoliploides son poliploides con cromosomas derivados de dos o más taxones divergentes.
Al igual que en la autopoliploidía, esto ocurre principalmente a través de la fusión de gametos no reducidos (2 n ), que puede tener lugar antes o después de la hibridación . En el primer caso, los gametos no reducidos de cada taxón diploide, o los gametos reducidos de dos taxones autotetraploides, se combinan para formar descendencia alopoliploide. En el último caso, uno o más híbridos diploides F 1 producen gametos no reducidos que se fusionan para formar progenie alopoliploide. [40] La hibridación seguida de duplicación del genoma puede ser una vía más común hacia la alopoliploidía porque los híbridos F 1 entre taxones a menudo tienen tasas relativamente altas de formación de gametos no reducidos: la divergencia entre los genomas de los dos taxones da como resultado un apareamiento anormal entre cromosomas homólogos o la no disyunción durante la meiosis. [40] En este caso, la alopoliploidía puede realmente restaurar el emparejamiento meiótico bivalente normal al proporcionar a cada cromosoma homólogo su propio homólogo. Si la divergencia entre cromosomas homólogos es uniforme en los dos subgenomas, esto puede dar como resultado teóricamente una restauración rápida del emparejamiento bivalente y la herencia disómica después de la alopoliploidización. Sin embargo, el emparejamiento multivalente es común en muchos alopoliploides recientemente formados, por lo que es probable que la mayor parte de la estabilización meiótica ocurra gradualmente a través de la selección. [28] [34]
Debido a que el apareamiento entre cromosomas homólogos es raro en alopoliploides establecidos, estos pueden beneficiarse de la heterocigosidad fija de los alelos homólogos. [41] En ciertos casos, dicha heterocigosidad puede tener efectos heteróticos beneficiosos , ya sea en términos de aptitud en contextos naturales o rasgos deseables en contextos agrícolas. Esto podría explicar parcialmente la prevalencia de la alopoliploidía entre las especies de cultivos. Tanto el trigo panificable como el triticale son ejemplos de alopoliploides con seis conjuntos de cromosomas. El algodón , el maní o la quinua son alotetraploides con múltiples orígenes. En los cultivos brasicáceos , el Triángulo de U describe las relaciones entre las tres Brassicas diploides comunes ( B. oleracea , B. rapa y B. nigra) y tres alotetraploides (B. napus, B. juncea y B. carinata ) derivados de la hibridación entre las especies diploides. Existe una relación similar entre tres especies diploides de Tragopogon ( T. dubius , T. pratensis y T. porrifolius ) y dos especies alotetraploides ( T. mirus y T. miscellus ). [42] También se han observado patrones complejos de evolución alopoliploide en animales, como en el género de ranas Xenopus . [43]
Aneuploide
Los organismos en los que un cromosoma particular, o un segmento cromosómico, está subrepresentado o sobrerrepresentado se denominan aneuploides (de las palabras griegas que significan "no", "bueno" y "doblado"). La aneuploidía se refiere a un cambio numérico en una parte del conjunto de cromosomas, mientras que la poliploidía se refiere a un cambio numérico en el conjunto completo de cromosomas. [44]
Endopoliploidía
La poliploidía se produce en algunos tejidos de animales que, por lo demás, son diploides, como los tejidos musculares humanos . [45] Esto se conoce como endopoliploidía . Las especies cuyas células no tienen núcleo, es decir, los procariotas , pueden ser poliploides, como se observa en la gran bacteria Epulopiscium fishelsoni . [46] Por tanto, la ploidía se define con respecto a una célula.
Monoploide
Un monoploide tiene un solo juego de cromosomas y el término generalmente se aplica únicamente a células u organismos que normalmente son diploides. El término más general para estos organismos es haploide .
Términos temporales
Neopoliploidía
Un poliploide que se ha formado recientemente.
Mesopoliploidía
En la historia más reciente, se ha vuelto poliploide; no es tan nuevo como un neopoliploide ni tan antiguo como un paleopoliploide. Es un poliploide de mediana edad. A menudo, esto se refiere a la duplicación de todo el genoma seguida de niveles intermedios de diploidización.
Paleopoliploidía

Probablemente se produjeron duplicaciones de genomas antiguos en la historia evolutiva de toda la vida. Los eventos de duplicación que ocurrieron hace mucho tiempo en la historia de varios linajes evolutivos pueden ser difíciles de detectar debido a la diploidización posterior (de modo que un poliploide comienza a comportarse citogenéticamente como un diploide con el tiempo) a medida que las mutaciones y las traducciones de genes hacen que gradualmente una copia de cada cromosoma sea diferente de la otra copia. Con el tiempo, también es común que las copias duplicadas de genes acumulen mutaciones y se conviertan en pseudogenes inactivos. [47]
En muchos casos, estos eventos pueden inferirse solo a través de la comparación de genomas secuenciados . Ejemplos de duplicaciones de genomas antiguos inesperadas pero confirmadas recientemente incluyen la levadura de panadería ( Saccharomyces cerevisiae ), la hierba mostaza/berro de thale ( Arabidopsis thaliana ), el arroz ( Oryza sativa ) y dos rondas de duplicación del genoma completo (la hipótesis 2R ) en un ancestro evolutivo temprano de los vertebrados (que incluye el linaje humano ) y otro cerca del origen de los peces teleósteos . [48] Las angiospermas ( plantas con flores ) tienen paleopoliploidía en su ascendencia. Todos los eucariotas probablemente han experimentado un evento de poliploidía en algún momento de su historia evolutiva.
Otros términos similares
Cariotipo
Un cariotipo es el complemento cromosómico característico de una especie eucariota . [49] [50] La preparación y estudio de los cariotipos es parte de la citología y, más específicamente, de la citogenética .
Aunque la replicación y transcripción del ADN está altamente estandarizada en los eucariotas , no se puede decir lo mismo de sus cariotipos, que son muy variables entre especies en cuanto a número de cromosomas y organización detallada a pesar de estar construidos a partir de las mismas macromoléculas. En algunos casos, incluso hay una variación significativa dentro de las especies. Esta variación proporciona la base para una serie de estudios en lo que podría llamarse citología evolutiva.
Cromosomas homólogos
Los cromosomas homólogos son aquellos que se unen tras la hibridación interespecies y la alopoliploidización , y cuya relación era completamente homóloga en una especie ancestral. Por ejemplo, el trigo duro es el resultado de la hibridación interespecies de dos especies de gramíneas diploides Triticum urartu y Aegilops speltoides . Ambos ancestros diploides tenían dos juegos de 7 cromosomas, que eran similares en términos de tamaño y genes contenidos en ellos. El trigo duro contiene un genoma híbrido con dos juegos de cromosomas derivados de Triticum urartu y dos juegos de cromosomas derivados de Aegilops speltoides . Cada par de cromosomas derivado del progenitor Triticum urartu es homólogo al par de cromosomas opuesto derivado del progenitor Aegilops speltoides , aunque cada par de cromosomas es homólogo en sí mismo .
Ejemplos
Animales
Los ejemplos en animales son más comunes en no vertebrados [51] como platelmintos , sanguijuelas y camarones en salmuera . Dentro de los vertebrados, los ejemplos de poliploidía estable incluyen los salmónidos y muchos ciprínidos (es decir, la carpa ). [52] Algunos peces tienen hasta 400 cromosomas. [52] La poliploidía también ocurre comúnmente en anfibios; por ejemplo, el género biomédicamente importante Xenopus contiene muchas especies diferentes con hasta 12 juegos de cromosomas (dodecaploide). [53] Los lagartos poliploides también son bastante comunes. La mayoría son estériles y se reproducen por partenogénesis ; [ cita requerida ] otros, como Liolaemus chiliensis , mantienen la reproducción sexual. Las salamandras topo poliploides (en su mayoría triploides) son todas hembras y se reproducen por cleptogénesis , [54] "robando" espermatóforos de machos diploides de especies relacionadas para desencadenar el desarrollo de óvulos, pero sin incorporar el ADN de los machos a la descendencia.
Si bien algunos tejidos de los mamíferos, como las células hepáticas parenquimatosas , son poliploides, [55] [56] se conocen casos raros de mamíferos poliploides , pero la mayoría de las veces resultan en muerte prenatal . Un roedor octodóntido de las duras regiones desérticas de Argentina , conocido como la rata vizcacha de las llanuras ( Tympanoctomys barrerae ) ha sido reportado como una excepción a esta "regla". [57] Sin embargo, un análisis cuidadoso utilizando pinturas cromosómicas muestra que solo hay dos copias de cada cromosoma en T. barrerae , no las cuatro esperadas si realmente fuera un tetraploide. [58] Este roedor no es una rata , sino pariente de los conejillos de indias y las chinchillas . Su "nuevo" número diploide (2 n ) es 102 y, por lo tanto, sus células tienen aproximadamente el doble del tamaño normal. Su pariente vivo más cercano es Octomys mimax , la rata vizcacha andina de la misma familia, cuyo 2 n = 56. Por lo tanto, se supuso que un ancestro similar a Octomys produjo crías tetraploides (es decir, 2 n = 4 x = 112) que estaban, en virtud de sus cromosomas duplicados, reproductivamente aisladas de sus padres.
La poliploidía fue inducida en peces por Har Swarup (1956) utilizando un tratamiento de choque frío de los huevos cerca del momento de la fertilización, que produjo embriones triploides que maduraron con éxito. [59] [60] También se ha demostrado que el choque frío o térmico da como resultado gametos de anfibios no reducidos, aunque esto ocurre más comúnmente en los huevos que en los espermatozoides. [61] John Gurdon (1958) trasplantó núcleos intactos de células somáticas para producir huevos diploides en la rana, Xenopus (una extensión del trabajo de Briggs y King en 1952) que pudieron desarrollarse hasta la etapa de renacuajo. [62] El científico británico JBS Haldane elogió el trabajo por sus posibles aplicaciones médicas y, al describir los resultados, se convirtió en uno de los primeros en usar la palabra " clon " en referencia a los animales. El trabajo posterior de Shinya Yamanaka mostró cómo las células maduras pueden reprogramarse para volverse pluripotentes, extendiendo las posibilidades a las células no madre. Gurdon y Yamanaka recibieron conjuntamente el Premio Nobel en 2012 por este trabajo. [62]
Humanos

La verdadera poliploidía rara vez ocurre en humanos, aunque las células poliploides aparecen en tejidos altamente diferenciados , como el parénquima hepático , el músculo cardíaco, la placenta y la médula ósea. [63] [64] La aneuploidía es más común.
La poliploidía se presenta en los seres humanos en forma de triploidía , con 69 cromosomas (a veces llamada 69, XXX), y tetraploidía con 92 cromosomas (a veces llamada 92, XXXX). La triploidía, generalmente debida a polispermia , se presenta en alrededor del 2-3% de todos los embarazos humanos y en ~15% de los abortos espontáneos. [ cita requerida ] La gran mayoría de las concepciones triploides terminan en un aborto espontáneo ; las que sobreviven hasta el término suelen morir poco después del nacimiento. En algunos casos, la supervivencia después del nacimiento puede extenderse si hay mixoploidía con una población de células tanto diploides como triploides presentes. Ha habido un informe de un niño que sobrevivió hasta la edad de siete meses con síndrome de triploidía completo. No mostró un desarrollo neonatal mental o físico normal y murió a causa de una infección por Pneumocystis carinii , lo que indica un sistema inmunológico débil. [65]
La triploidía puede ser el resultado de la digynia (el conjunto haploide adicional proviene de la madre) o de la diandria (el conjunto haploide adicional proviene del padre). La diandria es causada principalmente por la reduplicación del conjunto haploide paterno de un solo espermatozoide, pero también puede ser la consecuencia de la fertilización dispérmica (dos espermatozoides) del óvulo. [66] La digynia es causada más comúnmente por el fracaso de una división meiótica durante la ovogénesis que conduce a un ovocito diploide o por el fracaso en la extrusión de un cuerpo polar del ovocito . La diandria parece predominar entre los abortos espontáneos tempranos , mientras que la digynia predomina entre los cigotos triploides que sobreviven hasta el período fetal. [67] Sin embargo, entre los abortos espontáneos tempranos, la digynia también es más común en aquellos casos de menos de 8+1 ⁄ 2 semanas de edad gestacional o aquellos en los que está presente un embrión. También hay dos fenotipos distintos en placentas y fetos triploides que dependen del origen del conjunto haploide adicional . En la diginia, normalmente hay un feto asimétrico de bajo crecimiento , con marcada hipoplasia suprarrenal y una placenta muy pequeña. [68] En la diandria, se desarrolla una mola hidatiforme parcial . [66] Estos efectos de origen parental reflejan los efectos de la impronta genómica . [ cita requerida ]
La tetraploidía completa se diagnostica con menos frecuencia que la triploidía, pero se observa en el 1-2% de los abortos espontáneos tempranos. Sin embargo, algunas células tetraploides se encuentran comúnmente en el análisis cromosómico en el diagnóstico prenatal y generalmente se consideran "inofensivas". No está claro si estas células tetraploides simplemente tienden a surgir durante el cultivo celular in vitro o si también están presentes en las células placentarias in vivo . En cualquier caso, hay muy pocos informes clínicos de fetos/bebés diagnosticados con mosaicismo tetraploide.
La mixoploidía se observa con bastante frecuencia en embriones humanos preimplantacionales e incluye poblaciones celulares mixtas haploides/diploides y diploides/tetraploides. Se desconoce si estos embriones no logran implantarse y, por lo tanto, rara vez se detectan en embarazos en curso o si simplemente existe un proceso selectivo que favorece a las células diploides.
Pez
Se produjo un evento de poliploidía dentro del linaje madre de los peces teleósteos . [48]
Plantas

La poliploidía es frecuente en las plantas; algunas estimaciones sugieren que entre el 30 y el 80 % de las especies de plantas vivas son poliploides, y muchos linajes muestran evidencia de poliploidía antigua ( paleopoliploidía ) en sus genomas. [69] [70] [71] [72] Enormes explosiones en la diversidad de especies de angiospermas parecen haber coincidido con el momento de las antiguas duplicaciones de genomas compartidas por muchas especies. [73] Se ha establecido que el 15 % de los eventos de especiación de angiospermas y el 31 % de los helechos están acompañados de un aumento de la ploidía. [74]
Las plantas poliploides pueden surgir espontáneamente en la naturaleza por varios mecanismos, incluyendo fallas meióticas o mitóticas, y fusión de gametos no reducidos (2 n ). [41] Tanto los autopoliploides (por ejemplo, la papa [75] ) como los alopoliploides (como la canola, el trigo y el algodón) se pueden encontrar entre especies de plantas tanto silvestres como domesticadas.
La mayoría de los poliploides muestran variaciones o morfologías novedosas en relación con sus especies parentales, que pueden contribuir a los procesos de especiación y explotación de nichos ecológicos. [70] [41] Los mecanismos que conducen a una variación novedosa en alopoliploides recién formados pueden incluir efectos de dosis génica (resultantes de copias más numerosas del contenido del genoma), la reunión de jerarquías reguladoras génicas divergentes, reordenamientos cromosómicos y remodelación epigenética , todos los cuales afectan el contenido génico y/o los niveles de expresión. [76] [77] [78] [79] Muchos de estos cambios rápidos pueden contribuir al aislamiento reproductivo y la especiación. Sin embargo, las semillas generadas a partir de cruces de interploidía , como entre poliploides y sus especies parentales, generalmente tienen un desarrollo aberrante del endospermo que perjudica su viabilidad, [80] [81] contribuyendo así a la especiación poliploide . Los poliploides también pueden cruzarse con diploides y producir semillas poliploides, como se observa en los complejos agámicos de Crepis . [82]
Algunas plantas son triploides. Como la meiosis se altera, estas plantas son estériles y todas tienen la misma constitución genética: entre ellas, el azafrán ( Crocus sativus ), que se propaga exclusivamente de forma vegetativa. También el arbusto extremadamente raro de Tasmania Lomatia tasmanica es una especie triploide estéril.
Hay pocas coníferas poliploides naturales . [83] Un ejemplo es la sequoia sempervirens , que es hexaploide (6 x ) con 66 cromosomas (2 n = 6 x = 66), aunque el origen no está claro. [84]
Las plantas acuáticas, especialmente las monocotiledóneas , incluyen una gran cantidad de poliploides. [85]
Cultivos
La inducción de poliploidía es una técnica común para superar la esterilidad de una especie híbrida durante el mejoramiento de plantas. Por ejemplo, el triticale es el híbrido de trigo ( Triticum turgidum ) y centeno ( Secale cereale ). Combina las características buscadas de los progenitores, pero los híbridos iniciales son estériles. Después de la poliploidización, el híbrido se vuelve fértil y, por lo tanto, puede propagarse para convertirse en triticale.
En algunas situaciones, se prefieren los cultivos poliploides porque son estériles. Por ejemplo, muchas variedades de frutas sin semillas no tienen semillas como resultado de la poliploidía. Dichos cultivos se propagan mediante técnicas asexuales, como el injerto .
La poliploidía en plantas de cultivo se induce más comúnmente mediante el tratamiento de las semillas con el químico colchicina .
Ejemplos
- Cultivos triploides: algunas variedades de manzana (como Belle de Boskoop , Jonagold , Mutsu , Ribston Pippin ), plátano , cítricos , jengibre , sandía , [86] azafrán , pulpa blanca de coco.
- Cultivos tetraploides: muy pocas variedades de manzana , trigo duro o macarrones , algodón , papa , canola / colza , puerro , tabaco , maní , kinnow , Pelargonium
- Cultivos hexaploides: crisantemo , trigo panificable , triticale , avena , kiwi [15]
- Cultivos octaploides: fresa , dalia , pensamientos , caña de azúcar , oca ( Oxalis tuberosa ) [87]
- Cultivos dodecaploides: algunos híbridos de caña de azúcar [88]
Algunos cultivos se encuentran en una variedad de ploidías: los tulipanes y los lirios se encuentran comúnmente como diploides y triploides; los daylilies ( cultivares de Hemerocallis ) están disponibles como diploides o tetraploides; las manzanas y las mandarinas kinnow pueden ser diploides, triploides o tetraploides.
Hongos
Además de las plantas y los animales, la historia evolutiva de varias especies de hongos está salpicada de eventos pasados y recientes de duplicación de todo el genoma (véase Albertin y Marullo 2012 [89] para una revisión). Se conocen varios ejemplos de poliploides:
- autopoliploide: los hongos acuáticos del género Allomyces , [90] algunas cepas de Saccharomyces cerevisiae utilizadas en panadería , [91] etc.
- alopoliploide: el extendido Cyathus stercoreus , [92] la levadura lager alotetraploide Saccharomyces pastorianus , [93] la levadura alotriploide causante del deterioro del vino Dekkera bruxellensis , [94] etc.
- paleopoliploide: el patógeno humano Rhizopus oryzae , [95] el género Saccharomyces , [96] etc.
Además, la poliploidía se asocia frecuentemente con la hibridación y la evolución reticular, que parecen ser muy frecuentes en varios taxones de hongos. De hecho, se ha evidenciado la especiación homoploide (especiación híbrida sin cambio en el número de cromosomas ) en algunas especies de hongos (como el basidiomiceto Microbotryum violaceum [97] ).
En cuanto a las plantas y los animales, los híbridos fúngicos y poliploides muestran modificaciones estructurales y funcionales en comparación con sus progenitores y contrapartes diploides. En particular, los resultados estructurales y funcionales de los genomas poliploides de Saccharomyces reflejan sorprendentemente el destino evolutivo de los poliploides de las plantas. Se han descrito grandes reordenamientos cromosómicos [98] que conducen a cromosomas quiméricos [99] , así como modificaciones genéticas más puntuales como la pérdida de genes. [100] Los homoealelos de la levadura alotetraploide S. pastorianus muestran una contribución desigual al transcriptoma . [101] También se observa diversificación fenotípica después de la poliploidización y/o hibridación en hongos, [102] produciendo el combustible para la selección natural y la posterior adaptación y especiación.
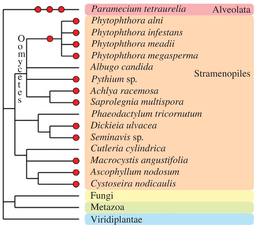
Cromalveolata
Otros taxones eucariotas han experimentado uno o más eventos de poliploidización durante su historia evolutiva (ver Albertin y Marullo, 2012 [89] para revisión). Los oomicetos , que no son miembros verdaderos de los hongos, contienen varios ejemplos de especies paleopoliploides y poliploides, como dentro del género Phytophthora . [103] Algunas especies de algas pardas ( Fucales , Laminariales [104] y diatomeas [105] ) contienen genomas poliploides aparentes. En el grupo Alveolata , la notable especie Paramecium tetraurelia experimentó tres rondas sucesivas de duplicación de todo el genoma [106] y se estableció como un modelo principal para los estudios paleopoliploides.
Bacteria
Cada bacteria Deinococcus radiodurans contiene de 4 a 8 copias de su cromosoma . [107] La exposición de D. radiodurans a la irradiación con rayos X o a la desecación puede romper sus genomas en cientos de fragmentos cortos aleatorios. Sin embargo, D. radiodurans es muy resistente a tales exposiciones. El mecanismo por el cual el genoma se restaura con precisión implica la recombinación homóloga mediada por RecA y un proceso conocido como recocido de cadena dependiente de síntesis extendida (SDSA) . [108]
Azotobacter vinelandii puede contener hasta 80 copias de cromosomas por célula. [109] Sin embargo, esto solo se observa en cultivos de rápido crecimiento, mientras que los cultivos cultivados en medios mínimos sintéticos no son poliploides. [110]
Arqueas
La arqueona Halobacterium salinarium es poliploide [111] y, al igual que Deinococcus radiodurans , es muy resistente a la irradiación con rayos X y a la desecación, condiciones que inducen la rotura de la doble cadena de ADN . [112] Aunque los cromosomas se rompen en muchos fragmentos, es posible regenerar cromosomas completos haciendo uso de fragmentos superpuestos. El mecanismo emplea una proteína de unión al ADN monocatenario y es probable que sea una reparación recombinatoria homóloga . [113]
Véase también
- Diploidización
- Genoma híbrido eucariota
- Ploidía
- Complejo poliploide
- Polisomía
- Silenciamiento recíproco
- Simpatía
Referencias
- ^ Solomon E (2014). Solomon/Martin/Martin/Berg, Biología . Cengage Learning. pág. 344. ISBN 978-1285423586.
- ^ Zhang S, Lin YH, Tarlow B, Zhu H (junio de 2019). "Los orígenes y funciones de la poliploidía hepática". Ciclo celular . 18 (12): 1302–1315. doi :10.1080/15384101.2019.1618123. PMC 6592246 . PMID 31096847.
- ^ Ohno S, Muramoto J, Christian L, Atkin NB (1967). "Relación diploide-tetraploide entre miembros del viejo mundo de la familia de peces Cyprinidae". Chromosoma . 23 (1): 1–9. doi :10.1007/BF00293307. S2CID 1181521.
- ^
- Manimekalai R, Suresh G, Govinda Kurup H, Athiappan S, Kandalam M (septiembre de 2020). "El papel de los métodos de genotipado NGS y SNP en los programas de mejoramiento de la caña de azúcar". Critical Reviews in Biotechnology . 40 (6). Taylor & Francis (T&F): 865–880. doi :10.1080/07388551.2020.1765730. PMID 32508157. S2CID 219537026.
- Esta reseña cita este estudio:
- Vilela MM, Del Bem LE, Van Sluys MA, de Setta N, Kitajima JP, Cruz GM, et al. (febrero de 2017). "El análisis de tres regiones homólogas/homólogas de la caña de azúcar sugiere eventos de poliploidización independientes de Saccharum officinarum y Saccharum spontaneum". Genome Biology and Evolution . 9 (2): 266–278. doi :10.1093/gbe/evw293. PMC 5381655 . PMID 28082603.
- ^ Vallejo-Marín M, Buggs RJ, Cooley AM, Puzey JR (junio de 2015). "Especiación por duplicación del genoma: Orígenes repetidos y composición genómica de la especie alopoliploide recientemente formada Mimulus peregrinus". Evolución; Revista Internacional de Evolución Orgánica . 69 (6): 1487–1500. doi :10.1111/evo.12678. PMC 5033005 . PMID 25929999.
- ^ Fessenden M. "Make Room for a New Bloom: New Flower Discovered" (Hagamos espacio para una nueva floración: se descubre una nueva flor). Scientific American . Consultado el 22 de febrero de 2017 .
- ^ Schmickl, Roswitha; Yant, Levi (abril de 2021). "Introgresión adaptativa: cómo la poliploidía reconfigura los paisajes del flujo genético". New Phytologist . 230 (2): 457–461. doi : 10.1111/nph.17204 . ISSN 0028-646X. PMID 33454987.
- ^ Marburger, Sarah; Monnahan, Patrick; Seear, Paul J.; Martin, Simon H.; Koch, Jordan; Paajanen, Pirita; Bohutínská, Magdalena; Higgins, James D.; Schmickl, Roswitha; Yant, Levi (18 de noviembre de 2019). "La introgresión interespecífica media la adaptación a la duplicación del genoma completo". Nature Communications . 10 (1): 5218. Bibcode :2019NatCo..10.5218M. doi :10.1038/s41467-019-13159-5. ISSN 2041-1723. PMC 6861236 . PMID 31740675.
- ^ Seear, Paul J.; France, Martin G.; Gregory, Catherine L.; Heavens, Darren; Schmickl, Roswitha; Yant, Levi; Higgins, James D. (15 de julio de 2020). "Un nuevo alelo de ASY3 se asocia con una mayor estabilidad meiótica en la Arabidopsis lyrata autotetraploide". PLOS Genetics . 16 (7): e1008900. doi : 10.1371/journal.pgen.1008900 . ISSN 1553-7404. PMC 7392332 . PMID 32667955.
- ^ Schmickl, Roswitha; Yant, Levi (abril de 2021). "Introgresión adaptativa: cómo la poliploidía reconfigura los paisajes del flujo genético". New Phytologist . 230 (2): 457–461. doi : 10.1111/nph.17204 . ISSN 0028-646X. PMID 33454987.
- ^ Bertolani R (2001). "Evolución de los mecanismos reproductivos en tardígrados: una revisión". Zoologischer Anzeiger . 240 (3–4): 247–252. Código Bibliográfico :2001ZooAn.240..247B. doi :10.1078/0044-5231-00032.
- ^ Stouder DJ, Bisson PA, Naiman RJ (1997). Salmón del Pacífico y sus ecosistemas: situación y opciones futuras. Springer. págs. 30-31. ISBN 978-0-412-98691-8. Recuperado el 9 de julio de 2013 .
- ^ Adams KL, Wendel JF (abril de 2005). "Poliploidía y evolución del genoma en plantas". Current Opinion in Plant Biology . 8 (2): 135–141. Bibcode :2005COPB....8..135A. doi :10.1016/j.pbi.2005.01.001. PMID 15752992.
- ^ "Triticum - una descripción general | Temas de ScienceDirect".
- ^ ab Crowhurst RN, Whittaker D, Gardner RC. "El origen genético del kiwi". Archivado desde el original el 25 de febrero de 2021. Consultado el 30 de agosto de 2008 .
- ^ Havelka, Miloš; Bytyutskyy, Dmytro; Symonová, Radka; Ráb, Petr; Flajšhans, Martin (11 de febrero de 2016). "El segundo recuento cromosómico más alto entre los vertebrados se observa en el esturión cultivado y está asociado con la plasticidad del genoma". Genética Selección Evolución . doi : 10.1186/s12711-016-0194-0 . PMC 4751722 . Consultado el 15 de septiembre de 2024 .
- ^ Aïnouche ML, Fortune PM, Salmon A, Parisod C, Grandbastien MA, Fukunaga K, et al. (2008). "Hibridación, poliploidía e invasión: lecciones de Spartina (Poaceae)". Invasiones biológicas . 11 (5): 1159–1173. doi :10.1007/s10530-008-9383-2. S2CID 25522023.
- ^
- Hussain F, Rana Z, Shafique H, Malik A, Hussain Z (2017). "Potencial fitofarmacológico de diferentes especies de Morus alba y sus fitoquímicos bioactivos: una revisión". Revista de biomedicina tropical del Pacífico asiático . 7 (10). Medknow : 950–956. doi : 10.1016/j.apjtb.2017.09.015 . ISSN 2221-1691.
- Al-Khayri JM, Jain SM, Johnson DV (2018). Al-Khayri JM, Jain SM, Johnson DV (eds.). Avances en estrategias de mejoramiento vegetal: frutas . Vol. 2. Springer International Publishing AG . págs. 89–130. doi :10.1007/978-3-319-91944-7. ISBN . 978-3-319-91943-0. Número de identificación del sujeto 51706250.
- Esta reseña y libro citan esta investigación.
- Zeng Q, Chen H, Zhang C, Han M, Li T, Qi X, et al. (2015). "Definición de ocho especies de morera en el género Morus mediante filogenia basada en espaciadores transcritos internos". PLOS ONE . 10 (8): e0135411. Bibcode :2015PLoSO..1035411Z. doi : 10.1371/journal.pone.0135411 . PMC 4534381 . PMID 26266951.
- ^ Soltis DE (1 de enero de 1984). "Autopoliploidía en Tolmiea menziesii (Saxifragaceae)". Revista americana de botánica . 71 (9): 1171-1174. doi :10.2307/2443640. JSTOR 2443640.
- ^ Drauch Schreier A, Gille D, Mahardja B, May B (noviembre de 2011). "Los marcadores neutrales confirman el origen octoploide y revelan autopoliploidía espontánea en el esturión blanco, Acipenser transmontanus". Revista de Ictiología Aplicada . 27 : 24–33. Bibcode :2011JApIc..27...24D. doi : 10.1111/j.1439-0426.2011.01873.x . ISSN 1439-0426.
- ^ ab Bretagnolle F, Thompson JD (enero de 1995). "Gametos con el número de cromosomas somáticos: mecanismos de su formación y papel en la evolución de plantas autopoliploides". The New Phytologist . 129 (1): 1–22. doi : 10.1111/j.1469-8137.1995.tb03005.x . PMID 33874422.
- ^ Müntzing A (marzo de 1936). "El significado evolutivo de la autopoliploidía". Hereditas . 21 (2–3): 363–378. doi :10.1111/j.1601-5223.1936.tb03204.x. ISSN 1601-5223.
- ^ Varoquaux F, Blanvillain R, Delseny M, Gallois P (junio de 2000). "Menos es mejor: nuevos enfoques para la producción de fruta sin semillas". Tendencias en biotecnología . 18 (6): 233–242. doi :10.1016/s0167-7799(00)01448-7. PMID 10802558. S2CID 29713625.
- ^ Cotter D, O'Donovan V, O'Maoiléidigh N, Rogan G, Roche N, Wilkins NP (junio de 2000). "Una evaluación del uso del salmón triploide del Atlántico ( Salmo salar L.) para minimizar el impacto del salmón de piscifactoría escapado en las poblaciones silvestres". Acuicultura . 186 (1–2): 61–75. Bibcode :2000Aquac.186...61C. doi :10.1016/S0044-8486(99)00367-1.
- ^ Lincoln RF, Scott AP (1983). "Producción de trucha arcoíris triploide exclusivamente hembra". Acuicultura . 30 (1–4): 375–380. Código Bibliográfico :1983Aquac..30..375L. doi :10.1016/0044-8486(83)90179-5.
- ^ Dermen H (mayo de 1951). "Brotes adventicios tetraploides y diploides: de una especie gigante de manzana McIntosh". Journal of Heredity . 42 (3): 145–149. doi :10.1093/oxfordjournals.jhered.a106189. ISSN 0022-1503.
- ^ Dwivedi SL, Upadhyaya HD, Stalker HT, Blair MW, Bertioli DJ, Nielen S, Ortiz R (enero de 2007). Janick J (ed.). Plant Breeding Reviews (PDF) . John Wiley & Sons. págs. 179–230. doi :10.1002/9780470380130.ch3. ISBN. 9780470380130.
- ^ ab Justin R (enero de 2002). "Neopoliploidía en plantas con flores". Revista anual de ecología y sistemática . 33 (1): 589–639. doi :10.1146/annurev.ecolsys.33.010802.150437. S2CID 41689384.
- ^ Yant, Levi; Hollister, Jesse D.; Wright, Kevin M.; Arnold, Brian J.; Higgins, James D.; Franklin, F. Chris H.; Bomblies, Kirsten (noviembre de 2013). "Adaptación meiótica a la duplicación del genoma en Arabidopsis arenosa". Current Biology . 23 (21): 2151–2156. Bibcode :2013CBio...23.2151Y. doi :10.1016/j.cub.2013.08.059. ISSN 0960-9822. PMC 3859316 . PMID 24139735.
- ^ Marburger, Sarah; Monnahan, Patrick; Seear, Paul J.; Martin, Simon H.; Koch, Jordan; Paajanen, Pirita; Bohutínská, Magdalena; Higgins, James D.; Schmickl, Roswitha; Yant, Levi (18 de noviembre de 2019). "La introgresión interespecífica media la adaptación a la duplicación del genoma completo". Nature Communications . 10 (1): 5218. Bibcode :2019NatCo..10.5218M. doi :10.1038/s41467-019-13159-5. ISSN 2041-1723. PMC 6861236 . PMID 31740675.
- ^ Marburger, Sarah; Monnahan, Patrick; Seear, Paul J.; Martin, Simon H.; Koch, Jordan; Paajanen, Pirita; Bohutínská, Magdalena; Higgins, James D.; Schmickl, Roswitha; Yant, Levi (18 de noviembre de 2019). "La introgresión interespecífica media la adaptación a la duplicación del genoma completo". Nature Communications . 10 (1): 5218. Bibcode :2019NatCo..10.5218M. doi :10.1038/s41467-019-13159-5. ISSN 2041-1723. PMC 6861236 . PMID 31740675.
- ^ Seear, Paul J.; France, Martin G.; Gregory, Catherine L.; Heavens, Darren; Schmickl, Roswitha; Yant, Levi; Higgins, James D. (15 de julio de 2020). Grelon, Mathilde (ed.). "Un nuevo alelo de ASY3 se asocia con una mayor estabilidad meiótica en la Arabidopsis lyrata autotetraploide". PLOS Genetics . 16 (7): e1008900. doi : 10.1371/journal.pgen.1008900 . ISSN 1553-7404. PMC 7392332 . PMID 32667955.
- ^ Parisod C, Holderegger R, Brochmann C (abril de 2010). "Consecuencias evolutivas de la autopoliploidía". The New Phytologist . 186 (1): 5–17. doi :10.1111/j.1469-8137.2009.03142.x. PMID 20070540.
- ^ ab Le Comber SC, Ainouche ML, Kovarik A, Leitch AR (abril de 2010). "Creación de un diploide funcional: de la herencia polisómica a la disómica". The New Phytologist . 186 (1): 113–122. doi :10.1111/j.1469-8137.2009.03117.x. PMID 20028473.
- ^ Stebbins GL (1947). Tipos de poliploides: su clasificación y significado . Avances en genética. Vol. 1. págs. 403–429. doi :10.1016/s0065-2660(08)60490-3. ISBN 9780120176014. Número de identificación personal 20259289.
- ^ Stebbins GL (1950). Variación y evolución en plantas . Oxford University Press.[ página necesaria ]
- ^ Ramsey J, Schemske DW (enero de 1998). "Vías, mecanismos y tasas de formación de poliploides en plantas con flores". Revista anual de ecología y sistemática . 29 (1): 467–501. doi :10.1146/annurev.ecolsys.29.1.467. S2CID 31637733.
- ^ Barker MS, Arrigo N, Baniaga AE, Li Z, Levin DA (abril de 2016). "Sobre la abundancia relativa de autopoliploides y alopoliploides". The New Phytologist . 210 (2): 391–398. doi : 10.1111/nph.13698 . PMID 26439879.
- ^ Doyle JJ, Sherman-Broyles S (enero de 2017). "Doble problema: taxonomía y definiciones de poliploidía". The New Phytologist . 213 (2): 487–493. doi : 10.1111/nph.14276 . PMID 28000935.
- ^ ab Ramsey J (enero de 1998). "Vías, mecanismos y tasas de formación de poliploides en plantas con flores". Revista anual de ecología y sistemática . 29 (1): 467–501. doi :10.1146/annurev.ecolsys.29.1.467. S2CID 31637733.
- ^ abc Comai L (noviembre de 2005). "Las ventajas y desventajas de ser poliploide". Nature Reviews. Genetics . 6 (11): 836–846. doi :10.1038/nrg1711. PMID 16304599. S2CID 3329282.
- ^ Ownbey M (enero de 1950). "Hibridación natural y anfiploidía en el género Tragopogon". American Journal of Botany . 37 (7): 487–499. doi :10.2307/2438023. JSTOR 2438023.
- ^ Schmid M, Evans BJ, Bogart JP (2015). "Poliploidía en anfibios". Investigación citogenética y genómica . 145 (3–4): 315–330. doi : 10.1159/000431388 . PMID: 26112701.
- ^ Griffiths AJ (1999). Introducción al análisis genético . San Francisco, CA: WH Freeman. ISBN 978-0-7167-3520-5.[ página necesaria ]
- ^ Parmacek MS, Epstein JA (julio de 2009). "Renovación de cardiomiocitos". The New England Journal of Medicine . 361 (1): 86–88. doi :10.1056/NEJMcibr0903347. PMC 4111249 . PMID 19571289.
- ^ Mendell JE, Clements KD, Choat JH, Angert ER (mayo de 2008). "Poliploidía extrema en una bacteria grande". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (18): 6730–6734. Bibcode :2008PNAS..105.6730M. doi : 10.1073/pnas.0707522105 . PMC 2373351 . PMID 18445653.
- ^ Edger PP, Pires JC (2009). "Duplicaciones de genes y genomas: el impacto de la sensibilidad a la dosis en el destino de los genes nucleares". Investigación cromosómica . 17 (5): 699–717. doi : 10.1007/s10577-009-9055-9 . PMID 19802709.
- ^ ab Clarke JT, Lloyd GT, Friedman M (octubre de 2016). "Poca evidencia de una evolución fenotípica mejorada en los primeros teleósteos en relación con su grupo hermano fósil viviente". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 113 (41): 11531–11536. Bibcode :2016PNAS..11311531C. doi : 10.1073/pnas.1607237113 . PMC 5068283 . PMID 27671652.
- ^ White MJ (1973). Los cromosomas (6.ª ed.). Londres: Chapman & Hall. pág. 28.
- ^ Stebbins GL (1950). "Capítulo XII: El cariotipo". Variación y evolución en plantas . Nueva York, NY: Columbia University Press.[ página necesaria ]
- ^ Otto SP, Whitton J (2000). "Incidencia y evolución de poliploides". Revisión anual de genética . 34 (1): 401–437. CiteSeerX 10.1.1.323.1059 . doi :10.1146/annurev.genet.34.1.401. PMID 11092833.
- ^ ab Smith LE (octubre de 2012). "Una sugerencia para los bibliotecarios médicos. 1920". Revista de la Asociación de Bibliotecarios Médicos . 100 (4 Suppl): B. doi :10.1023/B:RFBF.0000033049.00668.fe. PMC 3571666. PMID 23509424. S2CID 11649126 .
- ^ Cannatella DC, De Sa RO (1993). " Xenopus laevis como organismo modelo". Sociedad de Biólogos Sistemáticos . 42 (4): 476–507. doi :10.1093/sysbio/42.4.476.
- ^ Bogart JP, Bi K, Fu J, Noble DW, Niedzwiecki J (febrero de 2007). "Las salamandras unisexuales (género Ambystoma) presentan un nuevo modo reproductivo para los eucariotas". Genoma . 50 (2): 119–136. doi :10.1139/g06-152. PMID 17546077. S2CID 42528911.
- ^ Epstein CJ (febrero de 1967). "Tamaño celular, contenido nuclear y desarrollo de poliploidía en el hígado de mamíferos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 57 (2): 327–334. Bibcode :1967PNAS...57..327E. doi : 10.1073/pnas.57.2.327 . PMC 335509 . PMID 16591473.
- ^ Donne R, Saroul-Aïnama M, Cordier P, Celton-Morizur S, Desdouets C (julio de 2020). "Poliploidía en el desarrollo, homeostasis y enfermedad del hígado" (PDF) . Nature Reviews. Gastroenterología y hepatología . 17 (7): 391–405. doi :10.1038/s41575-020-0284-x. PMID 32242122. S2CID 214766734.
- ↑ Gallardo MH, González CA, Cebrián I (agosto de 2006). "Citogenética molecular y alotetraploidía en la rata vizcacha roja, Tympanoctomys barrerae (Rodentia, Octodontidae)". Genómica . 88 (2): 214–221. doi : 10.1016/j.ygeno.2006.02.010 . PMID 16580173.
- ^ Svartman M, Stone G, Stanyon R (abril de 2005). "La citogenética molecular descarta la poliploidía en mamíferos". Genomics . 85 (4): 425–430. doi :10.1016/j.ygeno.2004.12.004. PMID 15780745.
- ^ Swarup H (1956). "Producción de heteroploidía en el espinoso de tres espinas, Gasterosteus aculeatus (L.)". Nature . 178 (4542): 1124–1125. Código Bibliográfico :1956Natur.178.1124S. doi :10.1038/1781124a0. S2CID 4226624.
- ^ Swarup H (1959). "Producción de triploidía en Gasterosteus aculeatus (L.)". Revista de genética . 56 (2): 129–142. doi :10.1007/BF02984740. S2CID 42913498.
- ^ Mable BK, Alexandrou MA, Taylor MI (2011). "Duplicación del genoma en anfibios y peces: una síntesis extendida". Revista de zoología . 284 (3): 151–182. doi : 10.1111/j.1469-7998.2011.00829.x . S2CID 58937352.
- ^ ab "Premio Nobel de Fisiología o Medicina 2012 otorgado por el descubrimiento de que las células maduras pueden reprogramarse para convertirse en pluripotentes". ScienceDaily . 8 de octubre de 2012.
- ^ Velicky P, Meinhardt G, Plessl K, Vondra S, Weiss T, Haslinger P, et al. (octubre de 2018). "La amplificación del genoma y la senescencia celular son características del desarrollo de la placenta humana". PLOS Genetics . 14 (10): e1007698. doi : 10.1371/journal.pgen.1007698 . PMC 6200260 . PMID 30312291.
- ^ Winkelmann M, Pfitzer P, Schneider W (diciembre de 1987). "Importancia de la poliploidía en megacariocitos y otras células en la salud y las enfermedades tumorales". Klinische Wochenschrift . 65 (23): 1115-1131. doi :10.1007/BF01734832. PMID 3323647. S2CID 23496028.
- ^ "Triploidía". Organización Nacional de Enfermedades Raras . Consultado el 23 de diciembre de 2018 .
- ^ ab Baker P, Monga A, Baker P (2006). Ginecología por diez maestros . Londres: Arnold. ISBN 978-0-340-81662-2.
- ^ Brancati F, Mingarelli R, Dallapiccola B (diciembre de 2003). "Triploidía recurrente de origen materno". Revista europea de genética humana . 11 (12): 972–974. doi : 10.1038/sj.ejhg.5201076 . PMID 14508508. S2CID 28009278.
- ^ Wick JB, Johnson KJ, O'Brien J, Wick MJ (mayo de 2013). "Diagnóstico de triploidía en el segundo trimestre: una serie de cuatro casos". AJP Reports . 3 (1): 37–40. doi :10.1055/s-0032-1331378. PMC 3699153 . PMID 23943708.
- ^ Meyers LA, Levin DA (junio de 2006). "Sobre la abundancia de poliploides en plantas con flores". Evolución; Revista internacional de evolución orgánica . 60 (6): 1198–1206. doi : 10.1111/j.0014-3820.2006.tb01198.x . PMID 16892970.
- ^ ab Rieseberg LH, Willis JH (agosto de 2007). "Especiación de plantas". Science . 317 (5840): 910–914. Bibcode :2007Sci...317..910R. doi :10.1126/science.1137729. PMC 2442920 . PMID 17702935.
- ^ Otto SP (noviembre de 2007). "Las consecuencias evolutivas de la poliploidía". Cell . 131 (3): 452–462. doi : 10.1016/j.cell.2007.10.022 . PMID 17981114. S2CID 10054182.
- ^ Iniciativa de Mil Transcriptomas Vegetales (octubre de 2019). «Mil transcriptomas vegetales y la filogenómica de las plantas verdes». Nature . 574 (7780): 679–685. doi :10.1038/s41586-019-1693-2. PMC 6872490 . PMID 31645766.
- ^ De Bodt S, Maere S, Van de Peer Y (noviembre de 2005). "Duplicación del genoma y el origen de las angiospermas". Tendencias en ecología y evolución . 20 (11): 591–597. doi :10.1016/j.tree.2005.07.008. PMID 16701441.
- ^ Wood TE, Takebayashi N, Barker MS, Mayrose I, Greenspoon PB, Rieseberg LH (agosto de 2009). "La frecuencia de especiación poliploide en plantas vasculares". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 106 (33): 13875–13879. Bibcode :2009PNAS..10613875W. doi : 10.1073/pnas.0811575106 . JSTOR 40484335. PMC 2728988 . PMID 19667210.
- ^ Xu X, Pan S, Cheng S, Zhang B, Mu D, Ni P, et al. (julio de 2011). "Secuencia genómica y análisis del tubérculo de la patata". Nature . 475 (7355): 189–195. doi : 10.1038/nature10158 . PMID 21743474.
- ^ Osborn TC, Pires JC, Birchler JA, Auger DL, Chen ZJ, Lee HS, et al. (marzo de 2003). "Comprensión de los mecanismos de expresión de genes novedosos en poliploides". Tendencias en genética . 19 (3): 141–147. doi :10.1016/S0168-9525(03)00015-5. PMID 12615008.
- ^ Chen ZJ, Ni Z (marzo de 2006). "Mecanismos de reordenamientos genómicos y cambios en la expresión génica en poliploides de plantas". BioEssays . 28 (3): 240–252. doi :10.1002/bies.20374. PMC 1986666 . PMID 16479580.
- ^ Chen ZJ (2007). "Mecanismos genéticos y epigenéticos para la expresión génica y la variación fenotípica en poliploides de plantas". Revisión anual de biología vegetal . 58 : 377–406. doi :10.1146/annurev.arplant.58.032806.103835. PMC 1949485. PMID 17280525 .
- ^ Albertin W, Balliau T, Brabant P, Chèvre AM, Eber F, Malosse C, Thiellement H (junio de 2006). "Numerosas y rápidas modificaciones no estocásticas de productos genéticos en alotetraploides de Brassica napus recién sintetizados". Genética . 173 (2): 1101–1113. doi :10.1534/genetics.106.057554. PMC 1526534 . PMID 16624896.
- ^ Pennington PD, Costa LM, Gutierrez-Marcos JF, Greenland AJ, Dickinson HG (abril de 2008). "Cuando los genomas colisionan: desarrollo aberrante de semillas luego de cruces interploidía en maíz". Anales de Botánica . 101 (6): 833–843. doi :10.1093/aob/mcn017. PMC 2710208 . PMID 18276791.
- ^ von Wangenheim KH, Peterson HP (junio de 2004). "El desarrollo aberrante del endospermo en cruces interploidía revela un cronómetro de la diferenciación". Biología del desarrollo . 270 (2): 277–289. doi :10.1016/j.ydbio.2004.03.014. PMID 15183714.
- ^ Whitton J, Sears CJ, Maddison WP (diciembre de 2017). "La coexistencia de linajes asexuales, pero no sexuales, relacionados sugiere que la interferencia reproductiva limita la coexistencia". Actas. Ciencias biológicas . 284 (1868): 20171579. doi :10.1098/rspb.2017.1579. PMC 5740271. PMID 29212720 .
- ^ Halabi K, Shafir A, Mayrose I (junio de 2023). "PloiDB: La base de datos de ploidía de las plantas". The New Phytologist . 240 (3): 918–927. doi : 10.1111/nph.19057 . PMID 37337836.
- ^ Ahuja MR, Neale DB (2002). "Orígenes de la poliploidía en la sequoia costera ( Sequoia sempervirens (D. Don) Endl.) y relación de la sequoia costera con otros géneros de Taxodiaceae". Silvae Genetica . 51 : 2–3.
- ^ Les DH, Philbrick CT (1993). "Estudios de hibridación y variación del número de cromosomas en angiospermas acuáticas: implicaciones evolutivas". Botánica acuática . 44 (2–3): 181–228. Bibcode :1993AqBot..44..181L. doi :10.1016/0304-3770(93)90071-4.
- ^ "Las frutas sin semillas hacen que otras sean innecesarias". Archivado desde el original el 2019-02-03 . Consultado el 2022-03-07 .
- ^ Emshwiller E (2006). "Orígenes de los cultivos poliploides: el ejemplo del cultivo de tubérculos octaploides Oxalis tuberosa ". En Zeder MA, Decker-Walters D, Emshwiller E, Bradley D, Smith BD (eds.). Documentación de la domesticación: nuevos paradigmas genéticos y arqueológicos . Berkeley, CA: University of California Press.
- ^ Le Cunff L, Garsmeur O, Raboin LM, Pauquet J, Telismart H, Selvi A, et al. (septiembre de 2008). "Mapeo de lanzadera sinténica diploide/poliploide y desplazamiento cromosómico específico de haplotipo hacia un gen de resistencia a la roya (Bru1) en caña de azúcar altamente poliploide (2n aproximadamente 12x aproximadamente 115)". Genética . 180 (1): 649–660. doi :10.1534/genetics.108.091355. PMC 2535714 . PMID 18757946.
- ^ abc Albertin W, Marullo P (julio de 2012). "Poliploidía en hongos: evolución después de la duplicación del genoma completo". Actas. Ciencias Biológicas . 279 (1738): 2497–2509. doi :10.1098/rspb.2012.0434. PMC 3350714. PMID 22492065 .
- ^ Emerson R, Wilson CM (1954). "Híbridos interespecíficos y citogenética y citotaxonomía de Euallomyces". Mycologia . 46 (4): 393–434. doi :10.1080/00275514.1954.12024382. JSTOR 4547843.
- ^ Albertin W, Marullo P, Aigle M, Bourgais A, Bely M, Dillmann C, et al. (noviembre de 2009). "Evidencia de autotetraploidía asociada con aislamiento reproductivo en Saccharomyces cerevisiae: hacia una nueva especie domesticada". Journal of Evolutionary Biology . 22 (11): 2157–2170. doi : 10.1111/j.1420-9101.2009.01828.x . PMID 19765175.
- ^ Lu BC (1964). "Poliploidía en el basidiomiceto Cyathus stercoreus ". American Journal of Botany . 51 (3): 343–347. doi :10.2307/2440307. JSTOR 2440307.
- ^ Libkind D, Hittinger CT, Valério E, Gonçalves C, Dover J, Johnston M, et al. (agosto de 2011). "Domesticación de microbios e identificación del stock genético silvestre de levadura para elaboración de cerveza". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (35): 14539–14544. Bibcode :2011PNAS..10814539L. doi : 10.1073/pnas.1105430108 . PMC 3167505 . PMID 21873232.
- ^ Borneman AR, Zeppel R, Chambers PJ, Curtin CD (febrero de 2014). "Información sobre el panorama genómico de Dekkera bruxellensis: la genómica comparativa revela variaciones en la ploidía y el potencial de utilización de nutrientes entre los aislados del vino". PLOS Genetics . 10 (2): e1004161. doi : 10.1371/journal.pgen.1004161 . PMC 3923673 . PMID 24550744.
- ^ Ma LJ, Ibrahim AS, Skory C, Grabherr MG, Burger G, Butler M, et al. (julio de 2009). Madhani HD (ed.). "El análisis genómico del hongo de linaje basal Rhizopus oryzae revela una duplicación del genoma completo". PLOS Genetics . 5 (7): e1000549. doi : 10.1371/journal.pgen.1000549 . PMC 2699053 . PMID 19578406.
- ^ Wong S, Butler G, Wolfe KH (julio de 2002). "Gene order evolution and paleopolyploidy in hemiascomycete yeasts" (Evolución del orden genético y paleopoliploidía en levaduras hemiascomicetas). Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (14): 9272–9277. Bibcode :2002PNAS...99.9272W. doi : 10.1073/pnas.142101099 . JSTOR 3059188. PMC 123130 . PMID 12093907.
- ^ Devier B, Aguileta G, Hood ME, Giraud T (2009). "Uso de filogenias de genes de receptores de feromonas en el complejo de especies Microbotryum violaceum para investigar la posible especiación por hibridación". Mycologia . 102 (3): 689–696. doi :10.3852/09-192. PMID 20524600. S2CID 9072505.
- ^ Dunn B, Sherlock G (octubre de 2008). "Reconstrucción de los orígenes del genoma y evolución de la levadura lager híbrida Saccharomyces pastorianus". Genome Research . 18 (10): 1610–1623. doi :10.1101/gr.076075.108. PMC 2556262 . PMID 18787083.
- ^ Nakao Y, Kanamori T, Itoh T, Kodama Y, Rainieri S, Nakamura N, et al. (Abril de 2009). "Secuencia del genoma de la levadura cervecera lager, un híbrido entre especies". Investigación del ADN . 16 (2): 115-129. doi :10.1093/dnares/dsp003. PMC 2673734 . PMID 19261625.
- ^ Scannell DR, Byrne KP, Gordon JL, Wong S, Wolfe KH (marzo de 2006). "Múltiples rondas de especiación asociadas con pérdida recíproca de genes en levaduras poliploides". Nature . 440 (7082): 341–345. Bibcode :2006Natur.440..341S. doi :10.1038/nature04562. hdl : 2262/22660 . PMID 16541074. S2CID 94448.
- ^ Minato T, Yoshida S, Ishiguro T, Shimada E, Mizutani S, Kobayashi O, Yoshimoto H (marzo de 2009). "Perfiles de expresión de genes ortólogos de la levadura de fermentación baja Saccharomyces pastorianus utilizando microarreglos de oligonucleótidos". Levadura . 26 (3): 147–165. doi :10.1002/yea.1654. PMID 19243081. S2CID 23349682.
- ^ Lidzbarsky GA, Shkolnik T, Nevo E (junio de 2009). Idnurm A (ed.). "Respuesta adaptativa a agentes que dañan el ADN en poblaciones naturales de Saccharomyces cerevisiae de "Evolution Canyon", Mt. Carmel, Israel". PLOS ONE . 4 (6): e5914. Bibcode :2009PLoSO...4.5914L. doi : 10.1371/journal.pone.0005914 . PMC 2690839 . PMID 19526052.
- ^ Ioos R, Andrieux A, Marçais B, Frey P (julio de 2006). "Caracterización genética de la especie híbrida natural Phytophthora alni inferida a partir de análisis de ADN nuclear y mitocondrial" (PDF) . Genética y biología de hongos . 43 (7): 511–529. doi :10.1016/j.fgb.2006.02.006. PMID 16626980. S2CID 43275127.
- ^ Phillips N, Kapraun DF, Gómez Garreta A, Ribera Siguan MA, Rull JL, Salvador Soler N, et al. (2011). "Estimaciones del contenido de ADN nuclear en 98 especies de algas pardas (Phaeophyta)". AoB Plants . 2011 : plr001. doi :10.1093/aobpla/plr001. PMC 3064507 . PMID 22476472.
- ^ Chepurnov VA, Mann DG, Vyverman W, Sabbe K, Danielidis DB (2002). "Reproducción sexual, sistema de apareamiento y dinámica de protoplastos de Seminavis (Bacillariophyceae)". Revista de fisiología . 38 (5): 1004–1019. Código Bibliográfico :2002JPcgy..38.1004C. doi :10.1046/j.1529-8817.2002.t01-1-01233.x. S2CID 84299819.
- ^ Aury JM, Jaillon O, Duret L, Noel B, Jubin C, Porcel BM, et al. (noviembre de 2006). "Tendencias globales de duplicaciones de todo el genoma reveladas por el ciliado Paramecium tetraurelia". Nature . 444 (7116): 171–178. Bibcode :2006Natur.444..171A. doi : 10.1038/nature05230 . PMID 17086204.
- ^ Hansen MT (abril de 1978). "Multiplicidad de equivalentes genómicos en la bacteria resistente a la radiación Micrococcus radiodurans". Journal of Bacteriology . 134 (1): 71–75. doi :10.1128/JB.134.1.71-75.1978. PMC 222219 . PMID 649572.
- ^ Zahradka K, Slade D, Bailone A, Sommer S, Averbeck D, Petranovic M, et al. (octubre de 2006). "Reensamblaje de cromosomas destrozados en Deinococcus radiodurans". Nature . 443 (7111): 569–573. Bibcode :2006Natur.443..569Z. doi :10.1038/nature05160. PMID 17006450. S2CID 4412830.
- ^ Nagpal P, Jafri S, Reddy MA, Das HK (junio de 1989). "Cromosomas múltiples de Azotobacter vinelandii". Revista de bacteriología . 171 (6): 3133–3138. doi :10.1128/jb.171.6.3133-3138.1989. PMC 210026 . PMID 2785985.
- ^ Maldonado R, Jiménez J, Casadesús J (julio de 1994). "Cambios de ploidía durante el ciclo de crecimiento de Azotobacter vinelandii". Journal of Bacteriology . 176 (13): 3911–3919. doi :10.1128/jb.176.13.3911-3919.1994. PMC 205588 . PMID 8021173.
- ^ Soppa J (enero de 2011). "Ploidía y conversión génica en arqueas". Biochemical Society Transactions . 39 (1): 150–154. doi :10.1042/BST0390150. PMID 21265763. S2CID 31385928.
- ^ Kottemann M, Kish A, Iloanusi C, Bjork S, DiRuggiero J (junio de 2005). "Respuestas fisiológicas de la arqueona halófila Halobacterium sp. Cepa NRC1 a la desecación y la irradiación gamma" (PDF) . Extremófilos . 9 (3): 219–227. doi :10.1007/s00792-005-0437-4. PMID 15844015. S2CID 8391234.
- ^ DeVeaux LC, Müller JA, Smith J, Petrisko J, Wells DP, DasSarma S (octubre de 2007). "Mutantes extremadamente resistentes a la radiación de una arquea halófila con mayor expresión génica de la proteína de unión al ADN monocatenario (RPA)". Radiation Research . 168 (4): 507–514. Bibcode :2007RadR..168..507D. doi : 10.1667/RR0935.1 . PMID 17903038. S2CID 22393850.
Lectura adicional
- Snustad DP, Simmons MJ (2006). Principios de genética (4.ª ed.). Hoboken, Nueva Jersey: John Wiley & Sons. ISBN 978-0-471-69939-2.
- La Iniciativa Genómica de Arabidopsis (diciembre de 2000). «Análisis de la secuencia del genoma de la planta con flores Arabidopsis thaliana». Nature . 408 (6814): 796–815. Bibcode :2000Natur.408..796T. doi : 10.1038/35048692 . PMID 11130711.
- Eakin GS, Behringer RR (diciembre de 2003). "Desarrollo tetraploide en el ratón". Dinámica del desarrollo . 228 (4): 751–766. doi : 10.1002/dvdy.10363 . PMID 14648853.
- Gaeta RT, Pires JC, Iniguez-Luy F, Leon E, Osborn TC (noviembre de 2007). "Cambios genómicos en Brassica napus resintetizada y su efecto sobre la expresión génica y el fenotipo". The Plant Cell . 19 (11): 3403–3417. doi :10.1105/tpc.107.054346. PMC 2174891 . PMID 18024568.
- Gregory TR, Mable BK (2005). "Polyploidy in animals". En Gregory TR (ed.). The Evolution of the Genome . San Diego, California: Elsevier. págs. 427–517.
- Jaillon O, Aury JM, Brunet F, Petit JL, Stange-Thomann N, Mauceli E, et al. (octubre de 2004). "La duplicación del genoma en el pez teleósteo Tetraodon nigroviridis revela el protocariotipo vertebrado temprano". Nature . 431 (7011): 946–957. Bibcode :2004Natur.431..946J. doi : 10.1038/nature03025 . PMID 15496914.
- Paterson AH, Bowers JE, Van de Peer Y, Vandepoele K (marzo de 2005). "Duplicación antigua de genomas de cereales". The New Phytologist . 165 (3): 658–661. doi : 10.1111/j.1469-8137.2005.01347.x . PMID 15720677.
- Raes J, Vandepoele K, Simillion C, Saeys Y, Van de Peer Y (2003). "Investigación de eventos de duplicación antiguos en el genoma de Arabidopsis". Journal of Structural and Functional Genomics . 3 (1–4): 117–129. doi :10.1023/A:1022666020026. PMID 12836691. S2CID 9666357.
- Simillion C, Vandepoele K, Van Montagu MC, Zabeau M, Van de Peer Y (octubre de 2002). "El pasado de duplicación oculto de Arabidopsis thaliana". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 99 (21): 13627–13632. Bibcode :2002PNAS...9913627S. doi : 10.1073/pnas.212522399 . JSTOR 3073458. PMC 129725 . PMID 12374856.
- Soltis DE, Soltis PS , Schemske DW, Hancock JF, Thompson JN, Husband BC, Judd WS (2007). "Autopoliploidía en angiospermas: ¿Hemos subestimado enormemente el número de especies?". Taxon . 56 (1): 13–30. JSTOR 25065732.
- Soltis DE, Buggs RJ, Doyle JJ, Soltis PS (2010). "Lo que aún no sabemos sobre la poliploidía". Taxon . 59 (5): 1387–1403. doi :10.1002/tax.595006. JSTOR 20774036.
- Taylor JS, Braasch I, Frickey T, Meyer A, Van de Peer Y (marzo de 2003). "Duplicación del genoma, un rasgo compartido por 22.000 especies de peces con aletas radiadas". Genome Research . 13 (3): 382–390. doi :10.1101/gr.640303. PMC 430266 . PMID 12618368.
- Tate JA, Soltis DE, Soltis PS (2005). "Polyploidy in plants" (Poliploidía en plantas). En Gregory TR (ed.). The Evolution of the Genome (La evolución del genoma ). San Diego, California: Elsevier. pp. 371–426.
- Van de Peer Y, Taylor JS, Meyer A (2003). "¿Son todos los peces poliploides antiguos?". Journal of Structural and Functional Genomics . 3 (1–4): 65–73. doi :10.1023/A:1022652814749. PMID 12836686. S2CID 14092900.
- Van de Peer Y (2004). "El genoma del Tetraodon confirma los hallazgos de Takifugu: la mayoría de los peces son poliploides antiguos". Genome Biology . 5 (12): 250. doi : 10.1186/gb-2004-5-12-250 . PMC 545788 . PMID 15575976.
- Van de Peer Y, Meyer A (2005). "Duplicaciones de genes y genomas antiguos a gran escala". En Gregory TR (ed.). La evolución del genoma . San Diego, California: Elsevier. págs. 329–368.
- Wolfe KH, Shields DC (junio de 1997). "Evidencia molecular de una duplicación antigua de todo el genoma de la levadura". Nature . 387 (6634): 708–713. Bibcode :1997Natur.387..708W. doi : 10.1038/42711 . PMID 9192896. S2CID 4307263.
- Wolfe KH (mayo de 2001). "Los poliploides de ayer y el misterio de la diploidización". Nature Reviews. Genetics . 2 (5): 333–341. doi :10.1038/35072009. PMID 11331899. S2CID 20796914.
Enlaces externos
- La poliploidía en las páginas de biología de Kimball
- El portal de poliploidía es un proyecto editable por la comunidad con información, investigación, educación y una bibliografía sobre poliploidía.
