Haplogrupo
| Parte de una serie sobre |
| Genealogía genética |
|---|
| Conceptos |
| Temas relacionados |
Un haplotipo es un grupo de alelos en un organismo que se heredan juntos de un solo progenitor, [1] [2] y un haplogrupo ( haploide del griego : ἁπλοῦς , haploûs , "único, simple" y español: grupo ) es un grupo de haplotipos similares que comparten un ancestro común con una mutación de polimorfismo de un solo nucleótido . [3] Más específicamente, un haplotipo es una combinación de alelos en diferentes regiones cromosómicas que están estrechamente vinculados y que tienden a heredarse juntos. Como un haplogrupo consta de haplotipos similares, generalmente es posible predecir un haplogrupo a partir de haplotipos. Los haplogrupos pertenecen a una sola línea de descendencia . Como tal, la pertenencia a un haplogrupo, por parte de cualquier individuo, depende de una proporción relativamente pequeña del material genético que posee ese individuo.

Cada haplogrupo se origina a partir de un haplogrupo (o paragrupo ) anterior y sigue siendo parte de él . Por lo tanto, cualquier grupo relacionado de haplogrupos puede modelarse con precisión como una jerarquía anidada , en la que cada conjunto (haplogrupo) es también un subconjunto de un único conjunto más amplio (a diferencia, por supuesto, de los modelos biparentales, como los árboles genealógicos humanos). Los haplogrupos pueden dividirse a su vez en subclados.
Los haplogrupos se identifican normalmente con una letra inicial del alfabeto, y los refinamientos consisten en combinaciones adicionales de números y letras, como (por ejemplo) A → A1 → A1a . La nomenclatura alfabética fue publicada en 2002 por el Consorcio del Cromosoma Y. [ 4]
En genética humana , los haplogrupos que se estudian con más frecuencia son los haplogrupos del cromosoma Y (ADN-Y) y los haplogrupos del ADN mitocondrial (ADNmt) , cada uno de los cuales puede utilizarse para definir poblaciones genéticas . El ADN-Y se transmite únicamente a lo largo de la línea patrilineal , de padre a hijo, mientras que el ADNmt se transmite a lo largo de la línea matrilineal , de madre a descendencia de ambos sexos. Ninguno se recombina y, por lo tanto, el ADN-Y y el ADNmt cambian solo por mutación aleatoria en cada generación sin mezcla entre el material genético de los padres.
Formación de haplogrupos

Las mitocondrias son pequeños orgánulos que se encuentran en el citoplasma de las células eucariotas , como las de los humanos. Su función principal es proporcionar energía a la célula. Se cree que las mitocondrias son descendientes reducidos de bacterias simbióticas que alguna vez fueron de vida libre. Una indicación de que las mitocondrias alguna vez fueron de vida libre es que cada una contiene un ADN circular , llamado ADN mitocondrial (ADNmt), cuya estructura es más similar a la de las bacterias que a los organismos eucariotas (ver teoría endosimbiótica ). La abrumadora mayoría del ADN de un humano está contenido en los cromosomas en el núcleo de la célula, pero el ADNmt es una excepción. Un individuo hereda su citoplasma y los orgánulos contenidos en ese citoplasma exclusivamente del óvulo materno (célula del huevo); el esperma solo transmite el ADN cromosómico, todas las mitocondrias paternas se digieren en el ovocito . Cuando surge una mutación en una molécula de ADNmt, la mutación se transmite en una línea de descendencia femenina directa. Las mutaciones son cambios en las bases nitrogenadas de la secuencia de ADN . Los cambios aislados de la secuencia original se denominan polimorfismos de un solo nucleótido (SNP). [ dudoso – discutir ]
Los cromosomas Y humanos son cromosomas sexuales específicos de los hombres ; casi todos los seres humanos que poseen un cromosoma Y serán morfológicamente masculinos. Aunque los cromosomas Y están situados en el núcleo celular y emparejados con los cromosomas X , solo se recombinan con el cromosoma X en los extremos del cromosoma Y ; el 95% restante del cromosoma Y no se recombina. Por lo tanto, el cromosoma Y y cualquier mutación que surja en él se transmiten en una línea de descendencia masculina directa.
Otros cromosomas, los autosomas y los cromosomas X (cuando hay otro cromosoma X disponible para emparejarse con él), comparten su material genético durante la meiosis , el proceso de división celular que produce gametos . En efecto, esto significa que el material genético de estos cromosomas se mezcla en cada generación, por lo que cualquier nueva mutación se transmite aleatoriamente de padres a hijos.
La característica especial que presentan tanto los cromosomas Y como el ADNmt es que las mutaciones pueden acumularse a lo largo de un determinado segmento de ambas moléculas y estas mutaciones permanecen fijas en su lugar en el ADN. Además, también se puede inferir la secuencia histórica de estas mutaciones. Por ejemplo, si un conjunto de diez cromosomas Y (derivados de diez individuos diferentes) contiene una mutación, A, pero solo cinco de estos cromosomas contienen una segunda mutación, B, entonces debe ser el caso de que la mutación B se produjo después de la mutación A.
Además, los diez individuos que portan el cromosoma con la mutación A son descendientes directos de la línea masculina del mismo hombre que fue la primera persona que portó esta mutación. El primer hombre que portó la mutación B también fue descendiente directo de la línea masculina de este hombre, pero también es el antepasado directo de la línea masculina de todos los hombres que portan la mutación B. Las series de mutaciones como esta forman linajes moleculares. Además, cada mutación define un conjunto de cromosomas Y específicos llamado haplogrupo.
Todos los seres humanos que portan la mutación A forman un único haplogrupo, y todos los seres humanos que portan la mutación B son parte de este haplogrupo, pero la mutación B también define un haplogrupo más reciente (que es un subgrupo o subclado ) propio al que no pertenecen los seres humanos que solo portan la mutación A. Tanto el ADNmt como los cromosomas Y se agrupan en linajes y haplogrupos; estos a menudo se presentan como diagramas en forma de árbol.
Haplogrupos de ADN del cromosoma Y humano
Los haplogrupos de ADN del cromosoma Y (ADN-Y) humano se nombran de la A a la T y se subdividen mediante números y letras minúsculas. Las designaciones de los haplogrupos del cromosoma Y las establece el Consorcio del Cromosoma Y. [5]
Adán del cromosoma Y es el nombre dado por los investigadores al varón que es el ancestro patrilineal (linaje masculino) común más reciente de todos los seres humanos vivos.
Los principales haplogrupos del cromosoma Y y sus regiones geográficas de aparición (antes de la reciente colonización europea) incluyen:
Grupos sin mutación M168
- Haplogrupo A (M91) (África, especialmente los khoisan y los nilotes )
- Haplogrupo B (M60) (África, especialmente los pigmeos y hadzabe )
Grupos con mutación M168
(la mutación M168 ocurrió hace ~50.000 pb)
- Haplogrupo C (M130) (Oceanía, Asia del Norte, Central y Oriental, América del Norte y una presencia menor en América del Sur, Sudeste Asiático, Asia Meridional, Asia Occidental y Europa)
- Haplogrupos YAP+
- Haplogrupo DE (M1, M145, M203)
- Haplogrupo D (CTS3946) (Tíbet, Nepal, Japón, Islas Andamán, Asia Central y una presencia esporádica en Nigeria, Siria y Arabia Saudita)
- Haplogrupo E (M96)
- Haplogrupo E1b1a (V38) África occidental y regiones circundantes; anteriormente conocido como E3a
- Haplogrupo E1b1b (M215) Asociado con la difusión de las lenguas afroasiáticas; ahora concentrado en el norte de África y el Cuerno de África, así como en partes de Medio Oriente, el Mediterráneo y los Balcanes; anteriormente conocido como E3b
- Haplogrupo DE (M1, M145, M203)
Grupos con mutación M89
(la mutación M89 ocurrió hace ~45.000 pb)
- Haplogrupo F (M89) Oceanía, Europa, Asia, América del Norte y del Sur
- Haplogrupo FT (P14, M213) (China, Vietnam, [6] Singapur [7] )
- Haplogrupo G (M201) (presente entre muchos grupos étnicos de Eurasia, generalmente con baja frecuencia; más común en el Cáucaso, la meseta iraní y Anatolia; en Europa principalmente en Grecia, Italia, Iberia , el Tirol, Bohemia; raro en el norte de Europa)
- Haplogrupo H (L901/M2939)
- H1'3 (Z4221/M2826, Z13960)
- H1 (L902/M3061)
- H1a (M69/Página 45) India, Sri Lanka, Nepal, Pakistán, Irán, Asia Central
- H1b (B108) Se encontró en un individuo birmano en Myanmar . [7]
- H3 (Z5857) India, Sri Lanka, Pakistán, Bahrein, Qatar
- H1 (L902/M3061)
- H2 (P96) Anteriormente conocido como haplogrupo F3. Se encuentra con baja frecuencia en Europa y Asia occidental.
- H1'3 (Z4221/M2826, Z13960)
- Haplogrupo IJK (L15, L16)
Grupos con mutaciones L15 y L16
- Haplogrupo IJK (L15, L16)
- Haplogrupo IJ (S2, S22)
- Haplogrupo I (M170, P19, M258) (muy extendido en Europa, se encuentra con poca frecuencia en partes del Medio Oriente y prácticamente ausente en otros lugares [8] )
- Haplogrupo I1 (M253, M307, P30, P40) (Europa del Norte, dominante en Escandinavia)
- Haplogrupo I2 (S31) (Europa central y sudoriental, Cerdeña, Balcanes)
- Haplogrupo J (M304) (Oriente Medio, Turquía, Cáucaso, Italia, Grecia, Balcanes, África del Norte)
- Haplogrupo J* (se encuentra principalmente en Socotra , con algunas observaciones en Pakistán , Omán , Grecia , República Checa y entre los pueblos turcos )
- Haplogrupo J1 (M267) (principalmente asociado con pueblos semíticos en Medio Oriente, pero también se encuentra en Europa mediterránea, Etiopía, África del Norte, Pakistán, India y con pueblos del Cáucaso nororiental en Daguestán ; J1 con DYS388=13 está asociado con Anatolia oriental)
- Haplogrupo J2 (M172) (se encuentra principalmente en Asia occidental, Asia central, Irán, Italia, Grecia, los Balcanes y el norte de África)
- Haplogrupo I (M170, P19, M258) (muy extendido en Europa, se encuentra con poca frecuencia en partes del Medio Oriente y prácticamente ausente en otros lugares [8] )
- Haplogrupo K (M9, P128, P131, P132)
- Haplogrupo IJ (S2, S22)
Grupos con mutación M9
(La mutación M9 ocurrió hace unos 40.000 pb )
- Haplogrupo K
- Haplogrupo LT (L298/P326)
- Haplogrupo L (M11, M20, M22, M61, M185, M295) (Asia meridional, Asia central, Asia suroccidental y Mediterráneo)
- Haplogrupo T (M70, M184/USP9Y+3178, M193, M272) (África del Norte, Cuerno de África, Sudoeste de Asia, Mediterráneo, Sur de Asia); anteriormente conocido como Haplogrupo K2
- Haplogrupo K(xLT) (rs2033003/M526)
- Haplogrupo LT (L298/P326)
Grupos con mutación M526
- Haplogrupo M (P256) (Nueva Guinea, Melanesia, este de Indonesia)
- Haplogrupo NO (M214)
- Haplogrupo N (M231) (Eurasia más septentrional)
- Haplogrupo O (M175) (Asia oriental, Sudeste asiático, Pacífico sur, Asia meridional, Asia central)
- Haplogrupo O1 (F265)
- Haplogrupo O1a (M119)
- Haplogrupo O1b (P31, M268)
- Haplogrupo O2 (M122)
- Haplogrupo O1 (F265)
- Haplogrupo P-M45 (M45) (M45 apareció hace ~35.000 pb)
- Haplogrupo Q-M242 (M242) (Apareció hace unos 15 000–20 000 pb. Se encuentra en Asia y América)
- Haplogrupo Q-M3 (M3) (América del Norte, América Central y América del Sur)
- Haplogrupo R (M207)
- Haplogrupo R1 (M173)
- Haplogrupo R1a (M17) (Asia central, Asia meridional y Europa central, septentrional y oriental)
- Haplogrupo R1b (M343) (Europa, Cáucaso, Asia central, Asia meridional, África del Norte, África central)
- Haplogrupo R2 (M124) (Asia meridional, Cáucaso, Asia central)
- Haplogrupo R1 (M173)
- Haplogrupo Q-M242 (M242) (Apareció hace unos 15 000–20 000 pb. Se encuentra en Asia y América)
- Haplogrupo S (M230, P202, P204) (Nueva Guinea, Melanesia, este de Indonesia)
Haplogrupos del ADN mitocondrial humano
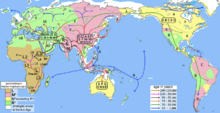
Los haplogrupos de ADNmt humano se clasifican con las letras: A , B , C , CZ , D , E , F , G , H , HV , I , J , pre- JT , JT , K , L0 , L1 , L2 , L3, L4, L5, L6 , M , N , O , P , Q , R , R0 , S , T , U , V , W , X , Y y Z. La versión más actualizada del árbol de ADNmt la mantiene Mannis van Oven en el sitio web PhyloTree. [9]
Eva mitocondrial es el nombre dado por los investigadores a la mujer que es el antepasado matrilineal (linaje femenino) común más reciente de todos los seres humanos vivos.
Definición de poblaciones

Los haplogrupos se pueden utilizar para definir poblaciones genéticas y suelen estar orientados geográficamente. Por ejemplo, las siguientes son divisiones comunes para los haplogrupos de ADNmt:
- Africano: L0 , L1 , L2 , L3 , L4 , L5 , L6
- Eurasia occidental: H , T , U , V , X , K , I , J , W (todos los haplogrupos de Eurasia occidental enumerados se derivan del macrohaplogrupo N ) [10]
- Eurasia oriental: A , B , C , D , E , F , G , Y , Z (nota: C, D, E, G y Z pertenecen al macrohaplogrupo M )
- Nativo americano: A , B , C , D , X
- Australo-melanesio : P , Q , S
Los haplogrupos mitocondriales se dividen en tres grupos principales, que se designan con las letras secuenciales L, M, N. La humanidad se dividió primero dentro del grupo L entre L0 y L1-6. L1-6 dio lugar a otros grupos L, uno de los cuales, L3, se dividió en el grupo M y N.
El grupo M comprende la primera ola de migración humana que se cree que evolucionó fuera de África, siguiendo una ruta hacia el este a lo largo de las zonas costeras del sur. En la actualidad, se encuentran linajes descendientes del haplogrupo M en toda Asia, América y Melanesia, así como en partes del Cuerno de África y el norte de África; casi ninguno se ha encontrado en Europa. El haplogrupo N puede representar otro macrolinaje que evolucionó fuera de África, dirigiéndose hacia el norte en lugar de hacia el este. Poco después de la migración, el gran grupo R se separó del N.
El haplogrupo R consta de dos subgrupos definidos en función de su distribución geográfica: uno se encuentra en el sudeste de Asia y Oceanía y el otro contiene casi todas las poblaciones europeas modernas. El haplogrupo N(xR), es decir, el ADNmt que pertenece al grupo N pero no a su subgrupo R, es típico de las poblaciones aborígenes australianas, aunque también está presente en frecuencias bajas entre muchas poblaciones de Eurasia y las Américas.
El tipo L está formado por casi todos los africanos.
El tipo M consta de:
M1 – Poblaciones etíopes, somalíes e indias. Probablemente debido al gran flujo genético entre el Cuerno de África y la península Arábiga (Arabia Saudita, Yemen, Omán), separadas únicamente por un estrecho angosto entre el mar Rojo y el golfo de Adén.
CZ – Muchos siberianos; rama C – Algunos amerindios; rama Z – Muchos saami, algunos coreanos, algunos chinos del norte y algunas poblaciones de Asia central.
D – Algunos amerindios, muchos siberianos y asiáticos del norte del este
E – Malayos, Borneo, Filipinas, aborígenes taiwaneses , Papúa Nueva Guinea
G – Muchos siberianos del noreste, asiáticos del norte y del este y asiáticos centrales
Q – Poblaciones melanesias, polinesias y neoguineanas
El tipo N consta de:
A – Se encuentra en muchos amerindios y algunos asiáticos orientales y siberianos.
I – 10% de frecuencia en el norte y este de Europa
S – Algunos indígenas australianos (pueblos de las Primeras Naciones de Australia)
W – Algunos europeos del este, asiáticos del sur y asiáticos del sur oriental
X – Algunos amerindios, siberianos del sur, asiáticos del suroeste y europeos del sur
Y – La mayoría de los Nivkhs y la gente de Nias ; muchos ainus, gente tungúsica y austronesios ; también se encuentra con baja frecuencia en algunas otras poblaciones de Siberia, Asia Oriental y Asia Central.
R – Gran grupo que se encuentra dentro del tipo N. Las poblaciones que lo componen pueden dividirse geográficamente en Eurasia occidental y Eurasia oriental. Casi todas las poblaciones europeas y una gran cantidad de la población de Oriente Medio en la actualidad se encuentran dentro de esta rama. Un porcentaje menor se encuentra dentro de otros grupos de tipo N (véase más arriba). A continuación se muestran los subclados de R :
B – Algunos chinos, tibetanos, mongoles, asiáticos centrales, coreanos, amerindios, siberianos del sur, japoneses, austronesios.
F – Se encuentra principalmente en el sudeste asiático, especialmente en Vietnam ; 8,3% en la isla de Hvar en Croacia. [11]
R0 – Se encuentra en Arabia y entre etíopes y somalíes; rama HV (rama H; rama V) – Europa, Asia occidental, norte de África;
Pre-JT: surgió en el Levante (área del Líbano moderno), se encuentra con una frecuencia del 25 % en las poblaciones beduinas; rama JT (rama J; rama T): norte, Europa del Este, Indo, Mediterráneo
U – Alta frecuencia en Eurasia occidental, subcontinente indio y Argelia, se encuentra desde la India hasta el Mediterráneo y el resto de Europa; U5 en particular muestra alta frecuencia en Escandinavia y los países bálticos, con la frecuencia más alta en el pueblo Sami .
Asignación geográfica de haplogrupos del cromosoma Y y del ADNmt
Aquí hay una lista de la asignación de haplogrupos geográficos del cromosoma Y y del ADNmt propuesta por Bekada et al. 2013. [12]
Cromosoma Y
Según SNPS, los haplogrupos que son la edad del primer evento de extinción tienden a ser alrededor de 45-50 kya. Los haplogrupos del segundo evento de extinción parecieron divergir 32-35 kya según Mal'ta . El evento de extinción de la zona cero parece ser Toba durante el cual el haplogrupo CDEF* pareció divergir en C, DE y F. C y F casi no tienen nada en común mientras que D y E tienen mucho en común. El evento de extinción #1 según las estimaciones actuales ocurrió después de Toba, aunque el ADN antiguo más antiguo podría empujar el evento de extinción de la zona cero mucho antes de Toba, y empujar el primer evento de extinción aquí de nuevo a Toba. Los haplogrupos con notas de eventos de extinción por ellos tienen un origen dudoso y esto se debe a que los eventos de extinción conducen a cuellos de botella severos, por lo que todas las notas de estos grupos son solo conjeturas. Tenga en cuenta que el recuento de SNP del ADN antiguo puede ser muy variable, lo que significa que, aunque todos estos grupos divergieron aproximadamente al mismo tiempo, nadie sabe cuándo. [13] [14]
| Origen | Haplogrupo | Marcador |
|---|---|---|
| Europa (¿Segunda extinción?) | I | M170, M253, P259, M227, M507 |
| Europa | Yo1b | P215, M438, P37.2, M359, P41.2 |
| Europa | yo1b2 | M26 |
| Europa | I1c | M223, M284, P78, P95 |
| Europa | J2a1 | M47 |
| Europa | J2a2 | M67, M166 |
| Europa | J2a2a | M92 |
| Europa | J2b | M12, M102, M280, M241 |
| Europa | R1b1b1a | M412, P310 |
| Europa | R1b1b1a1 | L11 |
| Europa | R1b1b1a1a | U106 |
| Europa | R1b1b1a1b | U198, P312, S116 |
| Europa | R1b1b1a1b1 | U152 |
| Europa | R1b1b1a1b2 | M529 |
| Europa | R1b1b1a1b3,4 | M65, M153 |
| Europa | R1b1b1a1b5 | SRY2627 |
| Asia meridional o Melanesia | C1 (antes conocida como CxC3) | Z1426 |
| Asia del Norte | C2 (antes conocido como C3) | M217+ |
| Indonesia o Asia del Sur | F | M89, M282 |
| Europa (Cáucaso) | GRAMO | M201, M285, P15, P16, M406 |
| Asia del Sur | yo | M69, M52, M82, M197, M370 |
| Europa o Oriente Medio | J1 | M304, M267, P58, M365, M368, M369 |
| Europa o Oriente Medio | J2 | M172, M410, M158, M319, DYS445=6, M339, M340 |
| Al oeste de Birmania en Eurasia (¿primer evento de extinción?) [15] | ||
| Indonesia (¿primer evento de extinción?) [15] | K2 (NOP) | M526 |
| Asia del Sur | yo | M11, M20, M27, M76, M317, M274, M349, M357 |
| Asia oriental, Asia sudoriental | norte | M231, M214, LLY22g, Tat, M178 |
| Asia Oriental, Sudeste Asiático, Sur de Asia | Oh | M175, M119 |
| Indonesia, Filipinas | P(xQR) | 92R7, M207, M173, M45 |
| Asia meridional, Siberia | División R y Q (QR) [15] | MEH2, M242, P36.2, M25, M346 |
| Oriente Medio, Europa, Siberia, Asia meridional | R1a1 | M420, M17, M198, M204, M458 |
| Anatolia, Sudeste de Europa ? | R1b | M173, M343, P25, M73 |
| Europa | R1b1b | M269 |
| Europa | R1b1b1 | L23 |
| Pakistán, India | R2 | M479, M124 |
| Oriente Medio | yo | M70 |
| África del Norte | E1b1b1 | M35 |
| África del Norte | E1b1b1a | M78 |
| Asia occidental | E1b1b1a2 | V13 |
| África del Norte | E1b1b1a1 | V12 |
| África del Norte | E1b1b1a1b | V32 |
| África del Norte | E1b1b1a3 | V22 |
| África del Norte | E1b1b1a4 | V65 |
| África del Norte | E1b1b1b | M81 |
| África del Norte | E1b1b1c | M123, M34 |
| África occidental, África del Norte | A | M91, M13 |
| África Oriental | B | M60, M181, SRY10831.1, M150, M109, M112 |
| Asia, África | Delaware | M1, YAP, M174, M40, M96, M75, M98 |
| Asia Oriental, Nepal | D | M174 |
| África Occidental (¿Primer evento de extinción?) | E1a | M33 |
| África Oriental (el primer evento de extinción es la división entre E1b1 y E1a, el segundo evento de extinción es la división entre E1b1b y E1b1a) | E1b1 | P2, M2, U175, M191 |
| Oriente Medio | J1 | P58 |
ADNmt
| Origen | Haplogrupo |
|---|---|
| Europa | H1 |
| Europa | H11a |
| Europa | H1a |
| Europa | H1b |
| Europa | H2a |
| Europa | H3 |
| Europa | H5a |
| Europa | H6a |
| Europa | H7 |
| Europa | HV0/HV0a/V |
| Europa | I4 |
| Europa | J1c7 |
| Europa | J2b1 |
| Europa | T2b* |
| Europa | T2b4 |
| Europa | T2e |
| Europa | U4c1 |
| Europa | U5* |
| Europa | U5a |
| Europa | U5a1b1 |
| Europa | U5b* |
| Europa | U5b1b* |
| Europa | U5b1c |
| Europa | U5b3 |
| Europa | X2c'e |
| Oriente Medio | I |
| Oriente Medio | A |
| Oriente Medio | B |
| Oriente Medio | C/Z |
| Oriente Medio | D/G/M9/E |
| India | F |
| Oriente Medio | H* |
| Oriente Medio | H13a1 |
| Oriente Medio | H14a |
| Oriente Medio | H20 |
| Oriente Medio | H2a1 |
| Oriente Medio | H4 |
| Oriente Medio | H6b |
| Oriente Medio | H8 |
| Oriente Medio | HV1 |
| Oriente Medio | Yo1 |
| Oriente Medio | J / J1c / J2 |
| Oriente Medio | J1a'b'e |
| Oriente Medio | J1b1a1 |
| Oriente Medio | J1b2a |
| Oriente Medio | J1d / J2b |
| Oriente Medio | J1d1 |
| Oriente Medio | J2a |
| Oriente Medio | J2a2a1 |
| Oriente Medio | K* |
| Oriente Medio | K1a* |
| Oriente Medio | K1b1* |
| Oriente Medio | N1a* |
| Oriente Medio | N1b |
| Oriente Medio | N1c |
| Oriente Medio | N2 |
| Oriente Medio | N9 |
| Oriente Medio | R* |
| Oriente Medio | R0a |
| Oriente Medio | yo |
| Oriente Medio | T1* |
| Asia occidental | T1a |
| Oriente Medio | T2 |
| Oriente Medio | T2c |
| Oriente Medio | T2i |
| Oriente Medio | U1* |
| Oriente Medio | U2* |
| Oriente Medio | U2e |
| Eurasia | U3* |
| Oriente Medio | U4 |
| Oriente Medio | U4a* |
| Oriente Medio | U7 |
| Oriente Medio | U8* |
| Oriente Medio | U9a |
| Oriente Medio | incógnita |
| Oriente Medio | X1a |
| Oriente Medio | X2b1 |
| África del Norte | L3e5 |
| África del Norte | M1 |
| África del Norte | M1a1 |
| África del Norte | U6a |
| África del Norte | U6a1'2'3 |
| África del Norte | U6b'c'd |
| África Oriental | L0* |
| África Oriental | L0a1 |
| África Oriental | L0a1b |
| África Oriental | L0a2* |
| África Oriental | Nivel 3c/nivel 4/nivel medio |
| África Oriental | L3d1a1 |
| África Oriental | L3d1d |
| África Oriental | L3e1* |
| África Oriental | L3f* |
| África Oriental | L3h1b* |
| África Oriental | L3i* |
| África Oriental | L3x* |
| África Oriental | L4a'b* |
| África Oriental | L5* |
| África Oriental | L6 |
| África Oriental | N* / M* / L3* |
| África occidental | L1b* |
| África occidental | L1b3 |
| África occidental | L1c* |
| África occidental | L1c2 |
| África occidental | L2* |
| África occidental | L2a |
| África occidental | L2a1* |
| África occidental | L2a1a2'3'4 |
| África occidental | L2a1b |
| África occidental | L2a1b'f |
| África occidental | L2a1c1'2 |
| África occidental | L2a1(16189) |
| África occidental | L2a2 |
| África occidental | L2b* |
| África occidental | L2c1'2 |
| África occidental | L2d |
| África occidental | Nivel 2e |
| África occidental | L3b |
| África occidental | L3b1a3 |
| África occidental | L3b(16124!) |
| África occidental | L3b2a |
| África occidental | L3d* |
| África occidental | L3e2'3'4 |
| África occidental | L3f1b* |
Véase también
- Proyecto Internacional HapMap
- Evolución molecular
- Filogenética molecular
- Genética evolutiva humana
- Raza (biología) / Raza (categorización humana)
- Genealogía genética
- Prueba de ADN genealógica
- Lista de temas de genealogía genética
- Lista de haplogrupos de personajes notables
Referencias
- ^ Por C. Barry Cox, Peter D. Moore, Richard Ladle. Wiley-Blackwell, 2016. ISBN 978-1-118-96858-1 p. 106. Biogeografía: un enfoque ecológico y evolutivo
- ^ Consejo editorial, V&S Publishers, 2012, ISBN 9381588643 pág. 137. Diccionario conciso de ciencias
- ^ Sociedad Internacional de Genealogía Genética 2015 Glosario de genética
- ^ Consorcio, El cromosoma Y (1 de febrero de 2002). "Un sistema de nomenclatura para el árbol de haplogrupos binarios del cromosoma Y humano". Genome Research . 12 (2). Cold Spring Harbor Laboratory: 339–348. doi :10.1101/gr.217602. ISSN 1088-9051. PMC 155271 . PMID 11827954.
- ^ "Consorcio del cromosoma Y". Archivado desde el original el 16 de enero de 2017. Consultado el 27 de julio de 2005 .
- ^ Poznik, G. David; Xue, Yali; Méndez, Fernando L.; et al. (2016). "Ráfagas puntuadas en la demografía masculina humana inferidas a partir de 1244 secuencias del cromosoma Y en todo el mundo". Nature Genetics . 48 (6): 593–599. doi :10.1038/ng.3559. PMC 4884158 . PMID 27111036.
- ^ ab Karmin, Monika; Saag, Lauri; Vicente, Mário; et al. (2015). "Un reciente cuello de botella en la diversidad del cromosoma Y coincide con un cambio global en la cultura". Genome Research . 25 (4): 459–466. doi :10.1101/gr.186684.114. PMC 4381518 . PMID 25770088.
- ^ Rootsi S, Magri C, Kivisild T, Benuzzi G, Help H, Bermisheva M, Kutuev I, Barać L, Pericić M, Balanovsky O, Pshenichnov A, Dion D, Grobei M, Zhivotovsky LA, Battaglia V, Achilli A, Al -Zahery N, Parik J, King R, Cinnioğlu C, Khusnutdinova E, Rudan P, Balanovska E, Scheffrahn W, Simonescu M, Brehm A, Goncalves R, Rosa A, Moisan JP, Chaventre A, Ferak V, Füredi S, Oefner PJ, Shen P, Beckman L, Mikerezi I, Terzić R, Primorac D, Cambon-Thomsen A, Krumina A, Torroni A, Underhill PA, Santachiara-Benerecetti AS, Villems R, Semino O (julio de 2004). "La filogeografía del haplogrupo I del cromosoma Y revela dominios distintos del flujo genético prehistórico en Europa" (PDF) . American Journal of Human Genetics . 75 (1): 128–37. doi :10.1086/422196. PMC 1181996 . PMID 15162323. Archivado del original (PDF) el 24 de junio de 2009. Recuperado el 8 de marzo de 2007 .
- ^ "PhyloTree.org".
- ^ Loogväli EL, Roostalu U, Malyarchuk BA, Derenko MV, Kivisild T, Metspalu E, et al. (2004). "Uniformidad desunida: un lienzo cladístico de varios colores del haplogrupo H de ADNmt en Eurasia". Mol. Biol. Evolución . 21 (11): 2012-21. doi : 10.1093/molbev/msh209 . PMID 15254257.
- ^ Tolk HV, Barac L, Pericic M, Klaric IM, Janicijevic B, Campbell H, Rudan I, Kivisild T, Villems R, Rudan P (septiembre de 2001). "La evidencia del haplogrupo F del ADNmt en una población europea y sus implicaciones etnohistóricas". Revista Europea de Genética Humana . 9 (9): 717–23. doi : 10.1038/sj.ejhg.5200709 . PMID 11571562.
- ^ Bekada A, Fregel R, Cabrera VM, Larruga JM, Pestano J, Benhamamouch S, González AM (2013). "Introducción de los perfiles argelinos de ADN mitocondrial y cromosoma Y en el paisaje del norte de África". PLOS ONE . 8 (2): e56775. Bibcode :2013PLoSO...856775B. doi : 10.1371/journal.pone.0056775 . PMC 3576335 . PMID 23431392.
- ^ "Los ancestros genéticos comunes vivieron durante aproximadamente el mismo período de tiempo". 1 de agosto de 2013. Consultado el 23 de enero de 2015 .
- ^ Raghavan M, Skoglund P, Graf KE, Metspalu M, Albrechtsen A, Moltke I, Rasmussen S, Stafford TW, Orlando L, Metspalu E, Karmin M, Tambets K, Rootsi S, Mägi R, Campos PF, Balanovska E, Balanovsky O, Khusnutdinova E, Litvinov S, Osipova LP, Fedorova SA, Voevoda MI, DeGiorgio M, Sicheritz-Ponten T, Brunak S, Demeshchenko S, Kivisild T, Villems R, Nielsen R, Jakobsson M, Willerslev E (enero de 2014). "El genoma siberiano del Paleolítico superior revela una doble ascendencia de los nativos americanos". Naturaleza . 505 (7481): 87–91. Código Bib :2014Natur.505...87R. doi :10.1038/nature12736. PMC 4105016. PMID 24256729 .
- ^ abc Karafet TM, Mendez FL, Sudoyo H, Lansing JS, Hammer MF (marzo de 2015). "Mejora de la resolución filogenética y rápida diversificación del haplogrupo K-M526 del cromosoma Y en el sudeste asiático". Revista Europea de Genética Humana . 23 (3): 369–73. doi :10.1038/ejhg.2014.106. PMC 4326703 . PMID 24896152.
Enlaces externos
General
- El Proyecto Genográfico
todos los haplogrupos de ADN
Cromosoma Y
- Mapas de haplogrupos del mundo (PDF)
Haplogrupos de ADN del cromosoma Y
- Consorcio del cromosoma Y
- Árbol de haplogrupos de ADN-Y de ISOGG
- Árbol Y de PhyloTree Una filogenia de referencia mínima para el cromosoma Y humano
- Predictor de haplogrupos
- El Consorcio del Cromosoma Y (2002), Un sistema de nomenclatura para el árbol de haplogrupos binarios del cromosoma Y humano, Genome Research, vol. 12 (2), 339–48, febrero de 2002. (El diagrama jerárquico detallado tiene conversiones de esquemas de nombres anteriores)
- Semino et al. (2000), El legado genético del Homo sapiens sapiens paleolítico en los europeos actuales, Science, Vol 290 (artículo que introdujo los haplogrupos "Eu").
- Atlas etnográfico y genográfico del ADN-Y y recopilación de datos de código abierto
Haplogrupos de ADN mitocondrial
- PhyloTree – El árbol filogenético de la variación global del ADN mitocondrial humano
- PhyloD3 – Árbol filogenético basado en D3.js basado en PhyloTree
- MitoTool: un servidor web para el análisis y la recuperación de variaciones de secuencias de ADN mitocondrial humano Archivado el 19 de junio de 2016 en Wayback Machine
- HaploGrep: clasificación automática de haplogrupos de ADN mitocondrial basada en PhyloTree Archivado el 12 de junio de 2016 en Wayback Machine
- HaploFind: un sistema rápido y automático de asignación de haplogrupos para el ADN mitocondrial humano Archivado el 11 de junio de 2016 en Wayback Machine
- Esqueleto gráfico del haplogrupo de mtADN
- La formación del panorama africano del ADNmt
- ¿Los cuatro clados del haplogrupo L2 de ADNmt evolucionan a ritmos diferentes?
Software
- Navegador de haplogrupos de ADN-Y
