Estructura genética del virus H5N1
Este artículo tiene varios problemas. Ayúdenos a mejorarlo o a discutir estos problemas en la página de discusión . ( Aprenda cómo y cuándo eliminar estos mensajes )
|
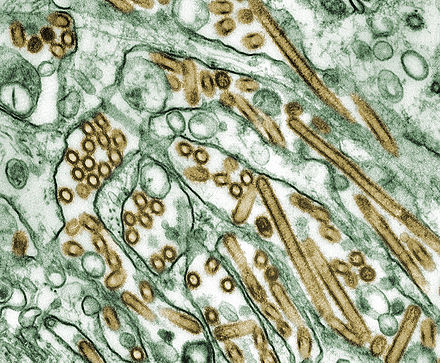 |
La estructura genética del H5N1 , un virus de influenza aviar altamente patógeno ([subtipo H5N1 del virus de influenza A]), se caracteriza por un genoma de ARN segmentado que consta de ocho segmentos genéticos que codifican varias proteínas virales esenciales para la replicación, la adaptación al huésped y la evasión inmunológica.
Virus
El virus de la influenza A subtipo H5N1 (A/H5N1) es un subtipo del virus de la influenza A , que causa influenza (gripe), predominantemente en aves. Es enzoótico (se mantiene en la población) en muchas poblaciones de aves, y también panzoótico (afecta a animales de muchas especies en un área extensa). [1] El virus A/H5N1 también puede infectar a mamíferos (incluidos los humanos) que han estado expuestos a aves infectadas; en estos casos, los síntomas suelen ser graves o fatales. Todos los subtipos del virus de la influenza A comparten la misma estructura genética y son potencialmente capaces de intercambiar material genético por medio de reordenamiento [2] [3]
El virus A/H5N1 se excreta en la saliva, la mucosidad y las heces de las aves infectadas; otros animales infectados pueden excretar virus de la gripe aviar en las secreciones respiratorias y otros fluidos corporales (como la leche). [4] El virus puede propagarse rápidamente a través de las bandadas de aves de corral y entre las aves silvestres. [4] Se estima que se han sacrificado 500 millones de aves de granja en un esfuerzo por contener el virus. [2]
Los síntomas de la influenza A/H5N1 varían según la cepa del virus que causa la infección y la especie de ave o mamífero afectado. [5] [6] La clasificación como influenza aviar de baja patogenicidad (LPAI) o influenza aviar de alta patogenicidad (HPAI) se basa en la gravedad de los síntomas en los pollos domésticos y no predice la gravedad de los síntomas en otras especies. [7] Los pollos infectados con el virus LPAI A/H5N1 presentan síntomas leves o son asintomáticos , mientras que HPAI A/H5N1 causa graves dificultades respiratorias, una caída significativa en la producción de huevos y muerte súbita. [8]
En los mamíferos, incluidos los humanos, la influenza A/H5N1 (ya sea LPAI o HPAI) es poco frecuente. Los síntomas de la infección varían de leves a graves, incluyendo fiebre, diarrea y tos. [6] Se han notificado infecciones humanas por el virus A/H5N1 en 23 países desde 1997, que provocaron neumonía grave y muerte en aproximadamente el 50% de los casos. [9] Entre 2003 y noviembre de 2024, la Organización Mundial de la Salud ha registrado 948 casos confirmados de influenza H5N1, que provocaron 464 muertes. [10] La tasa de mortalidad real puede ser menor porque algunos casos con síntomas leves pueden no haber sido identificados como H5N1. [11]
El virus de la influenza A/H5N1 se identificó por primera vez en aves de granja en el sur de China en 1996. [12] Entre 1996 y 2018, el A/H5N1 coexistió en poblaciones de aves con otros subtipos del virus, pero desde entonces, el subtipo altamente patógeno HPAI A(H5N1) se ha convertido en la cepa dominante en las poblaciones de aves de todo el mundo. [13] Algunas cepas de A/H5N1 que son altamente patógenas para los pollos se han adaptado para causar síntomas leves en patos y gansos, [14] [7] y pueden propagarse rápidamente a través de la migración de las aves. [15] Las especies de mamíferos que se han registrado con infección por H5N1 incluyen vacas, focas, cabras y zorrillos. [16]
Debido a la alta letalidad y virulencia del virus HPAI A(H5N1), su presencia mundial, su reservorio hospedador cada vez más diverso y sus importantes mutaciones en curso, el virus H5N1 se considera la mayor amenaza pandémica del mundo . [17] Las aves de corral domésticas podrían estar protegidas de cepas específicas del virus mediante la vacunación. [18] En caso de un brote grave de gripe H5N1 entre humanos, las agencias de salud han preparado vacunas "candidatas" que pueden usarse para prevenir la infección y controlar el brote; sin embargo, podría llevar varios meses aumentar la producción en masa. [4] [19] [20]
Nomenclatura
Debido a la alta variabilidad del virus, la subtipificación no es suficiente para identificar de forma única una cepa del virus de la influenza A. Para describir de forma inequívoca un aislado específico del virus, los investigadores utilizan la nomenclatura del virus de la influenza [21] , que describe, entre otras cosas, el subtipo, el año y el lugar de recolección. Algunos ejemplos incluyen: [22]
- A/Río de Janeiro/62434/2021 (H3N2) . [22]
- La A inicial indica que el virus es un virus de influenza A.
- Río de Janeiro indica el lugar de recolección. 62434 es un número de secuencia de laboratorio. 2021 (o simplemente 21 ) indica que la muestra se recolectó en 2021. No se menciona ninguna especie, por lo que, por defecto, la muestra se recolectó de un humano.
- (H3N2) indica el subtipo del virus.
- A/cerdo/Dakota del Sur/152B/2009 (H1N2) . [22]
- En este ejemplo se muestra un campo adicional antes del lugar: swine . Esto indica que la muestra se obtuvo de un cerdo.
- A/California/04/2009 A(H1N1)pdm09 . [22]
- Este ejemplo lleva una designación inusual en la última parte: en lugar del habitual (H1N1) , utiliza A(H1N1)pdm09 . Esto se hizo para distinguir el linaje del virus Pandemic H1N1/09 de los virus H1N1 más antiguos. [22]
Debido al impacto de la influenza aviar en granjas avícolas económicamente importantes , en 1981 se ideó un sistema de clasificación que dividía las cepas del virus aviar en altamente patógenas (y por lo tanto potencialmente requiriendo medidas de control enérgicas) o poco patógenas. La prueba para esto se basa únicamente en el efecto sobre los pollos: una cepa de virus es influenza aviar altamente patógena (HPAI) si el 75% o más de los pollos mueren después de ser infectados deliberadamente con ella. La clasificación alternativa es influenza aviar de baja patogenicidad (LPAI). [23] Este sistema de clasificación se ha modificado desde entonces para tener en cuenta la estructura de la proteína hemaglutinina del virus. [24] Otras especies de aves, especialmente aves acuáticas, pueden infectarse con el virus HPAI sin experimentar síntomas graves y pueden propagar la infección a grandes distancias; los síntomas exactos dependen de la especie de ave y la cepa del virus. [23] La clasificación de una cepa de virus aviar como HPAI o LPAI no predice cuán grave podría ser la enfermedad si infecta a humanos u otros mamíferos. [23] [25]
Desde 2006, la Organización Mundial de Sanidad Animal exige que se notifiquen todas las detecciones de LPAI H5 y H7 debido a su potencial de mutar en cepas altamente patógenas. [26]Estructura y genoma

Estructura
El virus de la influenza A tiene un genoma de ARN segmentado, monocatenario y de sentido negativo , encerrado en una envoltura lipídica. La partícula del virus (también llamada virión ) tiene un diámetro de entre 80 y 120 nanómetros, de modo que los viriones más pequeños adoptan una forma elíptica; los viriones más grandes tienen una forma filamentosa. [27]
Núcleo: el núcleo central del virión contiene el genoma viral de ARN, que está formado por ocho segmentos separados. [28] La nucleoproteína (NP) recubre el ARN viral para formar una ribonucleoproteína que asume una configuración helicoidal (espiral). Tres proteínas grandes (PB 1 , PB 2 y PA), que son responsables de la transcripción y replicación del ARN, están unidas a cada segmento de la RNP viral. [28] [29] [30]
Cápside - La proteína matriz M1 forma una capa entre la nucleoproteína y la envoltura, llamada cápside . [28] [29] [30]
Envoltura - La envoltura viral consiste en una bicapa lipídica derivada de la célula huésped. Dos proteínas virales; hemaglutinina (HA) y neuraminidasa (NA), se insertan en la envoltura y se exponen como espigas en la superficie del virión. Ambas proteínas son antigénicas ; el sistema inmunológico de un huésped puede reaccionar a ellas y producir anticuerpos en respuesta. La proteína M2 forma un canal iónico en la envoltura y es responsable de desenvolver el virión una vez que se ha unido a una célula huésped. [28] [29] [30]
Genoma
La siguiente tabla presenta un resumen conciso del genoma de la gripe y las principales funciones de las proteínas que codifica. Los segmentos se numeran convencionalmente del 1 al 8 en orden descendente de longitud. [31] [32] [33] [34]
| Segmento de ARN | Longitud | Proteína | Función |
| 1-PB2 | 2341 | PB2 (polimerasa básica 2) | Un componente de la ARN polimerasa viral . PB2 también inhibe la señalización JAK1/STAT para inhibir la respuesta inmune innata del huésped. |
| 2-PB1 | 2341 | PB1 (polimerasa básica 1) | Un componente de la ARN polimerasa viral. También degrada la proteína de señalización antiviral mitocondrial de la célula huésped. |
| PB1-F2 (polimerasa básica, 1-marco 2) | Proteína accesoria de la mayoría de los virus de la influenza aviar. No es necesaria para la replicación y el crecimiento del virus, pero interfiere en la respuesta inmunitaria del huésped. | ||
| 3-PA | 2233 | PA (ácido polimerasa) | Un componente de la ARN polimerasa viral |
| PAZ | Surge de un desplazamiento del marco de lectura ribosómico en el segmento PA. Inhibe las respuestas inmunitarias innatas del huésped, como la producción de citocinas e interferón . | ||
| 4-HA | 1775 | HA (hemaglutinina) | Parte de la envoltura viral, una proteína que une el virión a las células huésped, lo que permite que el material genético del ARN del virus lo invada. |
| 5- NP | 1565 | NP (Nucleoproteína) | La nucleoproteína se asocia con el ARN viral para formar una ribonucleoproteína (RNP). En la etapa temprana de la infección, el RNP se une a la importina-α de la célula huésped, que lo transporta al núcleo de la célula huésped, donde se transcribe y replica el ARN viral. En una etapa posterior de la infección, los segmentos de ARN viral recién fabricados se ensamblan con la proteína NP y la polimerasa (PB1, PB2 y PA) para formar el núcleo de un virión progenie. |
| 6- N/A | 1409 | NA (Neuraminidasa) | Parte de la envoltura viral. La NA permite que los viriones recién ensamblados escapen de la célula huésped y continúen propagando la infección. La NA también facilita el movimiento de partículas virales infecciosas a través del moco, lo que les permite llegar a las células epiteliales del huésped. |
| 7-M | 1027 | M1 (proteína de matriz 1) | Forma la cápside , que recubre las nucleoproteínas virales y sostiene la estructura de la envoltura viral. M1 también ayuda con la función de la proteína NEP. |
| M2 (proteína de matriz 2) | Forma un canal de protones en la envoltura viral, que se activa una vez que un virión se ha unido a una célula huésped. Esto desprotege al virus y expone su contenido infeccioso al citoplasma de la célula huésped. | ||
| 8-NS | 890 | NS1 (proteína no estructural 1) | Contrarresta la respuesta inmune natural del huésped e inhibe la producción de interferón. |
| NEP (proteína de exportación nuclear, anteriormente proteína no estructural NS2 2) | Coopera con la proteína M1 para mediar la exportación de copias de ARN viral desde el núcleo al citoplasma en la etapa tardía de la replicación viral. |
Tres proteínas virales (PB1, PB2 y PA) se asocian para formar la ARN polimerasa dependiente de ARN (RdRp), cuya función es transcribir y replicar el ARN viral.
Transcripción del ARN mensajero viral: el complejo RdRp transcribe los ARNm virales mediante un mecanismo llamado secuestro de la tapa . Consiste en el secuestro y la escisión de los pre-ARNm con tapa del huésped . El ARNm de la célula huésped se escinde cerca de la tapa para producir un cebador para la transcripción del ARNm viral de sentido positivo utilizando el ARN viral de sentido negativo como plantilla. [35] Luego, la célula huésped transporta el ARNm viral al citoplasma donde los ribosomas fabrican las proteínas virales. [31] [32] [33] [34]
Replicación del ARN viral - La replicación del genoma de la gripe implica dos pasos. En primer lugar, la RdRp transcribe el genoma viral de sentido negativo en un ARN complementario de sentido positivo (ARNc), luego los ARNc se utilizan como plantillas para transcribir nuevas copias de ARNv de sentido negativo. Estas se exportan desde el núcleo y se ensamblan cerca de la membrana celular para formar el núcleo de nuevos viriones. [31] [32] [33] [34]
Segmentos de genes que codifican la superficie
Todos los virus de influenza A tienen dos segmentos genéticos denominados HA y NA que codifican las proteínas antigénicas hemaglutina y neuraminidasa que se encuentran en la envoltura externa del virus.
JA
La HA codifica la hemaglutinina , que es una glicoproteína antigénica que se encuentra en la superficie de los virus de la gripe y es responsable de la unión del virus a la célula que está siendo infectada. La hemaglutinina forma picos en la superficie de los virus de la gripe que funcionan para unir los virus a las células . Esta unión es necesaria para la transferencia eficiente de los genes del virus de la gripe a las células, un proceso que puede ser bloqueado por anticuerpos que se unen a las proteínas de la hemaglutinina. Un factor genético que distingue entre los virus de la gripe humana y los virus de la gripe aviar es que la HA de la gripe aviar se une a los receptores de ácido siálico alfa 2-3 , mientras que la HA de la gripe humana se une a los receptores de ácido siálico alfa 2-6. [36]
N / A
La NA codifica la neuraminidasa , una enzima glucoproteínica antigénica que se encuentra en la superficie de los virus de la gripe . Ayuda a liberar la progenie de los virus de las células infectadas. Los medicamentos antivirales Tamiflu y Relenza actúan inhibiendo algunas cepas de la neuraminidasa . [37]
Matriz que codifica segmentos genéticos
METRO
M codifica las proteínas de la matriz (M1 y M2) que, junto con las dos proteínas de superficie ( hemaglutinina y neuraminidasa ), forman la cápside (capa protectora) del virus. Se codifica utilizando diferentes marcos de lectura del mismo segmento de ARN.
- La proteína matriz M1 forma la cápside , que recubre las nucleoproteínas virales y sostiene la estructura de la envoltura viral. M1 también colabora con la función de la proteína NEP.
- La proteína M2 forma un canal de protones en la envoltura viral que desprotege al virus, exponiendo así su contenido (los ocho segmentos de ARN) al citoplasma de la célula huésped. La proteína transmembrana M2 es un canal iónico necesario para una infección eficaz. [38]
Segmentos de genes que codifican nucleoproteínas.
notario público
NP codifica una proteína estructural que encapsida la cadena negativa del ARN viral . [39]
Nueva Zelanda
NS codifica dos proteínas no estructurales ( NS1 y proteína de exportación nuclear NEP, anteriormente llamada NS2).
- NS1 contrarresta la respuesta inmune natural del huésped e inhibe la producción de interferón.
- La NEP media la exportación de complejos de ribonucleoproteína (RNP) del virus de la influenza desde el núcleo al citoplasma, donde se ensamblan. [40]
Segmentos de genes que codifican la polimerasa
Pensilvania
PA codifica la proteína PA, que es un componente de la polimerasa viral .
PB1
PB1 codifica la proteína PB1 y la proteína PB1-F2.
- La proteína PB1 es un componente de la polimerasa viral .
- La proteína PB1-F2 está codificada por un marco de lectura abierto alternativo del segmento de ARN PB1
PB2
PB2 codifica la proteína PB2, que es un componente de la polimerasa viral .
Mutación
Véase también
Referencias
- ^ "Influenza (aviar y otras enfermedades zoonóticas)". who.int . Organización Mundial de la Salud . 3 de octubre de 2023 . Consultado el 6 de mayo de 2024 .
- ^ ab Bourk I (26 de abril de 2024). «'Sin precedentes': cómo la gripe aviar se convirtió en una pandemia animal». bbc.com . BBC . Consultado el 8 de mayo de 2024 .
- ^ Shao W, Li X, Goraya MU, Wang S, Chen JL (agosto de 2017). "Evolución del virus de la influenza A por mutación y reordenamiento". Revista Internacional de Ciencias Moleculares . 18 (8): 1650. doi : 10.3390/ijms18081650 . PMC 5578040 . PMID 28783091.
- ^ abc "Prevención y tratamiento antiviral de los virus de la gripe aviar en las personas | Influenza aviar (gripe)". cdc.gov . EE. UU.: Centros para el Control y la Prevención de Enfermedades . 2024-04-19 . Consultado el 2024-05-08 .
- ^ "Gripe aviar (influenza aviar)". betterhealth.vic.gov.au . Victoria, Australia: Departamento de Salud y Servicios Humanos . Consultado el 9 de mayo de 2024 .
- ^ ab "Influenza aviar: orientación, datos y análisis". gov.uk . 2021-11-18 . Consultado el 2024-05-09 .
- ^ ab "Influenza aviar en aves". cdc.gov . EE. UU.: Centros para el Control y la Prevención de Enfermedades. 2022-06-14 . Consultado el 2024-05-06 .
- ^ "Gripe aviar (influenza aviar): cómo detectarla y notificarla en aves de corral u otras aves cautivas". gov.uk . Reino Unido: Departamento de Medio Ambiente, Alimentación y Asuntos Rurales y Agencia de Sanidad Animal y Vegetal. 2022-12-13 . Consultado el 2024-05-06 .
- ^ "Virus de influenza tipo A". cdc.gov . EE. UU.: Centros para el Control y la Prevención de Enfermedades. 2024-02-01 . Consultado el 2024-05-03 .
- ^ "Virus de la influenza aviar A(H5N1)". www.who.int . Consultado el 28 de mayo de 2024 .
- ^ Li FC, Choi BC, Sly T, Pak AW (junio de 2008). "Detección de la tasa real de letalidad de la gripe aviar H5N1". J Epidemiol Community Health . 62 (6): 555–9. doi :10.1136/jech.2007.064030. PMID 18477756. S2CID 34200426.
- ^ "Aparición y evolución de la gripe aviar H5N1 | Influenza aviar (gripe)". cdc.gov . EE. UU.: Centros para el Control y la Prevención de Enfermedades. 2023-06-06 . Consultado el 2024-05-03 .
- ^ Huang P, Sun L, Li J, Wu Q, Rezaei N, Jiang S, et al. (junio de 2023). "La posible transmisión entre especies de los virus de la influenza aviar altamente patógena del subtipo H5 (HPAI H5) a los humanos requiere el desarrollo de vacunas antigripales universales y específicas para el subtipo H5". Cell Discovery . 9 (1): 58. doi :10.1038/s41421-023-00571-x. PMC 10275984 . PMID 37328456.
- ^ "Aspectos destacados en la historia de la influenza aviar (gripe aviar) Cronología – 2020-2024 | Influenza aviar (gripe)". cdc.gov . EE. UU.: Centros para el Control y la Prevención de Enfermedades. 2024-04-22 . Consultado el 2024-05-08 .
- ^ Caliendo V, Lewis NS, Pohlmann A, Baillie SR, Banyard AC, Beer M, et al. (julio de 2022). "Propagación transatlántica de la influenza aviar altamente patógena H5N1 por aves silvestres desde Europa hasta América del Norte en 2021". Scientific Reports . 12 (1): 11729. Bibcode :2022NatSR..1211729C. doi :10.1038/s41598-022-13447-z. PMC 9276711 . PMID 35821511.
- ^ "La gripe aviar es mala para las aves de corral y el ganado. Por qué no es una amenaza grave para la mayoría de nosotros, todavía". NBC News. 2024-05-02 . Consultado el 2024-05-09 .
- ^ McKie R (20 de abril de 2024). "Los científicos advierten que la próxima pandemia probablemente sea causada por el virus de la gripe". The Observer . ISSN 0029-7712 . Consultado el 9 de mayo de 2024 .
- ^ "Vacunación de aves de corral contra la gripe aviar altamente patógena: vacunas disponibles y estrategias de vacunación". efsa.europa.eu . 2023-10-10 . Consultado el 2024-05-09 .
- ^ "En caso de ser necesario, dos posibles vacunas contra la gripe aviar podrían estar disponibles en unas semanas". NBC News. 2024-05-01 . Consultado el 2024-05-09 .
- ^ "Influenza aviar (gripe aviar) | Agencia Europea de Medicamentos". ema.europa.eu . Consultado el 9 de mayo de 2024 .
- ^ "Una revisión del sistema de nomenclatura de los virus de la gripe: un memorando de la OMS". Boletín de la Organización Mundial de la Salud . 58 (4): 585–591. 1980. PMC 2395936 . PMID 6969132.
Este Memorándum fue redactado por los signatarios enumerados en la página 590 con ocasión de una reunión celebrada en Ginebra en febrero de 1980.
- ^ abcde «Nota técnica: Nomenclatura del virus de la influenza». Organización Panamericana de la Salud . 11 de enero de 2023. Archivado desde el original el 10 de agosto de 2023. Consultado el 27 de mayo de 2024 .
- ^ abc Alexander DJ, Brown IH (abril de 2009). "Historia de la influenza aviar altamente patógena". Revue Scientifique et Technique . 28 (1): 19–38. doi :10.20506/rst.28.1.1856. PMID 19618616.
- ^ "Ficha informativa sobre el virus A(H5N1)". www.ecdc.europa.eu . 2017-06-15 . Consultado el 2024-05-21 .
- ^ "Situación actual de la gripe aviar en humanos en Estados Unidos". Centros para el Control y la Prevención de Enfermedades (CDC) de Estados Unidos . 2024-04-05 . Consultado el 2024-05-22 .
- ^ "Plan nacional de vigilancia de la influenza aviar H5/H7". Departamento de Agricultura de los Estados Unidos . Servicio de Inspección de Sanidad Animal y Vegetal. Octubre de 2013.
- ^ Dadonaite B, Vijayakrishnan S, Fodor E, Bhella D, Hutchinson EC (agosto de 2016). "Virus filamentosos de la influenza". The Journal of General Virology . 97 (8): 1755–1764. doi :10.1099/jgv.0.000535. PMC 5935222 . PMID 27365089.
- ^ abcd Bouvier NM, Palese P (septiembre de 2008). "La biología de los virus de la gripe". Vaccine . 26 (Supl 4): D49–D53. doi :10.1016/j.vaccine.2008.07.039. PMC 3074182 . PMID 19230160.
- ^ abc Shaffer C (7 de marzo de 2018). «Estructura de la gripe A». News-Medical . Consultado el 18 de junio de 2024 .
- ^ abc «Virología de la gripe humana». Organización Mundial de la Salud . 13 de mayo de 2010. Consultado el 19 de junio de 2024 .
- ^ abc Krammer F, Smith GJ, Fouchier RA, Peiris M, Kedzierska K, Doherty PC, et al. (junio de 2018). "Influenza". Nature Reviews. Disease Primers . 4 (1): 3. doi :10.1038/s41572-018-0002-y. PMC 7097467. PMID 29955068 .
- ^ abc Jakob C, Paul-Stansilaus R, Schwemmle M, Marquet R, Bolte H (septiembre de 2022). "La red de empaquetamiento del genoma del virus de la influenza A: compleja, flexible y aún sin resolver". Nucleic Acids Research . 50 (16): 9023–9038. doi :10.1093/nar/gkac688. PMC 9458418 . PMID 35993811.
- ^ abc Dou D, Revol R, Östbye H, Wang H, Daniels R (20 de julio de 2018). "Entrada celular, replicación, ensamblaje y movimiento del virión del virus de la influenza A". Frontiers in Immunology . 9 : 1581. doi : 10.3389/fimmu.2018.01581 . PMC 6062596 . PMID 30079062.
- ^ abc Rashid F, Xie Z, Li M, Xie Z, Luo S, Xie L (13 de diciembre de 2023). "Roles y funciones de las proteínas IAV en la evasión inmunitaria del huésped". Frontiers in Immunology . 14 : 1323560. doi : 10.3389/fimmu.2023.1323560 . PMC 10751371 . PMID 38152399.
- ^ Decroly E, Canard B (junio de 2017). "Principios bioquímicos e inhibidores para interferir con las vías de protección viral". Current Opinion in Virology . 24 : 87–96. doi :10.1016/j.coviro.2017.04.003. PMC 7185569 . PMID 28527860.
- ^ Greninger A (16 de julio de 2004). "La definición y medición de la investigación peligrosa" (PDF) . Documento de trabajo del CISSM . Archivado desde el original (PDF) el 8 de noviembre de 2006. Consultado el 9 de diciembre de 2006 .
- ^ Scidev.net News Archivado el 5 de febrero de 2008 en Wayback Machine Artículo El 'bolsillo' de la proteína de la gripe aviar podría inspirar mejores medicamentos publicado el 16 de agosto de 2006
- ^ Replicación del virus de la influenza en Microbiología Médica, 4.ª edición, editado por Samuel Baron. 1996 Capítulo 58. ISBN 0-9631172-1-1 .
- ^ Steuler H, Schröder B, Bürger H, Scholtissek C (julio de 1985). "Secuencia del gen de la nucleoproteína de la influenza A/parrot/Ulster/73". Investigación de virus . 3 (1): 35–40. doi :10.1016/0168-1702(85)90039-5. PMID 4024728.
- ^ Paragas J, Talon J, O'Neill RE, Anderson DK, García-Sastre A, Palese P (agosto de 2001). "Las proteínas NEP (NS2) de los virus de la influenza B y C poseen actividades de exportación nuclear". Journal of Virology . 75 (16): 7375–7383. doi :10.1128/JVI.75.16.7375-7383.2001. PMC 114972 . PMID 11462009.
- ^ Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R (octubre de 2010). "Tasas de mutación viral". Revista de Virología . 84 (19): 9733–9748. doi :10.1128/JVI.00694-10. PMC 2937809 . PMID 20660197.
- ^ Kou Z, Lei FM, Yu J, Fan ZJ, Yin ZH, Jia CX y otros. (Diciembre de 2005). "Nuevo genotipo de virus de la influenza aviar H5N1 aislado de gorriones arbóreos en China". Revista de Virología . 79 (24): 15460–15466. doi :10.1128/JVI.79.24.15460-15466.2005. PMC 1316012 . PMID 16306617.
- ^ Red de Vigilancia del Programa Mundial de Influenza de la Organización Mundial de la Salud (octubre de 2005). "Evolución de los virus de la influenza aviar H5N1 en Asia". Enfermedades infecciosas emergentes . 11 (10): 1515–1521. doi :10.3201/eid1110.050644. PMC 3366754 . PMID 16318689. La figura 1 muestra una representación diagramática de la relación genética de los genes de hemaglutinina H5N1 asiáticos de varios aislamientos del virus.
- ^ CDC (15 de mayo de 2024). «Transmisión de virus de la gripe aviar entre animales y personas». Centros para el Control y la Prevención de Enfermedades . Consultado el 10 de junio de 2024 .
- ^ Taubenberger JK, Morens DM (abril de 2010). "Influenza: la pandemia pasada y futura". Public Health Reports . 125 (Supl 3): 16–26. doi :10.1177/00333549101250S305. PMC 2862331 . PMID 20568566.
- ^ Webster RG, Bean WJ, Gorman OT, Chambers TM, Kawaoka Y (marzo de 1992). "Evolución y ecología de los virus de la influenza A". Microbiological Reviews . 56 (1): 152–179. doi :10.1128/mr.56.1.152-179.1992. PMC 372859 . PMID 1579108.
- ^ "Ficha informativa sobre la gripe porcina en humanos y cerdos". Centro Europeo para el Control y Prevención de Enfermedades . 2017-06-15 . Consultado el 2024-06-13 .
- ^ Lee K, Fang J (2013). Diccionario histórico de la Organización Mundial de la Salud. Rowman & Littlefield. ISBN 9780810878587.
- ^ "70 años del SMVRG: el sistema mundial de vigilancia y respuesta a la gripe". Organización Mundial de la Salud . 19 de septiembre de 2022 . Consultado el 13 de junio de 2024 .
Lectura adicional
- Beigel JH, Farrar J, Han AM, Hayden FG, Hyer R, de Jong MD, et al. (Septiembre de 2005). "Infección por influenza aviar A (H5N1) en humanos". La Revista de Medicina de Nueva Inglaterra . 353 (13): 1374-1385. doi :10.1056/NEJMra052211. hdl : 10722/45195 . PMID 16192482.
- Ghedin E, Sengamalay NA, Shumway M, Zaborsky J, Feldblyum T, Subbu V, et al. (octubre de 2005). "La secuenciación a gran escala de la gripe humana revela la naturaleza dinámica de la evolución del genoma viral". Nature . 437 (7062): 1162–1166. Bibcode :2005Natur.437.1162G. doi : 10.1038/nature04239 . PMID 16208317.Presenta un resumen de lo que se ha descubierto en el Proyecto de Secuenciación del Genoma de la Influenza .
- Hiromoto Y, Yamazaki Y, Fukushima T, Saito T, Lindstrom SE, Omoe K, et al. (mayo de 2000). "Caracterización evolutiva de los seis genes internos del virus de la influenza A humana H5N1". The Journal of General Virology . 81 (Pt 5): 1293–1303. doi :10.1099/0022-1317-81-5-1293 (inactivo el 1 de noviembre de 2024). PMID 10769072.
{{cite journal}}: CS1 maint: DOI inactive as of November 2024 (link)
Enlaces externos
- "Informe sobre la gripe". InfluenzaReport.com .
- Enlaces y descripciones de resúmenes y textos completos Esta bibliografía de publicaciones sobre influenza aviar fue compilada a través del esfuerzo cooperativo del Centro Nacional de Salud de Vida Silvestre del USGS y el Nodo de Información sobre Enfermedades de Vida Silvestre.
- Búsqueda de publicaciones de investigación sobre H5N1: Entez PubMed
- El "árbol de la vida" evolutivo del virus H5N1:
- Aquí se muestra el árbol filogenético del segmento del gen de la hemaglutinina del virus de la influenza. Cambios de aminoácidos en tres linajes (ave, cerdo, humano) del segmento proteico de la hemaglutinina del virus de la influenza HA1.
- Aquí está el árbol que muestra la evolución por reordenamiento del H5N1 de 1999 a 2004 que creó el genotipo Z en 2002.
- Aquí está el árbol que muestra la evolución por deriva antigénica desde 2002 que creó docenas de variedades altamente patógenas del genotipo Z del virus de la gripe aviar H5N1, algunas de las cuales están cada vez más adaptadas a los mamíferos.
- La OMS (PDF) contiene el último "árbol de la vida" evolutivo del virus H5N1 Artículo sobre las características antigénicas y genéticas de los virus H5N1 y de los virus candidatos a la vacuna contra el virus H5N1 desarrollados para su posible uso como vacunas prepandémicas publicado el 18 de agosto de 2006
- Base de datos del genoma La página contiene enlaces a la secuencia completa del genoma del virus de la influenza A (A/Goose/Guangdong/1/96(H5N1)).
Enlaces externos
- Base de datos de investigación sobre la influenza: base de datos de secuencias genómicas de la influenza e información relacionada.



