Fosfoinosítido 3-quinasa
| Fosfatidilinositol-4,5-bisfosfato 3-quinasa | |||||||||
|---|---|---|---|---|---|---|---|---|---|
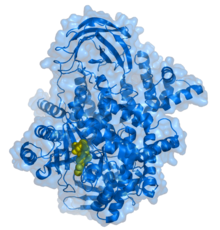 Inhibidor de PIK-93 (amarillo) unido a la subunidad gamma PI3K 110. [1] | |||||||||
| Identificadores | |||||||||
| Símbolo | PI3K | ||||||||
| Pfam | PF00454 | ||||||||
| Interprofesional | IPR000403 | ||||||||
| ELEGANTE | SM00146 | ||||||||
| PROSITIO | PDOC00710 | ||||||||
| SCOP2 | 3 gmm / ALCANCE / SUPFAM | ||||||||
| Superfamilia OPM | 265 | ||||||||
| Proteína OPM | 3 ml 9 | ||||||||
| |||||||||
| Fosfoinosítido 3-quinasa | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| N.º CE | 2.7.1.137 | ||||||||
| N.º CAS | 115926-52-8 | ||||||||
| Bases de datos | |||||||||
| IntEnz | Vista de IntEnz | ||||||||
| BRENDA | Entrada de BRENDA | ||||||||
| Expasí | Vista de NiceZyme | ||||||||
| BARRIL | Entrada de KEGG | ||||||||
| MetaCiclo | vía metabólica | ||||||||
| PRIAMO | perfil | ||||||||
| Estructuras del PDB | RCSB AP APBE APSUMA | ||||||||
| |||||||||
Las fosfoinosítido 3-quinasas ( PI3Ks ), también llamadas fosfatidilinositol 3-quinasas , son una familia de enzimas involucradas en funciones celulares como el crecimiento celular, proliferación, diferenciación, motilidad, supervivencia y tráfico intracelular, que a su vez están involucradas en el cáncer.
Las PI3K son una familia de enzimas transductoras de señales intracelulares relacionadas capaces de fosforilar el grupo hidroxilo de la posición 3 del anillo de inositol del fosfatidilinositol (PtdIns). [2] La vía, con el oncogén PIK3CA y el gen supresor de tumores PTEN , está implicada en la sensibilidad de los tumores cancerosos a la insulina y al IGF1 , y en la restricción calórica . [3] [4]
Descubrimiento
El descubrimiento de PI3Ks por Lewis Cantley y colegas comenzó con su identificación de una fosfoinosítido quinasa previamente desconocida asociada con la proteína T media del polioma . [5] Observaron una especificidad de sustrato única y propiedades cromatográficas de los productos de la lípido quinasa, lo que llevó al descubrimiento de que esta fosfoinosítido quinasa tenía la capacidad sin precedentes de fosforilar fosfoinosítidos en la posición 3' del anillo de inositol. [6] Posteriormente, Cantley y colegas demostraron que in vivo la enzima prefiere PtdIns(4,5)P2 como sustrato, produciendo el nuevo fosfoinosítido PtdIns(3,4,5)P3 [7] previamente identificado en neutrófilos. [8]
Clases
La familia PI3K se divide en cuatro clases diferentes: Clase I , Clase II , Clase III y Clase IV. Las clasificaciones se basan en la estructura primaria, la regulación y la especificidad del sustrato lipídico in vitro . [9]
Clase I
Las PI3K de clase I catalizan la conversión de fosfatidilinositol (4,5)-bisfosfato (PI(4,5)P 2 ) en fosfatidilinositol (3,4,5)-trisfosfato (PI(3,4,5)P 3 ) in vivo. Si bien in vitro también se ha demostrado que convierten fosfatidilinositol (PI) en fosfatidilinositol 3-fosfato (PI3P) y fosfatidilinositol 4-fosfato (PI4P) en fosfatidilinositol (3,4)-bisfosfato (PI(3,4)P 2 ), estas reacciones están fuertemente desfavorecidas in vivo. [10] [11] [12] [13] La PI3K es activada por receptores acoplados a proteína G y receptores de tirosina quinasa . [9]
Las PI3K de clase I son moléculas heterodímeras compuestas de una subunidad reguladora y una subunidad catalítica ; se dividen además entre los subconjuntos IA y IB según la similitud de secuencia. Las PI3K de clase IA se componen de un heterodímero entre una subunidad catalítica p110 y una subunidad reguladora más corta (a menudo p85). [14] Hay cinco variantes de la subunidad reguladora: las tres variantes de empalme p85α, p55α y p50α , p85β y p55γ . También hay tres variantes de la subunidad catalítica p110 denominadas subunidad catalítica p110α, β o δ. Las tres primeras subunidades reguladoras son todas variantes de empalme del mismo gen ( Pik3r1 ), las otras dos se expresan por otros genes (Pik3r2 y Pik3r3, p85β y p55γ, respectivamente). La subunidad reguladora más expresada es p85α; las tres subunidades catalíticas se expresan por genes separados ( Pik3ca , Pik3cb y Pik3cd para p110α , p110β y p110δ , respectivamente). Las dos primeras isoformas de p110 (α y β) se expresan en todas las células, pero p110δ se expresa principalmente en leucocitos , y se ha sugerido que evolucionó en paralelo con el sistema inmunológico adaptativo. Las subunidades reguladora p101 y catalítica p110γ comprenden las PI3K de clase IB y están codificadas por un solo gen cada una ( Pik3cg para p110γ y Pik3r5 para p101).
Las subunidades p85 contienen dominios SH2 y SH3 ( Online Mendelian Inheritance in Man (OMIM): 171833). Los dominios SH2 se unen preferentemente a residuos de tirosina fosforilados en el contexto de la secuencia de aminoácidos YXXM. [15] [16]
Clases II y III

Las PI3K de clase II y III se diferencian de las de clase I por su estructura y función. La característica distintiva de las PI3K de clase II es el dominio C2 C-terminal. Este dominio carece de residuos Asp críticos para coordinar la unión de Ca 2+ , lo que sugiere que las PI3K de clase II se unen a los lípidos de una manera independiente del Ca 2+ .
La clase II comprende tres isoformas catalíticas (C2α, C2β y C2γ), pero, a diferencia de las clases I y III, no contiene proteínas reguladoras. La clase II cataliza la producción de PI(3)P a partir de PI y PI(3,4)P 2 a partir de PI(4)P; sin embargo, se sabe poco sobre su papel en las células inmunes. Sin embargo, se ha demostrado que PI(3,4)P 2 desempeña un papel en la fase de invaginación de la endocitosis mediada por clatrina. [17] C2α y C2β se expresan en todo el cuerpo, pero la expresión de C2γ se limita a los hepatocitos .
Las PI3K de clase III producen solo PI(3)P a partir de PI [9] pero son más similares a la clase I en estructura, ya que existen como heterodímeros de una subunidad catalítica ( Vps34 ) y una subunidad reguladora (Vps15/p150). La clase III parece estar involucrada principalmente en el tráfico de proteínas y vesículas. Sin embargo, hay evidencia que demuestra que pueden contribuir a la efectividad de varios procesos importantes para las células inmunes, en particular la fagocitosis .
Clase IV
Un grupo de enzimas más distantemente relacionadas a veces se denomina PI3K de clase IV. Está compuesto por la ataxia telangiectasia mutada (ATM), la ataxia telangiectasia y relacionada con Rad3 (ATR), la proteína quinasa dependiente de ADN (DNA-PK) y la diana de rapamicina en mamíferos (mTOR). Son proteínas serina/treonina quinasas.
Genes humanos
| grupo | gene | proteína | alias | Número CE |
|---|---|---|---|---|
| Catalizador de clase 1 | PIK3CA | PI3K, polipéptido alfa catalítico | p110-α | 2.7.1.153 |
| PIK3CB | PI3K, polipéptido beta catalítico | p110-β | ||
| PIK3CG | PI3K, polipéptido catalítico gamma | p110-γ | ||
| PIK3CD | PI3K, polipéptido catalítico delta | p110-δ | ||
| clase 1 reglamentaria | PIK3R1 | PI3K, subunidad reguladora 1 (alfa) | p85-α, p55-α, p50-α (variantes de empalme) | N / A |
| PIK3R2 | PI3K, subunidad reguladora 2 (beta) | p85-β | ||
| PIK3R3 | PI3K, subunidad reguladora 3 (gamma) | p55-γ | ||
| PIK3R4 | PI3K, subunidad reguladora 4 | pág. 150 | ||
| PIK3R5 | PI3K, subunidad reguladora 5 | pág. 101 | ||
| PIK3R6 | PI3K, subunidad reguladora 6 | pág. 87 | ||
| Clase 2 | PIK3C2A | PI3K, clase 2, polipéptido alfa | PI3K-C2α | 2.7.1.154 |
| PIK3C2B | PI3K, clase 2, polipéptido beta | PI3K-C2β | ||
| PIK3C2G | PI3K, clase 2, polipéptido gamma | PI3K-C2γ | ||
| Clase 3 | PIK3C3 | PI3K, clase 3 | Vps34 | 2.7.1.137 |
Mecanismo
Los diversos fosfoinosítidos 3-fosforilados que son producidos por PI3Ks ( PtdIns3P , PtdIns(3,4)P2 , PtdIns(3,5)P2 y PtdIns(3,4,5)P3 ) funcionan en un mecanismo por el cual un grupo variado de proteínas de señalización, que contienen dominios PX , dominios de homología de pleckstrina (dominios PH), dominios FYVE u otros dominios de unión a fosfoinosítidos, son reclutados a varias membranas celulares.
Función
Las PI3K se han relacionado con un grupo extraordinariamente diverso de funciones celulares, que incluyen el crecimiento celular, la proliferación, la diferenciación, la motilidad, la supervivencia y el tráfico intracelular. Muchas de estas funciones se relacionan con la capacidad de las PI3K de clase I para activar la proteína quinasa B (PKB, también conocida como Akt) como en la vía PI3K/AKT/mTOR . Las isoformas p110δ y p110γ regulan diferentes aspectos de las respuestas inmunitarias. Las PI3K también son un componente clave de la vía de señalización de la insulina . Por lo tanto, existe un gran interés en el papel de la señalización de PI3K en la diabetes mellitus . La PI3K también está involucrada en la señalización de la interleucina (IL4) [ cita requerida ]
Mecanismo
El dominio de homología de pleckstrina de AKT se une directamente a PtdIns(3,4,5)P3 y PtdIns(3,4)P2 , que son producidos por PI3Ks activados. [18] Dado que PtdIns(3,4,5)P3 y PtdIns(3,4)P2 están restringidos a la membrana plasmática, esto da como resultado la translocación de AKT a la membrana plasmática. Del mismo modo, la quinasa dependiente de fosfoinosítido-1 (PDK1 o, raramente denominada como PDPK1) también contiene un dominio de homología de pleckstrina que se une directamente a PtdIns(3,4,5)P3 y PtdIns(3,4)P2, lo que hace que también se transloque a la membrana plasmática tras la activación de PI3K. La interacción de PDK1 activado y AKT permite que AKT sea fosforilado por PDK1 en la treonina 308, lo que conduce a la activación parcial de AKT. La activación completa de AKT ocurre tras la fosforilación de la serina 473 por el complejo TORC2 de la proteína quinasa mTOR .
Se ha demostrado que la vía PI3K/AKT es necesaria para una gama extremadamente diversa de actividades celulares, en particular la proliferación y supervivencia celular. Por ejemplo, se ha demostrado que participa en la protección de los astrocitos frente a la apoptosis inducida por ceramidas. [19]
Se han identificado muchas otras proteínas que están reguladas por PtdIns(3,4,5)P3, incluidas la tirosina quinasa de Bruton (BTK), el receptor general de fosfoinosítidos-1 (GRP1) y la transferasa de N-acetilglucosamina ligada a O (O-GlcNAc) .
PtdIns(3,4,5)P3 también activa factores de intercambio de nucleótidos de guanina (GEF) que activan la GTPasa Rac1, [20] lo que conduce a la polimerización de actina y al reordenamiento del citoesqueleto. [21]
Cánceres
La PI3K p110α de clase IA está mutada en muchos cánceres. Muchas de estas mutaciones hacen que la quinasa sea más activa. Es la quinasa más mutada en el glioblastoma , el tumor cerebral primario más maligno. [22] La fosfatasa PtdIns(3,4,5) P 3 PTEN que antagoniza la señalización de PI3K está ausente en muchos tumores. Además, el receptor del factor de crecimiento epidérmico EGFR que funciona aguas arriba de PI3K se activa o sobreexpresa por mutación en el cáncer. [22] [23] Por lo tanto, la actividad de PI3K contribuye significativamente a la transformación celular y al desarrollo del cáncer . Se ha demostrado que las células B malignas mantienen una actividad "tónica" del eje PI3K/Akt a través de la regulación positiva de una proteína adaptadora GAB1, y esto también permite que las células B sobrevivan a la terapia dirigida con inhibidores de BCR. [ cita requerida ]
Aprendizaje y memoria
Las PI3K también han sido implicadas en la potenciación a largo plazo (PLP). Aún se debate si son necesarias para la expresión o la inducción de la LTP. En las neuronas CA1 del hipocampo de ratón , ciertas PI3K forman complejos con receptores AMPA y se compartimentan en la densidad postsináptica de las sinapsis glutamatérgicas . [24] Las PI3K se fosforilan con la actividad CaMKII dependiente del receptor NMDA , [25] y luego facilita la inserción de las subunidades GluR1 del receptor AMPA-R en la membrana plasmática. Esto sugiere que las PI3K son necesarias para la expresión de la LTP. Además, los inhibidores de PI3K eliminaron la expresión de la LTP en el CA1 del hipocampo de rata, pero no afectan su inducción. [26] Cabe destacar que la dependencia de la expresión de la LTP en la fase tardía de las PI3K parece disminuir con el tiempo. [27]
Sin embargo, otro estudio encontró que los inhibidores de PI3K suprimieron la inducción, pero no la expresión, de LTP en CA1 del hipocampo de ratón. [28] La vía PI3K también recluta muchas otras proteínas aguas abajo, incluyendo mTOR , [29] GSK3β , [30] y PSD-95 . [29] La vía PI3K-mTOR conduce a la fosforilación de p70S6K , una quinasa que facilita la actividad traduccional, [31] [32] lo que sugiere además que las PI3K son necesarias para la fase de síntesis de proteínas de la inducción de LTP.
Las PI3K interactúan con el sustrato del receptor de insulina (IRS) para regular la captación de glucosa a través de una serie de eventos de fosforilación.
PI 3-quinasas como proteínas quinasas
Muchas PI3K parecen tener una actividad de serina/treonina quinasa in vitro ; sin embargo, no está claro si esto tiene algún papel in vivo . [ cita requerida ]
Inhibición
Todas las PI3K son inhibidas por los fármacos wortmanina y LY294002 , aunque ciertos miembros de la familia PI3K de clase II muestran una sensibilidad reducida. La wortmanina muestra una mayor eficacia que LY294002 en las posiciones de mutación de puntos calientes (GLU542, GLU545 y HIS1047) [33] [34]
Inhibidores de PI3K como agentes terapéuticos
Como la wortmanina y el LY294002 son inhibidores de amplio espectro de las PI3K y de varias proteínas no relacionadas en concentraciones más altas, son demasiado tóxicos para ser utilizados como agentes terapéuticos. [ cita requerida ] Por ello, varias compañías farmacéuticas han desarrollado inhibidores específicos de la isoforma de PI3K. A partir de enero de 2019, la FDA aprobó tres inhibidores de PI3K para uso clínico de rutina en humanos: el inhibidor de PIK3CD idelalisib (julio de 2014, NDA 206545), el inhibidor dual de PIK3CA y PIK3CD copanlisib (septiembre de 2017, NDA 209936) y el inhibidor dual de PIK3CD y PIK3CG duvelisib (septiembre de 2018, NDA 211155). La inhibición co-dirigida de la vía con otras vías como MAPK o PIM se ha destacado como una estrategia terapéutica anticancerígena prometedora, que podría ofrecer beneficios sobre el enfoque monoterapéutico al eludir la señalización compensatoria, retardar el desarrollo de resistencia y potencialmente permitir la reducción de la dosis. [35] [36] [37] [38] [39]
Véase también
Referencias
- ^ PDB : 2chz ; Knight ZA, Gonzalez B, Feldman ME, Zunder ER, Goldenberg DD, Williams O, et al. (Mayo de 2006). "Un mapa farmacológico de la familia PI3-K define un papel para p110alpha en la señalización de insulina". Cell . 125 (4): 733–47. doi :10.1016/j.cell.2006.03.035. PMC 2946820 . PMID 16647110.
- ^ "mioinositol". Archivado desde el original el 6 de agosto de 2011. Consultado el 28 de enero de 2006 .
- ^ Giese N (2009). "La vía celular en exceso previene la respuesta del cáncer a la restricción dietética". PhysOrg.com . Consultado el 22 de abril de 2009 .
- ^ Kalaany NY, Sabatini DM (abril de 2009). "Los tumores con activación de PI3K son resistentes a la restricción dietética". Nature . 458 (7239): 725–31. Bibcode :2009Natur.458..725K. doi :10.1038/nature07782. PMC 2692085 . PMID 19279572.
- ^ Whitman M, Kaplan DR, Schaffhausen B, Cantley L, Roberts TM (1985). "Asociación de la actividad de la fosfatidilinositol quinasa con células T medias de polioma competentes para la transformación". Nature . 315 (6016): 239–42. Bibcode :1985Natur.315..239W. doi :10.1038/315239a0. PMID 2987699. S2CID 4337999.
- ^ Whitman M, Downes CP, Keeler M, Keller T, Cantley L (abril de 1988). "La fosfatidilinositol quinasa de tipo I produce un nuevo fosfolípido de inositol, el fosfatidilinositol-3-fosfato". Nature . 332 (6165): 644–6. Bibcode :1988Natur.332..644W. doi :10.1038/332644a0. PMID 2833705. S2CID 4326568.
- ^ Auger KR, Serunian LA, Soltoff SP, Libby P, Cantley LC (abril de 1989). "La fosforilación de tirosina dependiente de PDGF estimula la producción de nuevos polifosfoinosítidos en células intactas". Cell . 57 (1): 167–75. doi :10.1016/0092-8674(89)90182-7. PMID 2467744. S2CID 22154860.
- ^ Traynor-Kaplan AE, Harris AL, Thompson BL, Taylor P, Sklar LA (julio de 1988). "Un fosfolípido que contiene tetrakisfosfato de inositol en neutrófilos activados". Nature . 334 (6180): 353–6. Bibcode :1988Natur.334..353T. doi :10.1038/334353a0. PMID 3393226. S2CID 4263472.
- ^ abc Leevers SJ, Vanhaesebroeck B, Waterfield MD (abril de 1999). "Señalización a través de las fosfoinosítido 3-quinasas: los lípidos ocupan un lugar central". Current Opinion in Cell Biology . 11 (2): 219–25. doi :10.1016/S0955-0674(99)80029-5. PMID 10209156.
- ^ Fruman DA, Chiu H, Hopkins BD, Bagrodia S, Cantley LC, Abraham RT (agosto de 2017). "La vía PI3K en enfermedades humanas". Cell . 170 (4): 605–635. doi :10.1016/j.cell.2017.07.029. PMC 5726441 . PMID 28802037.
- ^ Jean S, Kiger AA (marzo de 2014). "Clases de fosfoinosítido 3-quinasas de un vistazo". Journal of Cell Science . 127 (Pt 5): 923–8. doi :10.1242/jcs.093773. PMC 3937771 . PMID 24587488.
- ^ Vanhaesebroeck B, Stephens L, Hawkins P (febrero de 2012). "Señalización de PI3K: el camino hacia el descubrimiento y la comprensión". Nature Reviews. Molecular Cell Biology . 13 (3): 195–203. doi :10.1038/nrm3290. PMID 22358332. S2CID 6999833.
- ^ Okkenhaug K (enero de 2013). "Señalización por la familia de la fosfoinosítido 3-quinasa en células inmunes". Revisión anual de inmunología . 31 (2): 675–704. doi :10.1146/annurev-immunol-032712-095946. PMC 4516760 . PMID 23330955.
- ^ Carpenter CL, Duckworth BC, Auger KR, Cohen B, Schaffhausen BS, Cantley LC (noviembre de 1990). "Purificación y caracterización de la fosfoinosítido 3-quinasa de hígado de rata". The Journal of Biological Chemistry . 265 (32): 19704–11. doi : 10.1016/S0021-9258(17)45429-9 . PMID 2174051.
- ^ Songyang Z, Shoelson SE, Chaudhuri M, Gish G, Pawson T, Haser WG, et al. (marzo de 1993). "Los dominios SH2 reconocen secuencias de fosfopéptidos específicos". Cell . 72 (5): 767–78. doi : 10.1016/0092-8674(93)90404-E . PMID 7680959.
- ^ Yoakim M, Hou W, Songyang Z, Liu Y, Cantley L, Schaffhausen B (septiembre de 1994). "El análisis genético de un dominio SH2 de la fosfatidilinositol 3-quinasa revela determinantes de especificidad". Biología molecular y celular . 14 (9): 5929–38. doi :10.1128/MCB.14.9.5929. PMC 359119 . PMID 8065326.
- ^ Posor Y, Eichhorn-Grünig M, Haucke V (junio de 2015). "Fosfoinosítidos en endocitosis". Biochimica et Biophysica Acta (BBA) - Biología molecular y celular de los lípidos . 1851 (6): 794–804. doi :10.1016/j.bbalip.2014.09.014. PMID 25264171.
- ^ Franke TF, Kaplan DR, Cantley LC, Toker A (enero de 1997). "Regulación directa del producto del protooncogén Akt por fosfatidilinositol-3,4-bisfosfato". Science . 275 (5300): 665–8. doi :10.1126/science.275.5300.665. PMID 9005852. S2CID 31186873.
- ^ Gómez Del Pulgar T, De Ceballos ML, Guzmán M, Velasco G (septiembre de 2002). "Los cannabinoides protegen a los astrocitos de la apoptosis inducida por ceramida a través de la vía fosfatidilinositol 3-quinasa/proteína quinasa B". The Journal of Biological Chemistry . 277 (39): 36527–33. doi : 10.1074/jbc.M205797200 . PMID 12133838.
- ^ Welch HC, Coadwell WJ, Stephens LR, Hawkins PT (julio de 2003). "Activación de Rac dependiente de la fosfoinosítido 3-quinasa". FEBS Letters . 546 (1): 93–7. doi : 10.1016/s0014-5793(03)00454-x . PMID 12829242.
- ^ Jaffe AB, Hall A (2005). "GTPasas Rho: bioquímica y biología". Revisión anual de biología celular y del desarrollo . 21 : 247–69. doi :10.1146/annurev.cellbio.21.020604.150721. PMID 16212495.
- ^ ab Bleeker FE, Lamba S, Zanon C, Molenaar RJ, Hulsebos TJ, Troost D, et al. (septiembre de 2014). "Perfil mutacional de quinasas en glioblastoma". Cáncer BMC . 14 : 718. doi : 10.1186/1471-2407-14-718 . PMC 4192443 . PMID 25256166.
- ^ Bleeker FE, Molenaar RJ, Leenstra S (mayo de 2012). "Avances recientes en la comprensión molecular del glioblastoma". Journal of Neuro-Oncology . 108 (1): 11–27. doi :10.1007/s11060-011-0793-0. PMC 3337398 . PMID 22270850.
- ^ Man HY, Wang Q, Lu WY, Ju W, Ahmadian G, Liu L, et al. (mayo de 2003). "La activación de la PI3-quinasa es necesaria para la inserción del receptor AMPA durante la LTP de las mEPSC en neuronas hipocampales cultivadas". Neuron . 38 (4): 611–24. doi : 10.1016/S0896-6273(03)00228-9 . PMID 12765612.
- ^ Joyal JL, Burks DJ, Pons S, Matter WF, Vlahos CJ, White MF, Sacks DB (noviembre de 1997). "La calmodulina activa la fosfatidilinositol 3-quinasa". The Journal of Biological Chemistry . 272 (45): 28183–6. doi : 10.1074/jbc.272.45.28183 . PMID 9353264.
- ^ Sanna PP, Cammalleri M, Berton F, Simpson C, Lutjens R, Bloom FE, Francesconi W (mayo de 2002). "La fosfatidilinositol 3-quinasa es necesaria para la expresión, pero no para la inducción o el mantenimiento de la potenciación a largo plazo en la región CA1 del hipocampo". The Journal of Neuroscience . 22 (9): 3359–65. doi :10.1523/JNEUROSCI.22-09-03359.2002. PMC 6758361 . PMID 11978812.
- ^ Karpova A, Sanna PP, Behnisch T (febrero de 2006). "Participación de múltiples vías dependientes de la fosfatidilinositol 3-quinasa en la persistencia de la expresión de potenciación a largo plazo en fase tardía". Neurociencia . 137 (3): 833–41. doi :10.1016/j.neuroscience.2005.10.012. PMID 16326012. S2CID 38232127.
- ^ Opazo P, Watabe AM, Grant SG, O'Dell TJ (mayo de 2003). "La fosfatidilinositol 3-quinasa regula la inducción de la potenciación a largo plazo a través de mecanismos independientes de la quinasa relacionados con la señal extracelular". The Journal of Neuroscience . 23 (9): 3679–88. doi :10.1523/JNEUROSCI.23-09-03679.2003. PMC 6742185 . PMID 12736339.
- ^ ab Yang PC, Yang CH, Huang CC, Hsu KS (febrero de 2008). "La activación de la fosfatidilinositol 3-quinasa es necesaria para la modificación de la plasticidad sináptica del hipocampo inducida por el protocolo de estrés". The Journal of Biological Chemistry . 283 (5): 2631–43. doi : 10.1074/jbc.M706954200 . PMID 18057005.
- ^ Peineau S, Taghibiglou C, Bradley C, Wong TP, Liu L, Lu J, et al. (marzo de 2007). "LTP inhibe LTD en el hipocampo a través de la regulación de GSK3beta". Neuron . 53 (5): 703–17. doi : 10.1016/j.neuron.2007.01.029 . PMID 17329210. S2CID 6903401.
- ^ Toker A, Cantley LC (junio de 1997). "Señalización a través de los productos lipídicos de la fosfoinosítido-3-OH quinasa". Nature . 387 (6634): 673–6. Bibcode :1997Natur.387..673T. doi : 10.1038/42648 . PMID 9192891. S2CID 4347728.
- ^ Cammalleri M, Lütjens R, Berton F, King AR, Simpson C, Francesconi W, Sanna PP (noviembre de 2003). "Función restringida en el tiempo de la activación dendrítica de la vía mTOR-p70S6K en la inducción de la potenciación a largo plazo de fase tardía en el CA1". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 100 (24): 14368–73. Bibcode :2003PNAS..10014368C. doi : 10.1073/pnas.2336098100 . PMC 283598 . PMID 14623952.
- ^ Kumar DT, Doss CG (1 de enero de 2016). Investigación del efecto inhibidor de la wortmanina en la mutación de punto caliente en el codón 1047 del dominio de la quinasa PIK3CA: un enfoque de acoplamiento molecular y dinámica molecular . Vol. 102. págs. 267–97. doi :10.1016/bs.apcsb.2015.09.008. ISBN 9780128047958. Número de identificación personal 26827608.
{{cite book}}:|journal=ignorado ( ayuda ) - ^ Sudhakar N, Priya Doss CG, Thirumal Kumar D, Chakraborty C, Anand K, Suresh M (2 de enero de 2016). "Descifrando el impacto de las mutaciones somáticas en el exón 20 y el exón 9 del gen PIK3CA en tumores de mama entre mujeres indias a través de un enfoque de dinámica molecular". Revista de estructura y dinámica biomolecular . 34 (1): 29–41. doi :10.1080/07391102.2015.1007483. PMID 25679319. S2CID 205575161.
- ^ Malone T, Schäfer L, Simon N, Heavey S, Cuffe S, Finn S, et al. (marzo de 2020). "Perspectivas actuales sobre la focalización de las quinasas PIM para superar los mecanismos de resistencia a los fármacos y la evasión inmunitaria en el cáncer" (PDF) . Farmacología y terapéutica . 207 : 107454. doi :10.1016/j.pharmthera.2019.107454. PMID 31836451. S2CID 209357486.
- ^ Luszczak S, Kumar C, Sathyadevan VK, Simpson BS, Gately KA, Whitaker HC, Heavey S (2020). "Inhibición de la quinasa PIM: enfoques terapéuticos codirigidos en el cáncer de próstata". Transducción de señales y terapia dirigida . 5 : 7. doi :10.1038/s41392-020-0109-y. PMC 6992635. PMID 32025342 .
- ^ Heavey S, Dowling P, Moore G, Barr MP, Kelly N, Maher SG, et al. (enero de 2018). "Desarrollo y caracterización de un panel de fosfatidilinositol 3-quinasa: diana mamífera de líneas celulares de cáncer de pulmón resistentes al inhibidor de la rapamicina". Scientific Reports . 8 (1): 1652. Bibcode :2018NatSR...8.1652H. doi :10.1038/s41598-018-19688-1. PMC 5786033 . PMID 29374181.
- ^ Heavey S, Godwin P, Baird AM, Barr MP, Umezawa K, Cuffe S, et al. (octubre de 2014). "Dirección estratégica del eje PI3K-NFκB en NSCLC resistente al cisplatino". Biología y terapia del cáncer . 15 (10): 1367–77. doi :10.4161/cbt.29841. PMC 4130730 . PMID 25025901.
- ^ Heavey S, O'Byrne KJ, Gately K (abril de 2014). "Estrategias para la co-estrategia de la vía PI3K/AKT/mTOR en el CPCNP". Cancer Treatment Reviews . 40 (3): 445–56. doi :10.1016/j.ctrv.2013.08.006. PMID 24055012.
Lectura adicional
- Vanhaesebroeck B, Leevers SJ, Ahmadi K, Timms J, Katso R, Driscoll PC, et al. (2001). "Síntesis y función de los lípidos de inositol 3-fosforilados". Revisión anual de bioquímica . 70 : 535–602. doi :10.1146/annurev.biochem.70.1.535. PMID 11395417.[1]
- Schild C, Wirth M, Reichert M, Schmid RM, Saur D, Schneider G (diciembre de 2009). "La señalización de PI3K mantiene la expresión de c-myc para regular la transcripción de E2F1 en células de cáncer de páncreas". Molecular Carcinogenesis . 48 (12): 1149–58. doi :10.1002/mc.20569. PMID 19603422. S2CID 41545085.
- Williams R, Berndt A, Miller S, Hon WC, Zhang X (agosto de 2009). "Forma y flexibilidad en las fosfoinosítido 3-quinasas". Biochemical Society Transactions . 37 (Pt 4): 615–26. doi :10.1042/BST0370615. PMID 19614567.
- Quaresma AJ, Sievert R, Nickerson JA (abril de 2013). "Regulación de la exportación de ARNm por la vía de transducción de señales PI3 quinasa/AKT". Biología molecular de la célula . 24 (8): 1208–21. doi :10.1091/mbc.E12-06-0450. PMC 3623641 . PMID 23427269.
Enlaces externos
- Clase de motivo de recurso de motivo lineal eucariótico MOD_PIKK_1
- Proteopedia Phosphoinositide_3-Kinases para explorar la estructura en 3D interactivo
- PI-3+Kinase en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- Vía de señalización PI3K/Akt
