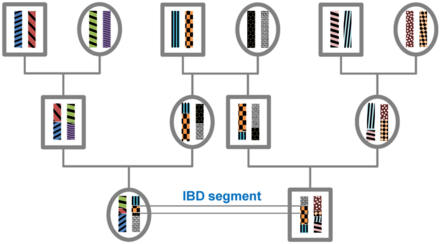
Identidad por descendencia
Un segmento de ADN es idéntico por estado (IBS) en dos o más individuos si tienen secuencias de nucleótidos idénticas en este segmento. Un segmento IBS es idéntico por descendencia ( IBD ) en dos o más individuos si lo han heredado de un ancestro común sin recombinación , es decir, el segmento tiene el mismo origen ancestral en estos individuos. Los segmentos de ADN que son IBD son IBS por definición, pero los segmentos que no son IBD pueden seguir siendo IBS debido a las mismas mutaciones en diferentes individuos o recombinaciones que no alteran el segmento. [ cita requerida ]

Teoría
Todos los individuos de una población finita están relacionados si se los rastrea lo suficiente y, por lo tanto, compartirán segmentos de sus genomas IBD. Durante la meiosis, los segmentos de IBD se dividen por recombinación. Por lo tanto, la longitud esperada de un segmento IBD depende del número de generaciones desde el ancestro común más reciente en el locus del segmento. La longitud de los segmentos IBD que resultan de un ancestro común n generaciones en el pasado (por lo tanto, involucrando 2 n meiosis) se distribuye exponencialmente con media 1/(2 n ) Morgans (M). [1] El número esperado de segmentos IBD disminuye con el número de generaciones desde el ancestro común en este locus. Para un segmento de ADN específico, la probabilidad de ser IBD disminuye como 2 −2 n ya que en cada meiosis la probabilidad de transmitir este segmento es 1/2. [2]
Aplicaciones
Los segmentos de IBD identificados se pueden utilizar para una amplia gama de propósitos. Como se señaló anteriormente, la cantidad (longitud y número) de IBD compartida depende de las relaciones familiares entre los individuos evaluados. Por lo tanto, una aplicación de la detección de segmentos de IBD es cuantificar el parentesco. [3] [4] [5] [6] La medición del parentesco se puede utilizar en genética forense , [7] pero también puede aumentar la información en el mapeo de ligamiento genético [3] [8] y ayudar a disminuir el sesgo por relaciones no documentadas en estudios de asociación estándar . [6] [9] Otra aplicación de IBD es la imputación de genotipos y la inferencia de fase de haplotipos . [10] [11] [12] Los segmentos compartidos largos de IBD, que están divididos por regiones cortas, pueden ser indicativos de errores de fase. [5] [13] : SI
Mapeo de la EII
El mapeo de la EII [3] es similar al análisis de ligamiento, pero se puede realizar sin un pedigrí conocido en una cohorte de individuos no relacionados. El mapeo de la EII puede verse como una nueva forma de análisis de asociación que aumenta el poder de mapear genes o regiones genómicas que contienen múltiples variantes de susceptibilidad a enfermedades raras. [6] [14]
Utilizando datos simulados, Browning y Thompson demostraron que el mapeo de IBD tiene mayor poder que las pruebas de asociación cuando múltiples variantes raras dentro de un gen contribuyen a la susceptibilidad a la enfermedad. [14] A través del mapeo de IBD, se encontraron regiones significativas de todo el genoma en poblaciones aisladas, así como en poblaciones exogámicas, mientras que las pruebas de asociación estándar fallaron. [11] [15] Houwen et al. utilizaron el intercambio de IBD para identificar la ubicación cromosómica de un gen responsable de la colestasis intrahepática recurrente benigna en una población de pescadores aislada. [16] Kenny et al. también utilizaron una población aislada para mapear con precisión una señal encontrada por un estudio de asociación de todo el genoma (GWAS) de los niveles de esterol vegetal (PPS) plasmático, una medida sustituta de la absorción de colesterol del intestino. [17] Francks et al. pudieron identificar un locus de susceptibilidad potencial para la esquizofrenia y el trastorno bipolar con datos de genotipo de muestras de casos y controles. [18] Lin et al. encontraron una señal de ligamiento significativa de todo el genoma en un conjunto de datos de pacientes con esclerosis múltiple . [19] Letouzé et al. utilizaron el mapeo de IBD para buscar mutaciones fundadoras en muestras de cáncer . [20]
La enfermedad inflamatoria intestinal en la genética de poblaciones
La detección de la selección natural en el genoma humano también es posible a través de la detección de segmentos IBD. La selección suele tender a aumentar el número de segmentos IBD entre los individuos de una población. Al buscar regiones con exceso de segmentos IBD compartidos, se pueden identificar regiones del genoma humano que han estado bajo una selección fuerte y muy reciente. [21] [22]
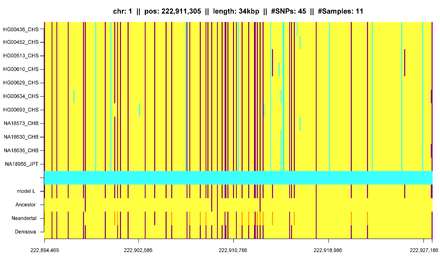
Además de eso, los segmentos de IBD pueden ser útiles para medir e identificar otras influencias en la estructura de la población. [6] [23] [24] [25] [26] Gusev et al. demostraron que los segmentos de IBD se pueden utilizar con modelos adicionales para estimar la historia demográfica, incluidos los cuellos de botella y la mezcla . [24] Utilizando modelos similares, Palamara et al. y Carmi et al. reconstruyeron la historia demográfica de los individuos judíos asquenazíes y masai kenianos . [25] [26] [27] Botigué et al. investigaron las diferencias en la ascendencia africana entre las poblaciones europeas. [28] Ralph y Coop utilizaron la detección de IBD para cuantificar la ascendencia común de diferentes poblaciones europeas [29] y Gravel et al. intentaron de manera similar sacar conclusiones de la historia genética de las poblaciones en las Américas. [30] Ringbauer et al. utilizaron la estructura geográfica de los segmentos de IBD para estimar la dispersión dentro de Europa del Este durante los últimos siglos. [31] Utilizando los datos de 1000 Genomas , Hochreiter encontró diferencias en la compartición de IBD entre poblaciones africanas, asiáticas y europeas, así como segmentos de IBD que se comparten con genomas antiguos como el neandertal o el denisovano . [13]
Métodos y software
Programas para la detección de segmentos de EII en individuos no relacionados:
- RAPID: Detección ultrarrápida de identidad por descendencia en cohortes a escala de biobanco mediante la transformación posicional de Burrows-Wheeler [32]
- Parente: identifica segmentos de IBD entre pares de individuos en datos de genotipo no escalonados [33]
- BEAGLE/fastIBD: encuentra segmentos de IBD entre pares de individuos en datos SNP de todo el genoma [34]
- BEAGLE/RefinedIBD: encuentra segmentos de IBD en pares de individuos utilizando un método de hash y evalúa su importancia a través de una razón de verosimilitud [35]
- IBDseq: detecta segmentos IBD por pares en datos de secuenciación [36]
- LÍNEA GERMINAL: descubre en tiempo lineal segmentos de IBD en pares de individuos [5]
- DASH: se basa en segmentos de IBD por pares para inferir grupos de individuos que probablemente compartan un solo haplotipo [15]
- PLINK: es un conjunto de herramientas para la asociación de todo el genoma y análisis de ligamiento basados en la población que incluye un método para la detección de segmentos de IBD por pares [6]
- Relacionar: estima la probabilidad de EII entre pares de individuos en un locus específico utilizando SNP [3]
- MCMC_IBDfinder: se basa en la cadena de Markov Monte Carlo (MCMC) para encontrar segmentos de IBD en múltiples individuos [37]
- IBD-Groupon: detecta segmentos de IBD por grupo basándose en relaciones de IBD por pares [38]
- HapFABIA: identifica segmentos de EII muy cortos caracterizados por variantes raras en datos de secuenciación grandes simultáneamente en múltiples individuos [13]
Véase también
- Mapeo de asociaciones
- Asociación genética
- Vinculación genética
- Estudio de asociación de todo el genoma
- Identidad por tipo
- Desequilibrio de ligamiento
- Genética de poblaciones
Referencias
- ^ Browning, SR (2008). "Estimación de la identidad por pares por descendencia a partir de datos de marcadores genéticos densos en una muestra poblacional de haplotipos". Genética . 178 (4): 2123–2132. doi :10.1534/genetics.107.084624. PMC 2323802 . PMID 18430938.
- ^ Thompson, EA (2008). "El proceso de la enfermedad inflamatoria intestinal a lo largo de cuatro cromosomas". Biología poblacional teórica . 73 (3): 369–373. doi :10.1016/j.tpb.2007.11.011. PMC 2518088 . PMID 18282591.
- ^ abcd Albrechtsen, A.; Sand Korneliussen, T.; Moltke, I.; Van Overseem Hansen, T.; Nielsen, FC; Nielsen, R. (2009). "Mapeo de parentesco y tramos de parentesco para datos de todo el genoma en presencia de desequilibrio de ligamiento". Epidemiología genética . 33 (3): 266–274. doi :10.1002/gepi.20378. PMID 19025785. S2CID 12029712.
- ^ Browning, SR; Browning, BL (2010). "Detección de alta resolución de identidad por descendencia en individuos no relacionados". The American Journal of Human Genetics . 86 (4): 526–539. doi :10.1016/j.ajhg.2010.02.021. PMC 2850444 . PMID 20303063.
- ^ abc Gusev, A.; Lowe, JK; Stoffel, M.; Daly, MJ; Altshuler, D.; Breslow, JL; Friedman, JM; Pe'Er, I. (2008). "Mapeo de parentesco oculto en toda la población y en todo el genoma". Genome Research . 19 (2): 318–326. doi :10.1101/gr.081398.108. PMC 2652213 . PMID 18971310.
- ^ abcde Purcell, S.; Neale, B.; Todd-Brown, K.; Thomas, L.; Ferreira, MAR; Bender, D.; Maller, J.; Sklar, P.; De Bakker, PIW; Daly, MJ; Sham, PC (2007). "PLINK: un conjunto de herramientas para la asociación del genoma completo y los análisis de ligamiento basados en la población". The American Journal of Human Genetics . 81 (3): 559–575. doi :10.1086/519795. PMC 1950838 . PMID 17701901.
- ^ Ian W. Evett; Bruce S. Weir (enero de 1998). Interpretación de la evidencia de ADN: genética estadística para científicos forenses. Sinauer Associates, Incorporated. ISBN 978-0-87893-155-2.
- ^ Leutenegger, A.; Prum, B.; Genin, E.; Verny, C.; Lemainque, A.; Clergetdarpoux, F.; Thompson, E. (2003). "Estimación del coeficiente de endogamia mediante el uso de datos genómicos". The American Journal of Human Genetics . 73 (3): 516–523. doi :10.1086/378207. PMC 1180677 . PMID 12900793.
- ^ Voight, BF; Pritchard, JK (2005). "Confusión a partir de la relación críptica en estudios de asociación de casos y controles". PLOS Genetics . 1 (3): e32. doi : 10.1371/journal.pgen.0010032 . PMC 1200427 . PMID 16151517.
- ^ Kong, A.; Masón, G.; Frigge, ML; Gylfason, A.; Zusmanovich, P.; Thorleifsson, G.; Olason, PI; Ingason, A.; Steinberg, S.; Rafnar, T.; Sulem, P.; Mouy, M.; Jonsson, F.; Thorsteinsdóttir, U.; Gudbjartsson, DF; Stefansson, H.; Stefansson, K. (2008). "Detección de compartición por descendencia, fase de largo alcance e imputación de haplotipos". Genética de la Naturaleza . 40 (9): 1068-1075. doi :10.1038/ng.216. PMC 4540081 . PMID 19165921.
- ^ ab Gusev, A.; Shah, MJ; Kenny, EE; Ramachandran, A.; Lowe, JK; Salit, J.; Lee, CC; Levandowsky, EC; Weaver, TN; Doan, QC; Peckham, HE; McLaughlin, SF; Lyons, MR; Sheth, VN; Stoffel, M.; De La Vega, FM; Friedman, JM; Breslow, JL; Pe'Er, I. (2011). "Secuenciación de todo el genoma de paso bajo e inferencia de variantes utilizando identidad por descendencia en una población humana aislada". Genética . 190 (2): 679–689. doi :10.1534/genetics.111.134874. PMC 3276614 . PMID 22135348.
- ^ Browning, BL; Browning, SR (2009). "Un enfoque unificado para la imputación de genotipos y la inferencia de fases de haplotipos para grandes conjuntos de datos de tríos e individuos no relacionados". The American Journal of Human Genetics . 84 (2): 210–223. doi :10.1016/j.ajhg.2009.01.005. PMC 2668004 . PMID 19200528.
- ^ abc Hochreiter, S. (2013). "HapFABIA: Identificación de segmentos muy cortos de identidad por descendencia caracterizados por variantes raras en datos de secuenciación de gran tamaño". Investigación de ácidos nucleicos . 41 (22): e202. doi :10.1093/nar/gkt1013. PMC 3905877 . PMID 24174545.
- ^ ab Browning, SR; Thompson, EA (2012). "Detección de asociaciones de variantes raras mediante mapeo de identidad por descendencia en estudios de casos y controles". Genética . 190 (4): 1521–1531. doi :10.1534/genetics.111.136937. PMC 3316661 . PMID 22267498.
- ^ ab Gusev, A.; Kenny, EE; Lowe, JK; Salit, J.; Saxena, R.; Kathiresan, S.; Altshuler, DM; Friedman, JM; Breslow, JL; Pe'Er, I. (2011). "DASH: un método para el mapeo de haplotipos idénticos por descendencia descubre una asociación con variación reciente". The American Journal of Human Genetics . 88 (6): 706–717. doi :10.1016/j.ajhg.2011.04.023. PMC 3113343 . PMID 21620352.
- ^ Houwen, RHJ; Baharloo, S.; Blankenship, K.; Raeymaekers, P.; Juyn, J.; Sandkuijl, LA; Freimer, NB (1994). "Cribado genómico mediante la búsqueda de segmentos compartidos: mapeo de un gen para la colestasis intrahepática recurrente benigna". Nature Genetics . 8 (4): 380–386. doi :10.1038/ng1294-380. hdl : 1765/55192 . PMID 7894490. S2CID 8131209.
- ^ Kenny, EE; Gusev, A.; Riegel, K.; Lutjohann, D.; Lowe, JK; Salit, J.; Maller, JB; Stoffel, M.; Daly, MJ; Altshuler, DM; Friedman, JM; Breslow, JL; Pe'Er, I.; Sehayek, E. (2009). "El análisis sistemático de haplotipos resuelve un locus complejo de esteroles vegetales plasmáticos en la isla micronesia de Kosrae". Actas de la Academia Nacional de Ciencias . 106 (33): 13886–13891. Bibcode :2009PNAS..10613886K. doi : 10.1073/pnas.0907336106 . PMC 2728990 . PMID 19667188.
- ^ Francks, C.; Tozzi, F.; Farmer, A.; Vincent, JB; Rujescu, D.; St Clair, D.; Muglia, P. (2008). "El análisis de vinculación basado en la población de cohortes de casos y controles de esquizofrenia y trastorno bipolar identifica un locus de susceptibilidad potencial en 19q13". Psiquiatría molecular . 15 (3): 319–325. doi : 10.1038/mp.2008.100 . hdl : 11858/00-001M-0000-0012-C935-9 . PMID 18794890.
- ^ Lin, R.; Charlesworth, J.; Stankovich, J.; Perreau, VM; Brown, MA; Anzgene, BV; Taylor, BV (2013). Toland, Amanda Ewart (ed.). "Mapeo de identidad por descendencia para detectar variantes raras que confieren susceptibilidad a la esclerosis múltiple". PLOS ONE . 8 (3): e56379. Bibcode :2013PLoSO...856379L. doi : 10.1371/journal.pone.0056379 . PMC 3589405 . PMID 23472070.
- ^ Letouzé, E.; Sow, A.; Petel, F.; Rosati, R.; Figueiredo, BC; Burnichon, N.; Gimenez-Roqueplo, AP; Lalli, E.; De Reyniès, AL (2012). Mailund, Thomas (ed.). "Identidad por mapeo de descendencia de mutaciones fundadoras en cáncer utilizando datos de SNP tumorales de alta resolución". PLOS ONE . 7 (5): e35897. Bibcode :2012PLoSO...735897L. doi : 10.1371/journal.pone.0035897 . PMC 3342326 . PMID 22567117.
- ^ Albrechtsen, A.; Moltke, I.; Nielsen, R. (2010). "Selección natural y distribución de la identidad por descendencia en el genoma humano". Genética . 186 (1): 295–308. doi :10.1534/genetics.110.113977. PMC 2940294 . PMID 20592267.
- ^ Han, L.; Abney, M. (2011). "Estimación de identidad por descendencia con datos de genotipos densos de todo el genoma". Epidemiología genética . 35 (6): 557–567. doi :10.1002/gepi.20606. PMC 3587128 . PMID 21769932.
- ^ Cockerham, CC; Weir, BS (1983). "Varianza de la endogamia real". Biología de poblaciones teórica . 23 (1): 85–109. doi :10.1016/0040-5809(83)90006-0. PMID 6857551.
- ^ ab Gusev, A.; Palamara, PF; Aponte, G.; Zhuang, Z.; Darvasi, A.; Gregersen, P.; Pe'Er, I. (2011). "La arquitectura de haplotipos de largo alcance compartidos dentro y entre poblaciones". Biología molecular y evolución . 29 (2): 473–486. doi :10.1093/molbev/msr133. PMC 3350316 . PMID 21984068.
- ^ ab Palamara, PF; Lencz, T.; Darvasi, A.; Pe'Er, I. (2012). "Distribuciones de longitud de identidad por descendencia revelan una historia demográfica a escala fina". The American Journal of Human Genetics . 91 (5): 809–822. doi :10.1016/j.ajhg.2012.08.030. PMC 3487132 . PMID 23103233.
- ^ ab Palamara, PF; Pe'Er, I. (2013). "Inferencia de tasas históricas de migración a través del intercambio de haplotipos". Bioinformática . 29 (13): i180–i188. doi :10.1093/bioinformatics/btt239. PMC 3694674 . PMID 23812983.
- ^ Carmi, S.; Palamara, PF; Vacic, V.; Lencz, T.; Darvasi, A.; Pe'Er, I. (2013). "La varianza de la identidad compartida por descendencia en el modelo de Wright-Fisher". Genética . 193 (3): 911–928. arXiv : 1206.4745 . doi :10.1534/genetics.112.147215. PMC 3584006 . PMID 23267057.
- ^ Botigue, LR; Henn, BM; Gravel, S.; Maples, BK; Gignoux, CR; Corona, E.; Atzmon, G.; Burns, E.; Ostrer, H.; Flores, C.; Bertranpetit, J.; Comas, D.; Bustamante, CD (2013). "El flujo genético del norte de África contribuye a la diversidad genética humana diferencial en el sur de Europa". Actas de la Academia Nacional de Ciencias . 110 (29): 11791–11796. Bibcode :2013PNAS..11011791B. doi : 10.1073/pnas.1306223110 . PMC 3718088 . PMID 23733930.
- ^ Ralph, P.; Coop, G. (2013). Tyler-Smith, Chris (ed.). "La geografía de la ascendencia genética reciente en toda Europa". PLOS Biology . 11 (5): e1001555. doi : 10.1371/journal.pbio.1001555 . PMC 3646727 . PMID 23667324.
- ^ Gravel, S.; Zakharia, F.; Moreno-Estrada, A.; Byrnes, JK; Muzzio, M.; Rodriguez-Flores, JL; Kenny, EE; Gignoux, CR; Maples, BK; Guiblet, W.; Dutil, J.; Via, M.; Sandoval, K.; Bedoya, G.; 1000 Genomes, TK; Oleksyk, A.; Ruiz-Linares, EG; Burchard, JC; Martinez-Cruzado, CD; Bustamante, CD (2013). Williams, Scott M (ed.). "Reconstrucción de las migraciones de los nativos americanos a partir de datos de todo el genoma y todo el exoma". PLOS Genetics . 9 (12): e1004023. arXiv : 1306.4021 . doi : 10.1371/journal.pgen.1004023 . PMC 3873240. PMID 24385924 .
{{cite journal}}: CS1 maint: nombres numéricos: lista de autores ( enlace ) - ^ Ringbauer, Harald; Coop, Graham; Barton, Nicholas H. (1 de marzo de 2017). "Inferencia de la demografía reciente a partir del aislamiento por distancia de bloques de secuencias compartidas de larga duración". Genética . 205 (3): 1335–1351. doi :10.1534/genetics.116.196220. ISSN 0016-6731. PMC 5340342 . PMID 28108588.
- ^ Naseri A, Liu X, Zhang S, Zhi D. Identidad ultrarrápida por detección de descendencia en cohortes a escala de biobanco utilizando la transformada posicional Burrows-Wheeler BioRxiv 2017.
- ^ Rodriguez JM, Batzoglou S, Bercovici S. Un método preciso para inferir parentesco en grandes conjuntos de datos de genotipos no en fase mediante una prueba de razón de verosimilitud integrada. RECOMB 2013, LNBI 7821:212-229.
- ^ Browning, BL; Browning, SR (2011). "Un método rápido y potente para detectar la identidad por descendencia". The American Journal of Human Genetics . 88 (2): 173–182. doi :10.1016/j.ajhg.2011.01.010. PMC 3035716 . PMID 21310274.
- ^ Browning, BL; Browning, SR (2013). "Mejora de la precisión y eficiencia de la detección de identidad por descendencia en datos de población". Genética . 194 (2): 459–471. doi :10.1534/genetics.113.150029. PMC 3664855 . PMID 23535385.
- ^ Browning, BL; Browning, SR (2013). "Detección de identidad por descendencia y estimación de tasas de error de genotipo en datos de secuencia". The American Journal of Human Genetics . 93 (5): 840–851. doi :10.1016/j.ajhg.2013.09.014. PMC 3824133 . PMID 24207118.
- ^ Moltke, I.; Albrechtsen, A.; Hansen, TVO; Nielsen, FC; Nielsen, R. (2011). "Un método para detectar regiones de IBD simultáneamente en múltiples individuos, con aplicaciones a la genética de enfermedades". Genome Research . 21 (7): 1168–1180. doi :10.1101/gr.115360.110. PMC 3129259 . PMID 21493780.
- ^ He, D. (2013). "IBD-Groupon: Un método eficiente para detectar regiones de identidad por descendencia grupales simultáneamente en múltiples individuos en función de relaciones IBD por pares". Bioinformática . 29 (13): i162–i170. doi :10.1093/bioinformatics/btt237. PMC 3694672 . PMID 23812980.