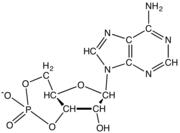
- AMPc , una molécula de señalización de nucleótidos cíclicos con un único fosfato unido a las posiciones 5 y 3.
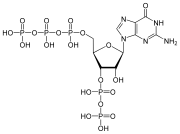
- pppGpp , una molécula de señalización de nucleótidos con fosfatos 5' y 3'.
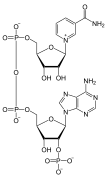
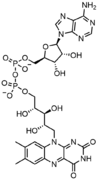
- FAD , un cofactor enzimático dinucleótido en el que uno de los azúcares ribosa adopta una configuración lineal en lugar de un anillo.
Nucleótido
| Parte de una serie sobre |
| Genética |
|---|
 |
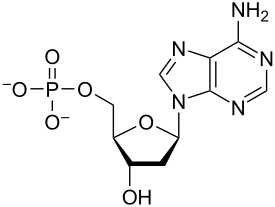
Los nucleótidos son moléculas orgánicas compuestas por una base nitrogenada, un azúcar pentosa y un fosfato . Actúan como unidades monoméricas de los polímeros de ácidos nucleicos : ácido desoxirribonucleico (ADN) y ácido ribonucleico (ARN), ambos biomoléculas esenciales en todas las formas de vida de la Tierra . Los nucleótidos se obtienen en la dieta y también se sintetizan a partir de nutrientes comunes en el hígado . [1]
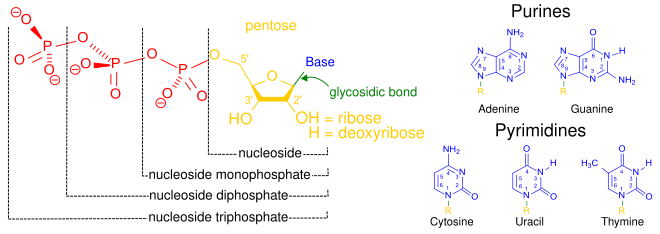
Los nucleótidos están compuestos por tres subunidades moleculares: una nucleobase , un azúcar de cinco carbonos ( ribosa o desoxirribosa ) y un grupo fosfato que consta de uno a tres fosfatos . Las cuatro nucleobases del ADN son guanina , adenina , citosina y timina ; en el ARN, se utiliza uracilo en lugar de timina.
Los nucleótidos también desempeñan un papel central en el metabolismo a un nivel celular fundamental. Proporcionan energía química, en forma de trifosfatos de nucleósidos , trifosfato de adenosina (ATP), trifosfato de guanosina (GTP), trifosfato de citidina (CTP) y trifosfato de uridina (UTP), en toda la célula para las muchas funciones celulares que demandan energía, incluyendo: la síntesis de aminoácidos , proteínas y membrana celular , el movimiento de la célula y partes celulares (tanto interna como intercelularmente), la división celular, etc. [2] Además, los nucleótidos participan en la señalización celular ( monofosfato de guanosina cíclico o cGMP y monofosfato de adenosina cíclico o cAMP) y se incorporan a cofactores importantes de reacciones enzimáticas (p. ej., coenzima A , FAD , FMN , NAD y NADP + ).
En bioquímica experimental , los nucleótidos pueden marcarse radiactivamente utilizando radionúclidos para producir radionucleótidos.
Los 5-nucleótidos también se utilizan en potenciadores del sabor como aditivos alimentarios para realzar el sabor umami , a menudo en forma de extracto de levadura. [3]
Estructura

Un nucleótido está compuesto por tres subunidades químicas distintivas: una molécula de azúcar de cinco carbonos, una nucleobase (las dos juntas se denominan nucleósido monofosfato ), y un grupo fosfato . Cuando se unen los tres, un nucleótido también se denomina "nucleósido monofosfato " , "nucleósido difosfato " o "nucleósido trifosfato ", según la cantidad de fosfatos que componen el grupo fosfato. [4]
En los ácidos nucleicos , los nucleótidos contienen una base de purina o de pirimidina (es decir, la molécula de nucleobase, también conocida como base nitrogenada) y se denominan ribonucleótidos si el azúcar es ribosa, o desoxirribonucleótidos si el azúcar es desoxirribosa. Las moléculas de fosfato individuales conectan repetidamente las moléculas del anillo de azúcar en dos monómeros de nucleótidos adyacentes, conectando así los monómeros de nucleótidos de un ácido nucleico de extremo a extremo en una cadena larga. Estas uniones de cadenas de moléculas de azúcar y fosfato crean una cadena de "columna vertebral" para una hélice simple o doble . En cualquier cadena, la orientación química ( direccionalidad ) de las uniones de cadenas va desde el extremo 5' hasta el extremo 3' ( léase : extremo primo 5 a extremo primo 3), refiriéndose a los cinco sitios de carbono en las moléculas de azúcar en nucleótidos adyacentes. En una doble hélice, las dos hebras están orientadas en direcciones opuestas, lo que permite el apareamiento de bases y la complementariedad entre los pares de bases, todo lo cual es esencial para replicar o transcribir la información codificada que se encuentra en el ADN. [ cita requerida ]
Los ácidos nucleicos son macromoléculas poliméricas ensambladas a partir de nucleótidos, las unidades monoméricas de los ácidos nucleicos . Las bases púricas adenina y guanina y la base pirimidínica citosina se encuentran tanto en el ADN como en el ARN, mientras que las bases pirimidínicas timina (en el ADN) y uracilo (en el ARN) se encuentran en una sola. La adenina forma un par de bases con la timina con dos enlaces de hidrógeno, mientras que la guanina se empareja con la citosina con tres enlaces de hidrógeno.
Además de ser bloques de construcción para la construcción de polímeros de ácidos nucleicos, los nucleótidos singulares desempeñan funciones en el almacenamiento y provisión de energía celular, señalización celular, como fuente de grupos fosfato utilizados para modular la actividad de proteínas y otras moléculas de señalización, y como cofactores enzimáticos , a menudo llevando a cabo reacciones redox . Los nucleótidos cíclicos de señalización se forman uniendo el grupo fosfato dos veces a la misma molécula de azúcar , uniendo los grupos hidroxilo 5' y 3' del azúcar. [2] Algunos nucleótidos de señalización difieren de la configuración estándar de un solo grupo fosfato, en que tienen múltiples grupos fosfato unidos a diferentes posiciones en el azúcar. [5] Los cofactores de nucleótidos incluyen una gama más amplia de grupos químicos unidos al azúcar a través del enlace glucosídico , incluyendo nicotinamida y flavina , y en el último caso, el azúcar ribosa es lineal en lugar de formar el anillo visto en otros nucleótidos.
Síntesis
Los nucleótidos se pueden sintetizar por diversos medios, tanto in vitro como in vivo . [ cita requerida ]
In vitro, se pueden utilizar grupos protectores durante la producción de nucleótidos en el laboratorio. Un nucleósido purificado se protege para crear un fosforamidito , que luego se puede utilizar para obtener análogos que no se encuentran en la naturaleza y/o para sintetizar un oligonucleótido . [ cita requerida ]
In vivo, los nucleótidos pueden sintetizarse de novo o reciclarse a través de vías de rescate . [1] Los componentes utilizados en la síntesis de nucleótidos de novo se derivan de precursores biosintéticos del metabolismo de carbohidratos y aminoácidos , y de amoníaco y dióxido de carbono. Recientemente también se ha demostrado que el metabolismo del bicarbonato celular puede regularse mediante la señalización de mTORC1. [6] El hígado es el principal órgano de síntesis de novo de los cuatro nucleótidos. La síntesis de novo de pirimidinas y purinas sigue dos vías diferentes. Las pirimidinas se sintetizan primero a partir de aspartato y carbamoil-fosfato en el citoplasma hasta la estructura de anillo precursora común, el ácido orótico, sobre el que se une covalentemente una unidad de ribosilo fosforilada. Las purinas, sin embargo, se sintetizan primero a partir de la plantilla de azúcar sobre la que se produce la síntesis del anillo. Como referencia, la síntesis de los nucleótidos de purina y pirimidina se lleva a cabo por varias enzimas en el citoplasma de la célula, no dentro de un orgánulo específico . Los nucleótidos sufren una degradación tal que las partes útiles se pueden reutilizar en reacciones de síntesis para crear nuevos nucleótidos. [ cita requerida ]
Síntesis de ribonucleótidos de pirimidina
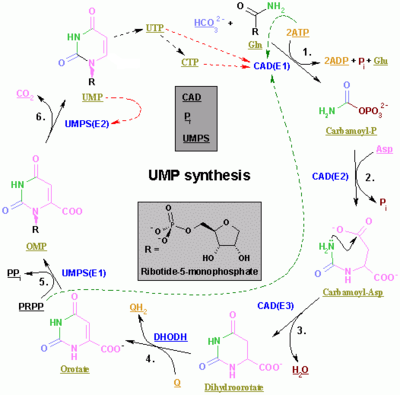
La síntesis de las pirimidinas CTP y UTP ocurre en el citoplasma y comienza con la formación de carbamoil fosfato a partir de glutamina y CO 2 . A continuación, la aspartato carbamoiltransferasa cataliza una reacción de condensación entre aspartato y carbamoil fosfato para formar ácido carbamoil aspártico , que se cicla en ácido 4,5-dihidroorótico por acción de la dihidroorotasa . Este último se convierte en orotato por acción de la dihidroorotato oxidasa . La reacción neta es:
- ( S )-Dihidroorotato + O 2 → Orotato + H 2 O 2
El orotato está unido covalentemente a una unidad de ribosilo fosforilada. El enlace covalente entre la ribosa y la pirimidina se produce en la posición C 1 [7] de la unidad de ribosa , que contiene un pirofosfato , y N 1 del anillo de pirimidina. La orotato fosforribosiltransferasa (PRPP transferasa) cataliza la reacción neta que produce monofosfato de orotidina (OMP):
- Orotato + 5-Fosfo-α-D-ribosa 1-difosfato (PRPP) → Orotidina 5'-fosfato + Pirofosfato
La orotidina 5'-monofosfato es descarboxilada por la orotidina-5'-fosfato descarboxilasa para formar uridina monofosfato (UMP). La transferasa PRPP cataliza las reacciones de ribosilación y descarboxilación, formando UMP a partir del ácido orótico en presencia de PRPP. Es a partir del UMP que se derivan otros nucleótidos de pirimidina. El UMP es fosforilado por dos quinasas a uridina trifosfato (UTP) a través de dos reacciones secuenciales con ATP. Primero, se produce el difosfato a partir de UDP, que a su vez es fosforilado a UTP. Ambos pasos son alimentados por la hidrólisis del ATP:
- ATP + UMP → ADP + UDP
- UDP + ATP → UTP + ADP
Posteriormente, el CTP se forma mediante la aminación del UTP por la actividad catalítica de la CTP sintetasa . La glutamina es el donante de NH3 y la reacción también se ve impulsada por la hidrólisis del ATP :
- UTP + Glutamina + ATP + H 2 O → CTP + ADP + Pi
El monofosfato de citidina (CMP) se deriva del trifosfato de citidina (CTP) con la consiguiente pérdida de dos fosfatos. [8] [9]
Síntesis de ribonucleótidos de purina
Los átomos que se utilizan para construir los nucleótidos de purina provienen de una variedad de fuentes:
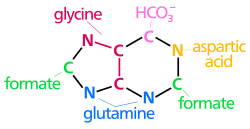
N 1 surge del grupo amina de Asp
C 2 y C 8 se originan del formato
N 3 y N 9 son aportados por el grupo amida de Gln
C 4 , C 5 y N 7 se derivan de Gly
C 6 proviene de HCO 3 − (CO 2 )

La síntesis de novo de nucleótidos de purina , mediante la cual estos precursores se incorporan al anillo de purina, se lleva a cabo mediante una vía de 10 pasos hasta el intermediario de ramificación IMP , el nucleótido de la base hipoxantina . Posteriormente, se sintetizan AMP y GMP a partir de este intermediario mediante vías separadas de dos pasos. Por lo tanto, las fracciones de purina se forman inicialmente como parte de los ribonucleótidos en lugar de como bases libres .
En la síntesis de IMP participan seis enzimas, tres de ellas multifuncionales:
La vía comienza con la formación de PRPP . PRPS1 es la enzima que activa R5P , que se forma principalmente por la vía de las pentosas fosfato , a PRPP al reaccionar con ATP . La reacción es inusual porque un grupo pirofosforilo se transfiere directamente del ATP al C1 de R5P y el producto tiene la configuración α sobre C1. Esta reacción también se comparte con las vías para la síntesis de Trp , His y los nucleótidos de pirimidina . Al estar en una encrucijada metabólica importante y requerir mucha energía, esta reacción está altamente regulada.
En la primera reacción exclusiva de la biosíntesis de nucleótidos de purina, la PPAT cataliza el desplazamiento del grupo pirofosfato de PRPP (PP i ) por un nitrógeno de amida donado desde glutamina (N), glicina (N&C), aspartato (N), ácido fólico (C 1 ) o CO 2 . Este es el paso comprometido en la síntesis de purina. La reacción ocurre con la inversión de la configuración sobre la ribosa C 1 , formando así β - 5-fosforibosilamina (5-PRA) y estableciendo la forma anomérica del futuro nucleótido.
A continuación, se incorpora una glicina alimentada por hidrólisis de ATP, y el grupo carboxilo forma un enlace amino con el NH2 previamente introducido. Luego se añade una unidad de un carbono de la coenzima del ácido fólico N10 - formil-THF al grupo amino de la glicina sustituida seguido del cierre del anillo de imidazol. A continuación, se transfiere un segundo grupo NH2 desde la glutamina al primer carbono de la unidad de glicina. Al mismo tiempo se añade una carboxilación del segundo carbono de la unidad de glicina. Este nuevo carbono se modifica mediante la adición de una tercera unidad NH2 , esta vez transferida desde un residuo de aspartato. Finalmente, se añade una segunda unidad de un carbono de formil-THF al grupo nitrógeno y el anillo se cierra covalentemente para formar el precursor común de la purina, el monofosfato de inosina (IMP).
El monofosfato de inosina se convierte en monofosfato de adenosina en dos pasos. En primer lugar, la hidrólisis de GTP alimenta la adición de aspartato al IMP por la adenilosuccinato sintasa, sustituyendo el oxígeno del carbonilo por un nitrógeno y formando el intermediario adenilosuccinato. A continuación, el fumarato se escinde formando monofosfato de adenosina. Este paso es catalizado por la adenilosuccinato liasa.
El monofosfato de inosina se convierte en monofosfato de guanosina por oxidación del IMP formando xantilato, seguido de la inserción de un grupo amino en C 2 . El NAD + es el aceptor de electrones en la reacción de oxidación. La transferencia del grupo amida desde la glutamina es impulsada por la hidrólisis del ATP.
Degradación de pirimidinas y purinas
En los seres humanos, los anillos de pirimidina (C, T, U) pueden degradarse completamente a CO2 y NH3 ( excreción de urea). Dicho esto, los anillos de purina (G, A) no pueden. En cambio, se degradan al ácido úrico, metabólicamente inerte , que luego se excreta del cuerpo. El ácido úrico se forma cuando el GMP se divide en la base guanina y ribosa. La guanina se desamina a xantina que, a su vez, se oxida a ácido úrico. Esta última reacción es irreversible. De manera similar, el ácido úrico se puede formar cuando el AMP se desamina a IMP del que se elimina la unidad de ribosa para formar hipoxantina. La hipoxantina se oxida a xantina y, finalmente, a ácido úrico. En lugar de la secreción de ácido úrico, la guanina y el IMP se pueden utilizar para fines de reciclaje y síntesis de ácidos nucleicos en presencia de PRPP y aspartato (donante de NH3 ) . [ cita requerida ]
Síntesis prebiótica de nucleótidos
Las teorías sobre el origen de la vida requieren el conocimiento de las vías químicas que permiten la formación de los bloques fundamentales de la vida en condiciones prebióticas plausibles. La hipótesis del mundo del ARN sostiene que en la sopa primordial existían ribonucleótidos flotantes , las moléculas fundamentales que se combinan en serie para formar ARN . Las moléculas complejas como el ARN deben haber surgido de pequeñas moléculas cuya reactividad estaba gobernada por procesos fisicoquímicos. El ARN está compuesto de nucleótidos de purina y pirimidina , ambos necesarios para la transferencia confiable de información y, por lo tanto, la evolución darwiniana . Becker et al. demostraron cómo los nucleósidos de pirimidina pueden sintetizarse a partir de moléculas pequeñas y ribosa , impulsados únicamente por ciclos húmedos-secos. [10] Los nucleósidos de purina pueden sintetizarse por una vía similar. Los 5'-mono- y di-fosfatos también se forman selectivamente a partir de minerales que contienen fosfato, lo que permite la formación simultánea de polirribonucleótidos con bases de purina y pirimidina. De esta manera, se puede establecer una red de reacción hacia los bloques de construcción del ARN de purina y pirimidina a partir de moléculas atmosféricas o volcánicas simples. [10]
Par de bases no naturales (UBP)
Un par de bases no naturales (UBP) es una subunidad diseñada (o nucleobase ) de ADN que se crea en un laboratorio y no se produce en la naturaleza. [11] Los ejemplos incluyen d5SICS y dNaM . Estos nucleótidos artificiales que llevan nucleobases hidrofóbicas , presentan dos anillos aromáticos fusionados que forman un complejo (d5SICS–dNaM) o par de bases en el ADN. [12] [13] Se ha inducido a E. coli a replicar un plásmido que contiene UBP a lo largo de múltiples generaciones. [14] Este es el primer ejemplo conocido de un organismo vivo que transmite un código genético expandido a generaciones posteriores. [12] [15]
Aplicaciones médicas de los nucleótidos sintéticos
Las aplicaciones de los nucleótidos sintéticos varían ampliamente e incluyen el diagnóstico de enfermedades, el tratamiento o la medicina de precisión.
- Agentes antivirales o antirretrovirales: se han utilizado varios derivados de nucleótidos en el tratamiento contra la infección por hepatitis y VIH . [16] [17] Los ejemplos de inhibidores directos de la transcriptasa inversa análogos de nucleósidos ( NRTI ) incluyen tenofovir disoproxil , tenofovir alafenamida y sofosbuvir . Por otro lado, agentes como mericitabina , lamivudina , entecavir y telbivudina primero deben sufrir metabolización a través de fosforilación para activarse.
- Oligonucleótidos antisentido (ASO) : los oligonucleótidos sintéticos se han utilizado en el tratamiento de enfermedades hereditarias raras, ya que pueden unirse a transcripciones de ARN específicas y, en última instancia, modular la expresión de proteínas. La atrofia muscular espinal , la esclerosis lateral amiotrófica , la hipercolesterolemia familiar homocigótica y la hiperoxaluria primaria tipo 1 son todas susceptibles de tratamiento con ASO. [18] La aplicación de oligonucleótidos es una nueva frontera en la medicina de precisión y el manejo de afecciones que no tienen tratamiento.
- ARN guía sintético (gRNA) : los nucleótidos sintéticos se pueden utilizar para diseñar gRNA que son esenciales para el correcto funcionamiento de las tecnologías de edición genética como CRISPR-Cas9 .
Unidad de longitud
El nucleótido (abreviado "nt") es una unidad de longitud común para los ácidos nucleicos monocatenarios, de forma similar a cómo el par de bases es una unidad de longitud para los ácidos nucleicos bicatenarios. [19]
Códigos abreviados para bases degeneradas
La IUPAC ha designado los símbolos para los nucleótidos. [20] Aparte de las cinco bases (A, G, C, T/U), a menudo se utilizan bases degeneradas especialmente para diseñar cebadores de PCR . Estos códigos de nucleótidos se enumeran aquí. Algunas secuencias de cebadores también pueden incluir el carácter "I", que codifica el nucleótido no estándar inosina . La inosina se encuentra en los ARNt y se emparejará con adenina, citosina o timina. Sin embargo, este carácter no aparece en la siguiente tabla porque no representa una degeneración. Si bien la inosina puede cumplir una función similar a la degeneración "D", es un nucleótido real, en lugar de una representación de una mezcla de nucleótidos que cubre cada posible emparejamiento necesario.
| Símbolo [20] | Descripción | Bases representadas | ||||
|---|---|---|---|---|---|---|
| A | una denine | A | 1 | |||
| do | citosina | do | ||||
| GRAMO | guanina | GRAMO | ||||
| yo | t himina | yo | ||||
| tú | Eres rápido | tú | ||||
| Yo | débil | A | yo | 2 | ||
| S | fuerte | do | GRAMO | |||
| METRO | un mínimo | A | do | |||
| K | k -eto | GRAMO | yo | |||
| R | purina | A | GRAMO | |||
| Y | p y rimidina | do | yo | |||
| B | no A ( B viene después de A) | do | GRAMO | yo | 3 | |
| D | no C ( D viene después de C) | A | GRAMO | yo | ||
| yo | no G ( H viene después de G) | A | do | yo | ||
| V | no T ( V viene después de T y U) | A | do | GRAMO | ||
| norte | cualquier base (no un hueco) | A | do | GRAMO | yo | 4 |
Véase también
- Biología
- Cromosoma
- Gene
- Genética
- Análogo de ácido nucleico : compuesto análogo al ARN y al ADN naturales.
- Secuencia de ácidos nucleicos – Sucesión de nucleótidos en un ácido nucleico
- Nucleobase : compuestos biológicos que contienen nitrógeno y que forman nucleósidos.Pages displaying short descriptions of redirect targets
Referencias
- ^ ab Zaharevitz DW, Anderson LW, Malinowski NM, Hyman R, Strong JM, Cysyk RL (noviembre de 1992). "Contribución de la síntesis de novo y de rescate al conjunto de nucleótidos de uracilo en tejidos y tumores de ratón in vivo". Revista Europea de Bioquímica . 210 (1): 293–6. doi : 10.1111/j.1432-1033.1992.tb17420.x . PMID 1446677.
- ^ ab Alberts B, Johnson A, Lewis J, Raff M, Roberts K y Walter P (2002). Biología molecular de la célula (4.ª ed.). Garland Science. ISBN 0-8153-3218-1 . págs. 120–121.
- ^ Abd El-Aleem FS, Taher MS, Lotfy SN, El-Massry KF, Fadel HH (18 de diciembre de 2017). "Influencia de los 5-nucleótidos extraídos en los compuestos aromáticos y la aceptabilidad del sabor de la sopa de carne real". Revista internacional de propiedades alimentarias . 20 (sup1): S1182–S1194. doi : 10.1080/10942912.2017.1286506 . S2CID 100497537.
- ^ Wiley (9 de septiembre de 2005). Enciclopedia de ciencias de la vida (1.ª edición). Wiley. doi :10.1002/9780470015902.a0001333.pub3. ISBN 978-0-470-01617-6.
- ^ Smith AD, ed. (2000). Diccionario Oxford de bioquímica y biología molecular (edición revisada). Oxford: Oxford University Press. pág. 460.
- ^ Ali E, Liponska A, O'Hara B, Amici D, Torno M, Gao P, et al. (junio de 2022). "El eje mTORC1-SLC4A7 estimula la importación de bicarbonato para mejorar la síntesis de nucleótidos de novo". Molecular Cell . 82 (1): 3284–3298.e7. doi :10.1016/j.molcel.2022.06.008. PMC 9444906 . PMID 35772404.
- ^ Consulte la nomenclatura IUPAC de química orgánica para obtener detalles sobre la numeración de residuos de carbono.
- ^ Jones ME (1980). "Biosíntesis de nucleótidos de pirimidina en animales: genes, enzimas y regulación de la biosíntesis de UMP". Revisión anual de bioquímica . 49 (1): 253–79. doi :10.1146/annurev.bi.49.070180.001345. PMID 6105839.
- ^ McMurry JE, Begley TP (2005). La química orgánica de las vías biológicas . Roberts & Company. ISBN 978-0-9747077-1-6.
- ^ ab Becker S, Feldmann J, Wiedemann S, Okamura H, Schneider C, Iwan K, et al. (octubre de 2019). "Síntesis prebióticamente plausible unificada de ribonucleótidos de ARN de pirimidina y purina". Science . 366 (6461): 76–82. Bibcode :2019Sci...366...76B. doi :10.1126/science.aax2747. PMID 31604305. S2CID 203719976.
- ^ Malyshev DA, Dhami K, Quach HT, Lavergne T, Ordoukhanian P, Torkamani A, et al. (julio de 2012). "La replicación eficiente e independiente de la secuencia de ADN que contiene un tercer par de bases establece un alfabeto genético funcional de seis letras". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (30): 12005–10. Bibcode :2012PNAS..10912005M. doi : 10.1073/pnas.1205176109 . PMC 3409741 . PMID 22773812.
- ^ ab Malyshev DA, Dhami K, Lavergne T, Chen T, Dai N, Foster JM, et al. (mayo de 2014). "Un organismo semisintético con un alfabeto genético expandido". Nature . 509 (7500): 385–8. Bibcode :2014Natur.509..385M. doi :10.1038/nature13314. PMC 4058825 . PMID 24805238.
- ^ Callaway E (7 de mayo de 2014). "Los científicos crean el primer organismo vivo con ADN 'artificial'". Nature News . Huffington Post . Consultado el 8 de mayo de 2014 .
- ^ Fikes BJ (8 de mayo de 2014). "Vida diseñada con código genético expandido". San Diego Union Tribune . Consultado el 8 de mayo de 2014 .
- ^ Sample I (7 de mayo de 2014). «Primeras formas de vida que transmiten ADN artificial diseñado por científicos estadounidenses». The Guardian . Consultado el 8 de mayo de 2014 .
- ^ Ramesh D, Vijayakumar BG, Kannan T (diciembre de 2020). "Potencial terapéutico del uracilo y sus derivados para contrarrestar trastornos patogénicos y fisiológicos". Revista Europea de Química Medicinal . 207 : 112801. doi :10.1016/j.ejmech.2020.112801. PMID 32927231. S2CID 221724578.
- ^ Ramesh D, Vijayakumar BG, Kannan T (mayo de 2021). "Avances en análogos de nucleótidos y nucleósidos para abordar las infecciones por el virus de la inmunodeficiencia humana y el virus de la hepatitis". ChemMedChem . 16 (9): 1403–1419. doi :10.1002/cmdc.202000849. PMID 33427377. S2CID 231576801. Archivado desde el original el 14 de diciembre de 2021 . Consultado el 13 de marzo de 2021 .
- ^ Lauffer MC, van Roon-Mom W, Aartsma-Rus A (enero de 2024). "Posibilidades y limitaciones de las terapias con oligonucleótidos antisentido para el tratamiento de trastornos monogénicos". Medicina de las comunicaciones . 4 (1): 6. doi :10.1038/s43856-023-00419-1. PMC 10770028 . PMID 38182878.
- ^ "Diccionario de términos de biología: nt". GenScript . Consultado el 31 de julio de 2023 .
- ^ ab Nomenclature Committee of the International Union of Biochemistry (NC-IUB) (1984). "Nomenclatura para bases incompletamente especificadas en secuencias de ácidos nucleicos" . Consultado el 4 de febrero de 2008 .
Lectura adicional
- Sigel A, Operschall BP, Sigel H (2017). "Capítulo 11. Formación de complejos de plomo (II) con nucleótidos y sus constituyentes". En Astrid S, Helmut S, Sigel RK (eds.). Plomo: sus efectos sobre el medio ambiente y la salud . Iones metálicos en las ciencias de la vida. Vol. 17. de Gruyter. págs. 319–402. doi :10.1515/9783110434330-011. ISBN . 9783110434330. Número de identificación personal 28731304.
- Freisinger E, Sigel RK (julio de 2007). "De los nucleótidos a las ribozimas: una comparación de sus propiedades de unión a iones metálicos" (PDF) . Coordination Chemistry Reviews . 251 (13–14): 1834–1851. doi :10.1016/j.ccr.2007.03.008.
- Comisión de Nomenclatura Bioquímica (CBN) de la IUPAC-IUB (14 de febrero de 1971). "Abreviaturas y símbolos para ácidos nucleicos, polinucleótidos y sus constituyentes". Journal of Molecular Biology . 55 (3): 299–310. doi :10.1016/0022-2836(71)90319-6. PMID 5551389.
- Favre HA, Powell WH, eds. (2014). "Capítulo P-10 Estructuras parentales de productos naturales y compuestos relacionados" (PDF) . Nomenclatura de la química orgánica: recomendaciones de la IUPAC y nombres preferidos 2013. Cambridge: Royal Soc. of Chemistry. ISBN 978-0-85404-182-4.
- Bender H, ed. (2003). "Nucleotide Structure". Clackamas Community College . Archivado desde el original el 2006-09-01 . Consultado el 2020-04-21 .