Dominio CBS
| Dominio CBS | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
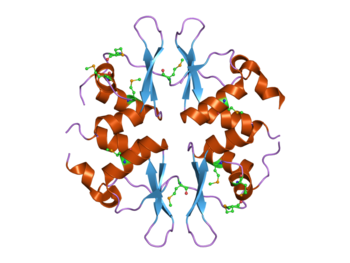 Estructura de la proteína SNF4 de levadura que contiene cuatro dominios CBS. [1] Esta proteína es parte del complejo de proteína quinasa activada por AMP (AMPK). | |||||||||||
| Identificadores | |||||||||||
| Símbolo | CBS | ||||||||||
| Pfam | PF00571 | ||||||||||
| Interprofesional | IPR000644 | ||||||||||
| ELEGANTE | CBS | ||||||||||
| PROSITIO | PS51371 | ||||||||||
| SCOP2 | 1zfj / ALCANCE / SUPFAM | ||||||||||
| Diligenciamiento de conflictos | cd02205 | ||||||||||
| |||||||||||
En biología molecular, el dominio CBS es un dominio proteico que se encuentra en una variedad de proteínas en todas las especies, desde bacterias hasta humanos. Se identificó por primera vez como una región de secuencia conservada en 1997 y se nombró así por la cistationina beta sintasa , una de las proteínas en las que se encuentra. [2] Los dominios CBS también se encuentran en una amplia variedad de otras proteínas, como la inosina monofosfato deshidrogenasa , [3] los canales de cloruro dependientes de voltaje [4] [5] [6] [7] [8] y la proteína quinasa activada por AMP (AMPK). [9] [10] Los dominios CBS regulan la actividad de los dominios enzimáticos y transportadores asociados en respuesta a la unión de moléculas con grupos adenosilo como AMP y ATP , o s-adenosilmetionina . [11]
Estructura
El dominio CBS está compuesto por un patrón de estructura secundaria beta-alfa-beta-beta-alfa que se pliega en una estructura terciaria globular que contiene una lámina β antiparalela de tres cadenas con dos hélices α en un lado. Los dominios CBS siempre se encuentran en pares en las secuencias de proteínas y cada par de estos dominios se asocia estrechamente en una disposición pseudodímera a través de sus láminas β formando un llamado par CBS o dominio Bateman . [12] [13] Estos pares de dominios CBS pueden asociarse de manera cabeza a cabeza (es decir, códigos PDB 3KPC , 1PVM , 2OOX ) o de manera cabeza a cola (es decir, códigos PDB 1O50 , 1PBJ ) formando una estructura compacta similar a un disco. Al hacerlo, forman hendiduras que constituyen las regiones de unión del ligando canónico. [14] [15] [16] [17] [18] En principio, el número de sitios de unión canónicos coincide con el número de dominios CBS dentro de la molécula y tradicionalmente se numeran de acuerdo con el dominio CBS que contiene cada uno de los residuos de aspartato conservados que potencialmente interactúan con la ribosa de los nucleótidos. [19] Sin embargo, no todas estas cavidades podrían necesariamente unir nucleótidos o ser funcionales. Recientemente, también se ha descrito un sitio no canónico para AMP en la proteína MJ1225 de M. jannaschii , aunque su papel funcional aún se desconoce. [20]

Unión de ligando
Se ha demostrado que los dominios CBS se unen a grupos adenosilo en moléculas como AMP y ATP , [11] o s-adenosilmetionina, [21] pero también pueden unirse a iones metálicos como Mg 2+ . [22] [23] Al unirse a estos diferentes ligandos, los dominios CBS regulan la actividad de los dominios enzimáticos asociados. [24] Los mecanismos moleculares subyacentes a esta regulación recién están comenzando a dilucidarse. [16] [17] [21] [22] [25] En este momento, se han propuesto dos tipos diferentes de mecanismos. El primero afirma que la porción nucleotídica del ligando no induce esencialmente ningún cambio en la estructura de la proteína, siendo el potencial electrostático en el sitio de unión la propiedad más significativa de la unión del nucleótido de adenosina. [17] [26] Esta respuesta "estática" estaría involucrada en procesos en los que la regulación por carga energética sería ventajosa. [17] [26] Por el contrario, el segundo tipo de mecanismo (denominado "dinámico") implica cambios conformacionales dramáticos en la estructura de la proteína tras la unión del ligando y se ha informado para el dominio citosólico del transportador de Mg 2+ MgtE de Thermus thermophilus , [22] la proteína de función desconocida MJ0100 de M. jannaschii [21] [27] y la región reguladora de la pirofosfatasa de Clostridium perfringens . [28]
Dominios asociados
Los dominios CBS se encuentran a menudo en proteínas que contienen otros dominios. Estos dominios suelen ser enzimáticos , transportadores de membrana o dominios de unión al ADN. Sin embargo, también se encuentran a menudo proteínas que contienen solo dominios CBS, particularmente en procariotas. Estas proteínas con dominio CBS independientes pueden formar complejos al unirse a otras proteínas, como las quinasas, con las que interactúan y las regulan.

Mutaciones que conducen a enfermedades
Las mutaciones en algunas proteínas humanas que contienen el dominio CBS conducen a enfermedades genéticas. [3] Por ejemplo, las mutaciones en la proteína cistationina beta sintasa conducen a un trastorno hereditario del metabolismo llamado homocistinuria (OMIM: 236200). [29] Se ha demostrado que las mutaciones en la subunidad gamma de la enzima AMPK conducen a la miocardiopatía hipertrófica familiar con síndrome de Wolff-Parkinson-White (OMIM: 600858). Las mutaciones en los dominios CBS de la enzima IMPDH conducen a la afección ocular retinitis pigmentosa (OMIM: 180105).
Los seres humanos tienen varios genes de canales de cloruro dependientes del voltaje , y las mutaciones en los dominios CBS de varios de ellos se han identificado como la causa de enfermedades genéticas. Las mutaciones en CLCN1 provocan miotonía (OMIM: 160800), [30] las mutaciones en CLCN2 pueden provocar epilepsia generalizada idiopática (OMIM: 600699), las mutaciones en CLCN5 pueden provocar enfermedad de Dent (OMIM: 300009), las mutaciones en CLCN7 pueden provocar osteopetrosis (OMIM: 259700), [31] y las mutaciones en CLCNKB pueden provocar síndrome de Bartter (OMIM: 241200).
Referencias
- ^ PDB : 2nye ; Rudolph MJ, Amodeo GA, Iram SH, Hong SP, Pirino G, Carlson M, Tong L (enero de 2007). "Estructura del dominio Bateman2 de Snf4 de levadura: asociación dimérica y relevancia para la unión de AMP". Estructura . 15 (1): 65–74. doi : 10.1016/j.str.2006.11.014 . PMID 17223533.
- ^ Bateman A (enero de 1997). "La estructura de un dominio común a las arqueobacterias y la proteína de la enfermedad de homocistinuria". Trends Biochem. Sci . 22 (1): 12–3. doi :10.1016/S0968-0004(96)30046-7. PMID 9020585.
- ^ ab Ignoul S, Eggermont J (diciembre de 2005). "Dominios CBS: estructura, función y patología en proteínas humanas". Am. J. Physiol., Cell Physiol . 289 (6): C1369–78. doi :10.1152/ajpcell.00282.2005. PMID 16275737.
- ^ Ponting CP (marzo de 1997). "Dominios CBS en los canales de cloruro CIC implicados en la miotonía y la nefrolitiasis (cálculos renales)". J. Mol. Med . 75 (3): 160–3. PMID 9106071.
- ^ Meyer S, Dutzler R (febrero de 2006). "Estructura cristalina del dominio citoplasmático del canal de cloruro ClC-0". Estructura . 14 (2): 299–307. doi : 10.1016/j.str.2005.10.008 . PMID 16472749.
- ^ Yusef YR, Zúñiga L, Catalán M, Niemeyer MI, Cid LP, Sepúlveda FV (abril de 2006). "Eliminación de la compuerta en el canal de cloruro ClC-2 dependiente de voltaje mediante mutaciones puntuales que afectan al poro y al dominio C-terminal CBS-2". J. Physiol . 572 (Pt 1): 173–81. doi :10.1113/jphysiol.2005.102392. PMC 1779660 . PMID 16469788.
- ^ Markovic S, Dutzler R (junio de 2007). "La estructura del dominio citoplasmático del canal de cloruro ClC-Ka revela una interfaz de interacción conservada". Structure . 15 (6): 715–25. doi : 10.1016/j.str.2007.04.013 . PMID 17562318.
- ^ Meyer S, Savaresi S, Forster IC, Dutzler R (enero de 2007). "Reconocimiento de nucleótidos por el dominio citoplasmático del transportador de cloruro humano ClC-5". Nat. Struct. Mol. Biol . 14 (1): 60–7. doi :10.1038/nsmb1188. PMID 17195847. S2CID 20733119.
- ^ Day P, Sharff A, Parra L, et al. (mayo de 2007). "Estructura de un par de dominios CBS de la subunidad reguladora gamma1 de la AMPK humana en complejo con AMP y ZMP". Acta Crystallogr. D . 63 (Pt 5): 587–96. doi :10.1107/S0907444907009110. PMID 17452784.
- ^ Rudolph MJ, Amodeo GA, Iram SH, et al. (enero de 2007). "Estructura del dominio Bateman2 de Snf4 de levadura: asociación dimérica y relevancia para la unión de AMP". Structure . 15 (1): 65–74. doi : 10.1016/j.str.2006.11.014 . PMID 17223533.
- ^ ab Kemp BE (enero de 2004). "Los dominios de Bateman y los derivados de adenosina forman un contrato vinculante". J. Clin. Invest . 113 (2): 182–4. doi :10.1172/JCI20846. PMC 311445 . PMID 14722609.
- ^ Kemp BE (enero de 2004). "Los dominios de Bateman y los derivados de adenosina forman un contrato vinculante". J. Clin. Invest . 113 (2): 182–4. doi :10.1172/JCI20846. PMC 311445 . PMID 14722609.
- ^ Zhang R, Evans G, Rotella FJ, Westbrook EM, Beno D, Huberman E, Joachimiak A, Collart FR (abril de 1999). "Características y estructura cristalina de la inosina-5'-monofosfato deshidrogenasa bacteriana". Bioquímica . 38 (15): 4691–700. CiteSeerX 10.1.1.488.2542 . doi :10.1021/bi982858v. PMID 10200156.
- ^ Rudolph MJ, Amodeo GA, Iram SH, Hong SP, Pirino G, Carlson M, Tong L (enero de 2007). "Estructura del dominio Bateman2 de Snf4 de levadura: asociación dimérica y relevancia para la unión de AMP". Estructura . 15 (1): 65–74. doi : 10.1016/j.str.2006.11.014 . PMID 17223533.
- ^ Meyer S, Savaresi S, Forster IC, Dutzler R (enero de 2007). "Reconocimiento de nucleótidos por el dominio citoplasmático del transportador de cloruro humano ClC-5". Nat. Struct. Mol. Biol . 14 (1): 60–7. doi :10.1038/nsmb1188. PMID 17195847. S2CID 20733119.
- ^ ab Amodeo GA, Rudolph MJ, Tong L (septiembre de 2007). "Estructura cristalina del núcleo heterotrimérico del homólogo SNF1 de AMPK de Saccharomyces cerevisiae". Nature . 449 (7161): 492–5. doi :10.1038/nature06127. PMID 17851534. S2CID 4342092.
- ^ abcd Townley R, Shapiro L (marzo de 2007). "Estructuras cristalinas del sensor de adenilato de la proteína quinasa activada por AMP de la levadura de fisión". Science . 315 (5819): 1726–9. doi : 10.1126/science.1137503 . PMID 17289942. S2CID 38983201.
- ^ Jin X, Townley R, Shapiro L (octubre de 2007). "Información estructural sobre la regulación de la AMPK: el ADP entra en juego". Structure . 15 (10): 1285–95. doi : 10.1016/j.str.2007.07.017 . PMID 17937917.
- ^ Kemp BE, Oakhill JS, Scott JW (octubre de 2007). "Estructura y regulación de AMPK desde tres ángulos". Structure . 15 (10): 1161–3. doi : 10.1016/j.str.2007.09.006 . PMID 17937905.
- ^ Gómez-García I, Oyenarte I, Martínez-Cruz LA (mayo de 2010). "La estructura cristalina de la proteína MJ1225 de Methanocaldococcus jannaschii muestra una fuerte conservación de las características estructurales clave observadas en la gamma-AMPK eucariota". J Mol Biol . 399 (1): 53–70. doi :10.1016/j.jmb.2010.03.045. PMID 20382158.
- ^ abc Lucas M, Encinar JA, Arribas EA, Oyenarte I, García IG, Kortazar D, Fernández JA, Mato JM, Martínez-Chantar ML, Martínez-Cruz LA (febrero de 2010). "La unión de S-metil-5'-tioadenosina y S-adenosil-L-metionina a la proteína MJ0100 desencadena un cambio conformacional de abierto a cerrado en su par de motivos CBS". J. Mol. Biol . 396 (3): 800–20. doi :10.1016/j.jmb.2009.12.012. PMID 20026078.
- ^ abc Ishitani R, Sugita Y, Dohmae N, Furuya N, Hattori M, Nureki O (octubre de 2008). "Mecanismo de detección de Mg2+ del transportador de Mg2+ MgtE investigado mediante un estudio de dinámica molecular". Proc . Natl. Sci. USA . 105 (40): 15393–8. doi : 10.1073/pnas.0802991105 . PMC 2563093. PMID 18832160.
- ^ Hattori M, Nureki O (marzo de 2008). "[Base estructural del mecanismo de homeostasis de Mg2 por el transportador de MgtE]". Tanpakushitsu Kakusan Koso (en japonés). 53 (3): 242–8. PMID 18326297.
- ^ Scott JW, Hawley SA, Green KA, et al. (enero de 2004). "Los dominios CBS forman módulos de detección de energía cuya unión de ligandos de adenosina se ve alterada por mutaciones de enfermedades". J. Clin. Invest . 113 (2): 274–84. doi :10.1172/JCI19874. PMC 311435. PMID 14722619 .
- ^ Tuominen H, Salminen A, Oksanen E, Jämsen J, Heikkilä O, Lehtiö L, Magretova NN, Goldman A, Baykov AA, Lahti R (mayo de 2010). "Estructuras cristalinas de los dominios CBS y DRTGG de la región reguladora de la pirofosfatasa de Clostridiumperfringens complejada con el inhibidor, AMP, y el activador, tetrafosfato de diadenosina". J. Mol. Biol . 398 (3): 400–13. doi :10.1016/j.jmb.2010.03.019. PMID 20303981.
- ^ ab Xiao B, Heath R, Saiu P, Leiper FC, Leone P, Jing C, Walker PA, Haire L, Eccleston JF, Davis CT, Martin SR, Carling D, Gamblin SJ (septiembre de 2007). "Base estructural para la unión de AMP a la proteína quinasa activada por AMP de mamíferos". Naturaleza . 449 (7161): 496–500. doi : 10.1038/naturaleza06161. PMID 17851531. S2CID 4345919.
- ^ Lucas M, Kortazar D, Astigarraga E, et al. (octubre de 2008). "Purificación, cristalización y análisis preliminar de difracción de rayos X del par de dominio CBS de la proteína MJ0100 de Methanococcus jannaschii". Acta Crystallographica Sección F. 64 (Parte 10): 936–41. doi :10.1107/S1744309108027930. PMC 2564890 . PMID 18931440.
- ^ Tuominen H, Salminen A, Oksanen E, et al. (mayo de 2010). "Estructuras cristalinas de los dominios CBS y DRTGG de la región reguladora de la pirofosfatasa de Clostridium perfringens complejada con el inhibidor, AMP, y el activador, tetrafosfato de diadenosina". J Mol Biol . 398 (3): 400–413. doi :10.1016/j.jmb.2010.03.019. PMID 20303981.
- ^ Shan X, Dunbrack RL, Christopher SA, Kruger WD (marzo de 2001). "Las mutaciones en el dominio regulador de la cistationina beta sintasa pueden suprimir funcionalmente las mutaciones derivadas del paciente en cis". Hum. Mol. Genet . 10 (6): 635–43. doi : 10.1093/hmg/10.6.635 . PMID 11230183.
- ^ Pusch M (abril de 2002). "Miotonía causada por mutaciones en el gen del canal de cloruro muscular CLCN1". Hum. Mutat . 19 (4): 423–34. doi : 10.1002/humu.10063 . PMID 11933197.
- ^ Cleiren E, Bénichou O, Van Hul E, et al. (diciembre de 2001). "La enfermedad de Albers-Schönberg (osteopetrosis autosómica dominante, tipo II) es resultado de mutaciones en el gen del canal de cloruro ClCN7". Hum. Mol. Genet . 10 (25): 2861–7. doi : 10.1093/hmg/10.25.2861 . PMID 11741829.