ADN antiguo

El ADN antiguo ( aDNA ) es ADN aislado de fuentes antiguas (normalmente especímenes , pero también ADN ambiental ). [1] [2] Debido a los procesos de degradación (incluyendo reticulación , desaminación y fragmentación ) [3] el ADN antiguo está más degradado en comparación con el material genético contemporáneo. [4] Se ha recuperado material genético de material esquelético paleo/arqueológico e histórico, tejidos momificados , colecciones de archivo de especímenes médicos no congelados, restos de plantas preservadas, hielo y de núcleos de permafrost , sedimentos marinos y lacustres y tierra de excavación .
Incluso en las mejores condiciones de conservación, existe un límite superior de 0,4 a 1,5 millones de años para que una muestra contenga suficiente ADN para las tecnologías de secuenciación. [5] El ADN más antiguo secuenciado a partir de especímenes físicos proviene de molares de mamut en Siberia de más de 1 millón de años. [6] En 2022, se recuperó material genético de dos millones de años de sedimentos en Groenlandia , y actualmente se considera el ADN más antiguo descubierto hasta ahora. [7] [8]
Historia de los estudios del ADN antiguo
Década de 1980
El primer estudio de lo que se llamaría ADNa se llevó a cabo en 1984, cuando Russ Higuchi y sus colegas de la Universidad de California, Berkeley informaron que los rastros de ADN de un espécimen de museo de Quagga no solo permanecieron en el espécimen más de 150 años después de la muerte del individuo, sino que también se podían extraer y secuenciar. [9] Durante los siguientes dos años, a través de investigaciones en especímenes momificados naturales y artificialmente, Svante Pääbo confirmó que este fenómeno no se limitaba a especímenes de museo relativamente recientes, sino que aparentemente podía replicarse en una variedad de muestras humanas momificadas que databan de hace varios miles de años. [10] [11] [12]
Los laboriosos procesos que se requerían en ese momento para secuenciar dicho ADN (a través de la clonación bacteriana ) fueron un freno efectivo para el estudio del ADN antiguo (aDNA) y el campo de la museomística . Sin embargo, con el desarrollo de la reacción en cadena de la polimerasa (PCR) a fines de la década de 1980, el campo comenzó a progresar rápidamente. [13] [14] [15] La amplificación por PCR de doble cebador de aDNA (jumping-PCR) puede producir artefactos de secuencia altamente sesgados y no auténticos. Se utilizó la estrategia de PCR anidada de múltiples cebadores para superar esas deficiencias.
Década de 1990
La era posterior a la PCR anunció una ola de publicaciones, ya que numerosos grupos de investigación afirmaron haber tenido éxito en el aislamiento de ADNa. Pronto se publicó una serie de hallazgos increíbles que afirmaban que se podía extraer ADN auténtico de especímenes que tenían millones de años, en los reinos de lo que Lindahl (1993b) ha denominado ADN antediluviano . [16] La mayoría de estas afirmaciones se basaban en la recuperación de ADN de organismos conservados en ámbar . Se decía que se habían extraído secuencias de insectos como abejas sin aguijón, [17] [18] termitas [19] y mosquitos de la madera [20] , así como de plantas [21] y bacterias [22] del ámbar dominicano que databa de la época del Oligoceno . Fuentes aún más antiguas de gorgojos libaneses envueltos en ámbar , que datan de la época del Cretácico , también habrían producido ADN auténtico. [23] Las afirmaciones de recuperación de ADN no se limitaban al ámbar.
Se publicaron informes de varios restos de plantas preservadas en sedimentos que datan del Mioceno . [24] [25] Luego, en 1994, Woodward et al. informaron lo que en ese momento se llamó los resultados más emocionantes hasta la fecha [26] : secuencias de citocromo b mitocondrial que aparentemente habían sido extraídas de huesos de dinosaurio que databan de hace más de 80 millones de años. Cuando en 1995 dos estudios más informaron secuencias de ADN de dinosaurio extraídas de un huevo del Cretácico, [27] [28] parecía que el campo revolucionaría el conocimiento del pasado evolutivo de la Tierra. Incluso estas edades extraordinarias fueron superadas por la supuesta recuperación de secuencias halobacterianas de 250 millones de años de antigüedad de la halita . [29] [30]
El desarrollo de una mejor comprensión de la cinética de la conservación del ADN, los riesgos de contaminación de las muestras y otros factores que complicaban la situación llevaron a que el campo viera estos resultados con más escepticismo. Numerosos intentos cuidadosos de reproducir muchos de los hallazgos fracasaron, y todas las afirmaciones de la década sobre la existencia de ADNa con varios millones de años de antigüedad terminarían siendo descartadas por no ser auténticas. [31]
Década de 2000
La amplificación por extensión de un solo cebador se introdujo en 2007 para abordar el daño por modificación del ADN post mortem. [32] Desde 2009, el campo de los estudios de ADNa se ha revolucionado con la introducción de técnicas de investigación mucho más económicas. [33] El uso de técnicas de secuenciación de próxima generación (NGS) de alto rendimiento en el campo de la investigación del ADN antiguo ha sido esencial para reconstruir los genomas de organismos antiguos o extintos. Un método de preparación de bibliotecas de ADN monocatenario (ssDNA) ha despertado un gran interés entre los investigadores del ADN antiguo (aDNA). [34] [35]
Además de estas innovaciones técnicas, a principios de la década se empezaron a desarrollar mejores estándares y criterios para evaluar los resultados de ADN, así como una mejor comprensión de los posibles peligros. [31] [36]
El 7 de diciembre de 2022, un estudio publicado en Nature informó que se encontró material genético de dos millones de años en Groenlandia, y actualmente se considera el ADN más antiguo descubierto hasta ahora. [7] [8]
Problemas y errores
Procesos de degradación
Debido a los procesos de degradación (incluyendo la reticulación, la desaminación y la fragmentación), [3] el ADN antiguo es de menor calidad que el material genético moderno. [4] Las características de daño y la capacidad del ADN antiguo para sobrevivir a través del tiempo restringen los posibles análisis y establecen un límite superior a la edad de las muestras exitosas. [4] Existe una correlación teórica entre el tiempo y la degradación del ADN, [37] aunque las diferencias en las condiciones ambientales complican las cosas. Es poco probable que las muestras sometidas a diferentes condiciones se alineen de manera predecible con una relación uniforme entre edad y degradación. [38] Los efectos ambientales pueden incluso importar después de la excavación, ya que las tasas de descomposición del ADN pueden aumentar, [39] particularmente en condiciones de almacenamiento fluctuantes. [40] Incluso en las mejores condiciones de conservación, existe un límite superior de 0,4 a 1,5 millones de años para que una muestra contenga suficiente ADN para las tecnologías de secuenciación contemporáneas. [5]
Las investigaciones sobre la descomposición del ADN mitocondrial y nuclear en los huesos de moa han modelado la degradación del ADN mitocondrial hasta una longitud media de 1 par de bases después de 6.830.000 años a -5 °C. [4] La cinética de descomposición se ha medido mediante experimentos de envejecimiento acelerado, lo que demuestra además la fuerte influencia de la temperatura y la humedad de almacenamiento en la descomposición del ADN. [41] El ADN nuclear se degrada al menos dos veces más rápido que el ADNmt. Los primeros estudios que informaron sobre la recuperación de ADN mucho más antiguo, por ejemplo de restos de dinosaurios del Cretácico , pueden haberse originado en la contaminación de la muestra.
Límite de edad
Una revisión crítica de la literatura sobre ADN antiguo a través del desarrollo del campo destaca que pocos estudios han logrado amplificar el ADN de restos con más de varios cientos de miles de años de antigüedad. [42] Una mayor apreciación de los riesgos de contaminación ambiental y estudios sobre la estabilidad química del ADN han suscitado inquietudes sobre los resultados informados anteriormente. Más tarde se reveló que el supuesto ADN de dinosaurio era el cromosoma Y humano . [43] El ADN informado de halobacterias encapsuladas ha sido criticado en función de su similitud con las bacterias modernas, lo que sugiere contaminación, [36] o pueden ser el producto de una actividad metabólica de bajo nivel y a largo plazo . [44]
El ADNa puede contener una gran cantidad de mutaciones post mortem , que aumentan con el tiempo. Algunas regiones del polinucleótido son más susceptibles a esta degradación, lo que permite que los datos de secuencia erróneos eludan los filtros estadísticos utilizados para verificar la validez de los datos. [31] Debido a los errores de secuenciación, se debe tener mucho cuidado al interpretar el tamaño de la población. [45] Las sustituciones resultantes de la desaminación de los residuos de citosina están ampliamente sobrerrepresentadas en las secuencias de ADN antiguas. La codificación incorrecta de C a T y de G a A explica la mayoría de los errores. [46]
Contaminación
Otro problema con las muestras de ADN antiguo es la contaminación por ADN humano moderno y por ADN microbiano (la mayor parte del cual también es antiguo). [47] [48] En los últimos años han surgido nuevos métodos para prevenir la posible contaminación de muestras de ADNa, incluyendo la realización de extracciones en condiciones extremadamente estériles, el uso de adaptadores especiales para identificar moléculas endógenas de la muestra (distinguidas de las introducidas durante el análisis) y la aplicación de bioinformática a las secuencias resultantes basándose en lecturas conocidas para aproximar las tasas de contaminación. [49] [50]
Autenticación de ADNa
El desarrollo en el campo del ADNa en la década de 2000 aumentó la importancia de autenticar el ADN recuperado para confirmar que es realmente antiguo y no el resultado de una contaminación reciente. A medida que el ADN se degrada con el tiempo, los nucleótidos que lo componen pueden cambiar, especialmente en los extremos de las moléculas de ADN. La desaminación de la citosina a uracilo en los extremos de las moléculas de ADN se ha convertido en una forma de autenticación. Durante la secuenciación del ADN, las ADN polimerasas incorporarán una adenina (A) frente al uracilo (U), lo que dará lugar a sustituciones de citosina (C) a timina (T) en los datos del ADNa. [51] Estas sustituciones aumentan en frecuencia a medida que la muestra envejece. La medición de frecuencia del nivel de CT, daño del ADN antiguo, se puede realizar utilizando varios programas como mapDamage2.0 o PMDtools [52] [53] y de forma interactiva en metaDMG. [54] Debido a la despurinización hidrolítica, el ADN se fragmenta en piezas más pequeñas, lo que da lugar a roturas de cadena sencilla. Combinado con el patrón de daño, esta corta longitud de fragmento también puede ayudar a diferenciar entre el ADN moderno y el antiguo. [55] [56]
ADNa no humano
A pesar de los problemas asociados con el ADN "antediluviano", se ha publicado una amplia y cada vez mayor gama de secuencias de ADNa de una variedad de taxones animales y vegetales . Los tejidos examinados incluyen restos animales momificados artificial o naturalmente, [9] [57] huesos, [58] [59] [60] [61] conchas, [62] paleofaeces, [63] [64] especímenes conservados en alcohol, [65] basureros de roedores, [66] restos vegetales secos, [67] [68] y, recientemente, extracciones de ADN animal y vegetal directamente de muestras de suelo . [69]
En junio de 2013, un grupo de investigadores que incluía a Eske Willerslev , Marcus Thomas Pius Gilbert y Orlando Ludovic del Centro de Geogenética del Museo de Historia Natural de Dinamarca en la Universidad de Copenhague , anunció que habían secuenciado el ADN de un caballo de entre 560 y 780 mil años de antigüedad, utilizando material extraído de un hueso de la pata encontrado enterrado en el permafrost en el territorio del Yukón de Canadá. [70] [71] [72] Un equipo alemán también informó en 2013 sobre el genoma mitocondrial reconstruido de un oso, Ursus deningeri , de más de 300.000 años de antigüedad, lo que demuestra que el ADN antiguo auténtico se puede conservar durante cientos de miles de años fuera del permafrost. [73] La secuencia de ADN de ADN nuclear aún más antiguo se informó en 2021 a partir de los dientes preservados en permafrost de dos mamuts siberianos , ambos de más de un millón de años. [6] [74]
En 2016, los investigadores midieron el ADN de los cloroplastos en núcleos de sedimentos marinos y encontraron ADN de diatomeas que databa de hace 1,4 millones de años. [75] Este ADN tenía una vida media significativamente más larga que la de investigaciones anteriores, de hasta 15.000 años. El equipo de Kirkpatrick también descubrió que el ADN solo se desintegraba a un ritmo de vida media hasta unos 100.000 años, momento en el que seguía un ritmo de desintegración más lento, de ley de potencia. [75]
ADNa humano
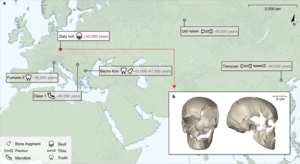
Debido al considerable interés antropológico , arqueológico y público que despiertan los restos humanos, estos han recibido una atención considerable de la comunidad del ADN. También existen problemas de contaminación más profundos, ya que los especímenes pertenecen a la misma especie que los investigadores que recogen y evalúan las muestras.
Fuentes
Debido a la preservación morfológica en momias, muchos estudios de los años 1990 y 2000 utilizaron tejido momificado como fuente de ADN humano antiguo. Los ejemplos incluyen tanto especímenes preservados naturalmente, como el Ötzi el Hombre de Hielo congelado en un glaciar [77] y cuerpos preservados a través de una rápida desecación a gran altitud en los Andes, [12] [78] así como varios tejidos preservados tratados químicamente como las momias del antiguo Egipto. [79] Sin embargo, los restos momificados son un recurso limitado. La mayoría de los estudios de ADNa humano se han centrado en extraer ADN de dos fuentes mucho más comunes en el registro arqueológico : huesos y dientes . El hueso que se utiliza con más frecuencia para la extracción de ADN es el hueso petroso de la oreja, ya que su estructura densa proporciona buenas condiciones para la preservación del ADN. [80] Varias otras fuentes también han producido ADN, incluyendo paleofaeces , [81] y cabello . [82] [83] La contaminación sigue siendo un problema importante cuando se trabaja con material humano antiguo.
Se ha recuperado con éxito ADN de patógenos antiguos de muestras que datan de más de 5000 años en humanos y de hasta 17 000 años atrás en otras especies. Además de las fuentes habituales de tejido momificado, huesos y dientes, estos estudios también han examinado una variedad de otras muestras de tejido, incluida la pleura calcificada , [84] tejido incluido en parafina , [85] [86] y tejido fijado con formalina . [87] Se han desarrollado herramientas computacionales eficientes para análisis de ADNa de patógenos y microorganismos a pequeña (QIIME [88] ) y gran escala (FALCON [89] ).
Resultados
Sin embargo, tomando medidas preventivas en sus procedimientos contra dicha contaminación, un estudio de 2012 analizó muestras de huesos de un grupo neandertal en la cueva de El Sidrón, encontrando nuevos conocimientos sobre el parentesco potencial y la diversidad genética del ADNa. [90] En noviembre de 2015, los científicos informaron haber encontrado un diente de 110.000 años de antigüedad que contenía ADN del homínido denisovano , una especie extinta de humano del género Homo . [91] [92]
La investigación ha añadido nueva complejidad al poblamiento de Eurasia. Un estudio de 2018 [93] mostró que una migración masiva de la Edad del Bronce había afectado en gran medida la composición genética de las Islas Británicas, trayendo consigo la cultura del vaso campaniforme de Europa continental.
También se han revelado nuevos datos sobre los vínculos entre los antepasados de los asiáticos centrales y los pueblos indígenas de las Américas. En África, el ADN más antiguo se degrada rápidamente debido al clima tropical más cálido, aunque en septiembre de 2017 se informó de la existencia de muestras de ADN antiguo, de hasta 8.100 años de antigüedad. [94]
Además, el ADN antiguo ha ayudado a los investigadores a estimar la divergencia humana moderna. [95] Al secuenciar los genomas africanos de tres cazadores-recolectores de la Edad de Piedra (de 2000 años de antigüedad) y cuatro agricultores de la Edad de Hierro (de 300 a 500 años de antigüedad), Schlebusch y sus colegas pudieron retrasar la fecha de la divergencia más temprana entre poblaciones humanas a entre 350.000 y 260.000 años atrás.
A partir de 2021, los genomas humanos completamente reconstruidos más antiguos tienen aproximadamente 45.000 años . [96] [76] Estos datos genéticos proporcionan información sobre la migración y la historia genética, por ejemplo, de Europa , incluido el mestizaje entre humanos arcaicos y modernos, como una mezcla común entre los humanos modernos europeos iniciales y los neandertales. [97] [76] [98]
Investigadores especializados en ADN antiguo
Véase también
Referencias
- ^ Pevsner J (2015). Bioinformática y genómica funcional (3.ª ed.). Wiley-Blackwell. ISBN 978-1118581780.
- ^ Jones M (2016). Descifrando el pasado: cómo los arqueólogos están reescribiendo la historia humana con ADN antiguo . Arcade. ISBN 978-1628724479.
- ^ ab Anderson LA (mayo de 2023). "Un marco químico para la preservación de células y tejidos blandos de vertebrados fósiles". Earth-Science Reviews . 240 : 104367. Bibcode :2023ESRv..24004367A. doi : 10.1016/j.earscirev.2023.104367 . S2CID 257326012.
- ^ abcd Allentoft ME, Collins M, Harker D, Haile J, Oskam CL, Hale ML, et al. (diciembre de 2012). "La vida media del ADN en el hueso: medición de la cinética de descomposición en 158 fósiles datados". Actas. Ciencias biológicas . 279 (1748): 4724–33. doi :10.1098/rspb.2012.1745. PMC 3497090. PMID 23055061 .
- ^ ab Willerslev E, Hansen AJ, Rønn R, Brand TB, Barnes I, Wiuf C, et al. (enero de 2004). "Persistencia a largo plazo del ADN bacteriano" (PDF) . Current Biology . 14 (1): R9-10. Bibcode :2004CBio...14...R9W. doi : 10.1016/j.cub.2003.12.012 . PMID 14711425. S2CID 12227538.
- ^ ab van der Valk T, Pečnerová P, Díez-Del-Molino D, Bergström A, Oppenheimer J, Hartmann S, et al. (marzo de 2021). "El ADN de un millón de años arroja luz sobre la historia genómica de los mamuts". Nature . 591 (7849): 265–269. Bibcode :2021Natur.591..265V. doi :10.1038/s41586-021-03224-9. PMC 7116897 . PMID 33597750.
- ^ ab Zimmer C (7 de diciembre de 2022). "El ADN más antiguo conocido ofrece una visión de un Ártico que alguna vez fue exuberante: en el permafrost de Groenlandia, los científicos descubrieron material genético de hace dos millones de años de decenas de especies de plantas y animales, incluidos mastodontes, gansos, lemmings y hormigas". The New York Times . Consultado el 7 de diciembre de 2022 .
- ^ ab Kjær KH, Winther Pedersen M, De Sanctis B, De Cahsan B, Korneliussen TS, Michelsen CS, et al. (diciembre de 2022). "Un ecosistema de 2 millones de años en Groenlandia descubierto por ADN ambiental". Naturaleza . 612 (7939): 283–291. Código Bib :2022Natur.612..283K. doi :10.1038/s41586-022-05453-y. PMC 9729109 . PMID 36477129.
- ^ ab Higuchi R, Bowman B, Freiberger M, Ryder OA, Wilson AC (1984). "Secuencias de ADN del quagga, un miembro extinto de la familia de los caballos". Nature . 312 (5991): 282–4. Bibcode :1984Natur.312..282H. doi :10.1038/312282a0. PMID 6504142. S2CID 4313241.
- ^ Pääbo S (1985a). "Preservación del ADN en momias del antiguo Egipto". J. Archaeol. Sci . 12 (6): 411–17. Código Bibliográfico :1985JArSc..12..411P. doi :10.1016/0305-4403(85)90002-0.
- ^ Pääbo S (1985b). "Clonación molecular del ADN de momias del Antiguo Egipto". Nature . 314 (6012): 644–5. Código Bibliográfico :1985Natur.314..644P. doi :10.1038/314644a0. PMID 3990798. S2CID 1358295.
- ^ ab Pääbo S (1986). "Investigaciones genéticas moleculares de restos humanos antiguos". Simposios de Cold Spring Harbor sobre biología cuantitativa . 51 (parte 1): 441–6. doi :10.1101/SQB.1986.051.01.053. PMID 3107879.
- ^ Mullis KB, Faloona FA (1987). "Síntesis específica de ADN in vitro mediante una reacción en cadena catalizada por polimerasa". ADN recombinante, parte F. Métodos en enzimología. Vol. 155. págs. 335–50. doi :10.1016/0076-6879(87)55023-6. ISBN 978-0-12-182056-5. Número PMID 3431465.
- ^ Raxworthy CJ, Smith BT (noviembre de 2021). "Explotación de ADN histórico en los museos: avances y desafíos en la museomística". Tendencias en ecología y evolución . 36 (11): 1049–1060. Bibcode :2021TEcoE..36.1049R. doi : 10.1016/j.tree.2021.07.009 . PMID 34456066. S2CID 239687836.
- ^ Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, et al. (enero de 1988). "Amplificación enzimática de ADN dirigida por cebadores con una ADN polimerasa termoestable". Science . 239 (4839): 487–491. Bibcode :1988Sci...239..487S. doi :10.1126/science.239.4839.487. PMID 2448875.
- ^ Lindahl T (octubre de 1993). "Recuperación del ADN antediluviano". Nature . 365 (6448): 700. Bibcode :1993Natur.365..700L. doi : 10.1038/365700a0 . PMID 8413647. S2CID 4365447.
- ^ Cano RJ, Poinar H, Poinar Jr GO (1992a). "Aislamiento y caracterización parcial del ADN de la abeja Problebeia dominicana (Apidae:Hymenoptera) en ámbar de entre 25 y 40 millones de años". Med Sci Res . 20 : 249–51.
- ^ Cano RJ, Poinar HN, Roubik DW, Poinar Jr GO (1992b). "Amplificación enzimática y secuenciación de nucleótidos de porciones del gen 18S rRNA de la abeja Problebeia dominicana (Apidae:Hymenoptera) aislada de ámbar dominicano de entre 25 y 40 millones de años". Med Sci Res . 20 : 619–22.
- ^ Matson E, Ottesen E, Leadbetter J (2007). "Extracción de ADN de los microbios intestinales de la termita (Zootermopsis nevadensis)". Journal of Visualized Experiments (4): 195. doi :10.3791/195. PMC 2556161. PMID 18979000 .
- ^ DeSalle R, Grimaldi D (diciembre de 1994). "ADN muy antiguo". Current Opinion in Genetics & Development . 4 (6): 810–5. doi :10.1016/0959-437x(94)90064-7. PMID 7888749.
- ^ Poinar H, Cano R, Poinar G (1993). "ADN de una planta extinta". Nature . 363 (6431): 677. Bibcode :1993Natur.363..677P. doi : 10.1038/363677a0 . S2CID 4330200.
- ^ Cano RJ, Borucki MK, Higby-Schweitzer M, Poinar HN, Poinar GO, Pollard KJ (junio de 1994). "ADN de Bacillus en abejas fósiles: ¿una simbiosis antigua?". Applied and Environmental Microbiology . 60 (6): 2164–2167. Bibcode :1994ApEnM..60.2164C. doi : 10.1128/aem.60.6.2164-2167.1994 . PMC 201618 . PMID 8031102.
- ^ Cano RJ, Poinar HN, Pieniazek NJ, Acra A, Poinar GO (junio de 1993). "Amplificación y secuenciación de ADN de un gorgojo de entre 120 y 135 millones de años". Nature . 363 (6429): 536–538. Bibcode :1993Natur.363..536C. doi :10.1038/363536a0. PMID 8505978. S2CID 4243196.
- ^ Golenberg EM (septiembre de 1991). "Amplificación y análisis del ADN fósil de plantas del Mioceno". Philosophical Transactions of the Royal Society of London. Serie B, Biological Sciences . 333 (1268): 419–26, discusión 426–7. doi :10.1098/rstb.1991.0092. PMID 1684052.
- ^ Golenberg EM, Giannasi DE, Clegg MT, Smiley CJ, Durbin M, Henderson D, et al. (abril de 1990). "Secuencia de ADN de cloroplastos de una especie de Magnolia del Mioceno". Nature . 344 (6267): 656–8. Bibcode :1990Natur.344..656G. doi :10.1038/344656a0. PMID 2325772. S2CID 26577394.
- ^ Woodward SR, Weyand NJ, Bunnell M (noviembre de 1994). "Secuencia de ADN de fragmentos de hueso del período Cretácico". Science . 266 (5188): 1229–32. Bibcode :1994Sci...266.1229W. doi :10.1126/science.7973705. PMID 7973705.
- ^ An CC, Li Y, Zhu YX (1995). "Clonación molecular y secuenciación del 18S rDNA de un fósil de huevo de dinosaurio especializado encontrado en Xixia Henan, China". Acta Sci Nat Univ Pekinensis . 31 : 140–47.
- ^ Li Y, An CC, Zhu YX (1995). "Aislamiento de ADN y análisis de secuencia de ADN de dinosaurio de huevos de dinosaurio del Cretácico en Xixia Henan, China". Acta Sci Nat Univ Pekinensis . 31 : 148–52.
- ^ Vreeland RH, Rosenzweig WD, Powers DW (octubre de 2000). "Aislamiento de una bacteria halotolerante de 250 millones de años de antigüedad a partir de un cristal de sal primario". Nature . 407 (6806): 897–900. Bibcode :2000Natur.407..897V. doi :10.1038/35038060. PMID 11057666. S2CID 9879073.
- ^ Fish SA, Shepherd TJ, McGenity TJ, Grant WD (mayo de 2002). "Recuperación de fragmentos del gen del ARN ribosómico 16S de halita antigua". Nature . 417 (6887): 432–6. Bibcode :2002Natur.417..432F. doi :10.1038/417432a. PMID 12024211. S2CID 4423309.
- ^ abc Pääbo S, Poinar H, Serre D, Jaenicke-Despres V, Hebler J, Rohland N, et al. (2004). "Análisis genéticos a partir de ADN antiguo" (PDF) . Annual Review of Genetics . 38 (1): 645–79. doi :10.1146/annurev.genet.37.110801.143214. PMID 15568989. Archivado desde el original (PDF) el 17 de diciembre de 2008.
- ^ Brotherton P, Endicott P, Sanchez JJ, Beaumont M, Barnett R, Austin J, et al. (2007). "Una nueva caracterización de alta resolución del ADN antiguo revela que los eventos de modificación de bases de tipo C > U son la única causa de las lesiones de codificación errónea post mortem". Nucleic Acids Research . 35 (17): 5717–28. doi :10.1093/nar/gkm588. PMC 2034480 . PMID 17715147.
- ^ Reich 2018.
- ^ Wales N, Carøe C, Sandoval-Velasco M, Gamba C, Barnett R, Samaniego JA, et al. (diciembre de 2015). "Nuevos conocimientos sobre la preparación de bibliotecas de ADN monocatenario frente a bicatenario para ADN antiguo". BioTechniques . 59 (6): 368–71. doi : 10.2144/000114364 . PMID 26651516.
- ^ Bennett EA, Massilani D, Lizzo G, Daligault J, Geigl EM, Grange T (junio de 2014). "Construcción de bibliotecas para genómica antigua: ¿cadena simple o doble?". BioTechniques . 56 (6): 289–90, 292–6, 298, passim. doi : 10.2144/000114176 . PMID 24924389.
- ^ ab Nicholls H (febrero de 2005). "El ADN antiguo alcanza la mayoría de edad". PLOS Biology . 3 (2): e56. doi : 10.1371/journal.pbio.0030056 . PMC 548952 . PMID 15719062.
- ^ Hebsgaard MB, Phillips MJ, Willerslev E (mayo de 2005). "ADN geológicamente antiguo: ¿hecho o artefacto?". Tendencias en microbiología . 13 (5): 212–20. doi :10.1016/j.tim.2005.03.010. PMID 15866038.
- ^ Hansen AJ, Mitchell DL, Wiuf C, Paniker L, Brand TB, Binladen J, et al. (junio de 2006). "Los enlaces cruzados, en lugar de las roturas de cadenas, determinan el acceso a secuencias de ADN antiguas a partir de sedimentos congelados". Genética . 173 (2): 1175–9. doi :10.1534/genetics.106.057349. PMC 1526502 . PMID 16582426.
- ^ Pruvost M, Schwarz R, Correia VB, Champlot S, Braguier S, Morel N, et al. (enero de 2007). "Los huesos fósiles recién excavados son los mejores para la amplificación del ADN antiguo". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (3): 739–44. Bibcode :2007PNAS..104..739P. doi : 10.1073/pnas.0610257104 . PMC 1783384 . PMID 17210911.
- ^ Burger J, Hummel S, Hermann B, Henke W (junio de 1999). "Preservación del ADN: un estudio de ADN microsatélite en restos esqueléticos antiguos". Electroforesis . 20 (8): 1722–8. doi :10.1002/(sici)1522-2683(19990101)20:8<1722::aid-elps1722>3.0.co;2-4. PMID 10435438. S2CID 7325310.
- ^ Grass RN, Heckel R, Puddu M, Paunescu D, Stark WJ (febrero de 2015). "Conservación química robusta de información digital sobre ADN en sílice con códigos de corrección de errores". Angewandte Chemie . 54 (8): 2552–5. doi :10.1002/anie.201411378. PMID 25650567.
- ^ Willerslev E, Hansen AJ, Binladen J, Brand TB, Gilbert MT, Shapiro B, et al. (mayo de 2003). "Diversos registros genéticos de plantas y animales de sedimentos del Holoceno y el Pleistoceno". Science . 300 (5620): 791–5. Bibcode :2003Sci...300..791W. doi : 10.1126/science.1084114 . PMID 12702808. S2CID 1222227.
- ^ Zischler H, Höss M, Handt O, von Haeseler A, van der Kuyl AC, Goudsmit J (mayo de 1995). "Detección de ADN de dinosaurio". Ciencia . 268 (5214): 1192–3, respuesta del autor 1194. Bibcode :1995Sci...268.1191B. doi : 10.1126/ciencia.7605504 . PMID 7605504.
- ^ Johnson SS, Hebsgaard MB, Christensen TR, Mastepanov M, Nielsen R, Munch K, et al. (septiembre de 2007). "Las bacterias antiguas muestran evidencia de reparación del ADN". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (36): 14401–5. Bibcode :2007PNAS..10414401J. doi : 10.1073/pnas.0706787104 . PMC 1958816 . PMID 17728401.
- ^ Johnson PL, Slatkin M (enero de 2008). "Explicación del sesgo por error de secuenciación en estimaciones genéticas de poblaciones" (Texto completo gratuito) . Biología molecular y evolución . 25 (1): 199–206. doi : 10.1093/molbev/msm239 . PMID 17981928.
- ^ Briggs AW, Stenzel U, Johnson PL, Green RE, Kelso J, Prüfer K, et al. (septiembre de 2007). "Patrones de daño en secuencias de ADN genómico de un neandertal". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 104 (37): 14616–21. Bibcode :2007PNAS..10414616B. doi : 10.1073/pnas.0704665104 . PMC 1976210 . PMID 17715061.
- ^ Gansauge MT, Meyer M (septiembre de 2014). "Enriquecimiento selectivo de moléculas de ADN dañadas para la secuenciación de genomas antiguos". Genome Research . 24 (9): 1543–9. doi :10.1101/gr.174201.114. PMC 4158764 . PMID 25081630.
- ^ Pratas D, Hosseini M, Grilo G, Pinho AJ, Silva RM, Caetano T, et al. (septiembre de 2018). "Análisis de la composición metagenómica de una mandíbula de oso polar antigua secuenciada de Svalbard". Genes . 9 (9): 445. doi : 10.3390/genes9090445 . PMC 6162538 . PMID 30200636.
- ^ Slatkin M, Racimo F (junio de 2016). "ADN antiguo e historia humana". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 113 (23): 6380–7. Bibcode :2016PNAS..113.6380S. doi : 10.1073/pnas.1524306113 . PMC 4988579 . PMID 27274045.
- ^ Borry M, Hübner A, Rohrlach AB, Warinner C (27 de julio de 2021). "PyDamage: identificación y estimación automatizada de daños antiguos para contigs en el ensamblaje de novo de ADN antiguo". PeerJ . 9 : e11845. doi : 10.7717/peerj.11845 . PMC 8323603 . PMID 34395085.
- ^ Dabney J, Meyer M, Pääbo S (julio de 2013). "Daños en el ADN antiguo". Cold Spring Harbor Perspectives in Biology . 5 (7): a012567. doi :10.1101/cshperspect.a012567. PMC 3685887 . PMID 23729639.
- ^ Jónsson H, Ginolhac A, Schubert M, Johnson PL, Orlando L (julio de 2013). "mapDamage2.0: estimaciones bayesianas rápidas y aproximadas de parámetros de daño del ADN antiguo". Bioinformática . 29 (13): 1682–1684. doi :10.1093/bioinformatics/btt193. PMC 3694634 . PMID 23613487.
- ^ Skoglund P, Northoff BH, Shunkov MV, Derevianko AP, Pääbo S, Krause J, et al. (febrero de 2014). "Separación del ADN antiguo endógeno de la contaminación moderna en un neandertal siberiano". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 111 (6): 2229–2234. Bibcode :2014PNAS..111.2229S. doi : 10.1073/pnas.1318934111 . PMC 3926038 . PMID 24469802.
- ^ Michelsen C, Pedersen MW, Fernandez-Guerra A, Zhao L, Petersen TC, Korneliussen TS (9 de diciembre de 2022). "metaDMG: un conjunto de herramientas rápido y preciso sobre daños al ADN antiguo para datos metagenómicos". bioRxiv : 2022.12.06.519264. doi :10.1101/2022.12.06.519264. S2CID 254536966.
- ^ Krause J, Briggs AW, Kircher M, Maricic T, Zwyns N, Derevianko A, et al. (febrero de 2010). "Un genoma completo de ADNmt de un humano moderno temprano de Kostenki, Rusia". Current Biology . 20 (3): 231–236. Bibcode :2010CBio...20..231K. doi : 10.1016/j.cub.2009.11.068 . PMID 20045327. S2CID 16440465.
- ^ Pochon Z, Bergfeldt N, Kırdök E, Vicente M, Naidoo T, van der Valk T, et al. (octubre de 2023). "aMeta: un flujo de trabajo de creación de perfiles metagenómicos antiguos preciso y eficiente en términos de memoria". Genome Biology . 24 (1): 242. doi :10.1101/2022.10.03.510579. PMC 10591440 . PMID 37872569. S2CID 252763827.
- ^ Thomas RH, Schaffner W, Wilson AC, Pääbo S (agosto de 1989). "Filogenia del ADN del lobo marsupial extinto". Nature . 340 (6233): 465–7. Código Bibliográfico :1989Natur.340..465T. doi :10.1038/340465a0. PMID 2755507. S2CID 4310500.
- ^ Hagelberg E, Sykes B, Hedges R (noviembre de 1989). "ADN óseo antiguo amplificado". Nature . 342 (6249): 485. Bibcode :1989Natur.342..485H. doi : 10.1038/342485a0 . PMID 2586623. S2CID 13434992.
- ^ Cooper A, Mourer-Chauviré C, Chambers GK, von Haeseler A, Wilson AC, Pääbo S (septiembre de 1992). "Orígenes independientes de los moas y kiwis de Nueva Zelanda". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 89 (18): 8741–4. Bibcode :1992PNAS...89.8741C. doi : 10.1073/pnas.89.18.8741 . PMC 49996 . PMID 1528888.
- ^ Hagelberg E, Thomas MG, Cook CE, Sher AV, Baryshnikov GF, Lister AM (agosto de 1994). "ADN de huesos de mamut antiguos". Nature . 370 (6488): 333–4. Código Bibliográfico :1994Natur.370R.333H. doi :10.1038/370333b0. PMID 8047136. S2CID 8694387.
- ^ Hänni C, Laudet V, Stehelin D, Taberlet P (diciembre de 1994). "Rastreo de los orígenes del oso cavernario (Ursus spelaeus) mediante secuenciación de ADN mitocondrial". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 91 (25): 12336–40. Bibcode :1994PNAS...9112336H. doi : 10.1073/pnas.91.25.12336 . PMC 45432 . PMID 7991628.
- ^ Martin-Roy R, Thyrring J, Mata X, Bangsgaard P, Bennike O, Christiansen G, et al. (2024-05-06). Fernández Robledo JA (ed.). "Avanzando en análisis genómicos responsables de conchas de moluscos antiguos". MÁS UNO . 19 (5): e0302646. Código Bib : 2024PLoSO..1902646M. doi : 10.1371/journal.pone.0302646 . PMC 11073703 . PMID 38709766.
- ^ Poinar HN, Hofreiter M, Spaulding WG, Martin PS, Stankiewicz BA, Bland H, et al. (julio de 1998). "Coproscopia molecular: estiércol y dieta del perezoso terrestre extinto Nothrotheriops shastensis". Science . 281 (5375): 402–6. Bibcode :1998Sci...281..402P. doi :10.1126/science.281.5375.402. PMID 9665881.
- ^ Hofreiter M, Poinar HN, Spaulding WG, Bauer K, Martin PS, Possnert G, et al. (diciembre de 2000). "Un análisis molecular de la dieta de los perezosos terrestres durante la última glaciación". Ecología molecular . 9 (12): 1975–84. Bibcode :2000MolEc...9.1975H. doi :10.1046/j.1365-294X.2000.01106.x. PMID 11123610. S2CID 22685601.
- ^ Junqueira AC, Lessinger AC, Azeredo-Espin AM (marzo de 2002). "Métodos para la recuperación de secuencias de ADN mitocondrial de especímenes de museo de moscas causantes de miasis". Entomología médica y veterinaria . 16 (1): 39–45. doi : 10.1046/j.0269-283x.2002.00336.x . PMID 11963980.
- ^ Kuch M, Rohland N, Betancourt JL, Latorre C, Steppan S, Poinar HN (mayo de 2002). "Análisis molecular de un basurero de roedores de 11.700 años de antigüedad del desierto de Atacama, Chile". Ecología molecular . 11 (5): 913–24. Bibcode :2002MolEc..11..913K. doi :10.1046/j.1365-294X.2002.01492.x. PMID 11975707. S2CID 10538371.
- ^ Goloubinoff P, Pääbo S, Wilson AC (marzo de 1993). "Evolución del maíz inferida a partir de la diversidad de secuencias de un segmento del gen Adh2 de especímenes arqueológicos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 90 (5): 1997–2001. Bibcode :1993PNAS...90.1997G. doi : 10.1073/pnas.90.5.1997 . PMC 46007 . PMID 8446621.
- ^ Dumolin-Lapègue S, Pemonge MH, Gielly L, Taberlet P, Petit RJ (diciembre de 1999). "Amplificación del ADN del roble a partir de madera antigua y moderna". Ecología molecular . 8 (12): 2137–40. Bibcode :1999MolEc...8.2137D. doi :10.1046/j.1365-294x.1999.00788.x. PMID 10632865. S2CID 41967121.
- ^ Willerslev E, Cooper A (enero de 2005). "ADN antiguo". Actas. Ciencias biológicas . 272 (1558): 3–16. doi :10.1098/rspb.2004.2813. PMC 1634942. PMID 15875564 .
- ^ Erika Check Hayden (26 de junio de 2013). "Los primeros caballos surgieron hace 4 millones de años". Nature . doi :10.1038/nature.2013.13261.
- ^ Lee JL (7 de noviembre de 2017). «Secuencian el genoma más antiguo del mundo a partir de ADN de caballo de 700.000 años». National Geographic . Consultado el 19 de mayo de 2019 .
- ^ Orlando L, Ginolhac A, Zhang G, Froese D, Albrechtsen A, Stiller M, et al. (julio de 2013). "Recalibración de la evolución de Equus utilizando la secuencia del genoma de un caballo del Pleistoceno medio temprano". Nature . 499 (7456): 74–8. Bibcode :2013Natur.499...74O. doi :10.1038/nature12323. PMID 23803765. S2CID 4318227.
- ^ Dabney J, Knapp M, Glocke I, Gansauge MT, Weihmann A, Nickel B, et al. (septiembre de 2013). "Secuencia completa del genoma mitocondrial de un oso cavernario del Pleistoceno medio reconstruida a partir de fragmentos de ADN ultracortos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 110 (39): 15758–63. Bibcode :2013PNAS..11015758D. doi : 10.1073/pnas.1314445110 . PMC 3785785 . PMID 24019490.
- ^ Callaway E (febrero de 2021). «Los genomas de mamut de un millón de años rompen el récord del ADN antiguo más antiguo». Nature . 590 (7847): 537–538. Bibcode :2021Natur.590..537C. doi : 10.1038/d41586-021-00436-x . ISSN 0028-0836. PMID 33597786.
- ^ ab Kirkpatrick JB, Walsh EA, D'Hondt S (8 de julio de 2016). "Persistencia y descomposición del ADN fósil en sedimentos marinos en escalas de tiempo de cientos de miles de años a millones de años". Geología . 44 (8): 615–18. Bibcode :2016Geo....44..615K. doi : 10.1130/g37933.1 . ISSN 0091-7613.
- ^ abc Prüfer K, Posth C, Yu H, Stoessel A, Spyrou MA, Deviese T, et al. (junio de 2021). "Una secuencia del genoma de un cráneo humano moderno de más de 45.000 años de antigüedad de Zlatý kůň en Chequia". Nature Ecology & Evolution . 5 (6): 820–825. Bibcode :2021NatEE...5..820P. doi : 10.1038/s41559-021-01443-x . PMC 8175239 . PMID 33828249.
 Disponible bajo CC BY 4.0.
Disponible bajo CC BY 4.0. - ^ Handt O, Richards M, Trommsdorff M, Kilger C, Simanainen J, Georgiev O, et al. (junio de 1994). "Análisis genético molecular del Hombre de hielo tirolés". Science . 264 (5166): 1775–8. Bibcode :1994Sci...264.1775H. doi :10.1126/science.8209259. PMID 8209259.
- ^ Montiel R, Malgosa A, Francalacci P (octubre de 2001). "Autentificación del ADN mitocondrial humano antiguo". Human Biology . 73 (5): 689–713. doi :10.1353/hub.2001.0069. PMID 11758690. S2CID 39302526.
- ^ Hänni C, Laudet V, Coll J, Stehelin D (julio de 1994). "Una variante inusual de la secuencia de ADN mitocondrial de una momia egipcia". Genomics . 22 (2): 487–9. doi :10.1006/geno.1994.1417. PMID 7806242.
- ^ Pinhasi R, Fernandes D, Sirak K, Novak M, Connell S, Alpaslan-Roodenberg S, et al. (18 de junio de 2015). "Rendimiento óptimo de ADN antiguo de la parte del oído interno del hueso petroso humano". PLOS ONE . 10 (6): e0129102. Bibcode :2015PLoSO..1029102P. doi : 10.1371/journal.pone.0129102 . PMC 4472748 . PMID 26086078.
- ^ Poinar HN, Kuch M, Sobolik KD, Barnes I, Stankiewicz AB, Kuder T, et al. (abril de 2001). "Un análisis molecular de la diversidad dietética de tres nativos americanos arcaicos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 98 (8): 4317–22. Bibcode :2001PNAS...98.4317P. doi : 10.1073/pnas.061014798 . PMC 31832 . PMID 11296282.
- ^ Baker LE (2001). Análisis de secuencias y haplotipos de ADN mitocondrial de muestras históricas de tallos capilares de los pueblos Choctaw y Menominee (tesis doctoral). Universidad de Tennessee, Knoxville.
- ^ Gilbert MT, Wilson AS, Bunce M, Hansen AJ, Willerslev E, Shapiro B, et al. (junio de 2004). "ADN mitocondrial antiguo del cabello". Current Biology . 14 (12): R463-4. Bibcode :2004CBio...14.R463G. doi : 10.1016/j.cub.2004.06.008 . PMID 15203015.
- ^ Donoghue HD, Spigelman M, Zias J, Gernaey-Child AM, Minnikin DE (1998). "ADN del complejo Mycobacterium tuberculosis en pleura calcificada de restos de 1400 años de antigüedad". Lett Appl Microbiol . 27 (5): 265–69. doi :10.1046/j.1472-765x.1998.t01-8-00449.x (inactivo 2024-05-17). PMID 9830142.
{{cite journal}}: CS1 maint: DOI inactivo a partir de mayo de 2024 ( enlace ) - ^ Jackson PJ, Hugh-Jones ME, Adair DM, Green G, Hill KK, Kuske CR, et al. (febrero de 1998). "Análisis de PCR de muestras de tejido de las víctimas del ántrax de Sverdlovsk en 1979: la presencia de múltiples cepas de Bacillus anthracis en diferentes víctimas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 95 (3): 1224–9. Bibcode :1998PNAS...95.1224J. doi : 10.1073/pnas.95.3.1224 . PMC 18726 . PMID 9448313.
- ^ Basler CF, Reid AH, Dybing JK, Janczewski TA, Fanning TG, Zheng H, et al. (febrero de 2001). "Secuencia del segmento del gen no estructural (NS) del virus de la gripe pandémica de 1918 y caracterización de virus recombinantes que portan los genes NS de 1918". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 98 (5): 2746–51. Bibcode :2001PNAS...98.2746B. doi : 10.1073/pnas.031575198 . PMC 30210 . PMID 11226311.
- ^ Taubenberger JK, Reid AH, Krafft AE, Bijwaard KE, Fanning TG (marzo de 1997). "Caracterización genética inicial del virus de la gripe "española" de 1918". Science . 275 (5307): 1793–6. doi :10.1126/science.275.5307.1793. PMID 9065404. S2CID 8976173.
- ^ QIIME
- ^ Pratas D, Pinho AJ, Silva RM, Rodrigues JM, Hosseini M, Caetano T, et al. (febrero de 2018). "FALCON: un método para inferir la composición metagenómica del ADN antiguo". bioRxiv . doi : 10.1101/267179 .
- ^ Lalueza-Fox C, Rosas A, de la Rasilla M (enero de 2012). "Investigaciones paleogenéticas en el yacimiento neandertal de El Sidrón". Anales de anatomía - Anatomischer Anzeiger . Número especial: ADN antiguo. 194 (1): 133–7. doi :10.1016/j.aanat.2011.01.014. hdl :10261/79609. PMID 21482084.
- ^ Zimmer C (16 de noviembre de 2015). "En un diente, ADN de unos primos muy antiguos, los denisovanos". The New York Times . Consultado el 16 de noviembre de 2015 .
- ^ Sawyer S, Renaud G, Viola B, Hublin JJ, Gansauge MT, Shunkov MV, et al. (diciembre de 2015). "Secuencias de ADN nuclear y mitocondrial de dos individuos denisovanos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 112 (51): 15696–700. Bibcode :2015PNAS..11215696S. doi : 10.1073/pnas.1519905112 . PMC 4697428 . PMID 26630009.
- ^ Olalde I, Brace S, Allentoft ME, Armit I, Kristiansen K, Booth T, et al. (marzo de 2018). "El fenómeno Beaker y la transformación genómica del noroeste de Europa". Nature . 555 (7695): 190–196. Bibcode :2018Natur.555..190O. doi :10.1038/nature25738. PMC 5973796 . PMID 29466337.
- ^ Zimmer C (21 de septiembre de 2017). "Pistas sobre el misterioso pasado de África encontradas en esqueletos antiguos". The New York Times . Consultado el 21 de septiembre de 2017 .
- ^ Schlebusch CM, Malmström H, Günther T, Sjödin P, Coutinho A, Edlund H, et al. (noviembre de 2017). "Los genomas antiguos del sur de África estiman que la divergencia de los humanos modernos se produjo hace entre 350.000 y 260.000 años". Science . 358 (6363): 652–655. Bibcode :2017Sci...358..652S. doi : 10.1126/science.aao6266 . PMID 28971970.
- ^ "La ascendencia neandertal identifica el genoma humano moderno más antiguo". phys.org . Consultado el 10 de mayo de 2021 .
- ^ "Los humanos más antiguos conocidos de Europa se aparearon con neandertales con una frecuencia sorprendente". Noticias de ciencia . 7 de abril de 2021 . Consultado el 10 de mayo de 2021 .
- ^ Hajdinjak M, Mafessoni F, Skov L, Vernot B, Hübner A, Fu Q, et al. (abril de 2021). "Los humanos del Paleolítico Superior inicial en Europa tenían ascendencia neandertal reciente". Nature . 592 (7853): 253–257. Bibcode :2021Natur.592..253H. doi : 10.1038/s41586-021-03335-3 . PMC 8026394 . PMID 33828320.
 Disponible bajo CC BY 4.0.
Disponible bajo CC BY 4.0.
Lectura adicional
- Reich D (2018). Quiénes somos y cómo llegamos aquí: el ADN antiguo y la nueva ciencia del pasado humano . Pantheon Books . ISBN 978-1101870327.
- Diamond J (20 de abril de 2018). "Una nueva versión de nuestra historia de origen". The New York Times . Consultado el 23 de abril de 2018 .
- Orlando L (junio de 2014). "Un genoma mitocondrial de 400.000 años cuestiona las relaciones filogenéticas entre los homínidos arcaicos: utilizando los últimos avances en genómica antigua, se ha descifrado la secuencia del genoma mitocondrial de un homínido de 400.000 años". BioEssays . 36 (6): 598–605. doi :10.1002/bies.201400018. PMID 24706482. S2CID 35786511.
- Jones E (2022). ADN antiguo: la creación de una ciencia de celebridades . Yale University Press . ISBN 978-0-300-24012-2.
Enlaces externos
- "ADN antiguo". Viajes ancestrales . Archivado desde el original el 3 de octubre de 2016.
- El famoso mtADN, wiki de isogg
- ADNmt antiguo, wiki isogg
- ADN antiguo Archivado el 6 de marzo de 2020 en Wayback Machine , y-str.org
- Evidencia del pasado: un mapa y estado de los restos antiguos: muestras de EE. UU., aquí no hay datos de secuencia.
- "Desvelando el misterio de la momia: usando ADN". Archivado desde el original el 14 de diciembre de 2009– no hay datos sobre el YDNA, solo sobre el mtDNA

