Diseño de fármacos
El diseño de fármacos , a menudo denominado diseño racional de fármacos o simplemente diseño racional , es el proceso inventivo de encontrar nuevos medicamentos basándose en el conocimiento de un objetivo biológico . [1] El fármaco es más comúnmente una pequeña molécula orgánica que activa o inhibe la función de una biomolécula como una proteína , lo que a su vez produce un beneficio terapéutico para el paciente . En el sentido más básico, el diseño de fármacos implica el diseño de moléculas que sean complementarias en forma y carga al objetivo biomolecular con el que interactúan y, por lo tanto, se unirán a él. El diseño de fármacos con frecuencia, pero no necesariamente, se basa en técnicas de modelado por computadora . [2] Este tipo de modelado a veces se denomina diseño de fármacos asistido por computadora . Finalmente, el diseño de fármacos que se basa en el conocimiento de la estructura tridimensional del objetivo biomolecular se conoce como diseño de fármacos basado en la estructura . [2] Además de las moléculas pequeñas, los productos biofarmacéuticos que incluyen péptidos [3] [4] y especialmente anticuerpos terapéuticos son una clase cada vez más importante de medicamentos y también se han desarrollado métodos computacionales para mejorar la afinidad, selectividad y estabilidad de estas terapias basadas en proteínas. [5]
Definición
La frase "diseño de fármacos" es similar al diseño de ligandos (es decir, el diseño de una molécula que se unirá firmemente a su objetivo). [6] Aunque las técnicas de diseño para la predicción de la afinidad de unión son razonablemente exitosas, hay muchas otras propiedades, como la biodisponibilidad , la vida media metabólica y los efectos secundarios , que primero deben optimizarse antes de que un ligando pueda convertirse en un fármaco seguro y eficaz. Estas otras características a menudo son difíciles de predecir con técnicas de diseño racionales.
Debido a las altas tasas de deserción, especialmente durante las fases clínicas del desarrollo de fármacos , se está prestando más atención en las primeras etapas del proceso de diseño de fármacos a la selección de fármacos candidatos cuyas propiedades fisicoquímicas se prevé que resulten en menos complicaciones durante el desarrollo y, por lo tanto, tengan más probabilidades de conducir a un fármaco aprobado y comercializado. [7] Además, los experimentos in vitro complementados con métodos computacionales se utilizan cada vez más en el descubrimiento temprano de fármacos para seleccionar compuestos con perfiles ADME (absorción, distribución, metabolismo y excreción) y toxicológicos más favorables . [8]
Objetivos de los fármacos
Un objetivo biomolecular (más comúnmente una proteína o un ácido nucleico ) es una molécula clave involucrada en una vía metabólica o de señalización particular que está asociada con una condición o patología de enfermedad específica o con la infectividad o supervivencia de un patógeno microbiano . Los objetivos potenciales de los fármacos no son necesariamente causantes de enfermedades, pero por definición deben ser modificadores de la enfermedad. [9] En algunos casos, se diseñarán moléculas pequeñas para mejorar o inhibir la función del objetivo en la vía específica modificadora de la enfermedad. Se diseñarán moléculas pequeñas (por ejemplo, agonistas , antagonistas , agonistas inversos o moduladores de receptores ; activadores o inhibidores de enzimas; o abridores o bloqueadores de canales iónicos ) [10] que sean complementarias al sitio de unión del objetivo. [11] Las moléculas pequeñas (fármacos) se pueden diseñar de manera que no afecten a ninguna otra molécula importante "fuera del objetivo" (a menudo denominadas antiobjetivos ) ya que las interacciones de los fármacos con moléculas fuera del objetivo pueden provocar efectos secundarios indeseables . [12] Debido a las similitudes en los sitios de unión, los objetivos estrechamente relacionados identificados a través de la homología de secuencia tienen la mayor probabilidad de reactividad cruzada y, por lo tanto, el mayor potencial de efectos secundarios.
En la mayoría de los casos, los fármacos son pequeñas moléculas orgánicas producidas mediante síntesis química, pero los fármacos basados en biopolímeros (también conocidos como biofármacos ) producidos mediante procesos biológicos son cada vez más comunes. [13] Además, las tecnologías de silenciamiento de genes basadas en ARNm pueden tener aplicaciones terapéuticas. [14] Por ejemplo, las nanomedicinas basadas en ARNm pueden agilizar y acelerar el proceso de desarrollo de fármacos, lo que permite la expresión transitoria y localizada de moléculas inmunoestimulantes. [15] El ARNm transcrito in vitro (IVT) permite la administración a varios tipos de células accesibles a través de la sangre o vías alternativas. El uso de ARNm IVT sirve para transmitir información genética específica a las células de una persona, con el objetivo principal de prevenir o alterar una enfermedad en particular. [16]
Descubrimiento de fármacos
Descubrimiento fenotípico de fármacos
El descubrimiento fenotípico de fármacos es un método tradicional de descubrimiento de fármacos, también conocido como farmacología avanzada o farmacología clásica. Utiliza el proceso de cribado fenotípico en colecciones de pequeñas moléculas sintéticas, productos naturales o extractos dentro de bibliotecas químicas para identificar sustancias que exhiben efectos terapéuticos beneficiosos. Este método consiste en descubrir primero la actividad funcional in vivo o in vitro de los fármacos (como los fármacos de extracto o los productos naturales) y luego realizar la identificación del objetivo. El descubrimiento fenotípico utiliza un enfoque práctico e independiente del objetivo para generar pistas iniciales, con el objetivo de descubrir compuestos farmacológicamente activos y terapias que operan a través de nuevos mecanismos farmacológicos. [17] Este método permite la exploración de fenotipos de enfermedades para encontrar tratamientos potenciales para afecciones con orígenes desconocidos, complejos o multifactoriales, donde la comprensión de los objetivos moleculares es insuficiente para una intervención eficaz. [18]
Descubrimiento racional de fármacos
El diseño racional de fármacos (también llamado farmacología inversa ) comienza con la hipótesis de que la modulación de un objetivo biológico específico puede tener valor terapéutico. Para que una biomolécula sea seleccionada como objetivo de un fármaco, se requieren dos datos esenciales. El primero es la evidencia de que la modulación del objetivo modificará la enfermedad. Este conocimiento puede provenir, por ejemplo, de estudios de vinculación de enfermedades que muestran una asociación entre mutaciones en el objetivo biológico y ciertos estados patológicos. [19] El segundo es que el objetivo sea capaz de unirse a una molécula pequeña y que su actividad pueda ser modulada por la molécula pequeña. [20]
Una vez que se ha identificado un objetivo adecuado, normalmente se clona , se produce y se purifica . La proteína purificada se utiliza luego para establecer un ensayo de detección . Además, se puede determinar la estructura tridimensional del objetivo.
La búsqueda de pequeñas moléculas que se unan al objetivo se inicia con la selección de bibliotecas de compuestos farmacológicos potenciales. Esto se puede hacer utilizando el ensayo de selección ("pantalla húmeda"). Además, si se dispone de la estructura del objetivo, se puede realizar una selección virtual de los fármacos candidatos. Idealmente, los compuestos farmacológicos candidatos deberían ser " similares a los fármacos ", es decir, deberían poseer propiedades que se prevea que conduzcan a una biodisponibilidad oral , una estabilidad química y metabólica adecuada y efectos tóxicos mínimos. [21] Hay varios métodos disponibles para estimar la similitud con los fármacos, como la Regla de los Cinco de Lipinski y una variedad de métodos de puntuación, como la eficiencia lipofílica . [22] También se han propuesto varios métodos para predecir el metabolismo de los fármacos en la literatura científica. [23]
Debido a la gran cantidad de propiedades de los fármacos que deben optimizarse simultáneamente durante el proceso de diseño, a veces se emplean técnicas de optimización multiobjetivo . [24] Finalmente, debido a las limitaciones de los métodos actuales para la predicción de la actividad, el diseño de fármacos todavía depende en gran medida de la serendipia [25] y la racionalidad limitada . [26]
Diseño de fármacos asistido por ordenador
El objetivo más fundamental en el diseño de fármacos es predecir si una molécula dada se unirá a un objetivo y, de ser así, con qué fuerza. La mecánica molecular o la dinámica molecular se utilizan con mayor frecuencia para estimar la fuerza de la interacción intermolecular entre la molécula pequeña y su objetivo biológico. Estos métodos también se utilizan para predecir la conformación de la molécula pequeña y para modelar los cambios conformacionales en el objetivo que pueden ocurrir cuando la molécula pequeña se une a él. [3] [4] Los métodos semiempíricos , de química cuántica ab initio o la teoría funcional de la densidad se utilizan a menudo para proporcionar parámetros optimizados para los cálculos de mecánica molecular y también proporcionar una estimación de las propiedades electrónicas (potencial electrostático, polarizabilidad , etc.) del candidato a fármaco que influirán en la afinidad de unión. [27]
También se pueden utilizar métodos de mecánica molecular para proporcionar una predicción semicuantitativa de la afinidad de unión. Además, se puede utilizar una función de puntuación basada en el conocimiento para proporcionar estimaciones de la afinidad de unión. Estos métodos utilizan regresión lineal , aprendizaje automático , redes neuronales u otras técnicas estadísticas para derivar ecuaciones predictivas de afinidad de unión ajustando las afinidades experimentales a las energías de interacción derivadas computacionalmente entre la molécula pequeña y el objetivo. [28] [29]
Lo ideal sería que el método computacional pudiera predecir la afinidad antes de sintetizar un compuesto, por lo que en teoría solo se necesita sintetizar un compuesto, lo que ahorraría mucho tiempo y dinero. La realidad es que los métodos computacionales actuales son imperfectos y, en el mejor de los casos, solo brindan estimaciones cualitativamente precisas de la afinidad. En la práctica, se requieren varias iteraciones de diseño, síntesis y pruebas antes de descubrir un fármaco óptimo. Los métodos computacionales han acelerado el descubrimiento al reducir el número de iteraciones necesarias y, a menudo, han proporcionado estructuras novedosas. [30] [31]
El diseño de fármacos asistido por computadora se puede utilizar en cualquiera de las siguientes etapas del descubrimiento de fármacos:
- Identificación de resultados mediante cribado virtual (diseño basado en estructura o ligando)
- Optimización de afinidad y selectividad hit-to-lead (diseño basado en estructura, QSAR , etc.)
- Optimización de otras propiedades farmacéuticas manteniendo la afinidad

Para superar la predicción insuficiente de la afinidad de unión calculada por las funciones de puntuación recientes, se utilizan para el análisis la interacción proteína-ligando y la información de la estructura 3D del compuesto. Para el diseño de fármacos basado en la estructura, se han desarrollado varios análisis posteriores a la selección centrados en la interacción proteína-ligando para mejorar el enriquecimiento y extraer de forma eficaz candidatos potenciales:
- Puntuación de consenso [32] [33]
- Selección de candidatos mediante votación de múltiples funciones de puntuación
- Puede perderse la relación entre la información estructural proteína-ligando y el criterio de puntuación
- Análisis de conglomerados [34] [35]
- Representar y agrupar candidatos según la información 3D proteína-ligando
- Necesita una representación significativa de las interacciones proteína-ligando.
Tipos
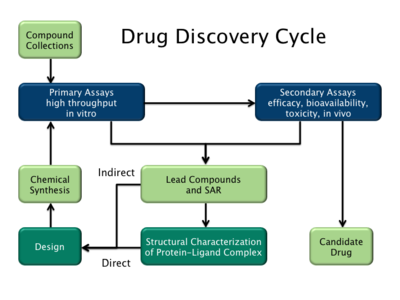
Existen dos tipos principales de diseño de fármacos. El primero se denomina diseño de fármacos basado en ligandos y el segundo, diseño de fármacos basado en la estructura. [2]
Basado en ligando
El diseño de fármacos basado en ligandos (o diseño indirecto de fármacos ) se basa en el conocimiento de otras moléculas que se unen al objetivo biológico de interés. Estas otras moléculas pueden utilizarse para derivar un modelo de farmacóforo que define las características estructurales mínimas necesarias que debe poseer una molécula para unirse al objetivo. [36] Se puede construir un modelo del objetivo biológico basándose en el conocimiento de lo que se une a él, y este modelo a su vez puede utilizarse para diseñar nuevas entidades moleculares que interactúen con el objetivo. Alternativamente, se puede derivar una relación cuantitativa estructura-actividad (QSAR), en la que se correlaciona entre las propiedades calculadas de las moléculas y su actividad biológica determinada experimentalmente . Estas relaciones QSAR a su vez pueden utilizarse para predecir la actividad de nuevos análogos. [37]
Basado en la estructura
El diseño de fármacos basado en la estructura (o diseño directo de fármacos ) se basa en el conocimiento de la estructura tridimensional del objetivo biológico obtenido a través de métodos como la cristalografía de rayos X o la espectroscopia RMN . [38] Si no se dispone de una estructura experimental de un objetivo, puede ser posible crear un modelo de homología del objetivo basado en la estructura experimental de una proteína relacionada. Utilizando la estructura del objetivo biológico, se pueden diseñar fármacos candidatos que se predice que se unirán con alta afinidad y selectividad al objetivo utilizando gráficos interactivos y la intuición de un químico medicinal . Alternativamente, se pueden utilizar varios procedimientos computacionales automatizados para sugerir nuevos candidatos a fármacos. [39]
Los métodos actuales para el diseño de fármacos basados en la estructura se pueden dividir en tres categorías principales. [40] El primer método es la identificación de nuevos ligandos para un receptor determinado mediante la búsqueda en grandes bases de datos de estructuras 3D de moléculas pequeñas para encontrar aquellas que se ajusten al bolsillo de unión del receptor utilizando programas rápidos de acoplamiento aproximado . Este método se conoce como cribado virtual .
Una segunda categoría es el diseño de novo de nuevos ligandos. En este método, las moléculas de ligando se construyen dentro de las limitaciones de la cavidad de unión mediante el ensamblaje de pequeñas piezas de manera gradual. Estas piezas pueden ser átomos individuales o fragmentos moleculares. La principal ventaja de este método es que se pueden sugerir estructuras nuevas, que no están contenidas en ninguna base de datos. [41] [42] [43] Un tercer método es la optimización de ligandos conocidos mediante la evaluación de análogos propuestos dentro de la cavidad de unión. [40]
Identificación del sitio de enlace
La identificación del sitio de unión es el primer paso en el diseño basado en la estructura. [20] [44] Si la estructura del objetivo o un homólogo suficientemente similar se determina en presencia de un ligando unido, entonces el ligando debería ser observable en la estructura, en cuyo caso la ubicación del sitio de unión es trivial. Sin embargo, puede haber sitios de unión alostéricos desocupados que puedan ser de interés. Además, puede ser que solo estén disponibles estructuras de apoproteína (proteína sin ligando) y la identificación confiable de sitios desocupados que tienen el potencial de unir ligandos con alta afinidad no sea trivial. En resumen, la identificación del sitio de unión generalmente se basa en la identificación de superficies cóncavas en la proteína que pueden acomodar moléculas del tamaño de un fármaco que también poseen "puntos calientes" apropiados ( superficies hidrofóbicas , sitios de unión de hidrógeno , etc.) que impulsan la unión del ligando. [20] [44]
Funciones de puntuación
El diseño de fármacos basado en la estructura intenta utilizar la estructura de las proteínas como base para diseñar nuevos ligandos aplicando los principios del reconocimiento molecular . La unión selectiva de alta afinidad al objetivo es generalmente deseable ya que conduce a fármacos más eficaces con menos efectos secundarios. Por lo tanto, uno de los principios más importantes para diseñar u obtener nuevos ligandos potenciales es predecir la afinidad de unión de un determinado ligando a su objetivo (y antiobjetivos conocidos ) y utilizar la afinidad predicha como criterio de selección. [45]
Böhm desarrolló una de las primeras funciones de puntuación empírica de uso general para describir la energía de enlace de los ligandos a los receptores. [46] [47] Esta función de puntuación empírica tomó la forma:
dónde:
- ΔG 0 – desplazamiento derivado empíricamente que corresponde en parte a la pérdida general de entropía traslacional y rotacional del ligando al unirse.
- ΔG hb – contribución del enlace de hidrógeno
- ΔG iónico : contribución de las interacciones iónicas
- ΔG lip – contribución de las interacciones lipofílicas donde |A lipo | es el área superficial de contacto lipofílico entre el ligando y el receptor
- ΔG rot – penalización de entropía debido a la congelación de un elemento rotatorio en el enlace del ligando al momento de la unión
Una ecuación termodinámica "maestra" más general es la siguiente: [48]
dónde:
- Desolvatación: penalización entálpica por eliminar el ligando del solvente
- Movimiento: penalización entrópica por reducir los grados de libertad cuando un ligando se une a su receptor.
- Configuración: energía de tensión conformacional necesaria para poner al ligando en su conformación "activa"
- Interacción: ganancia entálpica para “resolver” el ligando con su receptor.
La idea básica es que la energía libre de enlace total se puede descomponer en componentes independientes que se sabe que son importantes para el proceso de enlace. Cada componente refleja un cierto tipo de alteración de la energía libre durante el proceso de enlace entre un ligando y su receptor objetivo. La ecuación maestra es la combinación lineal de estos componentes. De acuerdo con la ecuación de energía libre de Gibbs, se construyó la relación entre la constante de equilibrio de disociación, K d , y los componentes de la energía libre.
Se utilizan varios métodos computacionales para estimar cada uno de los componentes de la ecuación maestra. Por ejemplo, el cambio en el área de superficie polar tras la unión del ligando se puede utilizar para estimar la energía de desolvatación. El número de enlaces rotatorios congelados tras la unión del ligando es proporcional al término de movimiento. La energía configuracional o de deformación se puede estimar utilizando cálculos de mecánica molecular . Finalmente, la energía de interacción se puede estimar utilizando métodos como el cambio en la superficie no polar, potenciales derivados estadísticamente de la fuerza media , el número de enlaces de hidrógeno formados, etc. En la práctica, los componentes de la ecuación maestra se ajustan a los datos experimentales utilizando regresión lineal múltiple. Esto se puede hacer con un conjunto de entrenamiento diverso que incluya muchos tipos de ligandos y receptores para producir un modelo "global" menos preciso pero más general o un conjunto más restringido de ligandos y receptores para producir un modelo "local" más preciso pero menos general. [49]
Ejemplos
Un ejemplo particular de diseño racional de fármacos implica el uso de información tridimensional sobre biomoléculas obtenida a partir de técnicas como la cristalografía de rayos X y la espectroscopia de RMN. El diseño de fármacos asistido por ordenador, en particular, se vuelve mucho más manejable cuando existe una estructura de alta resolución de una proteína diana unida a un ligando potente. Este enfoque para el descubrimiento de fármacos se conoce a veces como diseño de fármacos basado en la estructura. El primer ejemplo inequívoco de la aplicación del diseño de fármacos basado en la estructura que conduce a un fármaco aprobado es el inhibidor de la anhidrasa carbónica dorzolamida , que fue aprobado en 1995. [50] [51]
Otro caso de estudio en el diseño racional de fármacos es el imatinib , un inhibidor de la tirosina quinasa diseñado específicamente para la proteína de fusión bcr-abl que es característica de las leucemias positivas para el cromosoma Filadelfia ( leucemia mieloide crónica y ocasionalmente leucemia linfocítica aguda ). El imatinib es sustancialmente diferente de los fármacos anteriores para el cáncer , ya que la mayoría de los agentes de quimioterapia simplemente se dirigen a las células que se dividen rápidamente, sin diferenciar entre las células cancerosas y otros tejidos. [52]
Otros ejemplos incluyen:
- Muchos de los antipsicóticos atípicos
- Cimetidina , el antagonista prototípico del receptor H2 a partir del cual se desarrollaron los miembros posteriores de la clase.
- AINE inhibidores selectivos de la COX-2
- Enfuvirtida , un péptido inhibidor de la entrada del VIH
- Medicamentos no benzodiazepínicos como zolpidem y zopiclona
- Raltegravir , un inhibidor de la integrasa del VIH [53]
- ISRS (inhibidores selectivos de la recaptación de serotonina), una clase de antidepresivos
- Zanamivir , un fármaco antiviral
Prueba de detección de drogas
Los tipos de cribado de fármacos incluyen el cribado fenotípico , el cribado de alto rendimiento y el cribado virtual . El cribado fenotípico se caracteriza por el proceso de cribado de fármacos utilizando modelos de enfermedades animales o celulares para identificar compuestos que alteran el fenotipo y producen efectos beneficiosos relacionados con la enfermedad. [54] [55] Las tecnologías emergentes en el cribado de alto rendimiento mejoran sustancialmente la velocidad de procesamiento y disminuyen el volumen de detección necesario. [56] El cribado virtual se completa por computadora, lo que permite que se pueda cribar una gran cantidad de moléculas con un ciclo corto y bajo costo. El cribado virtual utiliza una variedad de métodos computacionales que permiten a los químicos reducir bibliotecas virtuales extensas a tamaños más manejables. [57]
Estudios de caso
- Antagonistas de 5-HT3
- Agonistas del receptor de acetilcolina
- Antagonistas del receptor de angiotensina
- Inhibidores de la tirosina quinasa Bcr-Abl
- Antagonistas de los receptores cannabinoides
- Antagonistas del receptor CCR5
- Inhibidores de la ciclooxigenasa 2
- Inhibidores de la dipeptidil peptidasa-4
- Inhibidores de la proteasa del VIH
- Antagonistas del receptor NK1
- Inhibidores no nucleósidos de la transcriptasa inversa
- Inhibidores de la transcriptasa inversa nucleósidos y nucleótidos
- Inhibidores de la PDE5
- Inhibidores de la bomba de protones
- Inhibidores de la renina
- Triptanos
- Antagonistas de TRPV1
- Inhibidores de c-Met
Crítica
Se ha argumentado que la naturaleza altamente rígida y centrada del diseño racional de fármacos suprime la serendipia en el descubrimiento de fármacos. [58]
Véase también
Referencias
- ^ Madsen U, Krogsgaard-Larsen P, Liljefors T (2002). Libro de texto sobre diseño y descubrimiento de fármacos . Washington, DC: Taylor y Francis. ISBN 978-0-415-28288-8.
- ^ abc Reynolds CH, Merz KM, Ringe D, eds. (2010). Diseño de fármacos: enfoques basados en la estructura y el ligando (1.ª ed.). Cambridge, Reino Unido: Cambridge University Press. ISBN 978-0521887236.
- ^ ab Fosgerau K, Hoffmann T (enero de 2015). "Terapéutica peptídica: estado actual y direcciones futuras". Drug Discovery Today . 20 (1): 122–128. doi : 10.1016/j.drudis.2014.10.003 . PMID 25450771.
- ^ ab Ciemny M, Kurcinski M, Kamel K, Kolinski A, Alam N, Schueler-Furman O, Kmiecik S (agosto de 2018). "Acoplamiento proteína-péptido: oportunidades y desafíos". Drug Discovery Today . 23 (8): 1530–1537. doi : 10.1016/j.drudis.2018.05.006 . PMID 29733895.
- ^ Shirai H, Prades C, Vita R, Marcatili P, Popovic B, Xu J, et al. (noviembre de 2014). "Informática de anticuerpos para el descubrimiento de fármacos". Biochimica et Biophysica Acta (BBA) - Proteínas y Proteómica . 1844 (11): 2002-2015. doi :10.1016/j.bbapap.2014.07.006. PMID 25110827.
- ^ Tollenaere JP (abril de 1996). "El papel del diseño de ligandos basado en la estructura y el modelado molecular en el descubrimiento de fármacos". Pharmacy World & Science . 18 (2): 56–62. doi :10.1007/BF00579706. PMID 8739258. S2CID 21550508.
- ^ Waring MJ, Arrowsmith J, Leach AR, Leeson PD, Mandrell S, Owen RM, et al. (julio de 2015). "Análisis de la deserción de candidatos a fármacos de cuatro importantes compañías farmacéuticas". Nature Reviews. Drug Discovery . 14 (7): 475–486. doi :10.1038/nrd4609. PMID 26091267. S2CID 25292436.
- ^ Yu H, Adedoyin A (septiembre de 2003). "ADME-Tox en el descubrimiento de fármacos: integración de tecnologías experimentales y computacionales". Drug Discovery Today . 8 (18): 852–861. doi :10.1016/S1359-6446(03)02828-9. PMID 12963322.
- ^ Dixon SJ, Stockwell BR (diciembre de 2009). "Identificación de productos genéticos modificadores de enfermedades que pueden tratarse con fármacos". Current Opinion in Chemical Biology . 13 (5–6): 549–555. doi :10.1016/j.cbpa.2009.08.003. PMC 2787993 . PMID 19740696.
- ^ Imming P, Sinning C, Meyer A (octubre de 2006). "Fármacos, sus objetivos y la naturaleza y número de objetivos farmacológicos". Nature Reviews. Drug Discovery . 5 (10): 821–834. doi :10.1038/nrd2132. PMID 17016423. S2CID 8872470.
- ^ Anderson AC (septiembre de 2003). "El proceso de diseño de fármacos basado en la estructura". Química y biología . 10 (9): 787–797. doi : 10.1016/j.chembiol.2003.09.002 . PMID 14522049.
- ^ Recanatini M, Bottegoni G, Cavalli A (diciembre de 2004). "Detección in silico de dianas terapéuticas". Drug Discovery Today: Technologies . 1 (3): 209–215. doi :10.1016/j.ddtec.2004.10.004. PMID 24981487.
- ^ Wu-Pong S, Rojanasakul Y (2008). Diseño y desarrollo de fármacos biofarmacéuticos (2ª ed.). Totowa, Nueva Jersey Humana Press: Humana Press. ISBN 978-1-59745-532-9.
- ^ Scomparin A, Polyak D, Krivitsky A, Satchi-Fainaro R (noviembre de 2015). "Lograr la entrega exitosa de oligonucleótidos: desde la caracterización fisicoquímica hasta la evaluación in vivo". Avances en biotecnología . 33 (6 partes 3): 1294–1309. doi :10.1016/j.biotechadv.2015.04.008. PMID 25916823.
- ^ Youssef M, Hitti C, Puppin Chaves Fulber J, Kamen AA (octubre de 2023). "Habilitación de terapias de ARNm: panorama actual y desafíos en la fabricación". Biomoléculas . 13 (10): 1497. doi : 10.3390/biom13101497 . PMC 10604719 . PMID 37892179.
- ^ Sahin U, Karikó K, Türeci Ö (octubre de 2014). «Terapéutica basada en ARNm: desarrollo de una nueva clase de fármacos». Nature Reviews. Drug Discovery . 13 (10): 759–780. doi : 10.1038/nrd4278 . PMID: 25233993. S2CID : 27454546.
- ^ Swinney DC, Lee JA (2020). "Avances recientes en el descubrimiento de fármacos fenotípicos". F1000Research . 9 : F1000 Faculty Rev–944. doi : 10.12688/f1000research.25813.1 . PMC 7431967 . PMID 32850117.
- ^ Moffat JG, Vincent F, Lee JA, Eder J, Prunotto M (agosto de 2017). "Oportunidades y desafíos en el descubrimiento de fármacos fenotípicos: una perspectiva de la industria". Nature Reviews. Descubrimiento de fármacos . 16 (8): 531–543. doi : 10.1038/nrd.2017.111 . PMID 28685762. S2CID 6180139.
- ^ Ganellin CR, Jefferis R, Roberts SM (2013). "El proceso de descubrimiento de fármacos de moléculas pequeñas: desde la selección de objetivos hasta la selección de candidatos". Introducción a la investigación y el desarrollo de fármacos biológicos y de moléculas pequeñas: teoría y estudios de casos . Elsevier. ISBN 9780123971760.
- ^ abc Yuan Y, Pei J, Lai L (diciembre de 2013). "Detección de sitios de unión y predicción de la farmacogenibilidad de proteínas diana para el diseño de fármacos basado en la estructura". Current Pharmaceutical Design . 19 (12): 2326–2333. doi :10.2174/1381612811319120019. PMID 23082974.
- ^ Rishton GM (enero de 2003). "Nonleadlikeness y leadlikeness in biochemical screening" (Similares al plomo y no al plomo en el cribado bioquímico). Drug Discovery Today . 8 (2): 86–96. doi :10.1016/s1359644602025722. PMID 12565011.
- ^ Hopkins AL (2011). "Capítulo 25: Espacio farmacológico". En Wermuth CG (ed.). La práctica de la química medicinal (3.ª ed.). Academic Press. págs. 521–527. ISBN 978-0-12-374194-3.
- ^ Kirchmair J (2014). Predicción del metabolismo de fármacos . Métodos y principios de química medicinal de Wiley. Vol. 63. Wiley-VCH. ISBN 978-3-527-67301-8.
- ^ Nicolaou CA, Brown N (septiembre de 2013). "Métodos de optimización multiobjetivo en el diseño de fármacos". Drug Discovery Today: Technologies . 10 (3): e427–e435. doi :10.1016/j.ddtec.2013.02.001. PMID 24050140.
- ^ Ban TA (2006). "El papel de la serendipia en el descubrimiento de fármacos". Diálogos en neurociencia clínica . 8 (3): 335–344. doi :10.31887/DCNS.2006.8.3/tban. PMC 3181823 . PMID 17117615.
- ^ Ethiraj SK, Levinthal D (septiembre de 2004). "Racionalidad limitada y la búsqueda de una arquitectura organizacional: una perspectiva evolutiva sobre el diseño de las organizaciones y su capacidad de evolución". Administrative Science Quarterly . 49 (3). Sage Publications, Inc. en nombre de la Johnson Graduate School of Management, Cornell University: 404–437. doi :10.2307/4131441. JSTOR 4131441. S2CID 142910916. SSRN 604123.
- ^ Lewis RA (2011). "Capítulo 4: El desarrollo de programas de modelado molecular: el uso y las limitaciones de los modelos físicos". En Gramatica P, Livingstone DJ, Davis AM (eds.). Estrategias de diseño de fármacos: enfoques cuantitativos . RSC Drug Discovery. Royal Society of Chemistry. págs. 88–107. doi :10.1039/9781849733410-00088. ISBN . 978-1849731669.
- ^ Rajamani R, Good AC (mayo de 2007). "Pose de clasificación en el descubrimiento y optimización de líderes basados en la estructura: tendencias actuales en el desarrollo de funciones de puntuación". Current Opinion in Drug Discovery & Development . 10 (3): 308–315. PMID 17554857.
- ^ de Azevedo WF, Dias R (diciembre de 2008). "Métodos computacionales para el cálculo de la afinidad de unión a ligandos". Current Drug Targets . 9 (12): 1031–1039. doi :10.2174/138945008786949405. PMID 19128212.
- ^ Singh J, Chuaqui CE, Boriack-Sjodin PA, Lee WC, Pontz T, Corbley MJ, et al. (diciembre de 2003). "Exitosa detección virtual basada en la forma: el descubrimiento de un potente inhibidor de la quinasa del receptor de TGFbeta tipo I (TbetaRI)". Bioorganic & Medicinal Chemistry Letters . 13 (24): 4355–4359. doi :10.1016/j.bmcl.2003.09.028. PMID 14643325.
- ^ Becker OM, Dhanoa DS, Marantz Y, Chen D, Shacham S, Cheruku S, et al. (junio de 2006). "Un descubrimiento basado en un modelo 3D integrado in silico de un agonista de amidosulfonamida 5-HT1A novedoso, potente y selectivo (PRX-00023) para el tratamiento de la ansiedad y la depresión". Journal of Medicinal Chemistry . 49 (11): 3116–3135. doi :10.1021/jm0508641. PMID 16722631.
- ^ Liang S, Meroueh SO, Wang G, Qiu C, Zhou Y (mayo de 2009). "Puntuación de consenso para enriquecer estructuras casi nativas a partir de señuelos de acoplamiento proteína-proteína". Proteins . 75 (2): 397–403. doi :10.1002/prot.22252. PMC 2656599 . PMID 18831053.
- ^ Oda A, Tsuchida K, Takakura T, Yamaotsu N, Hirono S (2006). "Comparación de estrategias de puntuación de consenso para evaluar modelos computacionales de complejos proteína-ligando". Revista de información y modelado químico . 46 (1): 380–391. doi :10.1021/ci050283k. PMID 16426072.
- ^ Deng Z, Chuaqui C, Singh J (enero de 2004). "Huella de interacción estructural (SIFt): un nuevo método para analizar interacciones tridimensionales de unión proteína-ligando". Journal of Medicinal Chemistry . 47 (2): 337–344. doi :10.1021/jm030331x. PMID 14711306.
- ^ Amari S, Aizawa M, Zhang J, Fukuzawa K, Mochizuki Y, Iwasawa Y, et al. (2006). "VISCANA: análisis de agrupamiento visualizado de la interacción proteína-ligando basado en el método de orbital molecular de fragmentos ab initio para la detección de ligandos virtuales". Journal of Chemical Information and Modeling . 46 (1): 221–230. doi :10.1021/ci050262q. PMID 16426058.
- ^ Guner OF (2000). Percepción, desarrollo y uso de farmacóforos en el diseño de fármacos . La Jolla, California: International University Line. ISBN 978-0-9636817-6-8.
- ^ Tropsha A (2010). "QSAR en el descubrimiento de fármacos". En Reynolds CH, Merz KM, Ringe D (eds.). Diseño de fármacos: enfoques basados en la estructura y el ligando (1.ª ed.). Cambridge, Reino Unido: Cambridge University Press. págs. 151–164. ISBN 978-0521887236.
- ^ Leach AR, Harren J (2007). Descubrimiento de fármacos basado en estructuras . Berlín: Springer. ISBN 978-1-4020-4406-9.
- ^ Mauser H, Guba W (mayo de 2008). "Desarrollos recientes en diseño de novo y salto de andamiaje". Current Opinion in Drug Discovery & Development . 11 (3): 365–374. PMID 18428090.
- ^ ab Klebe G (2000). "Desarrollos recientes en el diseño de fármacos basado en la estructura". Revista de Medicina Molecular . 78 (5): 269–281. doi :10.1007/s001090000084. PMID 10954199. S2CID 21314020.
- ^ Wang R, Gao Y, Lai L (2000). "LigBuilder: un programa multipropósito para el diseño de fármacos basado en la estructura". Journal of Molecular Modeling . 6 (7–8): 498–516. doi :10.1007/s0089400060498. S2CID 59482623.
- ^ Schneider G, Fechner U (agosto de 2005). "Diseño de novo basado en computadora de moléculas similares a fármacos". Nature Reviews. Drug Discovery . 4 (8): 649–663. doi :10.1038/nrd1799. PMID 16056391. S2CID 2549851.
- ^ Jorgensen WL (marzo de 2004). "Los múltiples roles de la computación en el descubrimiento de fármacos". Science . 303 (5665): 1813–1818. Bibcode :2004Sci...303.1813J. doi :10.1126/science.1096361. PMID 15031495. S2CID 1307935.
- ^ ab Leis S, Schneider S, Zacharias M (2010). "Predicción in silico de sitios de unión en proteínas". Química medicinal actual . 17 (15): 1550–1562. doi :10.2174/092986710790979944. PMID 20166931.
- ^ Warren GL, Warren SD (2011). "Capítulo 16: Puntuación de interacciones fármaco-receptor". En Gramatica P, Livingstone DJ, Davis AM (eds.). Estrategias de diseño de fármacos: enfoques cuantitativos . RSC Drug Discovery. Royal Society of Chemistry. págs. 440–457. doi :10.1039/9781849733410-00440. ISBN . 978-1849731669.
- ^ Böhm HJ (junio de 1994). "El desarrollo de una función de puntuación empírica simple para estimar la constante de unión para un complejo proteína-ligando de estructura tridimensional conocida". Journal of Computer-Aided Molecular Design . 8 (3): 243–256. Bibcode :1994JCAMD...8..243B. doi :10.1007/BF00126743. PMID 7964925. S2CID 2491616.
- ^ Liu J, Wang R (marzo de 2015). "Clasificación de las funciones de puntuación actuales". Revista de información y modelado químico . 55 (3): 475–482. doi :10.1021/ci500731a. PMID 25647463.
- ^ Murcko MA (diciembre de 1995). "Métodos computacionales para predecir la energía libre de enlace en complejos ligando-receptor". Journal of Medicinal Chemistry . 38 (26): 4953–4967. doi :10.1021/jm00026a001. PMID 8544170.
- ^ Gramatica P (2011). "Capítulo 17: Modelado de sustancias químicas en el medio ambiente". En Gramatica P, Livingstone DJ, Davis AM (eds.). Estrategias de diseño de fármacos: enfoques cuantitativos . RSC Drug Discovery. Royal Society of Chemistry. pág. 466. doi :10.1039/9781849733410-00458. ISBN . 978-1849731669.
- ^ Greer J, Erickson JW, Baldwin JJ, Varney MD (abril de 1994). "Aplicación de las estructuras tridimensionales de las moléculas diana de proteínas en el diseño de fármacos basado en la estructura". Journal of Medicinal Chemistry . 37 (8): 1035–1054. doi :10.1021/jm00034a001. PMID 8164249.
- ^ Timmerman H, Gubernator K, Böhm HJ, Mannhold R, Kubinyi H (1998). Diseño de ligando basado en la estructura (métodos y principios en química medicinal) . Weinheim: Wiley-VCH. ISBN 978-3-527-29343-8.
- ^ Capdeville R, Buchdunger E, Zimmermann J, Matter A (julio de 2002). "Glivec (STI571, imatinib), un fármaco anticanceroso dirigido y desarrollado racionalmente". Nature Reviews. Drug Discovery . 1 (7): 493–502. doi :10.1038/nrd839. PMID 12120256. S2CID 2728341.
- ^ "El papel de AutoDock en el desarrollo del primer inhibidor de la integrasa del VIH aprobado clínicamente". Nota de prensa . The Scripps Research Institute. 17 de diciembre de 2007.
- ^ Prior M, Chiruta C, Currais A, Goldberg J, Ramsey J, Dargusch R, et al. (julio de 2014). "Regreso al futuro con el cribado fenotípico". ACS Chemical Neuroscience . 5 (7): 503–513. doi :10.1021/cn500051h. PMC 4102969 . PMID 24902068.
- ^ Kotz J (abril de 2012). "Cribado fenotípico, segunda parte". Science-Business EXchange . 5 (15): 380. doi : 10.1038/scibx.2012.380 . ISSN 1945-3477. S2CID 72519717.
- ^ Hertzberg RP, Pope AJ (agosto de 2000). "Cribado de alto rendimiento: nueva tecnología para el siglo XXI". Current Opinion in Chemical Biology . 4 (4): 445–451. doi :10.1016/S1367-5931(00)00110-1. PMID 10959774.
- ^ Walters WP, Stahl MT, Murcko MA (abril de 1998). "Cribado virtual: una descripción general". Drug Discovery Today . 3 (4): 160–178. doi :10.1016/S1359-6446(97)01163-X.
- ^ Klein DF (marzo de 2008). "La pérdida de la serendipia en la psicofarmacología". JAMA . 299 (9): 1063–1065. doi :10.1001/jama.299.9.1063. PMID 18319418.
Enlaces externos
- Drug+Design en los encabezamientos de temas médicos (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- [Organización de diseño de medicamentos](https://www.drugdesign.org/chapters/drug-design/)

![{\displaystyle {\begin{array}{lll}\Delta G_{\text{bind}}=-RT\ln K_{\text{d}}\\[1.3ex]K_{\text{d}}={\dfrac {[{\text{Ligando}}][{\text{Receptor}}]}{[{\text{Complejo}}]}}\\[1.3ex]\Delta G_{\text{bind}}=\Delta G_{\text{desolvatación}}+\Delta G_{\text{movimiento}}+\Delta G_{\text{configuración}}+\Delta G_{\text{interacción}}\end{array}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ba49ddd9dec7415d129787213744ca1afcd2d021)