Coronavirus
| Ortocoronavirinae | |
|---|---|
 | |
| Miembro del grupo SARS-CoV-2 Clave de la ilustración:
| |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Virus de la ortiga |
| Filo: | Pisuviricota |
| Clase: | Pisoniviricetes |
| Orden: | Nidovirus |
| Familia: | Coronavirus |
| Subfamilia: | Ortocoronavirinae |
| Géneros [1] | |
Información: | |
| Sinónimos [2] [3] | |
| |
Los coronavirus son un grupo de virus ARN relacionados que causan enfermedades en mamíferos y aves. En humanos y aves, causan infecciones del tracto respiratorio que pueden ir de leves a letales. Las enfermedades leves en humanos incluyen algunos casos de resfriado común (que también es causado por otros virus, predominantemente rinovirus ), mientras que las variedades más letales pueden causar SARS , MERS y COVID-19 . En vacas y cerdos causan diarrea , mientras que en ratones causan hepatitis y encefalomielitis .
Los coronavirus constituyen la subfamilia Orthocoronavirinae , en la familia Coronaviridae , orden Nidovirales y reino Riboviria . [3] [4] Son virus envueltos con un genoma de ARN monocatenario de sentido positivo y una nucleocápside de simetría helicoidal. [5] El tamaño del genoma de los coronavirus varía de aproximadamente 26 a 32 kilobases , uno de los más grandes entre los virus de ARN. [6] Tienen picos característicos en forma de maza que se proyectan desde su superficie, que en las micrografías electrónicas crean una imagen que recuerda a la corona estelar , de la que deriva su nombre. [7]
Etimología
El nombre "coronavirus" se deriva del latín corona , que significa "corona" o "guirnalda", un préstamo del griego κορώνη korṓnē , "guirnalda, corona". [8] [9] El nombre fue acuñado por June Almeida y David Tyrrell, quienes observaron y estudiaron por primera vez los coronavirus humanos. [10] La palabra fue utilizada por primera vez en forma impresa en 1968 por un grupo informal de virólogos en la revista Nature para designar a la nueva familia de virus. [7] El nombre se refiere a la apariencia característica de los viriones (la forma infecciosa del virus) por microscopía electrónica , que tienen una franja de proyecciones superficiales grandes y bulbosas que crean una imagen que recuerda a la corona o halo solar . [7] [10] Esta morfología es creada por los peplómeros de la espiga viral , que son proteínas en la superficie del virus. [11]
El nombre científico Coronavirus fue aceptado como nombre de género por el Comité Internacional para la Nomenclatura de Virus (posteriormente renombrado Comité Internacional de Taxonomía de Virus ) en 1971. [12] A medida que aumentó el número de nuevas especies, el género se dividió en cuatro géneros, a saber, Alphacoronavirus , Betacoronavirus , Deltacoronavirus y Gammacoronavirus en 2009. [13] El nombre común coronavirus se utiliza para referirse a cualquier miembro de la subfamilia Orthocoronavirinae . [4] A partir de 2020, 45 especies están reconocidas oficialmente. [14]
Historia

Los primeros informes de una infección por coronavirus en animales ocurrieron a fines de la década de 1920, cuando surgió una infección respiratoria aguda de pollos domésticos en América del Norte. [15] Arthur Schalk y MC Hawn en 1931 realizaron el primer informe detallado que describía una nueva infección respiratoria de pollos en Dakota del Norte . La infección de los polluelos recién nacidos se caracterizó por jadeo y apatía con altas tasas de mortalidad del 40 al 90%. [16] Leland David Bushnell y Carl Alfred Brandly aislaron el virus que causó la infección en 1933. [17] El virus se conocía entonces como virus de la bronquitis infecciosa (IBV). Charles D. Hudson y Fred Robert Beaudette cultivaron el virus por primera vez en 1937. [18] El espécimen llegó a conocerse como la cepa Beaudette. A fines de la década de 1940, se descubrieron dos coronavirus animales más, JHM que causa enfermedad cerebral (encefalitis murina) y el virus de la hepatitis del ratón (MHV) que causa hepatitis en ratones. [19] En ese momento no se sabía que estos tres virus diferentes estaban relacionados. [20] [12]
Los coronavirus humanos fueron descubiertos en la década de 1960 [21] [22] utilizando dos métodos diferentes en el Reino Unido y los Estados Unidos. [23] EC Kendall, Malcolm Bynoe y David Tyrrell , que trabajaban en la Unidad de Resfriado Común del Consejo de Investigación Médica Británico, recolectaron un virus del resfriado común único designado B814 en 1961. [24] [25] [26] El virus no podía cultivarse utilizando técnicas estándar que habían cultivado con éxito rinovirus , adenovirus y otros virus del resfriado común conocidos. En 1965, Tyrrell y Bynoe cultivaron con éxito el nuevo virus pasándolo en serie a través de un cultivo de órganos de tráquea embrionaria humana . [27] El nuevo método de cultivo fue introducido en el laboratorio por Bertil Hoorn. [28] El virus aislado cuando se inoculó intranasalmente en voluntarios causó un resfriado y fue inactivado por éter , lo que indicó que tenía una envoltura lipídica . [24] [29] Dorothy Hamre y John Procknow de la Universidad de Chicago aislaron un nuevo resfriado en estudiantes de medicina en 1962. Aislaron y cultivaron el virus en un cultivo de tejido renal , denominándolo 229E. El nuevo virus causó un resfriado en voluntarios y, al igual que el B814, fue inactivado por éter. [30] [31]

En 1967, la viróloga escocesa June Almeida, del St Thomas' Hospital de Londres, en colaboración con Tyrrell, comparó las estructuras de IBV, B814 y 229E. [32] [33] Mediante microscopía electrónica, se demostró que los tres virus estaban relacionados morfológicamente por su forma general y sus distintivas espigas en forma de maza . [34] Un grupo de investigación del Instituto Nacional de Salud ese mismo año pudo aislar a otro miembro de este nuevo grupo de virus mediante cultivo de órganos y denominó una de las muestras OC43 (OC por cultivo de órganos). [35] Al igual que B814, 229E y IBV, el nuevo virus del resfriado OC43 tenía espigas distintivas en forma de maza cuando se observaba con el microscopio electrónico. [36] [37]
Pronto se demostró que los nuevos virus del resfriado similares al IBV también estaban morfológicamente relacionados con el virus de la hepatitis del ratón. [19] Este nuevo grupo de virus recibió el nombre de coronavirus por su apariencia morfológica distintiva. [7] El coronavirus humano 229E y el coronavirus humano OC43 continuaron estudiándose en las décadas posteriores. [38] [39] La cepa de coronavirus B814 se perdió. No se sabe qué coronavirus humano actual era. [40] Desde entonces se han identificado otros coronavirus humanos, incluido el SARS-CoV en 2003, el HCoV NL63 en 2003, el HCoV HKU1 en 2004, el MERS-CoV en 2013 y el SARS-CoV-2 en 2019. [41] También se ha identificado una gran cantidad de coronavirus animales desde la década de 1960. [42]
Microbiología
Estructura
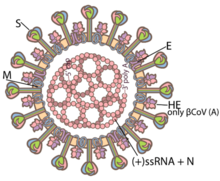
Los coronavirus son partículas grandes, aproximadamente esféricas, con proyecciones superficiales únicas. [43] Su tamaño es muy variable, con diámetros promedio de 80 a 120 nm . Se conocen tamaños extremos de 50 a 200 nm de diámetro. [44] La masa molecular total es en promedio de 40.000 kDa . Están encerrados en una envoltura incrustada con varias moléculas de proteína. [45] La envoltura de bicapa lipídica, las proteínas de membrana y la nucleocápside protegen al virus cuando está fuera de la célula huésped. [46]
La envoltura viral está formada por una bicapa lipídica en la que se anclan las proteínas estructurales de membrana (M), envoltura (E) y espícula (S) . [47] La relación molar de E:S:M en la bicapa lipídica es de aproximadamente 1:20:300. [48] Las proteínas E y M son las proteínas estructurales que se combinan con la bicapa lipídica para dar forma a la envoltura viral y mantener su tamaño. [49] Las proteínas S son necesarias para la interacción con las células huésped. Pero el coronavirus humano NL63 es peculiar en el sentido de que su proteína M tiene el sitio de unión para la célula huésped, y no su proteína S. [50] El diámetro de la envoltura es de 85 nm. La envoltura del virus en las micrografías electrónicas aparece como un par distinto de capas densas en electrones (capas que son relativamente opacas al haz de electrones utilizado para escanear la partícula del virus). [51] [49]
La proteína M es la principal proteína estructural de la envoltura que proporciona la forma general y es una proteína de membrana tipo III . Consta de 218 a 263 residuos de aminoácidos y forma una capa de 7,8 nm de espesor. [45] Tiene tres dominios, un ectodominio N-terminal corto, un dominio transmembrana de triple extensión y un endodominio C-terminal . El dominio C-terminal forma una red similar a una matriz que se suma al espesor adicional de la envoltura. Diferentes especies pueden tener glicanos unidos a N u O en su dominio amino-terminal de proteína. La proteína M es crucial durante las etapas de ensamblaje, gemación , formación de la envoltura y patogénesis del ciclo de vida del virus. [52]
Las proteínas E son proteínas estructurales menores y muy variables en diferentes especies. [44] Solo hay alrededor de 20 copias de la molécula de proteína E en una partícula de coronavirus. [48] Tienen un tamaño de 8,4 a 12 kDa y están compuestas de 76 a 109 aminoácidos. [44] Son proteínas integrales (es decir, incrustadas en la capa lipídica) y tienen dos dominios, a saber, un dominio transmembrana y un dominio C-terminal extramembrana. Son casi completamente α-helicoidales, con un solo dominio transmembrana α-helicoidal, y forman canales iónicos pentaméricos (de cinco moléculas) en la bicapa lipídica. Son responsables del ensamblaje del virión, el tráfico intracelular y la morfogénesis (gemación). [45]

Las espigas son la característica más distintiva de los coronavirus y son responsables de la superficie similar a una corona o halo. En promedio, una partícula de coronavirus tiene 74 espigas superficiales. [53] Cada espiga tiene aproximadamente 20 nm de largo y está compuesta por un trímero de la proteína S. La proteína S está compuesta a su vez por una subunidad S1 y S2 . La proteína S homotrimérica es una proteína de fusión de clase I que media la unión al receptor y la fusión de la membrana entre el virus y la célula huésped. La subunidad S1 forma la cabeza de la espiga y tiene el dominio de unión al receptor (RBD). La subunidad S2 forma el tallo que ancla la espiga en la envoltura viral y, al activarse la proteasa, permite la fusión. Las dos subunidades permanecen unidas de forma no covalente mientras están expuestas en la superficie viral hasta que se adhieren a la membrana de la célula huésped. [45] En un estado funcionalmente activo, tres S1 están unidas a dos subunidades S2. El complejo de subunidades se divide en subunidades individuales cuando el virus se une y se fusiona con la célula huésped bajo la acción de proteasas como la familia de catepsinas y la proteasa transmembrana serina 2 (TMPRSS2) de la célula huésped. [54]
Las proteínas S1 son los componentes más críticos en términos de infección. También son los componentes más variables, ya que son responsables de la especificidad de la célula huésped. Poseen dos dominios principales llamados dominio N-terminal (S1-NTD) y dominio C-terminal (S1-CTD), los cuales sirven como dominios de unión al receptor. Los NTD reconocen y se unen a los azúcares en la superficie de la célula huésped. Una excepción es el NTD del MHV que se une a un receptor proteico, la molécula de adhesión celular relacionada con el antígeno carcinoembrionario 1 (CEACAM1). Los S1-CTD son responsables de reconocer diferentes receptores proteicos como la enzima convertidora de angiotensina 2 (ACE2), la aminopeptidasa N (APN) y la dipeptidil peptidasa 4 (DPP4). [45]
Un subconjunto de coronavirus (específicamente los miembros del subgrupo A de betacoronavirus ) también tiene una proteína de superficie más corta en forma de espiga llamada hemaglutinina esterasa (HE). [42] Las proteínas HE se presentan como homodímeros compuestos por alrededor de 400 residuos de aminoácidos y tienen un tamaño de 40 a 50 kDa. Aparecen como pequeñas proyecciones superficiales de 5 a 7 nm de largo incrustadas entre las espigas. Ayudan en la unión y el desprendimiento de la célula huésped. [55]
Dentro de la envoltura, se encuentra la nucleocápside , que se forma a partir de múltiples copias de la proteína nucleocápside (N), que están unidas al genoma de ARN monocatenario de sentido positivo en una conformación continua de tipo cuentas en una cuerda. [49] [56] La proteína N es una fosfoproteína de 43 a 50 kDa de tamaño, y se divide en tres dominios conservados. La mayoría de la proteína está formada por los dominios 1 y 2, que suelen ser ricos en argininas y lisinas . El dominio 3 tiene un extremo carboxiterminal corto y tiene una carga neta negativa debido al exceso de residuos de aminoácidos ácidos sobre básicos. [44]
Genoma

Los coronavirus contienen un genoma de ARN monocatenario de sentido positivo . El tamaño del genoma de los coronavirus varía de 26,4 a 31,7 kilobases . [6] El tamaño del genoma es uno de los más grandes entre los virus de ARN. El genoma tiene una tapa metilada de 5′ y una cola poliadenilada de 3′ . [49]
La organización del genoma de un coronavirus es: 5′-líder-UTR -replicasa (ORF1ab)-espícula (S)-envoltura (E)-membrana (M)-nucleocápside (N) -3′UTR -poli (A) cola. Los marcos de lectura abiertos 1a y 1b, que ocupan los dos primeros tercios del genoma, codifican la poliproteína replicasa (pp1ab). La poliproteína replicasa se autoescinde para formar 16 proteínas no estructurales (nsp1–nsp16). [49]
Los marcos de lectura posteriores codifican las cuatro proteínas estructurales principales: proteína de la espícula , proteína de la envoltura , proteína de la membrana y proteína de la nucleocápside . [57] Entre estos marcos de lectura se encuentran los marcos de lectura de las proteínas accesorias. La cantidad de proteínas accesorias y su función es única según el coronavirus específico. [49]
Ciclo de replicación
Entrada de celda
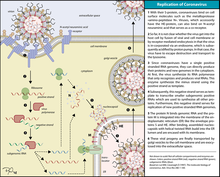
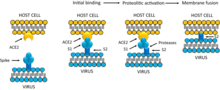
La infección comienza cuando la proteína de la espícula viral se une a su receptor complementario en la célula huésped. Después de la unión, una proteasa de la célula huésped corta y activa la proteína de la espícula unida al receptor. Dependiendo de la proteasa de la célula huésped disponible, la escisión y la activación permiten que el virus ingrese a la célula huésped por endocitosis o fusión directa de la envoltura viral con la membrana del huésped . [58]
Los coronavirus pueden ingresar a las células fusionándose con su envoltura lipídica en la membrana celular en la superficie celular o mediante internalización a través de endocitosis. [59]
Traducción del genoma
Al entrar en la célula huésped , la partícula del virus no tiene cubierta y su genoma entra en el citoplasma celular . El genoma del ARN del coronavirus tiene una tapa metilada en 5′ y una cola poliadenilada en 3′, lo que le permite actuar como un ARN mensajero y ser traducido directamente por los ribosomas de la célula huésped . Los ribosomas del huésped traducen los marcos de lectura abiertos superpuestos iniciales ORF1a y ORF1b del genoma del virus en dos grandes poliproteínas superpuestas, pp1a y pp1ab. [49]
La poliproteína pp1ab de mayor tamaño es el resultado de un desplazamiento del marco ribosómico -1 causado por una secuencia resbaladiza (UUUAAAC) y un pseudonudo de ARN corriente abajo al final del marco de lectura abierto ORF1a. [60] El desplazamiento del marco ribosómico permite la traducción continua de ORF1a seguido de ORF1b. [49]
Las poliproteínas tienen sus propias proteasas , PLpro (nsp3) y 3CLpro (nsp5), que escinden las poliproteínas en diferentes sitios específicos. La escisión de la poliproteína pp1ab produce 16 proteínas no estructurales (nsp1 a nsp16). Las proteínas del producto incluyen varias proteínas de replicación como la ARN polimerasa dependiente de ARN ( nsp12 ), la ARN helicasa (nsp13) y la exorribonucleasa (nsp14). [49]
Replicasa-transcriptasa
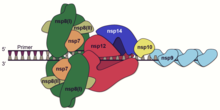
Varias proteínas no estructurales se unen para formar un complejo multiproteico de replicasa-transcriptasa (RTC). La principal proteína de replicasa-transcriptasa es la ARN polimerasa dependiente de ARN (RdRp). Está directamente involucrada en la replicación y transcripción de ARN a partir de una cadena de ARN. Las otras proteínas no estructurales del complejo ayudan en el proceso de replicación y transcripción. La proteína no estructural exorribonucleasa , por ejemplo, proporciona fidelidad adicional a la replicación al proporcionar una función de corrección de errores de la que carece la ARN polimerasa dependiente de ARN. [61]
Replicación – Una de las principales funciones del complejo es replicar el genoma viral. RdRp media directamente la síntesis de ARN genómico de sentido negativo a partir del ARN genómico de sentido positivo. A esto le sigue la replicación del ARN genómico de sentido positivo a partir del ARN genómico de sentido negativo. [49]


Transcripción – La otra función importante del complejo es transcribir el genoma viral. RdRp media directamente la síntesis de moléculas de ARN subgenómico de sentido negativo a partir del ARN genómico de sentido positivo. Este proceso es seguido por la transcripción de estas moléculas de ARN subgenómico de sentido negativo a sus ARNm de sentido positivo correspondientes . [49] Los ARNm subgenómicos forman un " conjunto anidado " que tienen una cabeza 5' común y un extremo 3' parcialmente duplicado. [62]
Recombinación – El complejo replicasa-transcriptasa también es capaz de recombinación genética cuando al menos dos genomas virales están presentes en la misma célula infectada. [62] La recombinación de ARN parece ser una fuerza impulsora importante en la determinación de la variabilidad genética dentro de una especie de coronavirus, la capacidad de una especie de coronavirus para saltar de un huésped a otro y, con poca frecuencia, en la determinación de la aparición de nuevos coronavirus. [63] El mecanismo exacto de recombinación en los coronavirus no está claro, pero probablemente implica un cambio de plantilla durante la replicación del genoma. [63]
Montaje y liberación
El ARN genómico de sentido positivo replicado se convierte en el genoma de los virus de la progenie . Los ARNm son transcripciones genéticas del último tercio del genoma del virus después del marco de lectura superpuesto inicial. Estos ARNm son traducidos por los ribosomas del huésped en las proteínas estructurales y muchas proteínas accesorias. [49] La traducción del ARN ocurre dentro del retículo endoplasmático . Las proteínas estructurales virales S, E y M se mueven a lo largo de la vía secretora hacia el compartimento intermedio de Golgi . Allí, las proteínas M dirigen la mayoría de las interacciones proteína-proteína necesarias para el ensamblaje de los virus después de su unión a la nucleocápside . Luego, los virus de la progenie se liberan de la célula huésped por exocitosis a través de vesículas secretoras. Una vez liberados, los virus pueden infectar otras células huésped. [64]
Transmisión
Los portadores infectados pueden diseminar virus al medio ambiente. La interacción de la proteína de la espícula del coronavirus con su receptor celular complementario es fundamental para determinar el tropismo tisular , la infectividad y el rango de especies del virus liberado. [65] [66] Los coronavirus se dirigen principalmente a las células epiteliales . [42] Se transmiten de un huésped a otro, dependiendo de la especie de coronavirus, ya sea por vía de aerosol , fómite o fecal-oral . [67]
Los coronavirus humanos infectan las células epiteliales del tracto respiratorio , mientras que los coronavirus animales generalmente infectan las células epiteliales del tracto digestivo . [42] El coronavirus del SARS , por ejemplo, infecta las células epiteliales humanas de los pulmones a través de una ruta de aerosol [68] al unirse al receptor de la enzima convertidora de angiotensina 2 (ACE2). [69] El coronavirus de gastroenteritis transmisible (TGEV) infecta las células epiteliales porcinas del tracto digestivo a través de una ruta fecal-oral [67] al unirse al receptor de alanina aminopeptidasa (APN). [49]
Clasificación

Los coronavirus forman la subfamilia Orthocoronavirinae, [2] [3] [4] que es una de las dos subfamilias de la familia Coronaviridae , orden Nidovirales y reino Riboviria . [42] [70] Se dividen en cuatro géneros: Alphacoronavirus , Betacoronavirus , Gammacoronavirus y Deltacoronavirus . Los alfacoronavirus y betacoronavirus infectan a los mamíferos, mientras que los gammacoronavirus y deltacoronavirus infectan principalmente a las aves. [71] [72]
- Género: Alphacoronavirus ; [67]
- Especies: Alphacoronavirus 1 ( TGEV , coronavirus felino , coronavirus canino ), coronavirus humano 229E , coronavirus humano NL63 , coronavirus 1 de murciélago Miniopterus , coronavirus HKU8 de murciélago Miniopterus , virus de la diarrea epidémica porcina , coronavirus HKU2 de murciélago Rhinolophus , coronavirus 512 de murciélago Scotophilus
- Género Betacoronavirus ; [68]
- Especies: Betacoronavirus 1 ( coronavirus bovino , coronavirus humano OC43 ), coronavirus 1 del erizo , coronavirus humano HKU1 , coronavirus relacionado con el síndrome respiratorio de Oriente Medio , coronavirus murino , coronavirus del murciélago Pipistrellus HKU5 , coronavirus del murciélago Rousettus HKU9 , coronavirus relacionado con el síndrome respiratorio agudo severo ( SARS-CoV-1 , SARS-CoV-2 ), coronavirus del murciélago Tylonycteris HKU4
- Género Gammacoronavirus ; [18]
- Género Deltacoronavirus
- Especie: Coronavirus bulbul HKU11 , Coronavirus porcino HKU15
Origen

Se estima que el ancestro común más reciente (MRCA) de todos los coronavirus existió tan recientemente como 8000 a. C. , aunque algunos modelos lo ubican tan atrás como 55 millones de años o más, lo que implica una coevolución a largo plazo con especies de murciélagos y aves. [73] El ancestro común más reciente de la línea alfacoronavirus se ha ubicado alrededor de 2400 a. C., de la línea betacoronavirus a 3300 a. C., de la línea gammacoronavirus a 2800 a. C. y la línea deltacoronavirus a alrededor de 3000 a. C. Los murciélagos y las aves, como vertebrados voladores de sangre caliente , son un reservorio natural ideal para el acervo genético de coronavirus (siendo los murciélagos el reservorio de alfacoronavirus y betacoronavirus, y las aves el reservorio de gammacoronavirus y deltacoronavirus). La gran cantidad y la gama global de especies de murciélagos y aves que albergan virus han permitido una amplia evolución y diseminación de coronavirus. [74]
Muchos coronavirus humanos tienen su origen en los murciélagos. [75] El coronavirus humano NL63 compartió un ancestro común con un coronavirus de murciélago (ARCoV.2) entre 1190 y 1449 d. C. [76] El coronavirus humano 229E compartió un ancestro común con un coronavirus de murciélago (GhanaGrp1 Bt CoV) entre 1686 y 1800 d. C. [77] Más recientemente, el coronavirus de alpaca y el coronavirus humano 229E divergieron en algún momento antes de 1960. [78] El MERS-CoV surgió en los humanos a partir de murciélagos a través del huésped intermediario de los camellos. [79] El MERS-CoV, aunque relacionado con varias especies de coronavirus de murciélago, parece haber divergido de estos hace varios siglos. [80] El coronavirus de murciélago más estrechamente relacionado y el SARS-CoV divergieron en 1986. [81] Los ancestros del SARS-CoV infectaron por primera vez a los murciélagos de nariz de hoja del género Hipposideridae ; Posteriormente, se propagaron a los murciélagos de herradura de la especie Rhinolophidae , luego a las civetas palmeras asiáticas y, finalmente, a los humanos. [82] [83]
A diferencia de otros betacoronavirus, se cree que el coronavirus bovino de la especie Betacoronavirus 1 y el subgénero Embecovirus se originó en roedores y no en murciélagos. [75] [84] En la década de 1790, el coronavirus equino divergió del coronavirus bovino después de un salto entre especies . [85] Más tarde, en la década de 1890, el coronavirus humano OC43 divergió del coronavirus bovino después de otro evento de derrame entre especies. [86] [85] Se especula que la pandemia de gripe de 1890 puede haber sido causada por este evento de derrame, y no por el virus de la influenza , debido al momento relacionado, los síntomas neurológicos y el agente causal desconocido de la pandemia. [87] Además de causar infecciones respiratorias, también se sospecha que el coronavirus humano OC43 desempeña un papel en las enfermedades neurológicas . [88] En la década de 1950, el coronavirus humano OC43 comenzó a divergir en sus genotipos actuales . [89] Filogenéticamente, el virus de la hepatitis del ratón ( coronavirus murino ), que infecta el hígado y el sistema nervioso central del ratón , [90] está relacionado con el coronavirus humano OC43 y el coronavirus bovino. El coronavirus humano HKU1, al igual que los virus mencionados anteriormente, también tiene su origen en roedores. [75]
Infección en humanos

Los coronavirus varían significativamente en cuanto a sus factores de riesgo. Algunos pueden matar a más del 30% de las personas infectadas, como el MERS-CoV , y otros son relativamente inofensivos, como el resfriado común. [49] Los coronavirus pueden causar resfriados con síntomas importantes, como fiebre y dolor de garganta por la inflamación de las adenoides . [91] Los coronavirus pueden causar neumonía (ya sea neumonía viral directa o neumonía bacteriana secundaria ) y bronquitis (ya sea bronquitis viral directa o bronquitis bacteriana secundaria). [92] El coronavirus humano descubierto en 2003, el SARS-CoV , que causa el síndrome respiratorio agudo severo (SARS), tiene una patogenia única porque causa infecciones del tracto respiratorio superior e inferior . [92]
Se conocen seis especies de coronavirus humanos, cada una de las cuales se subdivide en dos cepas diferentes, lo que da un total de siete cepas de coronavirus humanos.

Cuatro coronavirus humanos producen síntomas que generalmente son leves, aunque se sostiene que podrían haber sido más agresivos en el pasado: [93]
- Coronavirus humano OC43 (HCoV-OC43), β-CoV
- Coronavirus humano HKU1 (HCoV-HKU1), β-CoV
- Coronavirus humano 229E (HCoV-229E), α-CoV
- Coronavirus humano NL63 (HCoV-NL63), α-CoV–
Tres coronavirus humanos producen síntomas potencialmente graves:
- Coronavirus del síndrome respiratorio agudo severo (SARS-CoV), β-CoV (identificado en 2003)
- Coronavirus relacionado con el síndrome respiratorio de Oriente Medio (MERS-CoV), β-CoV (identificado en 2012)
- Coronavirus 2 del síndrome respiratorio agudo severo (SARS-CoV-2), β-CoV (identificado en 2019)
Estos causan las enfermedades comúnmente llamadas SARS , MERS y COVID-19 respectivamente.
Resfriado común
Aunque el resfriado común suele ser causado por rinovirus , [94] en aproximadamente el 15% de los casos la causa es un coronavirus. [95] Los coronavirus humanos HCoV-OC43, HCoV-HKU1, HCoV-229E y HCoV-NL63 circulan continuamente en la población humana en adultos y niños en todo el mundo y producen los síntomas generalmente leves del resfriado común. [88] Los cuatro coronavirus leves tienen una incidencia estacional que ocurre en los meses de invierno en climas templados . [96] [97] No hay preponderancia en ninguna estación en climas tropicales . [98]
Síndrome respiratorio agudo severo (SARS)
| MERS-CoV | SARS-CoV | SARS-CoV-2 | |
|---|---|---|---|
| Enfermedad | MERS | SARS | COVID-19 |
| Brotes | Brote de MERS de 2012 | 2002–2004 | Pandemia de COVID-19 |
| Epidemiología | |||
| Fecha del primer caso identificado | Junio de 2012 | Noviembre de 2002 | Diciembre de 2019 [99] |
| Ubicación del primer caso identificado | Yeda , Arabia Saudita | Shunde , China | Wuhan , China |
| Edad promedio | 56 | 44 [100] [a] | 56 [101] |
| Relación sexual (M:F) | 3.3:1 | 0,8:1 [102] | 1.6:1 [101] |
| Casos confirmados | 2494 | 8096 [103] | 676.609.955 [104] [b] |
| Fallecidos | 858 | 774 [103] | 6.881.955 [104] [b] |
| Tasa de letalidad | 37% | 9,2% | 1,02% [104] |
| Síntomas | |||
| Fiebre | 98% | 99–100% | 87,9% [105] |
| Tos seca | 47% | 29–75% | 67,7% [105] |
| Disnea | 72% | 40–42% | 18,6% [105] |
| Diarrea | 26% | 20–25% | 3,7% [105] |
| Dolor de garganta | 21% | 13–25% | 13,9% [105] |
| Uso ventilatorio | 24,5% [106] | 14–20% | 4,1% [107] |
Notas
| |||
En 2003, tras el brote del síndrome respiratorio agudo severo (SARS) que había comenzado el año anterior en Asia y otros casos secundarios en otras partes del mundo, la Organización Mundial de la Salud (OMS) emitió un comunicado de prensa en el que afirmaba que un nuevo coronavirus identificado por varios laboratorios era el agente causante del SARS. El virus recibió el nombre oficial de coronavirus del SARS (SARS-CoV). Más de 8.000 personas de 29 países y territorios se infectaron y al menos 774 murieron. [108] [69]
Síndrome respiratorio del Oriente Medio (MERS)
En septiembre de 2012, se identificó un nuevo tipo de coronavirus, inicialmente llamado Novel Coronavirus 2012, y ahora oficialmente llamado coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV). [109] [110] La Organización Mundial de la Salud emitió una alerta mundial poco después. [111] La actualización de la OMS del 28 de septiembre de 2012 decía que el virus no parecía transmitirse fácilmente de persona a persona. [112] Sin embargo, el 12 de mayo de 2013, el Ministerio de Asuntos Sociales y Salud francés confirmó un caso de transmisión de persona a persona en Francia. [113] Además, el Ministerio de Salud de Túnez notificó casos de transmisión de persona a persona . Dos casos confirmados involucraban a personas que parecían haber contraído la enfermedad de su difunto padre, que se enfermó después de una visita a Qatar y Arabia Saudita. A pesar de esto, parece que el virus tuvo problemas para propagarse de persona a persona, ya que la mayoría de las personas infectadas no transmiten el virus. [114] Al 30 de octubre de 2013, había 124 casos y 52 muertes en Arabia Saudita. [115]
Después de que el Centro Médico Erasmus holandés secuenciara el virus, se le dio un nuevo nombre, Coronavirus Humano–Centro Médico Erasmus (HCoV-EMC). El nombre final del virus es coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV). Los únicos casos en Estados Unidos (ambos sobrevivieron) se registraron en mayo de 2014. [116]
En mayo de 2015, se produjo un brote de MERS-CoV en la República de Corea , cuando un hombre que había viajado a Oriente Medio visitó cuatro hospitales en el área de Seúl para tratar su enfermedad. Esto provocó uno de los mayores brotes de MERS-CoV fuera de Oriente Medio. [117] Hasta diciembre de 2019, se habían confirmado 2.468 casos de infección por MERS-CoV mediante pruebas de laboratorio, 851 de los cuales fueron mortales, una tasa de mortalidad de aproximadamente el 34,5 %. [118]
Enfermedad por coronavirus 2019 (COVID-19)
En diciembre de 2019, se informó de un brote de neumonía en Wuhan (China). [119] El 31 de diciembre de 2019, el brote se atribuyó a una nueva cepa de coronavirus, [120] a la que la Organización Mundial de la Salud le dio el nombre provisional de 2019-nCoV, [121] [122] [123] posteriormente rebautizada como SARS-CoV-2 por el Comité Internacional de Taxonomía de Virus .
Hasta el 10 de marzo de 2023, se habían confirmado al menos 6.881.955 [104] muertes y más de 676.609.955 [104] casos en la pandemia de COVID-19 . La cepa de Wuhan ha sido identificada como una nueva cepa de Betacoronavirus del grupo 2B con aproximadamente un 70% de similitud genética con el SARS-CoV. [124] El virus tiene una similitud del 96% con un coronavirus de murciélago, por lo que se sospecha ampliamente que también se origina en murciélagos. [125] [126]
Coronavirus HuPn-2018
Durante un estudio de vigilancia de muestras archivadas de pacientes con neumonía viral de Malasia, los virólogos identificaron una cepa de coronavirus canino que infectó a humanos en 2018.
Infección en animales
Los coronavirus han sido reconocidos como causantes de condiciones patológicas en medicina veterinaria desde la década de 1930. [19] Infectan a una variedad de animales, incluidos cerdos, ganado, caballos, camellos, gatos, perros, roedores, aves y murciélagos. [127] La mayoría de los coronavirus relacionados con animales infectan el tracto intestinal y se transmiten por vía fecal-oral. [128] Se han centrado importantes esfuerzos de investigación en dilucidar la patogénesis viral de estos coronavirus animales, especialmente por parte de virólogos interesados en enfermedades veterinarias y zoonóticas . [129]
Animales de granja
Los coronavirus infectan a las aves domésticas. [130] El virus de la bronquitis infecciosa (IBV), un tipo de coronavirus, causa bronquitis infecciosa aviar . [131] El virus es motivo de preocupación para la industria avícola debido a la alta mortalidad por infección, su rápida propagación y su efecto en la producción. [127] El virus afecta tanto a la producción de carne como a la de huevos y causa pérdidas económicas sustanciales. [132] En los pollos, el virus de la bronquitis infecciosa ataca no solo al tracto respiratorio sino también al tracto urogenital . El virus puede propagarse a diferentes órganos en todo el pollo. [131] El virus se transmite por aerosol y alimentos contaminados por heces. Existen diferentes vacunas contra el IBV que han ayudado a limitar la propagación del virus y sus variantes. [127] El virus de la bronquitis infecciosa es una de varias cepas de la especie Coronavirus aviar . [133] Otra cepa de coronavirus aviar es el coronavirus del pavo (TCV), que causa enteritis en los pavos . [127]
Los coronavirus también afectan a otras ramas de la ganadería, como la cría de cerdos y la ganadería. [127] El coronavirus del síndrome de diarrea aguda porcina (SADS-CoV), relacionado con el coronavirus de murciélago HKU2 , causa diarrea en cerdos. [134] El virus de la diarrea epidémica porcina (PEDV) es un coronavirus que ha surgido recientemente y que también causa diarrea en cerdos. [135] El virus de la gastroenteritis transmisible (TGEV), que es miembro de la especie Alphacoronavirus 1 , [136] es otro coronavirus que causa diarrea en cerdos jóvenes. [137] [138] En la industria ganadera, el coronavirus bovino (BCV), que es miembro de la especie Betacoronavirus 1 y está relacionado con HCoV-OC43, [139] es responsable de enteritis profusa grave en terneros jóvenes. [127]
Mascotas domésticas
Los coronavirus infectan a mascotas domésticas como gatos, perros y hurones. [130] Hay dos formas de coronavirus felino que son miembros de la especie Alphacoronavirus 1. [ 136] El coronavirus entérico felino es un patógeno de menor importancia clínica, pero la mutación espontánea de este virus puede provocar peritonitis infecciosa felina (PIF), una enfermedad con alta mortalidad. [127] Hay dos coronavirus diferentes que infectan a los perros. El coronavirus canino (CCoV), que es miembro de la especie Alphacoronavirus 1 , [136] causa una enfermedad gastrointestinal leve. [127] El coronavirus respiratorio canino (CRCoV), que es miembro de la especie Betacoronavirus 1 y está relacionado con HCoV-OC43, [139] causa enfermedad respiratoria. [127] De manera similar, hay dos tipos de coronavirus que infectan a los hurones. [140] El coronavirus entérico del hurón causa un síndrome gastrointestinal conocido como enteritis catarral epizoótica (ECE), y una versión sistémica más letal del virus (como la PIF en gatos) conocida como coronavirus sistémico del hurón (FSC). [141] [142]
Animales de laboratorio
Los coronavirus infectan a los animales de laboratorio. [127] El virus de la hepatitis del ratón (MHV), que es un miembro de la especie Coronavirus murino , [143] causa una enfermedad murina epidémica con alta mortalidad, especialmente entre colonias de ratones de laboratorio. [144] Antes del descubrimiento del SARS-CoV, el MHV era el coronavirus mejor estudiado tanto in vivo como in vitro, así como a nivel molecular. Algunas cepas de MHV causan una encefalitis desmielinizante progresiva en ratones que se ha utilizado como modelo murino para la esclerosis múltiple . [129] El virus de la sialodacrioadenitis (SDAV), que es una cepa de la especie Coronavirus murino , [143] es un coronavirus altamente infeccioso de ratas de laboratorio, que puede transmitirse entre individuos por contacto directo e indirectamente por aerosol. El coronavirus entérico del conejo causa enfermedad gastrointestinal aguda y diarrea en conejos europeos jóvenes . [127] Las tasas de mortalidad son altas. [145]
Prevención y tratamiento
Se han desarrollado varias vacunas que utilizan diferentes métodos contra el coronavirus humano SARS-CoV-2. [ 146 ] [147] También se han identificado dianas antivirales contra los coronavirus humanos, como las proteasas virales, las polimerasas y las proteínas de entrada. Se están desarrollando medicamentos que se dirigen a estas proteínas y a los diferentes pasos de la replicación viral. [148] [147]
Existen vacunas contra los coronavirus animales IBV, TGEV y Canine CoV, aunque su eficacia es limitada. En el caso de brotes de coronavirus animales altamente contagiosos, como PEDV, se pueden utilizar medidas como la destrucción de rebaños enteros de cerdos para prevenir la transmisión a otros rebaños. [49]
Véase también
Referencias
- ^ "Taxonomía de virus: versión 2018b". Comité Internacional de Taxonomía de Virus (ICTV) . Marzo de 2019. Archivado desde el original el 4 de marzo de 2018. Consultado el 24 de enero de 2020 .
- ^ ab "2017.012-015S" (xlsx) . Comité Internacional de Taxonomía de Virus (ICTV) . Octubre de 2018. Archivado desde el original el 14 de mayo de 2019. Consultado el 24 de enero de 2020 .
- ^ abc Fan Y, Zhao K, Shi ZL, Zhou P (marzo de 2019). "Coronavirus de murciélago en China". Viruses . 11 (3): 210. doi : 10.3390/v11030210 . PMC 6466186 . PMID 30832341.
- ^ Cherry J, Demmler-Harrison GJ, Kaplan SL, Steinbach WJ, Hotez PJ (2017). Feigin y Cherry's Textbook of Pediatric Infectious Diseases. Elsevier Health Sciences. pág. PT6615. ISBN 978-0-323-39281-5.
- ^ ab Woo PC, Huang Y, Lau SK, Yuen KY (agosto de 2010). "Análisis genómico y bioinformático del coronavirus". Viruses . 2 (8): 1804–20. doi : 10.3390/v2081803 . ISSN 1999-4915. PMC 3185738 . PMID 21994708.
Los coronavirus poseen los genomas más grandes [26,4 kb (ThCoV HKU12) a 31,7 kb (SW1)] entre todos los virus de ARN conocidos (Figura 1) [2,13,16].
- ^ abcd Almeida JD, Berry DM, Cunningham CH, Hamre D, Hofstad MS, Mallucci L, et al. (noviembre de 1968). "Virología: coronavirus". Nature . 220 (5168): 650. Bibcode :1968Natur.220..650.. doi : 10.1038/220650b0 . PMC 7086490 .
[H]ay también una "franja" característica de proyecciones de 200 A de largo, que son redondeadas o tienen forma de pétalo
... Esta apariencia, que recuerda a la corona solar, es compartida por el virus de la hepatitis del ratón y varios virus recientemente recuperados del hombre, a saber, la cepa B814, 229E y varios otros.
- ^ "Definición de coronavirus según Merriam-Webster". Merriam-Webster. Archivado desde el original el 23 de marzo de 2020. Consultado el 24 de marzo de 2020 .
- ^ "Definición de Corona según Merriam-Webster". Merriam-Webster. Archivado desde el original el 2020-03-24 . Consultado el 2020-03-24 .
- ^ ab Tyrrell DA, Fielder M (2002). Guerras frías: la lucha contra el resfriado común. Oxford University Press. pág. 96. ISBN 978-0-19-263285-2
Observamos más de cerca la apariencia de los nuevos virus y notamos que tenían una especie de halo que los rodeaba. Al recurrir a un diccionario, encontramos el equivalente en latín, corona, y así nació el nombre de coronavirus
. - ^ Sturman LS, Holmes KV (1 de enero de 1983). Lauffer MA, Maramorosch K (eds.). "La biología molecular de los coronavirus". Avances en la investigación de virus . 28 : 35-112. doi : 10.1016/s0065-3527(08)60721-6 . ISBN 978-0-12-039828-7. PMC 7131312 . PMID 6362367.
[E]stos virus mostraban una franja característica de peplómeros o espigas grandes y distintivos en forma de pétalos que se parecían a una corona, como la corona spinarum en el arte religioso; de ahí el nombre de coronavirus.
- ^ ab Lalchhandama K (2020). "Las crónicas de los coronavirus: la bronquitis, la hepatitis y el resfriado común". Science Vision . 20 (1): 43–53. doi : 10.33493/scivis.20.01.04 .
- ^ Carstens EB (2010). "Votación de ratificación de las propuestas taxonómicas ante el Comité Internacional de Taxonomía de Virus (2009)". Archivos de Virología . 155 (1): 133–46. doi :10.1007/s00705-009-0547-x. PMC 7086975 . PMID 19960211.
- ^ "Comité Internacional de Taxonomía de Virus (ICTV)". talk.ictvonline.org . Consultado el 14 de septiembre de 2020 .
- ^ Estola T (1970). "Coronavirus, un nuevo grupo de virus ARN animales". Enfermedades aviares . 14 (2): 330–336. doi :10.2307/1588476. ISSN 0005-2086. JSTOR 1588476. PMID 4316767.
- ^ Fabricant J (1998). "La historia temprana de la bronquitis infecciosa". Enfermedades aviares . 42 (4): 648–650. doi :10.2307/1592697. ISSN 0005-2086. JSTOR 1592697. PMID 9876830.
- ^ Bushnell LD, Brandly CA (1933). "Laringotraqueítis en pollitos". Ciencia avícola . 12 (1): 55–60. doi : 10.3382/ps.0120055 .
- ^ ab Decaro N (2011). "Gammacoronavirus". En Tidona C, Darai G (eds.). Gammacoronavirus‡: Coronaviridae . El índice Springer de virus . Saltador. págs. 403–413. doi :10.1007/978-0-387-95919-1_58. ISBN 978-0-387-95919-1. Número de pieza 7176155 .
- ^ abc McIntosh K (1974). "Coronavirus: una revisión comparativa". En Arber W, Haas R, Henle W, Hofschneider PH, Jerne NK, Koldovský P, Koprowski H, Maaløe O, Rott R (eds.). Temas actuales en microbiología e inmunología / Ergebnisse der Mikrobiologie und Immunitätsforschung . Berlín, Heidelberg: Springer. pag. 87. doi :10.1007/978-3-642-65775-7_3. ISBN 978-3-642-65775-7.
- ^ "Il était une fois les coronavirus". Réalités Biomédicales (en francés). 2020-03-27 . Consultado el 18 de abril de 2020 .
- ^ Kahn JS, McIntosh K (noviembre de 2005). "Historia y avances recientes en el descubrimiento del coronavirus". The Pediatric Infectious Disease Journal . 24 (11 Suppl): S223–7, discusión S226. doi : 10.1097/01.inf.0000188166.17324.60 . PMID 16378050.
- ^ Mahase E (abril de 2020). "El BMJ en 1965". BMJ . 369 : m1547. doi : 10.1136/bmj.m1547 . PMID 32299810.
- ^ Monto AS (1984). "Coronavirus". En Evans AS (ed.). Infecciones virales en humanos . Springer US. págs. 151–165. doi :10.1007/978-1-4684-4727-9_7. ISBN 978-1-4684-4727-9.
{{cite book}}:|work=ignorado ( ayuda ) - ^ ab Kendall EJ, Bynoe ML, Tyrrell DA (julio de 1962). "Aislamientos de virus de resfriados comunes ocurridos en una escuela residencial". British Medical Journal . 2 (5297): 82–6. doi :10.1136/bmj.2.5297.82. PMC 1925312 . PMID 14455113.
- ^ Richmond C (18 de junio de 2005). "David Tyrrell". BMJ: British Medical Journal . 330 (7505): 1451. doi :10.1136/bmj.330.7505.1451. PMC 558394 .
- ^ "Obituarios: Malcom Byone". British Medical Journal . 2 (5660): 827–829. 28 de junio de 1969. doi :10.1136/bmj.2.5660.827. S2CID 220187042.
- ^ Tyrrell DA, Bynoe ML (junio de 1965). "Cultivo de un nuevo tipo de virus del resfriado común en cultivos de órganos". British Medical Journal . 1 (5448): 1467–70. doi :10.1136/bmj.1.5448.1467. PMC 2166670 . PMID 14288084.
- ^ Tyrrell DA, Fielder M (2002). Guerras frías: la lucha contra el resfriado común. Oxford University Press. pp. 93–95. ISBN 978-0-19-263285-2.
- ^ Hagan WA, Bruner DW, Gillespie JH, Timoney JF, Scott FW, Barlough JE (1988). Hagan y Bruner, Microbiología y enfermedades infecciosas de los animales domésticos: con referencia a la etiología, epizootiología, patogénesis, inmunidad, diagnóstico y susceptibilidad a los antimicrobianos. Cornell University Press. pág. 440. ISBN 978-0-8014-1896-9.
- ^ Hamre D, Procknow JJ (enero de 1966). "Un nuevo virus aislado del tracto respiratorio humano". Actas de la Sociedad de Biología y Medicina Experimental . 121 (1): 190–3. doi :10.3181/00379727-121-30734. PMID 4285768. S2CID 1314901.
- ^ Knapp A. "La historia secreta del primer coronavirus". Forbes . Consultado el 6 de mayo de 2020 .
- ^ "La mujer que descubrió el primer coronavirus". BBC News . 14 de abril de 2020.
- ^ Almeida J (26 de junio de 2008). "June Almeida (née Hart)". BMJ . 336 (7659): 1511.1–1511. doi :10.1136/bmj.a434. ISSN 0959-8138. PMC 2440895 .
- ^ Almeida JD, Tyrrell DA (abril de 1967). "La morfología de tres virus respiratorios humanos no caracterizados previamente que crecen en cultivos de órganos". The Journal of General Virology . 1 (2): 175–8. doi : 10.1099/0022-1317-1-2-175 . PMID 4293939.
- ^ McIntosh K, Becker WB, Chanock RM (diciembre de 1967). "Crecimiento en el cerebro de ratones lactantes de virus "similares al IBV" de pacientes con enfermedad del tracto respiratorio superior". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 58 (6): 2268–73. Bibcode :1967PNAS...58.2268M. doi : 10.1073/pnas.58.6.2268 . PMC 223830 . PMID 4298953.
- ^ McIntosh K, Dees JH, Becker WB, Kapikian AZ, Chanock RM (abril de 1967). "Recuperación en cultivos de órganos traqueales de nuevos virus de pacientes con enfermedad respiratoria". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 57 (4): 933–40. Bibcode :1967PNAS...57..933M. doi : 10.1073/pnas.57.4.933 . PMC 224637 . PMID 5231356.
- ^ Times HM Jr (5 de mayo de 1967). "Seis virus recién descubiertos pueden explicar el resfriado; se cree que las cepas son similares al germen que causa una infección bronquial en pollos y que pertenece a un nuevo grupo". The New York Times . ISSN 0362-4331 . Consultado el 25 de abril de 2020 .
- ^ Myint SH (1995). "Infecciones por coronavirus en humanos". En Siddell SG (ed.). Coronaviridae . Los virus. Springer US. págs. 389–401. doi :10.1007/978-1-4899-1531-3_18. ISBN 978-1-4899-1531-3.S2CID80726096 .
- ^ Geller C, Varbanov M, Duval RE (noviembre de 2012). "Coronavirus humanos: perspectivas sobre la resistencia ambiental y su influencia en el desarrollo de nuevas estrategias antisépticas". Viruses . 4 (11): 3044–68. doi : 10.3390/v4113044 . PMC 3509683 . PMID 23202515.
- ^ Monto AS, Cowling BJ, Peiris JS (2014), Kaslow RA, Stanberry LR, Le Duc JW (eds.), "Coronavirus", Infecciones virales en humanos , Boston, MA: Springer US: 199–223, doi :10.1007/978-1-4899-7448-8_10, ISBN 978-1-4899-7447-1, PMC 7122465 ,
Las otras cepas OC y B814 que no pudieron adaptarse al cerebro de ratón también resistieron la adaptación al cultivo celular; estos virus distintos se han perdido desde entonces y es posible que hayan sido redescubiertos recientemente.
- ^ Zhu N, Zhang D, Wang W, Li X, Yang B, Song J, et al. (febrero de 2020). "Un nuevo coronavirus de pacientes con neumonía en China, 2019". The New England Journal of Medicine . 382 (8): 727–733. doi :10.1056/NEJMoa2001017. PMC 7092803 . PMID 31978945.
- ^ abcde de Groot RJ, Baker SC , Baric R, Enjuanes L, Gorbalenya AE, Holmes KV, et al. (2011). "Familia Coronaviridae". En King AM, Lefkowitz E, Adams MJ, Carstens EB, Comité Internacional de Taxonomía de Virus, Unión Internacional de Sociedades de Microbiología. División de Virología (eds.). Noveno Informe del Comité Internacional de Taxonomía de Virus . Oxford: Elsevier. págs. 806–28. doi :10.1016/B978-0-12-384684-6.00068-9. ISBN 978-0-12-384684-6. Número de identificación del sujeto 212719285.
- ^ Goldsmith CS, Tatti KM, Ksiazek TG, Rollin PE, Comer JA, Lee WW, et al. (febrero de 2004). "Caracterización ultraestructural del coronavirus del SARS". Enfermedades infecciosas emergentes . 10 (2): 320–6. doi :10.3201/eid1002.030913. PMC 3322934 . PMID 15030705.
Los viriones adquirieron una envoltura al gemar en las cisternas y formaron partículas principalmente esféricas, a veces pleomórficas, que promediaron 78 nm de diámetro (Figura 1A).
- ^ abcd Masters PS (2006). "La biología molecular de los coronavirus". Avances en la investigación de virus . 66 : 193–292. doi :10.1016/S0065-3527(06)66005-3. ISBN 978-0-12-039869-0. PMC 7112330 . PMID 16877062.
- ^ abcde Lalchhandama K (2020). "Las crónicas de los coronavirus: el microscopio electrónico, el donut y la púa". Visión científica . 20 (2): 78–92. doi : 10.33493/scivis.20.02.03 .
- ^ Neuman BW, Kiss G, Kunding AH, Bhella D, Baksh MF, Connelly S, et al. (abril de 2011). "Un análisis estructural de la proteína M en el ensamblaje y la morfología del coronavirus". Journal of Structural Biology . 174 (1): 11–22. doi :10.1016/j.jsb.2010.11.021. PMC 4486061 . PMID 21130884.
Véase la Figura 10.
- ^ Lai MM, Cavanagh D (1997). "La biología molecular de los coronavirus". Avances en la investigación de virus . 48 : 1–100. doi : 10.1016/S0065-3527(08)60286-9 . ISBN 978-0-12-039848-5. PMC 7130985 . PMID 9233431.
- ^ ab Godet M, L'Haridon R, Vautherot JF, Laude H (1992). "El ORF4 del coronavirus TGEV codifica una proteína de membrana que se incorpora a los viriones". Virology . 188 (2): 666–75. doi :10.1016/0042-6822(92)90521-p. PMC 7131960 . PMID 1316677.
- ^ abcdefghijklmno Fehr AR, Perlman S (2015). "Coronavirus: una descripción general de su replicación y patogénesis". En Maier HJ, Bickerton E, Britton P (eds.). Coronavirus . Métodos en biología molecular. Vol. 1282. Springer. págs. 1–23. doi :10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2438-7. PMC 4369385 . PMID 25720466.
Ver sección: Estructura del virión.
- ^ Naskalska A, Dabrowska A, Szczepanski A, Milewska A, Jasik KP, Pyrc K (octubre de 2019). "La proteína de membrana del coronavirus humano NL63 es responsable de la interacción con el receptor de adhesión". Journal of Virology . 93 (19). doi :10.1128/JVI.00355-19. PMC 6744225 . PMID 31315999.
- ^ Neuman BW, Adair BD, Yoshioka C, Quispe JD, Orca G, Kuhn P, et al. (agosto de 2006). "Arquitectura supramolecular del coronavirus del síndrome respiratorio agudo severo revelada por criomicroscopía electrónica". Journal of Virology . 80 (16): 7918–28. doi :10.1128/JVI.00645-06. PMC 1563832 . PMID 16873249.
Los diámetros de las partículas oscilaron entre 50 y 150 nm, excluyendo las espigas, con diámetros de partícula medios de 82 a 94 nm; Véase también la Figura
1 para la doble capa.
- ^ Schoeman D, Fielding BC (mayo de 2019). "Proteína de envoltura del coronavirus: conocimiento actual". Revista de Virología . 16 (1): 69. doi : 10.1186/s12985-019-1182-0 . PMC 6537279 . PMID 31133031.
- ^ Neuman BW, Kiss G, Kunding AH, Bhella D, Baksh MF, Connelly S, et al. (abril de 2011). "Un análisis estructural de la proteína M en el ensamblaje y la morfología del coronavirus". Journal of Structural Biology . 174 (1): 11–22. doi :10.1016/j.jsb.2010.11.021. PMC 4486061 . PMID 21130884.
- ^ J Alsaadi EA, Jones IM (abril de 2019). "Proteínas de unión a membrana de los coronavirus". Future Virology . 14 (4): 275–286. doi :10.2217/fvl-2018-0144. PMC 7079996 . PMID 32201500.
- ^ Zeng Q, Langereis MA, van Vliet AL, Huizinga EG, de Groot RJ (julio de 2008). "La estructura de la hemaglutinina-esterasa del coronavirus ofrece información sobre la evolución de los virus de la corona y la influenza". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (26): 9065–9. Código Bib : 2008PNAS..105.9065Z. doi : 10.1073/pnas.0800502105 . PMC 2449365 . PMID 18550812.
- ^ Chang CK, Hou MH, Chang CF, Hsiao CD, Huang TH (marzo de 2014). "La proteína de la nucleocápside del coronavirus del SARS: formas y funciones". Antiviral Research . 103 : 39–50. doi : 10.1016/j.antiviral.2013.12.009 . PMC 7113676 . PMID 24418573.
Véase la Figura 4c.
- ^ Snijder EJ, Bredenbeek PJ, Dobbe JC, Thiel V, Ziebuhr J, Poon LL, et al. (agosto de 2003). "Características únicas y conservadas del genoma y el proteoma del coronavirus del SARS, una escisión temprana del linaje del grupo 2 de coronavirus". Journal of Molecular Biology . 331 (5): 991–1004. doi : 10.1016/S0022-2836(03)00865-9 . PMC 7159028 . PMID 12927536.
Véase la Figura 1.
- ^ Simmons G, Zmora P, Gierer S, Heurich A, Pöhlmann S (diciembre de 2013). "Activación proteolítica de la proteína de pico del coronavirus SARS: enzimas de corte en la vanguardia de la investigación antiviral". Investigación antiviral . 100 (3): 605–14. doi :10.1016/j.antiviral.2013.09.028. PMC 3889862 . PMID 24121034.
Véase la Figura 2.
- ^ Szlachcic WJ, Dabrowska A, Milewska A, Ziojla N, Blaszczyk K, Barreto-Duran E, et al. (julio de 2022). "El SARS-CoV-2 infecta un modelo in vitro del páncreas humano en desarrollo mediante endocitosis". iCiencia . 25 (7): 104594. Código bibliográfico : 2022iSci...25j4594S. doi :10.1016/j.isci.2022.104594. PMC 9212970 . PMID 35756892.
- ^ Masters PS (1 de enero de 2006). "La biología molecular de los coronavirus". Avances en la investigación de virus . 66 . Academic Press: 193–292. doi : 10.1016/S0065-3527(06)66005-3 . ISBN 978-0-12-039869-0. PMC 7112330 . PMID 16877062.
Véase la Figura 8.
- ^ Sexton NR, Smith EC, Blanc H, Vignuzzi M, Peersen OB, Denison MR (agosto de 2016). "Identificación basada en homología de una mutación en la ARN polimerasa dependiente del ARN del coronavirus que confiere resistencia a múltiples mutágenos". Journal of Virology . 90 (16): 7415–28. doi :10.1128/JVI.00080-16. PMC 4984655 . PMID 27279608.
Finalmente, estos resultados, combinados con los de trabajos anteriores (33, 44), sugieren que los CoV codifican al menos tres proteínas implicadas en la fidelidad (nsp12-RdRp, nsp14-ExoN y nsp10), lo que respalda el ensamblaje de un complejo de fidelidad de replicasa multiproteica, como se describió anteriormente (38).
- ^ ab Payne S (1 de enero de 2017). "Capítulo 17 - Familia Coronaviridae". Viruses . Academic Press. págs. 149–158. doi :10.1016/B978-0-12-803109-4.00017-9. ISBN 978-0-12-803109-4.S2CID 91572610 .
- ^ ab Su S, Wong G, Shi W, Liu J, Lai AC, Zhou J, et al. (junio de 2016). "Epidemiología, recombinación genética y patogénesis de los coronavirus". Tendencias en microbiología . 24 (6): 490–502. doi :10.1016/j.tim.2016.03.003. PMC 7125511 . PMID 27012512.
- ^ Fehr AR, Perlman S (2015). "Coronavirus: una descripción general de su replicación y patogénesis". En Maier HJ, Bickerton E, Britton P (eds.). Coronavirus . Métodos en biología molecular. Vol. 1282. Springer. págs. 1–23. doi :10.1007/978-1-4939-2438-7_1. ISBN . 978-1-4939-2438-7. PMC 4369385 . PMID 25720466.
Consulte la sección: Ciclo de vida del coronavirus: ensamblaje y liberación
- ^ Masters PS (1 de enero de 2006). "La biología molecular de los coronavirus". Avances en la investigación de virus . 66 . Academic Press: 193–292. doi : 10.1016/S0065-3527(06)66005-3 . ISBN 978-0-12-039869-0. PMC 7112330 . PMID 16877062.
Sin embargo, la interacción entre la proteína S y el receptor sigue siendo el determinante principal, si no el único, del rango de especies hospedadoras del coronavirus y del tropismo tisular.
- ^ Cui J, Li F, Shi ZL (marzo de 2019). "Origen y evolución de los coronavirus patógenos". Nature Reviews. Microbiology . 17 (3): 181–92. doi : 10.1038/s41579-018-0118-9 . PMC 7097006 . PMID 30531947.
Diferentes cepas de SARS-CoV aisladas de varios hospedadores varían en sus afinidades de unión para la ECA2 humana y, en consecuencia, en su infectividad de las células humanas 76, 78 (Fig. 6b)
- ^ abc Decaro N (2011). "Alfacoronavirus‡: Coronaviridae". En Tidona C, Darai G (eds.). El índice Springer de virus . Saltador. págs. 371–383. doi :10.1007/978-0-387-95919-1_56. ISBN 978-0-387-95919-1. Número de pieza 7176201 .
- ^ ab Decaro N (2011). "Betacoronavirus‡: Coronaviridae". En Tidona C, Darai G (eds.). Índice Springer de virus . Springer. págs. 385–401. doi :10.1007/978-0-387-95919-1_57. ISBN . 978-0-387-95919-1. Número de pieza 7176184 .
- ^ ab Li F, Li W, Farzan M, Harrison SC (septiembre de 2005). "Estructura del dominio de unión al receptor de la proteína spike del coronavirus del SARS en complejo con el receptor". Science . 309 (5742): 1864–68. Bibcode :2005Sci...309.1864L. doi : 10.1126/science.1116480 . PMID 16166518. S2CID 12438123.
- ^ Comité Internacional de Taxonomía de Virus (24 de agosto de 2010). "ICTV Master Species List 2009—v10". Archivado desde el original (xls) el 15 de abril de 2013.
- ^ Wertheim JO, Chu DK, Peiris JS, Kosakovsky Pond SL, Poon LL (junio de 2013). "Un caso para el origen antiguo de los coronavirus". Journal of Virology . 87 (12): 7039–45. doi :10.1128/JVI.03273-12. PMC 3676139 . PMID 23596293.
Los alfacoronavirus y betacoronavirus se encuentran exclusivamente en mamíferos, mientras que los gammacoronavirus y deltacoronavirus infectan principalmente a las aves.
- ^ "Nextstrain, árbol filogenético de Beta-CoV". nextstrain.org .
- ^ Wertheim JO, Chu DK, Peiris JS, Kosakovsky Pond SL, Poon LL (junio de 2013). "Un caso sobre el antiguo origen de los coronavirus". Revista de Virología . 87 (12): 7039–45. doi :10.1128/JVI.03273-12. PMC 3676139 . PMID 23596293.
- ^ Woo PC, Lau SK, Lam CS, Lau CC, Tsang AK, Lau JH y otros. (Abril de 2012). "El descubrimiento de siete nuevos coronavirus de mamíferos y aves en el género deltacoronavirus respalda los coronavirus de murciélago como fuente genética de alfacoronavirus y betacoronavirus y los coronavirus aviares como fuente genética de gammacoronavirus y deltacoronavirus". Revista de Virología . 86 (7): 3995–4008. doi :10.1128/JVI.06540-11. PMC 3302495 . PMID 22278237.
- ^ abc Forni D, Cagliani R, Clerici M, Sironi M (enero de 2017). "Molecular Evolution of Human Coronavirus Genomes". Trends in Microbiology . 25 (1): 35–48. doi :10.1016/j.tim.2016.09.001. PMC 7111218 . PMID 27743750.
Específicamente, se cree que todos los HCoV tienen un origen en murciélagos, con la excepción de los beta-CoV de linaje A, que pueden tener reservorios en roedores [2].
- ^ Huynh J, Li S, Yount B, Smith A, Sturges L, Olsen JC, et al. (diciembre de 2012). "Evidencia que respalda un origen zoonótico de la cepa NL63 del coronavirus humano". Journal of Virology . 86 (23): 12816–25. doi :10.1128/JVI.00906-12. PMC 3497669 . PMID 22993147.
Si estas predicciones son correctas, esta observación sugiere que el HCoV-NL63 puede haberse originado en murciélagos entre 1190 y 1449 d. C.
- ^ Pfefferle S, Oppong S, Drexler JF, Gloza-Rausch F, Ipsen A, Seebens A, et al. (septiembre de 2009). "Parientes lejanos del coronavirus del síndrome respiratorio agudo severo y parientes cercanos del coronavirus humano 229E en murciélagos, Ghana". Enfermedades infecciosas emergentes . 15 (9): 1377–84. doi :10.3201/eid1509.090224. PMC 2819850 . PMID 19788804.
El ancestro común más reciente de hCoV-229E y GhanaBt-CoVGrp1 existió en ≈1686–1800 d. C.
- ^ Crossley BM, Mock RE, Callison SA, Hietala SK (diciembre de 2012). "Identificación y caracterización de un nuevo coronavirus respiratorio de alpaca más estrechamente relacionado con el coronavirus humano 229E". Viruses . 4 (12): 3689–700. doi : 10.3390/v4123689 . PMC 3528286 . PMID 23235471.
- ^ Forni D, Cagliani R, Clerici M, Sironi M (enero de 2017). "Evolución molecular de los genomas de coronavirus humanos". Tendencias en microbiología . 25 (1): 35–48. doi :10.1016/j.tim.2016.09.001. PMC 7111218 . PMID 27743750.
- ^ Lau SK, Li KS, Tsang AK, Lam CS, Ahmed S, Chen H, et al. (agosto de 2013). "La caracterización genética de los virus del linaje C de Betacoronavirus en murciélagos revela una marcada divergencia de secuencia en la proteína de la espícula del coronavirus de murciélago pipistrellus HKU5 en el pipistrello japonés: implicaciones para el origen del nuevo coronavirus del síndrome respiratorio de Oriente Medio". Journal of Virology . 87 (15): 8638–50. doi :10.1128/JVI.01055-13. PMC 3719811 . PMID 23720729.
- ^ Vijaykrishna D, Smith GJ, Zhang JX, Peiris JS, Chen H, Guan Y (abril de 2007). "Conocimientos evolutivos sobre la ecología de los coronavirus". Revista de Virología . 81 (8): 4012–20. doi :10.1128/jvi.02605-06. PMC 1866124 . PMID 17267506.
- ^ Gouilh MA, Puechmaille SJ, Gonzalez JP, Teeling E, Kittayapong P, Manuguerra JC (octubre de 2011). "Huellas de los ancestros del SARS-Coronavirus en colonias de murciélagos del sudeste asiático y la teoría del refugio". Infección, genética y evolución . 11 (7): 1690–702. Bibcode :2011InfGE..11.1690G. doi :10.1016/j.meegid.2011.06.021. PMC 7106191 . PMID 21763784.
- ^ Cui J, Han N, Streicker D, Li G, Tang X, Shi Z, et al. (octubre de 2007). "Relaciones evolutivas entre los coronavirus de murciélago y sus huéspedes". Enfermedades infecciosas emergentes . 13 (10): 1526–32. doi :10.3201/eid1310.070448. PMC 2851503 . PMID 18258002.
- ^ Lau SK, Woo PC, Li KS, Tsang AK, Fan RY, Luk HK, et al. (marzo de 2015). "El descubrimiento de un nuevo coronavirus, el coronavirus Rattus de China HKU24, en ratas noruegas respalda el origen murino del Betacoronavirus 1 y tiene implicaciones para el ancestro del linaje A del Betacoronavirus". Journal of Virology . 89 (6): 3076–92. doi :10.1128/JVI.02420-14. PMC 4337523 . PMID 25552712.
- ^ ab Bidokhti MR, Tråvén M, Krishna NK, Munir M, Belák S, Alenius S, et al. (septiembre de 2013). "Dinámica evolutiva de los coronavirus bovinos: el patrón de selección natural del gen de la espícula implica una evolución adaptativa de las cepas". The Journal of General Virology . 94 (Pt 9): 2036–2049. doi : 10.1099/vir.0.054940-0 . PMID 23804565.
Véase la Tabla 1
- ^ Vijgen L, Keyaerts E, Moës E, Thoelen I, Wollants E, Lemey P, et al. (febrero de 2005). "Secuencia genómica completa del coronavirus humano OC43: el análisis del reloj molecular sugiere un evento de transmisión de coronavirus zoonótico relativamente reciente". Journal of Virology . 79 (3): 1595–604. doi :10.1128/jvi.79.3.1595-1604.2005. PMC 544107 . PMID 15650185.
- ^ Vijgen L, Keyaerts E, Moës E, Thoelen I, Wollants E, Lemey P, et al. (febrero de 2005). "Secuencia genómica completa del coronavirus humano OC43: el análisis del reloj molecular sugiere un evento de transmisión de coronavirus zoonótico relativamente reciente". Journal of Virology . 79 (3): 1595–604. doi :10.1128/JVI.79.3.1595-1604.2005. PMC 544107 . PMID 15650185.
Sin embargo, es tentador especular sobre una hipótesis alternativa, que la pandemia de 1889-1890 puede haber sido el resultado de la transmisión interespecies de coronavirus bovinos a humanos, lo que resultó en la aparición posterior de HCoV-OC43.
- ^ ab Corman VM, Muth D, Niemeyer D, Drosten C (2018). "Huéspedes y fuentes de coronavirus humanos endémicos". Avances en la investigación de virus . 100 : 163–188. doi : 10.1016/bs.aivir.2018.01.001 . ISBN . 978-0-12-815201-0. PMC 7112090 . PMID 29551135.
- ^ Lau SK, Lee P, Tsang AK, Yip CC, Tse H, Lee RA, et al. (noviembre de 2011). "La epidemiología molecular del coronavirus humano OC43 revela la evolución de diferentes genotipos a lo largo del tiempo y la aparición reciente de un nuevo genotipo debido a la recombinación natural". Journal of Virology . 85 (21): 11325–37. doi :10.1128/JVI.05512-11. PMC 3194943 . PMID 21849456.
- ^ Schaumburg CS, Held KS, Lane TE (mayo de 2008). "Infección del sistema nervioso central por el virus de la hepatitis del ratón: un modelo de defensa, enfermedad y reparación". Frontiers in Bioscience . 13 (13): 4393–406. doi :10.2741/3012. PMC 5025298 . PMID 18508518.
- ^ Liu P, Shi L, Zhang W, He J, Liu C, Zhao C, et al. (noviembre de 2017). "Análisis de prevalencia y diversidad genética de coronavirus humanos entre niños transfronterizos". Revista de Virología . 14 (1): 230. doi : 10.1186/s12985-017-0896-0 . PMC 5700739 . PMID 29166910.
- ^ ab Forgie S, Marrie TJ (febrero de 2009). "Neumonía atípica asociada a la atención médica". Seminarios en medicina respiratoria y de cuidados críticos . 30 (1): 67–85. doi :10.1055/s-0028-1119811. PMID 19199189. S2CID 260316838.
- ^ King A (2020-05-02). "Un resfriado poco común". New Scientist . 246 (3280): 32–35. Código Bibliográfico :2020NewSc.246...32K. doi :10.1016/S0262-4079(20)30862-9. ISSN 0262-4079. PMC 7252012 . PMID 32501321.
- ^ Cecil RL, Goldman L, Schafer AI (2012). Goldman's Cecil Medicine, Expert Consult Premium Edition (24.ª ed.). Elsevier Health Sciences. págs. 2103–. ISBN 978-1-4377-1604-7. Archivado desde el original el 4 de mayo de 2016.
- ^ Pelczar (2010). Microbiología: enfoque basado en aplicaciones. McGraw-Hill Education (India) Pvt Limited. pág. 656. ISBN 978-0-07-015147-5. Archivado desde el original el 16 de mayo de 2016.
- ^ Charlton CL, Babady E, Ginocchio CC, Hatchette TF, Jerris RC, Li Y, et al. (enero de 2019). "Orientación práctica para laboratorios de microbiología clínica: virus que causan infecciones agudas del tracto respiratorio". Clinical Microbiology Reviews . 32 (1). doi :10.1128/CMR.00042-18. PMC 6302358 . PMID 30541871.
Véase la Figura 1.
- ^ Monto AS, DeJonge P, Callear AP, Bazzi LA, Capriola S, Malosh RE, et al. (abril de 2020). "Presentación y transmisión del coronavirus durante 8 años en la cohorte HIVE de hogares en Michigan". The Journal of Infectious Diseases . 222 : 9–16. doi :10.1093/infdis/jiaa161. PMC 7184402 . PMID 32246136.
- ^ Abdul-Rasool S, Fielding BC (mayo de 2010). "Comprensión del coronavirus humano HCoV-NL63". The Open Virology Journal . 4 : 76–84. doi : 10.2174/1874357901004010076 . PMC 2918871 . PMID 20700397.
- ^ Wang C, Horby PW, Hayden FG, Gao GF (febrero de 2020). "Un brote de nuevo coronavirus de preocupación sanitaria mundial". Lancet . 395 (10223): 470–473. doi : 10.1016/S0140-6736(20)30185-9 . PMC 7135038 . PMID 31986257.
- ^ Lau EH, Hsiung CA, Cowling BJ, Chen CH, Ho LM, Tsang T, et al. (marzo de 2010). "Un análisis epidemiológico comparativo del SARS en Hong Kong, Pekín y Taiwán". BMC Infectious Diseases . 10 : 50. doi : 10.1186/1471-2334-10-50 . PMC 2846944 . PMID 20205928.
- ^ ab "La vejez y la sepsis se relacionan con malos resultados en la COVID-19 y la muerte". CIDRAP , Universidad de Minnesota . 2020-03-10 . Consultado el 2020-03-29 .
- ^ Karlberg J, Chong DS, Lai WY (febrero de 2004). "¿Los hombres tienen una tasa de letalidad más alta de síndrome respiratorio agudo severo que las mujeres?". American Journal of Epidemiology . 159 (3): 229–31. doi :10.1093/aje/kwh056. PMC 7110237 . PMID 14742282.
- ^ ab "Resumen de los casos probables de SARS con inicio de la enfermedad entre el 1 de noviembre de 2002 y el 31 de julio de 2003". Organización Mundial de la Salud. Abril de 2004.
- ^ abcde "Panel de control de COVID-19 del Centro de Ciencias e Ingeniería de Sistemas (CSSE) de la Universidad Johns Hopkins (JHU)". ArcGIS . Universidad Johns Hopkins . Consultado el 10 de marzo de 2023 .
- ^ abcde "Informe de la misión conjunta OMS-China sobre la enfermedad por coronavirus 2019 (COVID-19)" (PDF) . Organización Mundial de la Salud. Febrero de 2020.
- ^ Oh MD, Park WB, Park SW, Choe PG, Bang JH, Song KH, et al. (marzo de 2018). "Síndrome respiratorio de Oriente Medio: lo que aprendimos del brote de 2015 en la República de Corea". The Korean Journal of Internal Medicine . 33 (2): 233–246. doi :10.3904/kjim.2018.031. PMC 5840604 . PMID 29506344.
- ^ Ñamendys-Silva SA (marzo de 2020). "Soporte respiratorio para pacientes con infección por COVID-19". The Lancet. Medicina respiratoria . 8 (4): e18. doi :10.1016/S2213-2600(20)30110-7. PMC 7129706 . PMID 32145829.
- ^ Pasley J. "Cómo el SARS aterrorizó al mundo en 2003, infectando a más de 8.000 personas y matando a 774". Business Insider . Consultado el 8 de noviembre de 2020 .
- ^ Doucleef M (26 de septiembre de 2012). "Los científicos profundizan en los genes de un virus similar al SARS". Associated Press. Archivado desde el original el 27 de septiembre de 2012. Consultado el 27 de septiembre de 2012 .
- ^ Falco M (24 de septiembre de 2012). "Nuevo virus similar al SARS plantea un misterio médico". CNN Health . Archivado desde el original el 1 de noviembre de 2013. Consultado el 16 de marzo de 2013 .
- ^ "Se encuentra un nuevo virus similar al SARS en Oriente Medio". Al-Jazeera . 24 de septiembre de 2012. Archivado desde el original el 9 de marzo de 2013 . Consultado el 16 de marzo de 2013 .
- ^ Kelland K (28 de septiembre de 2012). «Nuevo virus que no se propaga fácilmente entre personas: OMS». Reuters . Archivado desde el original el 24 de noviembre de 2012. Consultado el 16 de marzo de 2013 .
- ^ Nuevo coronavirus: punto de situación: Un nouveau cas d'infection confirmé Archivado el 8 de junio de 2013 en Wayback Machine (Nuevo coronavirus: informe de estado: un nuevo caso de infección confirmada) 12 de mayo de 2013, social-sante.gouv.fr
- ^ "Transmisión del MERS". Centros para el Control y la Prevención de Enfermedades (CDC) . 2 de agosto de 2019. Archivado desde el original el 7 de diciembre de 2019. Consultado el 10 de diciembre de 2019 .
- ^ "Infección por el nuevo coronavirus". Asociación Mundial de la Salud. 22 de mayo de 2013. Archivado desde el original el 7 de junio de 2013. Consultado el 23 de mayo de 2013 .
- ^ "MERS en los EE. UU." Centro para el Control de Enfermedades . 2 de agosto de 2019. Archivado desde el original el 15 de diciembre de 2019. Consultado el 10 de diciembre de 2019 .
- ^ Sang-Hun C (8 de junio de 2015). "El camino del virus MERS: un hombre, muchos hospitales de Corea del Sur". The New York Times . Archivado desde el original el 15 de julio de 2017. Consultado el 1 de marzo de 2017 .
- ^ "Coronavirus causante del síndrome respiratorio de Oriente Medio (MERS-CoV)". OMS . Archivado desde el original el 18 de octubre de 2019. Consultado el 10 de diciembre de 2019 .
- ^ El Consejo Editorial (29 de enero de 2020). "¿Está el mundo preparado para el coronavirus? La desconfianza en la ciencia y las instituciones podría ser un problema importante si el brote empeora". The New York Times . Consultado el 30 de enero de 2020 .
- ^ "Declaración de la OMS sobre el grupo de casos de neumonía en Wuhan (China)". www.who.int . 2020-01-09. Archivado desde el original el 2020-01-14 . Consultado el 2020-01-10 .
- ^ "Pruebas de laboratorio de casos humanos sospechosos de infección por el nuevo coronavirus (nCoV). Orientación provisional, 10 de enero de 2020" (PDF) . Archivado (PDF) desde el original el 20 de enero de 2020 . Consultado el 14 de enero de 2020 .
- ^ "Nuevo coronavirus 2019, Wuhan, China". www.cdc.gov (CDC) . 23 de enero de 2020. Archivado desde el original el 20 de enero de 2020. Consultado el 23 de enero de 2020 .
- ^ "Infección por el nuevo coronavirus 2019 (Wuhan, China): actualización del brote". Canada.ca . 2020-01-21.
- ^ Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R, Dar O, et al. (febrero de 2020). "La continua amenaza epidémica de 2019-nCoV de los nuevos coronavirus para la salud mundial: el último brote de nuevo coronavirus de 2019 en Wuhan, China". Revista Internacional de Enfermedades Infecciosas . 91 : 264–66. doi : 10.1016/j.ijid.2020.01.009 . PMC 7128332 . PMID 31953166.
- ^ Cohen J (26 de enero de 2020). "El mercado de mariscos de Wuhan puede no ser la fuente de la propagación global de un nuevo virus". ScienceMag Asociación Estadounidense para el Avance de la Ciencia. (AAAS) . Archivado desde el original el 27 de enero de 2020. Consultado el 29 de enero de 2020 .
- ^ Eschner K (28 de enero de 2020). «Todavía no estamos seguros de dónde vino realmente el COVID-19». Popular Science . Archivado desde el original el 30 de enero de 2020. Consultado el 30 de enero de 2020 .
- ^ abcdefghijk "Capítulo 24 - Coronaviridae". Fenner's Veterinary Virology (Quinta edición). Academic Press. 2017. págs. 435–461. doi :10.1016/B978-0-12-800946-8.00024-6. ISBN 978-0-12-800946-8.S2CID219575461 .
- ^ Murphy FA, Gibbs EP, Horzinek MC, Studdart MJ (1999). Virología veterinaria . Boston: Academic Press. págs. 495-508. ISBN. 978-0-12-511340-3.
- ^ ab Tirotta E, Carbajal KS, Schaumburg CS, Whitman L, Lane TE (julio de 2010). "Terapias de reemplazo celular para promover la remielinización en un modelo viral de desmielinización". Journal of Neuroimmunology . 224 (1–2): 101–07. doi :10.1016/j.jneuroim.2010.05.013. PMC 2919340 . PMID 20627412.
- ^ ab "Manual veterinario de Merck". Manual veterinario de Merck . Consultado el 8 de junio de 2020 .
- ^ ab Bande F, Arshad SS, Bejo MH, Moeini H, Omar AR (2015). "Progreso y desafíos hacia el desarrollo de vacunas contra la bronquitis infecciosa aviar". Journal of Immunology Research . 2015 : 424860. doi : 10.1155/2015/424860 . PMC 4411447 . PMID 25954763.
- ^ Cavanagh D (2007). "Virus de la bronquitis infecciosa aviar por coronavirus". Investigación veterinaria . 38 (2): 281–97. doi : 10.1051/vetres:2006055 . PMID 17296157.

- ^ "Navegador de taxonomía (coronavirus aviar)" www.ncbi.nlm.nih.gov . Consultado el 3 de junio de 2020 .
- ^ Zhou P, Fan H, Lan T, Yang XL, Shi WF, Zhang W, et al. (abril de 2018). "Síndrome de diarrea aguda porcina mortal causado por un coronavirus relacionado con HKU2 de origen en murciélagos". Nature . 556 (7700): 255–58. Bibcode :2018Natur.556..255Z. doi : 10.1038/s41586-018-0010-9 . PMC 7094983 . PMID 29618817.
- ^ Wei X, She G, Wu T, Xue C, Cao Y (febrero de 2020). "El PEDV ingresa a las células a través de la endocitosis mediada por clatrina, caveolas y balsa lipídica y se desplaza a través de la vía endo/lisosoma". Veterinary Research . 51 (1): 10. doi : 10.1186/s13567-020-0739-7 . PMC 7011528 . PMID 32041637.
- ^ abc "Navegador de taxonomía (Alphacoronavirus 1)". www.ncbi.nlm.nih.gov . Consultado el 8 de junio de 2020 .
- ^ Cruz JL, Sola I, Becares M, Alberca B, Plana J, Enjuanes L, et al. (junio de 2011). "El gen 7 del coronavirus contrarresta las defensas del huésped y modula la virulencia del virus". PLOS Pathogens . 7 (6): e1002090. doi : 10.1371/journal.ppat.1002090 . PMC 3111541 . PMID 21695242.
- ^ Cruz JL, Becares M, Sola I, Oliveros JC, Enjuanes L, Zúñiga S (septiembre de 2013). "La proteína 7 del alfacoronavirus modula la respuesta inmune innata del huésped". Revista de Virología . 87 (17): 9754–67. doi :10.1128/JVI.01032-13. PMC 3754097 . PMID 23824792.
- ^ ab "Navegador de taxonomía (Betacoronavirus 1)". www.ncbi.nlm.nih.gov . Consultado el 8 de junio de 2020 .
- ^ "Navegador de taxonomía (Alphacoronavirus)" www.ncbi.nlm.nih.gov . Consultado el 8 de junio de 2020 .
- ^ Murray J (16 de abril de 2014). "¿Qué hay de nuevo con la enfermedad similar a la PIF en hurones?" (xls) . Archivado desde el original el 24 de abril de 2014. Consultado el 24 de abril de 2014 .
- ^ "Enfermedades infecciosas de los hurones: animales exóticos y de laboratorio". Manual veterinario Merck . Consultado el 8 de junio de 2020 .
- ^ ab "Navegador de taxonomía (Embecovirus)". www.ncbi.nlm.nih.gov . Consultado el 8 de junio de 2020 .
- ^ Weiss SR, Navas-Martin S (diciembre de 2005). "Patogénesis del coronavirus y el patógeno emergente coronavirus causante del síndrome respiratorio agudo severo". Microbiology and Molecular Biology Reviews . 69 (4): 635–64. doi :10.1128/MMBR.69.4.635-664.2005. PMC 1306801 . PMID 16339739.
- ^ "Coronavirus entérico". Enfermedades de los animales de investigación . Archivado desde el original el 1 de julio de 2019. Consultado el 24 de enero de 2020 .
- ^ "Rastreador de vacunas y tratamientos contra la COVID-19 (seleccione la pestaña de vacunas o tratamientos, aplique filtros para ver los datos seleccionados)". Instituto Milken. 2020-11-03 . Consultado el 2020-11-03 .
- ^ ab "Rastreador de vacunas y terapias contra la COVID-19". BioRender. 2020-10-30 . Consultado el 2020-11-03 .
- ^ Dong L, Hu S, Gao J (2020). "Descubrimiento de fármacos para tratar la enfermedad por coronavirus 2019 (COVID-19)". Descubrimientos farmacológicos y terapias . 14 (1): 58–60. doi : 10.5582/ddt.2020.01012 . PMID 32147628.
Lectura adicional
- Acheson NH (2011). "Capítulo 14: Coronavirus". Fundamentos de virología molecular . Hoboken, NJ: John Wiley & Sons. págs. 159-171. ISBN 978-0-470-90059-8.
- Alwan A, Mahjour J, Memish ZA (2013). “Infección por el nuevo coronavirus: es hora de mantenerse a la vanguardia”. Revista de Salud del Mediterráneo Oriental . 19 (Supl 1): S3–4. doi : 10.26719/2013.19.supp1.S3 . PMID 23888787.[ enlace muerto ]
- Laude H, Rasschaert D, Delmas B, Godet M, Gelfi J, Charley B (junio de 1990). "Biología molecular del virus de la gastroenteritis transmisible". Microbiología veterinaria . 23 (1–4): 147–54. doi :10.1016/0378-1135(90)90144-K. PMC 7117338. PMID 2169670 .
- Sola I, Alonso S, Zúñiga S, Balasch M, Plana-Durán J, Enjuanes L (abril de 2003). "Diseñar el genoma del virus de la gastroenteritis transmisible como vector de expresión que induce inmunidad lactogénica". Revista de Virología . 77 (7): 4357–69. doi :10.1128/JVI.77.7.4357-4369.2003. PMC 150661 . PMID 12634392.
- Tajima M (1970). "Morfología del virus de la gastroenteritis transmisible del cerdo. Un posible miembro de los coronavirus. Breve informe". Archiv für die Gesamte Virusforschung . 29 (1): 105–08. doi :10.1007/BF01253886. PMC 7086923 . PMID 4195092. S2CID 42104521.






