Biología molecular
| Parte de una serie sobre |
| Biología |
|---|
 |
La biología molecular / m ə ˈ l ɛ k j ʊ l ər / es una rama de la biología que busca comprender la base molecular de la actividad biológica en y entre las células , incluida la síntesis, modificación, mecanismos e interacciones biomoleculares . [1] [2] [3]
Aunque las células y otras estructuras microscópicas se habían observado en organismos vivos desde el siglo XVIII, una comprensión detallada de los mecanismos e interacciones que rigen su comportamiento no surgió hasta el siglo XX, cuando las tecnologías utilizadas en física y química habían avanzado lo suficiente como para permitir su aplicación en las ciencias biológicas. El término "biología molecular" fue utilizado por primera vez en 1945 por el físico inglés William Astbury , quien lo describió como un enfoque centrado en discernir los fundamentos de los fenómenos biológicos, es decir, descubrir las estructuras y propiedades físicas y químicas de las moléculas biológicas, así como sus interacciones con otras moléculas y cómo estas interacciones explican las observaciones de la llamada biología clásica, que en cambio estudia los procesos biológicos a escalas mayores y niveles superiores de organización. [4] En 1953, Francis Crick , James Watson , Rosalind Franklin y sus colegas de la Unidad del Consejo de Investigación Médica del Laboratorio Cavendish fueron los primeros en describir el modelo de doble hélice para la estructura química del ácido desoxirribonucleico (ADN), que a menudo se considera un acontecimiento histórico para el campo naciente porque proporcionó una base fisicoquímica para comprender la idea previamente nebulosa de los ácidos nucleicos como la sustancia primaria de la herencia biológica. Propusieron esta estructura basándose en investigaciones previas realizadas por Franklin, que les fueron transmitidas por Maurice Wilkins y Max Perutz . [5] Su trabajo condujo al descubrimiento del ADN en otros microorganismos, plantas y animales. [6]
El campo de la biología molecular incluye técnicas que permiten a los científicos aprender sobre los procesos moleculares. [7] Estas técnicas se utilizan para seleccionar de manera eficiente nuevos medicamentos, diagnosticar enfermedades y comprender mejor la fisiología celular. [8] Algunas investigaciones clínicas y terapias médicas que surgen de la biología molecular están cubiertas por la terapia génica , mientras que el uso de la biología molecular o la biología celular molecular en medicina ahora se conoce como medicina molecular . [ cita requerida ]
Historia de la biología molecular


La biología molecular se encuentra en la intersección de la bioquímica y la genética ; a medida que estas disciplinas científicas surgieron y evolucionaron en el siglo XX, se hizo evidente que ambas buscaban determinar los mecanismos moleculares que subyacen a las funciones celulares vitales. [9] [10] Los avances en biología molecular han estado estrechamente relacionados con el desarrollo de nuevas tecnologías y su optimización. [11] La biología molecular ha sido dilucidada por el trabajo de muchos científicos y, por lo tanto, la historia del campo depende de la comprensión de estos científicos y sus experimentos. [ cita requerida ]
El campo de la genética surgió de los intentos de comprender el conjunto de reglas que subyacen a la reproducción y la herencia , y la naturaleza de las unidades hipotéticas de la herencia conocidas como genes . Gregor Mendel fue pionero en este trabajo en 1866, cuando describió por primera vez las leyes de la herencia que observó en sus estudios de cruces de apareamiento en plantas de guisantes. [12] Una de esas leyes de la herencia genética es la ley de la segregación , que establece que los individuos diploides con dos alelos para un gen en particular pasarán uno de estos alelos a su descendencia. [13] Debido a su trabajo crítico, el estudio de la herencia genética se conoce comúnmente como genética mendeliana . [14]
Un hito importante en la biología molecular fue el descubrimiento de la estructura del ADN . Este trabajo comenzó en 1869 por Friedrich Miescher , un bioquímico suizo que propuso por primera vez una estructura llamada nucleína , que ahora sabemos que es (ácido desoxirribonucleico), o ADN. [15] Descubrió esta sustancia única al estudiar los componentes de vendajes llenos de pus y notando las propiedades únicas de las "sustancias que contienen fósforo". [16] Otro contribuyente notable al modelo de ADN fue Phoebus Levene , quien propuso el "modelo de polinucleótido" del ADN en 1919 como resultado de sus experimentos bioquímicos en levadura. [17] En 1950, Erwin Chargaff amplió el trabajo de Levene y dilucidó algunas propiedades críticas de los ácidos nucleicos: primero, la secuencia de ácidos nucleicos varía entre especies. [18] Segundo, la concentración total de purinas (adenina y guanina) siempre es igual a la concentración total de pirimidinas (cisteína y timina). [15] Esto se conoce ahora como la regla de Chargaff. En 1953, James Watson y Francis Crick publicaron la estructura de doble hélice del ADN, [19] basándose en el trabajo de cristalografía de rayos X realizado por Rosalind Franklin que les fue transmitido por Maurice Wilkins y Max Perutz . [5] Watson y Crick describieron la estructura del ADN y conjeturaron sobre las implicaciones de esta estructura única para los posibles mecanismos de replicación del ADN. [19] Watson y Crick recibieron el Premio Nobel de Fisiología o Medicina en 1962, junto con Wilkins, por proponer un modelo de la estructura del ADN. [6]
En 1961, se demostró que cuando un gen codifica una proteína , tres bases secuenciales del ADN de un gen especifican cada aminoácido sucesivo de la proteína. [20] Por lo tanto, el código genético es un código de tripletes, donde cada triplete (llamado codón ) especifica un aminoácido particular. Además, se demostró que los codones no se superponen entre sí en la secuencia de ADN que codifica una proteína, y que cada secuencia se lee desde un punto de partida fijo. Durante 1962-1964, mediante el uso de mutantes letales condicionales de un virus bacteriano, [21] se lograron avances fundamentales en nuestra comprensión de las funciones e interacciones de las proteínas empleadas en la maquinaria de replicación del ADN , reparación del ADN , recombinación del ADN y en el ensamblaje de estructuras moleculares. [22]
El experimento de Griffith

En 1928, Frederick Griffith descubrió una propiedad virulenta en la bacteria neumococo , que estaba matando ratas de laboratorio. Según Mendel, que prevalecía en ese momento, la transferencia de genes solo podía ocurrir de células madre a células hijas. Griffith propuso otra teoría, afirmando que la transferencia de genes que ocurre en miembros de la misma generación se conoce como transferencia horizontal de genes (HGT). Este fenómeno ahora se conoce como transformación genética. [23]
El experimento de Griffith se centró en la bacteria neumococo, que tenía dos cepas diferentes, una virulenta y lisa y otra avirulenta y rugosa. La cepa lisa tenía un aspecto brillante debido a la presencia de un tipo de polisacárido específico: una cápsula de polímero de glucosa y ácido glucurónico. Debido a esta capa de polisacárido de la bacteria, el sistema inmunológico del huésped no puede reconocerla y mata al huésped. La otra cepa, avirulenta y rugosa, carece de esta cápsula de polisacárido y tiene un aspecto opaco y rugoso. [ cita requerida ]
Se sabe que la presencia o ausencia de cápsula en la cepa está determinada genéticamente. Las cepas lisas y rugosas se presentan en varios tipos diferentes, como SI, S-II, S-III, etc. y RI, R-II, R-III, etc. respectivamente. Todos estos subtipos de bacterias S y R difieren entre sí en el tipo de antígeno que producen. [6]
Experimento de Avery-MacLeod-McCarty
El experimento de Avery-MacLeod-McCarty fue un estudio histórico realizado en 1944 que demostró que el ADN, no las proteínas como se creía anteriormente, transporta la información genética en las bacterias. Oswald Avery , Colin Munro MacLeod y Maclyn McCarty utilizaron un extracto de una cepa de neumococo que podía causar neumonía en ratones. Demostraron que la transformación genética en las bacterias podía lograrse inyectándoles ADN purificado del extracto. Descubrieron que cuando digerían el ADN del extracto con DNasa , se perdía la transformación de bacterias inofensivas en virulentas. Esto proporcionó una evidencia sólida de que el ADN era el material genético, desafiando la creencia predominante de que las proteínas eran las responsables. Sentó las bases para el posterior descubrimiento de su estructura por Watson y Crick.
Experimento de Hershey-Chase

La confirmación de que el ADN es el material genético que causa la infección se obtuvo a partir del experimento de Hershey-Chase . Para el experimento se utilizaron bacterias E. coli y bacteriófagos. Este experimento también se conoce como experimento de la licuadora, ya que se utilizó una licuadora de cocina como pieza principal del aparato. Alfred Hershey y Martha Chase demostraron que el ADN inyectado por una partícula de fago en una bacteria contiene toda la información necesaria para sintetizar partículas de fago progenie. Utilizaron radioactividad para marcar la capa proteica del bacteriófago con azufre radiactivo y el ADN con fósforo radiactivo, en dos tubos de ensayo diferentes respectivamente. Después de mezclar el bacteriófago y la E. coli en el tubo de ensayo, comienza el período de incubación en el que el fago transforma el material genético en las células de E. coli . Luego se mezcla o agita la mezcla, lo que separa el fago de las células de E. coli . Se centrifuga toda la mezcla y se comprueba el sedimento que contiene células de E. coli y se descarta el sobrenadante. Las células de E. coli mostraron fósforo radiactivo, lo que indicó que el material transformado era ADN y no la capa de proteína.
El ADN transformado se une al ADN de E. coli y la radiactividad solo se ve en el ADN del bacteriófago. Este ADN mutado puede transmitirse a la siguiente generación y así surgió la teoría de la transducción. La transducción es un proceso en el que el ADN bacteriano transporta el fragmento de bacteriófagos y lo transmite a la siguiente generación. Este también es un tipo de transferencia horizontal de genes. [6]
Experimento de Meselson-Stahl

El experimento de Meselson-Stahl fue un hito en la biología molecular que proporcionó evidencia de la replicación semiconservativa del ADN. Realizado en 1958 por Matthew Meselson y Franklin Stahl , el experimento implicó el cultivo de bacterias E. coli en un medio que contenía el isótopo pesado del nitrógeno ( 15 N) durante varias generaciones. Esto hizo que todo el ADN bacteriano recién sintetizado se incorporara con el isótopo pesado.
Después de permitir que las bacterias se replicaran en un medio que contenía nitrógeno normal ( 14 N), se tomaron muestras en varios momentos. Luego, estas muestras se sometieron a centrifugación en un gradiente de densidad, que separó las moléculas de ADN en función de su densidad.
Los resultados mostraron que después de una generación de replicación en el medio 14 N, el ADN formó una banda de densidad intermedia entre la del ADN 15 N puro y la del ADN 14 N puro. Esto respaldó la replicación de ADN semiconservativa propuesta por Watson y Crick, donde cada hebra de la molécula de ADN parental sirve como plantilla para la síntesis de una nueva hebra complementaria, lo que da como resultado dos moléculas de ADN hija, cada una compuesta por una hebra parental y una hebra recién sintetizada.
El experimento de Meselson-Stahl proporcionó evidencia convincente de la replicación semiconservativa del ADN, que es fundamental para la comprensión de la genética y la biología molecular.
Biología molecular moderna
A principios de la década de 2020, la biología molecular entró en una era dorada definida por el desarrollo técnico tanto vertical como horizontal. En el plano vertical, las nuevas tecnologías están permitiendo el seguimiento en tiempo real de los procesos biológicos a nivel atómico. [24] Hoy en día, los biólogos moleculares tienen acceso a datos de secuenciación cada vez más asequibles a profundidades cada vez mayores, lo que facilita el desarrollo de nuevos métodos de manipulación genética en nuevos organismos no modelo. Del mismo modo, los biólogos moleculares sintéticos impulsarán la producción industrial de moléculas pequeñas y macro a través de la introducción de vías metabólicas exógenas en varias líneas celulares procariotas y eucariotas. [25]
En sentido horizontal, los datos de secuenciación son cada vez más asequibles y se utilizan en muchos campos científicos diferentes. Esto impulsará el desarrollo de industrias en países en desarrollo y aumentará la accesibilidad para investigadores individuales. Asimismo, los experimentos de edición genética CRISPR-Cas9 ahora pueden ser concebidos e implementados por individuos por menos de 10.000 dólares en organismos nuevos, lo que impulsará el desarrollo de aplicaciones industriales y médicas. [26]
Relación con otras ciencias biológicas

La siguiente lista describe un punto de vista sobre las relaciones interdisciplinarias entre la biología molecular y otros campos relacionados. [27]
- La biología molecular es el estudio de las bases moleculares de los fenómenos biológicos, centrándose en la síntesis, modificación, mecanismos e interacciones moleculares.
- La bioquímica es el estudio de las sustancias químicas y los procesos vitales que ocurren en los organismos vivos . Los bioquímicos se centran principalmente en el papel, la función y la estructura de biomoléculas como las proteínas , los lípidos , los carbohidratos y los ácidos nucleicos . [28]
- La genética es el estudio de cómo las diferencias genéticas afectan a los organismos. La genética intenta predecir cómo las mutaciones , los genes individuales y las interacciones genéticas pueden afectar la expresión de un fenotipo [29]
Aunque los investigadores practican técnicas específicas de la biología molecular, es común combinarlas con métodos de la genética y la bioquímica . Gran parte de la biología molecular es cuantitativa, y recientemente se ha realizado una cantidad significativa de trabajo utilizando técnicas de ciencias de la computación como la bioinformática y la biología computacional . La genética molecular , el estudio de la estructura y función de los genes, ha sido uno de los subcampos más destacados de la biología molecular desde principios de la década de 2000. Otras ramas de la biología se nutren de la biología molecular, ya sea estudiando directamente las interacciones de las moléculas por derecho propio, como en la biología celular y la biología del desarrollo , o indirectamente, donde se utilizan técnicas moleculares para inferir atributos históricos de poblaciones o especies , como en campos de la biología evolutiva como la genética de poblaciones y la filogenética . También existe una larga tradición de estudiar biomoléculas "desde cero", o molecularmente, en la biofísica . [30]
Técnicas de biología molecular

Clonación molecular

La clonación molecular se utiliza para aislar y luego transferir una secuencia de ADN de interés a un vector plasmídico. [31] Esta tecnología de ADN recombinante se desarrolló por primera vez en la década de 1960. [32] En esta técnica, una secuencia de ADN que codifica una proteína de interés se clona utilizando la reacción en cadena de la polimerasa (PCR) y/o enzimas de restricción en un plásmido ( vector de expresión ). El vector plasmídico generalmente tiene al menos 3 características distintivas: un origen de replicación, un sitio de clonación múltiple (MCS) y un marcador selectivo (generalmente resistencia a antibióticos ). Además, aguas arriba del MCS se encuentran las regiones promotoras y el sitio de inicio de la transcripción , que regulan la expresión del gen clonado.
Este plásmido se puede insertar en células bacterianas o animales. La introducción de ADN en células bacterianas se puede realizar por transformación a través de la captación de ADN desnudo, conjugación a través del contacto célula-célula o por transducción a través de un vector viral. La introducción de ADN en células eucariotas , como las células animales, por medios físicos o químicos se denomina transfección . Hay disponibles varias técnicas de transfección diferentes, como la transfección con fosfato de calcio, la electroporación , la microinyección y la transfección con liposomas . El plásmido puede integrarse en el genoma , lo que da como resultado una transfección estable, o puede permanecer independiente del genoma y expresarse temporalmente, lo que se denomina transfección transitoria. [33] [34]
El ADN que codifica una proteína de interés se encuentra ahora dentro de una célula y la proteína puede expresarse. Hay una variedad de sistemas, como promotores inducibles y factores de señalización celular específicos, disponibles para ayudar a expresar la proteína de interés en niveles altos. A continuación, se pueden extraer grandes cantidades de una proteína de la célula bacteriana o eucariota. La proteína puede analizarse para determinar su actividad enzimática en diversas situaciones, puede cristalizarse para estudiar su estructura terciaria o, en la industria farmacéutica, puede estudiarse la actividad de nuevos fármacos contra la proteína. [35]
Reacción en cadena de la polimerasa
La reacción en cadena de la polimerasa (PCR) es una técnica extremadamente versátil para copiar ADN. En resumen, la PCR permite copiar o modificar una secuencia específica de ADN de formas predeterminadas. La reacción es extremadamente poderosa y, en condiciones perfectas, podría amplificar una molécula de ADN para convertirla en 1.07 mil millones de moléculas en menos de dos horas. La PCR tiene muchas aplicaciones, incluido el estudio de la expresión genética, la detección de microorganismos patógenos, la detección de mutaciones genéticas y la introducción de mutaciones en el ADN. [36] La técnica de PCR se puede utilizar para introducir sitios de enzimas de restricción en los extremos de las moléculas de ADN o para mutar bases particulares del ADN; este último es un método conocido como mutagénesis dirigida al sitio . La PCR también se puede utilizar para determinar si un fragmento de ADN particular se encuentra en una biblioteca de ADNc . La PCR tiene muchas variaciones, como la PCR de transcripción inversa ( RT-PCR ) para la amplificación de ARN y, más recientemente, la PCR cuantitativa que permite la medición cuantitativa de moléculas de ADN o ARN. [37] [38]
.jpg/440px-Two_percent_Agarose_Gel_in_Borate_Buffer_cast_in_a_Gel_Tray_(Front,_angled).jpg)
Electroforesis en gel

La electroforesis en gel es una técnica que separa las moléculas por su tamaño utilizando un gel de agarosa o poliacrilamida. [39] Esta técnica es una de las principales herramientas de la biología molecular. El principio básico es que los fragmentos de ADN se pueden separar aplicando una corriente eléctrica a través del gel; debido a que la cadena principal del ADN contiene grupos fosfato con carga negativa, el ADN migrará a través del gel de agarosa hacia el extremo positivo de la corriente. [39] Las proteínas también se pueden separar en función de su tamaño utilizando un gel SDS-PAGE , o en función del tamaño y su carga eléctrica utilizando lo que se conoce como electroforesis en gel 2D . [40]

El ensayo de proteínas de Bradford
El ensayo de Bradford es una técnica de biología molecular que permite la cuantificación rápida y precisa de moléculas de proteína utilizando las propiedades únicas de un tinte llamado Azul Brillante de Coomassie G-250. [41] El Azul de Coomassie sufre un cambio de color visible de marrón rojizo a azul brillante al unirse a la proteína. [41] En su estado catiónico inestable, el Azul de Coomassie tiene una longitud de onda de fondo de 465 nm y emite un color marrón rojizo. [42] Cuando el Azul de Coomassie se une a la proteína en una solución ácida, la longitud de onda de fondo cambia a 595 nm y el tinte emite un color azul brillante. [42] Las proteínas en el ensayo se unen al azul de Coomassie en aproximadamente 2 minutos, y el complejo proteína-tinte es estable durante aproximadamente una hora, aunque se recomienda que las lecturas de absorbancia se tomen entre 5 y 20 minutos después del inicio de la reacción. [41] La concentración de proteína en el ensayo de Bradford se puede medir utilizando un espectrofotómetro de luz visible y, por lo tanto, no requiere un equipo extenso. [42]
Este método fue desarrollado en 1975 por Marion M. Bradford y ha permitido una cuantificación de proteínas significativamente más rápida y precisa en comparación con los métodos anteriores: el procedimiento de Lowry y el ensayo de biuret. [41] A diferencia de los métodos anteriores, el ensayo de Bradford no es susceptible a la interferencia de varias moléculas no proteicas, incluido el etanol, el cloruro de sodio y el cloruro de magnesio. [41] Sin embargo, es susceptible a la influencia de agentes tampón alcalinos fuertes, como el dodecil sulfato de sodio (SDS). [41]
Transferencia y sondeo de macromoléculas
Los términos northern , western y eastern blotting se derivan de lo que inicialmente fue una broma de biología molecular que jugó con el término Southern blotting , en honor a la técnica descrita por Edwin Southern para la hibridación del ADN borrado. Patricia Thomas, desarrolladora del blot de ARN que luego se conocería como northern blotting , en realidad no utilizó el término. [43]
Transferencia Southern
El Southern blot, que debe su nombre a su inventor, el biólogo Edwin Southern , es un método para detectar la presencia de una secuencia de ADN específica en una muestra de ADN. Las muestras de ADN antes o después de la digestión con enzimas de restricción (endonucleasas de restricción) se separan mediante electroforesis en gel y luego se transfieren a una membrana mediante transferencia por capilaridad . Luego, la membrana se expone a una sonda de ADN marcada que tiene una secuencia de bases complementaria a la secuencia del ADN de interés. [44] El Southern blot se usa con menos frecuencia en la ciencia de laboratorio debido a la capacidad de otras técnicas, como la PCR , para detectar secuencias de ADN específicas a partir de muestras de ADN. Sin embargo, estos blots todavía se usan para algunas aplicaciones, como la medición del número de copias de transgenes en ratones transgénicos o en la ingeniería de líneas de células madre embrionarias con genes knock out . [30]
Transferencia Northern
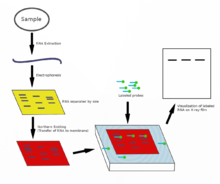
El Northern blot se utiliza para estudiar la presencia de moléculas de ARN específicas como comparación relativa entre un conjunto de diferentes muestras de ARN. Es esencialmente una combinación de electroforesis en gel de ARN desnaturalizante y un blot . En este proceso, el ARN se separa en función del tamaño y luego se transfiere a una membrana que luego se prueba con un complemento marcado de una secuencia de interés. Los resultados se pueden visualizar a través de una variedad de formas dependiendo de la etiqueta utilizada; sin embargo, la mayoría da como resultado la revelación de bandas que representan los tamaños del ARN detectado en la muestra. La intensidad de estas bandas está relacionada con la cantidad del ARN objetivo en las muestras analizadas. El procedimiento se utiliza comúnmente para estudiar cuándo y cuánta expresión génica está ocurriendo midiendo cuánto de ese ARN está presente en diferentes muestras, asumiendo que no ocurre ninguna regulación postranscripcional y que los niveles de ARNm reflejan niveles proporcionales de la proteína correspondiente que se está produciendo. Es una de las herramientas más básicas para determinar en qué momento y bajo qué condiciones se expresan ciertos genes en tejidos vivos. [45] [46]
Western blot
Un western blot es una técnica mediante la cual se pueden detectar proteínas específicas a partir de una mezcla de proteínas. [47] Los western blots se pueden utilizar para determinar el tamaño de las proteínas aisladas, así como para cuantificar su expresión. [48] En el western blot , las proteínas se separan primero por tamaño, en un gel fino intercalado entre dos placas de vidrio en una técnica conocida como SDS-PAGE . Las proteínas en el gel se transfieren luego a una membrana de soporte de fluoruro de polivinilideno (PVDF), nitrocelulosa, nailon u otra membrana de soporte. Esta membrana se puede sondear con soluciones de anticuerpos . Los anticuerpos que se unen específicamente a la proteína de interés se pueden visualizar mediante una variedad de técnicas, incluidos productos coloreados, quimioluminiscencia o autorradiografía . A menudo, los anticuerpos se marcan con enzimas. Cuando un sustrato quimioluminiscente se expone a la enzima, permite la detección. El uso de técnicas de western blot permite no solo la detección sino también el análisis cuantitativo. Se pueden utilizar métodos análogos al western blot para teñir directamente proteínas específicas en células vivas o secciones de tejido . [47] [49]
Transferencia oriental
La técnica de transferencia Eastern se utiliza para detectar modificaciones postraduccionales de proteínas. Las proteínas transferidas a la membrana de PVDF o nitrocelulosa se analizan para detectar modificaciones utilizando sustratos específicos. [50]
Microarrays

Un microarray de ADN es una colección de puntos adheridos a un soporte sólido, como un portaobjetos de microscopio , donde cada punto contiene uno o más fragmentos de oligonucleótidos de ADN monocatenario . Los arrays permiten colocar grandes cantidades de puntos muy pequeños (de 100 micrómetros de diámetro) en un solo portaobjetos. Cada punto tiene una molécula de fragmento de ADN que es complementaria a una sola secuencia de ADN . Una variación de esta técnica permite calificar la expresión genética de un organismo en una etapa particular del desarrollo ( perfil de expresión ). En esta técnica, el ARN de un tejido se aísla y se convierte en ADN complementario marcado (ADNc). Luego, este ADNc se hibrida con los fragmentos en el array y se puede realizar la visualización de la hibridación. Dado que se pueden hacer múltiples arrays con exactamente la misma posición de fragmentos, son particularmente útiles para comparar la expresión genética de dos tejidos diferentes, como un tejido sano y canceroso. Además, se pueden medir qué genes se expresan y cómo cambia esa expresión con el tiempo o con otros factores. Hay muchas formas diferentes de fabricar microarrays; Los más comunes son chips de silicio, portaobjetos de microscopio con puntos de ~100 micrómetros de diámetro, matrices personalizadas y matrices con puntos más grandes en membranas porosas (macroarrays). Puede haber desde 100 puntos hasta más de 10.000 en una matriz dada. Las matrices también se pueden hacer con moléculas distintas del ADN. [51] [52] [53] [54]
Oligonucleótido específico de alelo
El oligonucleótido específico de alelo (ASO) es una técnica que permite la detección de mutaciones de una sola base sin la necesidad de PCR o electroforesis en gel. Se exponen sondas cortas (de 20 a 25 nucleótidos de longitud) marcadas al ADN diana no fragmentado, la hibridación se produce con alta especificidad debido a la corta longitud de las sondas e incluso un solo cambio de base obstaculizará la hibridación. A continuación, se lava el ADN diana y se eliminan las sondas no hibridadas. A continuación, se analiza el ADN diana para detectar la presencia de la sonda mediante radiactividad o fluorescencia. En este experimento, como en la mayoría de las técnicas de biología molecular, se debe utilizar un control para garantizar el éxito de la experimentación. [55] [56]
En biología molecular, se desarrollan continuamente procedimientos y tecnologías y se abandonan las tecnologías más antiguas. Por ejemplo, antes de la llegada de la electroforesis en gel de ADN ( agarosa o poliacrilamida ), el tamaño de las moléculas de ADN se determinaba típicamente por la velocidad de sedimentación en gradientes de sacarosa , una técnica lenta y laboriosa que requería instrumentación costosa; antes de los gradientes de sacarosa, se utilizaba la viscosimetría . Aparte de su interés histórico, a menudo vale la pena conocer tecnologías más antiguas, ya que ocasionalmente es útil para resolver otro problema nuevo para el cual la técnica más nueva es inadecuada. [57]
Véase también
- Astrobiología
- Dogma central de la biología molecular
- Código genético
- Analizador Geniom RT , instrumento de diagnóstico
- Genoma
- Institutos de biología molecular
- Ingeniería molecular
- Modelado molecular
- Predicción de la interacción de proteínas
- Predicción de la estructura de las proteínas
- Proteoma
- Biología celular
Referencias
- ^ Alberts B, Johnson A, Lewis J, Morgan D, Raff M, Roberts K, Walter P (2014). Biología molecular de la célula, sexta edición. Garland Science. págs. 1–10. ISBN 978-1-317-56375-4.
- ^ Gannon F (febrero de 2002). "Biología molecular: ¿qué hay en un nombre?". EMBO Reports . 3 (2): 101. doi :10.1093/embo-reports/kvf039. PMC 1083977 . PMID 11839687.
- ^ "Biología molecular: últimas investigaciones y noticias | Nature". nature.com . Consultado el 7 de noviembre de 2021 .
- ^ Astbury, WT (junio de 1961). "¿Biología molecular o biología ultraestructural?". Nature . 190 (4781): 1124. Bibcode :1961Natur.190.1124A. doi : 10.1038/1901124a0 . ISSN 1476-4687. PMID 13684868. S2CID 4172248.
- ^ ab "Rosalind Franklin: una contribución crucial". nature.com .
- ^ abcd Verma, PS (2004). Biología celular, genética, biología molecular, evolución y ecología . S Chand and Company. ISBN 81-219-2442-1.OCLC 1045495545 .[ página necesaria ]
- ^ Morange, Michel (2016). "Historia de la biología molecular". Enciclopedia de ciencias de la vida . págs. 1–8. doi :10.1002/9780470015902.a0003079.pub3. ISBN 978-0-470-01617-6.
- ^ Bello, Elizabeth A.; Schwinn, Debra A. (1996-12-01). "Biología molecular y medicina: una introducción para el clínico". Anestesiología . 85 (6): 1462–1478. doi : 10.1097/00000542-199612000-00029 . ISSN 0003-3022. PMID 8968195. S2CID 29581630.
- ^ Bynum, William (febrero de 1999). "Una historia de la biología molecular". Nature Medicine . 5 (2): 140–140. doi :10.1038/5498. ISSN 1546-170X.
- ^ Morange, Michel (junio de 2021). Una historia de la biología . Princeton University Press. ISBN 978-0-691-18878-2.OCLC 1184123419 .[ página necesaria ]
- ^ Fields, Stanley (28 de agosto de 2001). "La interacción entre biología y tecnología". Actas de la Academia Nacional de Ciencias . 98 (18): 10051–10054. doi : 10.1073/pnas.191380098 . ISSN 0027-8424. PMC 56913 . PMID 11517346.
- ^ Ellis, TH Noel; Hofer, Julie MI; Timmerman-Vaughan, Gail M.; Coyne, Clarice J.; Hellens, Roger P. (noviembre de 2011). "Mendel, 150 años después". Tendencias en la ciencia de las plantas . 16 (11): 590–596. Bibcode :2011TPS....16..590E. doi :10.1016/j.tplants.2011.06.006. PMID 21775188.
- ^ "12.3C: Ley de segregación de Mendel". Biology LibreTexts . 2018-07-12 . Consultado el 2021-11-18 .
- ^ "Herencia mendeliana". Genome.gov . Consultado el 18 de noviembre de 2021 .
- ^ ab Pray, L (2008). «Descubrimiento de la estructura y función del ADN: Watson y Crick». Nature Education . 1 (1): 100 . Consultado el 21 de junio de 2024 .
- ^ George., Wolf (2003). Friedrich Miescher: el hombre que descubrió el ADN . OCLC 907773747.[ página necesaria ]
- ^ Levene, PA (1919). "Estructura del ácido nucleico de la levadura". Revista de química biológica . 43 (2): 379–382. doi : 10.1016/s0021-9258(18)86289-5 . ISSN 0021-9258.
- ^ Chargaff, Erwin (junio de 1950). "Especificidad química de los ácidos nucleicos y mecanismo de su degradación enzimática". Experientia . 6 (6): 201–209. doi :10.1007/bf02173653. PMID 15421335. S2CID 2522535.
- ^ ab Watson, JD ; Crick, FHC (abril de 1953). "Estructura molecular de los ácidos nucleicos: una estructura para el ácido nucleico desoxirribonucleico". Nature . 171 (4356): 737–738. Bibcode :1953Natur.171..737W. doi :10.1038/171737a0. ISSN 1476-4687. PMID 13054692. S2CID 4253007.
- ^ Crick, FHC; Barnett, Leslie; Brenner, S.; Watts-Tobin, RJ (1961). "Naturaleza general del código genético de las proteínas". Nature . 192 (4809). Springer Science and Business Media LLC: 1227–1232. Bibcode :1961Natur.192.1227C. doi :10.1038/1921227a0. ISSN 0028-0836. PMID 13882203. S2CID 4276146.
- ^ Epstein, RH; Bolle, A.; Steinberg, CM; Kellenberger, E.; Boy de la Tour, E.; et al. (1963-01-01). "Estudios fisiológicos de mutantes letales condicionales del bacteriófago T4D". Simposios de Cold Spring Harbor sobre biología cuantitativa . 28 . Laboratorio de Cold Spring Harbor: 375–394. doi :10.1101/sqb.1963.028.01.053. ISSN 0091-7451.
- ^ Edgar, Bob (1 de octubre de 2004). "El genoma del bacteriófago T4". Genética . 168 (2): 575–582. doi :10.1093/genetics/168.2.575. ISSN 1943-2631. PMC 1448817 . PMID 15514035.
- ^ Ravenhall, Matt; Škunca, Nives; Lassalle, Florent; Dessimoz, Christophe (mayo de 2015). "Inferir la transferencia horizontal de genes". PLOS Computational Biology . 11 (5): e1004095. Bibcode :2015PLSCB..11E4095R. doi : 10.1371/journal.pcbi.1004095 . PMC 4462595 . PMID 26020646.
- ^ Mojiri, Soheil; Isbaner, Sebastian; Mühle, Steffen; Jang, Hongje; Bae, Albert Johann; Gregor, Ingo; Gholami, Azam; Gholami, Azam; Enderlein, Jörg (1 de junio de 2021). "La microscopía rápida de contraste de fase multiplano revela la dinámica torsional en el movimiento flagelar". Biomedical Optics Express . 12 (6): 3169–3180. doi :10.1364/BOE.419099. ISSN 2156-7085. PMC 8221972 . PMID 34221652.
- ^ van Warmerdam, T. "Recursos para laboratorios de biología molecular". Yourbiohelper.com .
- ^ van Warmerdam, T. "Recursos de laboratorio de biología molecular". Yourbiohelper.com .
- ^ Lodish H, Berk A, Zipursky SL, Matsudaira P, Baltimore D, Darnell J (2000). Biología celular molecular (4ª ed.). Nueva York: Scientific American Books. ISBN 978-0-7167-3136-8.
- ^ Berg, Jeremy (2002). Bioquímica . Tymoczko, John L.; Stryer, Lubert (5.ª ed.). Nueva York: WH Freeman. ISBN 0-7167-3051-0.OCLC 48055706 .
- ^ Referencia, Genetics Home. "Ayúdame a entender la genética". Referencia de Genetics Home . Consultado el 31 de diciembre de 2016 .
- ^ ab Tian J, ed. (2013). Imágenes moleculares: fundamentos y aplicaciones. Springer-Verlag Berlin & Heidelberg GmbH & Co. K. pág. 542. ISBN 9783642343032. Recuperado el 8 de julio de 2019 .
- ^ "Fundamentos de la clonación molecular: pasado, presente y futuro | NEB". www.neb.com . Consultado el 25 de noviembre de 2021 .
- ^ "Fundamentos de la clonación molecular: pasado, presente y futuro | NEB". www.neb.com . Consultado el 4 de noviembre de 2021 .
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P. Aislamiento, clonación y secuenciación de ADN . Consultado el 31 de diciembre de 2016 .
- ^ Lessard, Juliane C. (1 de enero de 2013). "Clonación molecular". Métodos de Laboratorio en Enzimología: ADN . vol. 529, págs. 85–98. doi :10.1016/B978-0-12-418687-3.00007-0. ISBN 978-0-12-418687-3. ISSN 1557-7988. PMID 24011038.
- ^ Kokate C, Jalalpure SS, Hurakadle PJ (2016). Libro de texto de biotecnología farmacéutica. Clonación de expresión. Elsevier. p. 125. ISBN 9788131239872. Recuperado el 8 de julio de 2019 .
- ^ Lenstra, JA (julio de 1995). "Las aplicaciones de la reacción en cadena de la polimerasa en las ciencias de la vida". Biología celular y molecular (Noisy-Le-Grand, Francia) . 41 (5): 603–614. ISSN 0145-5680. PMID 7580841.
- ^ "Reacción en cadena de la polimerasa (PCR)". Centro Nacional de Información Biotecnológica . Biblioteca Nacional de Medicina de EE. UU . . Consultado el 31 de diciembre de 2016 .
- ^ "Hoja informativa sobre la reacción en cadena de la polimerasa (PCR)". Instituto Nacional de Investigación del Genoma Humano (NHGRI) . Consultado el 31 de diciembre de 2016 .
- ^ ab Lee, Pei Yun; Costumbrado, John; Hsu, Chih-Yuan; Kim, Yong Hoon (20 de abril de 2012). "Electroforesis en gel de agarosa para la separación de fragmentos de ADN". Journal of Visualized Experiments (62): 3923. doi :10.3791/3923. ISSN 1940-087X. PMC 4846332. PMID 22546956.
- ^ Lee PY, Costumbrado J, Hsu CY, Kim YH (abril de 2012). "Electroforesis en gel de agarosa para la separación de fragmentos de ADN". Journal of Visualized Experiments (62). doi :10.3791/3923. PMC 4846332. PMID 22546956 .
- ^ abcdef Bradford, Marion M. (mayo de 1976). "Un método rápido y sensible para la cuantificación de cantidades de microgramos de proteína utilizando el principio de unión proteína-colorante". Analytical Biochemistry . 72 (1–2): 248–254. doi :10.1016/0003-2697(76)90527-3. PMID 942051. S2CID 4359292.
- ^ abc "Determinación de proteínas por el método de Bradford". www.ruf.rice.edu . Consultado el 8 de noviembre de 2021 .
- ^ Thomas PS (septiembre de 1980). "Hibridación de ARN desnaturalizado y fragmentos pequeños de ADN transferidos a nitrocelulosa". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 77 (9): 5201–5. Bibcode :1980PNAS...77.5201T. doi : 10.1073/pnas.77.9.5201 . PMC 350025 . PMID 6159641.
- ^ Brown, Terry (1993). "Southern Blotting". Protocolos actuales en inmunología . 6 : Unidad 10.6A. doi :10.1002/0471142735.im1006as06. PMID 18432697.
- ^ Josefsen, Knud; Nielsen, Henrik (2011). "Análisis de transferencia Northern". ARN . Métodos en biología molecular. vol. 703, págs. 87-105. doi :10.1007/978-1-59745-248-9_7. ISBN 978-1-58829-913-0. Número de identificación personal 21125485.
- ^ He SL, Green R (1 de enero de 2013). "Northern blotting". Métodos de laboratorio en enzimología: ARN . Vol. 530. págs. 75–87. doi :10.1016/B978-0-12-420037-1.00003-8. ISBN 978-0-12-420037-1. PMC 4287216 . PMID 24034315.
- ^ ab Mahmood T, Yang PC (septiembre de 2012). "Western blot: técnica, teoría y resolución de problemas". Revista norteamericana de ciencias médicas . 4 (9): 429–34. doi : 10.4103/1947-2714.100998 . PMC 3456489 . PMID 23050259.
- ^ "Western blot | Aprende ciencias en Scitable". www.nature.com . Consultado el 25 de noviembre de 2021 .
- ^ Kurien BT, Scofield RH (abril de 2006). "Western blotting". Métodos . 38 (4): 283–93. doi :10.1016/j.ymeth.2005.11.007. PMID 16483794.
- ^ Thomas S, Thirumalapura N, Crossley EC, Ismail N, Walker DH (junio de 2009). "Modificaciones de proteínas antigénicas en Ehrlichia". Inmunología de parásitos . 31 (6): 296–303. doi :10.1111/j.1365-3024.2009.01099.x. PMC 2731653 . PMID 19493209.
- ^ "Microarrays". Centro Nacional de Información Biotecnológica . Biblioteca Nacional de Medicina de EE. UU . . Consultado el 31 de diciembre de 2016 .
- ^ Bumgarner, Roger (2013). "Descripción general de los microarrays de ADN: tipos, aplicaciones y su futuro". Protocolos actuales en biología molecular . 101 : Unidad 22.1. doi :10.1002/0471142727.mb2201s101. PMC 4011503. PMID 23288464 .
- ^ Govindarajan R, Duraiyan J, Kaliyappan K, Palanisamy M (agosto de 2012). "Microarray y sus aplicaciones". Journal of Pharmacy & Bioallied Sciences . 4 (Supl 2): S310-2. doi : 10.4103/0975-7406.100283 . PMC 3467903 . PMID 23066278.
- ^ Tarca AL, Romero R, Draghici S (agosto de 2006). "Análisis de experimentos de microarrays de perfiles de expresión génica". American Journal of Obstetrics and Gynecology . 195 (2): 373–88. doi :10.1016/j.ajog.2006.07.001. PMC 2435252 . PMID 16890548.
- ^ Cheng L, Zhang DY, eds. (2008). Patología genética molecular. Totowa, NJ: Humana. p. 96. ISBN 978-1-59745-405-6. Recuperado el 31 de diciembre de 2016 .
- ^ Leonard DG (2016). Patología molecular en la práctica clínica. Springer. pág. 31. ISBN 978-3-319-19674-9. Recuperado el 31 de diciembre de 2016 .
- ^ Tian J, ed. (2013). Imágenes moleculares: fundamentos y aplicaciones. Springer-Verlag Berlin & Heidelberg GmbH & Co.K. págs. 550, 552. ISBN 9783642343032. Recuperado el 8 de julio de 2019 .
Lectura adicional
- Cohen SN, Chang AC, Boyer HW, Helling RB (noviembre de 1973). "Construcción de plásmidos bacterianos biológicamente funcionales in vitro". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 70 (11): 3240–4. Bibcode :1973PNAS...70.3240C. doi : 10.1073/pnas.70.11.3240 . PMC 427208 . PMID 4594039.
- Rodgers M (junio de 1975). "El congreso de la caja de Pandora". Rolling Stone . Vol. 189. págs. 37–77.
- Roberts K, Raff M, Alberts B, Walter P, Lewis J, Johnson A (2002). Biología molecular de la célula. Garland Science. ISBN 978-0-8153-3218-3.


