Biología estructural
| Parte de una serie sobre |
| Bioquímica |
|---|
 |
La biología estructural , tal como la define el Journal of Structural Biology , se ocupa del análisis estructural del material vivo (formado, compuesto y/o mantenido y refinado por células vivas) en todos los niveles de organización. [1]
Los primeros biólogos estructurales a lo largo del siglo XIX y principios del XX fueron principalmente capaces de estudiar estructuras sólo hasta el límite de la agudeza visual del ojo desnudo y a través de lupas y microscopios ópticos. En el siglo XX, se desarrollaron una variedad de técnicas experimentales para examinar las estructuras 3D de las moléculas biológicas. Las técnicas más destacadas son la cristalografía de rayos X , la resonancia magnética nuclear y la microscopía electrónica . A través del descubrimiento de los rayos X y sus aplicaciones a los cristales de proteínas, la biología estructural se revolucionó, ya que ahora los científicos podían obtener las estructuras tridimensionales de las moléculas biológicas con detalle atómico. [2] Asimismo, la espectroscopia de RMN permitió obtener información sobre la estructura y la dinámica de las proteínas. [3] Finalmente, en el siglo XXI, la microscopía electrónica también experimentó una revolución drástica con el desarrollo de fuentes de electrones más coherentes, corrección de aberraciones para microscopios electrónicos y software de reconstrucción que permitió la implementación exitosa de la criomicroscopía electrónica de alta resolución, permitiendo así el estudio de proteínas individuales y complejos moleculares en tres dimensiones con una resolución de angstroms.
Con el desarrollo de estas tres técnicas, el campo de la biología estructural se expandió y también se convirtió en una rama de la biología molecular , la bioquímica y la biofísica que se ocupa de la estructura molecular de las macromoléculas biológicas (especialmente las proteínas , formadas por aminoácidos , el ARN o ADN , formado por nucleótidos , y las membranas , formadas por lípidos ), cómo adquieren las estructuras que tienen y cómo las alteraciones en sus estructuras afectan su función. [4] Este tema es de gran interés para los biólogos porque las macromoléculas llevan a cabo la mayoría de las funciones de las células , y es solo al enrollarse en formas tridimensionales específicas que son capaces de realizar estas funciones. Esta arquitectura, la " estructura terciaria " de las moléculas, depende de manera complicada de la composición básica de cada molécula, o " estructura primaria ". En resoluciones más bajas, herramientas como la tomografía FIB-SEM han permitido una mayor comprensión de las células y sus orgánulos en tres dimensiones, y cómo cada nivel jerárquico de varias matrices extracelulares contribuye a la función (por ejemplo, en el hueso). En los últimos años también se ha hecho posible predecir modelos moleculares físicos de alta precisión para complementar el estudio experimental de las estructuras biológicas. [5] Las técnicas computacionales como las simulaciones de dinámica molecular se pueden utilizar junto con estrategias de determinación de estructura empírica para ampliar y estudiar la estructura, la conformación y la función de las proteínas. [6]
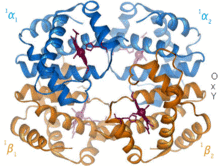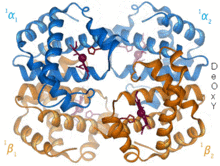

Historia
En 1912 Max Von Laue dirigió rayos X al sulfato de cobre cristalizado generando un patrón de difracción . [7] Estos experimentos llevaron al desarrollo de la cristalografía de rayos X y su uso en la exploración de estructuras biológicas. [5] En 1951, Rosalind Franklin y Maurice Wilkins utilizaron patrones de difracción de rayos X para capturar la primera imagen del ácido desoxirribonucleico (ADN). Francis Crick y James Watson modelaron la estructura de doble hélice del ADN utilizando esta misma técnica en 1953 y recibieron el Premio Nobel de Medicina junto con Wilkins en 1962. [8]
Los cristales de pepsina fueron las primeras proteínas que se cristalizaron para su uso en difracción de rayos X, por Theodore Svedberg, quien recibió el Premio Nobel de Química en 1962. [9] La primera estructura terciaria de la proteína , la de la mioglobina , fue publicada en 1958 por John Kendrew . [10] Durante este tiempo, el modelado de las estructuras de las proteínas se realizó utilizando modelos de madera de balsa o alambre . [11] Con la invención del software de modelado como CCP4 a fines de la década de 1970, [12] el modelado ahora se realiza con asistencia informática. Los desarrollos recientes en el campo han incluido la generación de láseres de electrones libres de rayos X , que permiten el análisis de la dinámica y el movimiento de las moléculas biológicas, [13] y el uso de la biología estructural para ayudar a la biología sintética . [14]
A finales de la década de 1930 y principios de la de 1940, la combinación del trabajo realizado por Isidor Rabi , Felix Bloch y Edward Mills Purcell condujo al desarrollo de la resonancia magnética nuclear (RMN). Actualmente, la RMN de estado sólido se utiliza ampliamente en el campo de la biología estructural para determinar la estructura y la naturaleza dinámica de las proteínas ( RMN de proteínas ). [15]
En 1990, Richard Henderson produjo la primera imagen tridimensional de alta resolución de la bacteriorodopsina utilizando microscopía electrónica criogénica (crio-EM). [16] Desde entonces, la crio-EM ha surgido como una técnica cada vez más popular para determinar estructuras tridimensionales de alta resolución de imágenes biológicas. [17]
Más recientemente, se han desarrollado métodos computacionales para modelar y estudiar estructuras biológicas. Por ejemplo, la dinámica molecular (MD) se utiliza comúnmente para analizar los movimientos dinámicos de las moléculas biológicas. En 1975, la primera simulación de un proceso de plegamiento biológico utilizando MD se publicó en Nature. [18] Recientemente, la predicción de la estructura de las proteínas se mejoró significativamente mediante un nuevo método de aprendizaje automático llamado AlphaFold . [19] Algunos afirman que los enfoques computacionales están comenzando a liderar el campo de la investigación de la biología estructural. [20]
Técnicas
Las biomoléculas son demasiado pequeñas para poder verlas en detalle incluso con los microscopios ópticos más avanzados . Los métodos que utilizan los biólogos estructurales para determinar sus estructuras generalmente implican mediciones de grandes cantidades de moléculas idénticas al mismo tiempo. Estos métodos incluyen:
- Espectrometría de masas
- Cristalografía macromolecular
- Difracción de neutrones
- Proteólisis
- Espectroscopia de resonancia magnética nuclear de proteínas (RMN)
- Resonancia paramagnética electrónica (EPR)
- Microscopía electrónica criogénica (cryoEM)
- Cristalografía electrónica y difracción de electrones en microcristales
- Dispersión de luz multiángulo
- Dispersión de ángulos pequeños
- Espectroscopia láser ultrarrápida
- Microespectroscopia anisotrópica de terahercios
- Espectroscopia infrarroja bidimensional
- Interferometría de polarización dual y dicroísmo circular
La mayoría de las veces, los investigadores los utilizan para estudiar los " estados nativos " de las macromoléculas. Pero también se utilizan variaciones de estos métodos para observar cómo las moléculas nacientes o desnaturalizadas asumen o recuperan sus estados nativos. Véase plegamiento de proteínas .
Un tercer enfoque que los biólogos estructurales adoptan para comprender la estructura es la bioinformática , que consiste en buscar patrones entre las diversas secuencias que dan lugar a formas particulares. Los investigadores a menudo pueden deducir aspectos de la estructura de las proteínas integrales de membrana basándose en la topología de membrana predicha por el análisis de hidrofobicidad . Véase predicción de la estructura de proteínas .
Aplicaciones
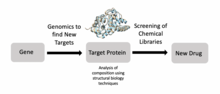
Los biólogos estructurales han hecho contribuciones significativas para comprender los componentes moleculares y los mecanismos subyacentes a las enfermedades humanas. Por ejemplo, la crio-EM y la ssNMR se han utilizado para estudiar la agregación de fibrillas amiloides, que están asociadas con la enfermedad de Alzheimer , la enfermedad de Parkinson y la diabetes tipo II . [21] Además de las proteínas amiloides, los científicos han utilizado la crio-EM para producir modelos de alta resolución de filamentos de tau en el cerebro de pacientes con Alzheimer que pueden ayudar a desarrollar mejores tratamientos en el futuro. [22] Las herramientas de biología estructural también se pueden utilizar para explicar las interacciones entre patógenos y huéspedes. Por ejemplo, las herramientas de biología estructural han permitido a los virólogos comprender cómo la envoltura del VIH permite que el virus evada las respuestas inmunitarias humanas. [23]
La biología estructural también es un componente importante del descubrimiento de fármacos . [24] Los científicos pueden identificar objetivos utilizando la genómica, estudiar esos objetivos utilizando la biología estructural y desarrollar fármacos que sean adecuados para esos objetivos. Específicamente, la RMN de ligando , la espectrometría de masas y la cristalografía de rayos X son técnicas comúnmente utilizadas en el proceso de descubrimiento de fármacos. Por ejemplo, los investigadores han utilizado la biología estructural para comprender mejor Met , una proteína codificada por un protooncogén que es un objetivo farmacológico importante en el cáncer . [25] Se han realizado investigaciones similares para los objetivos del VIH para tratar a las personas con SIDA . [24] Los investigadores también están desarrollando nuevos antimicrobianos para infecciones micobacterianas utilizando el descubrimiento de fármacos impulsado por la estructura. [24]
Véase también
- Estructura primaria
- Estructura secundaria
- Estructura terciaria
- Estructura cuaternaria
- Dominio estructural
- Motivo estructural
- Subunidad proteica
- Modelo molecular
- Cooperatividad
- Chaperonina
- Genómica estructural
- Estereoquímica
- Resolución (densidad electrónica)
- Proteopedia La enciclopedia colaborativa en 3D de proteínas y otras moléculas .
- Predicción de la estructura de las proteínas
Referencias
- ^ "Journal of Structural Biology | ScienceDirect.com de Elsevier". 18 de abril de 2024. Archivado desde el original el 18 de abril de 2024. Consultado el 14 de mayo de 2024 .
{{cite web}}: CS1 maint: bot: original URL status unknown (link) - ^ Curry, Stephen (3 de julio de 2015). "Biología estructural: un viaje de un siglo hacia un mundo invisible". Interdisciplinary Science Reviews . 40 (3): 308–328. Bibcode :2015ISRv...40..308C. doi :10.1179/0308018815Z.000000000120. ISSN 0308-0188. PMC 4697198 . PMID 26740732.
- ^ Campbell, Iain D. (1 de enero de 2013). "La evolución de la RMN de proteínas". Espectroscopia e imágenes biomédicas . 2 (4): 245–264. doi : 10.3233/BSI-130055 . ISSN 2212-8794.
- ^ Banaszak LJ (2000). Fundamentos de la biología estructural . Burlington: Elsevier. ISBN 9780080521848.
- ^ ab Jumper, John; Evans, Richard; Pritzel, Alexander; Green, Tim; Figurnov, Michael; Ronneberger, Olaf; Tunyasuvunakool, Kathryn; Bates, Russ; Žídek, Augustin; Potapenko, Anna; Bridgland, Alex; Meyer, Clemens; Kohl, Simon AA; Ballard, Andrew J.; Cowie, Andrew (15 de julio de 2021). "Predicción de la estructura de proteínas de alta precisión con AlphaFold". Nature . 596 (7873): 583–589. Bibcode :2021Natur.596..583J. doi :10.1038/s41586-021-03819-2. ISSN 1476-4687. PMC 8371605 . PMID 34265844.
- ^ Karplus M, McCammon JA (septiembre de 2002). "Simulaciones de dinámica molecular de biomoléculas". Nature Structural Biology . 9 (9): 646–652. doi :10.1038/nsb0902-646. PMID 12198485. S2CID 590640.
- ^ Curry S (julio de 2015). "Biología estructural: un viaje de un siglo hacia un mundo invisible". Interdisciplinary Science Reviews . 40 (3): 308–328. Bibcode :2015ISRv...40..308C. doi : 10.1179/0308018815z.000000000120 . PMC 4697198 . PMID 26740732.
- ^ "El Premio Nobel de Fisiología o Medicina 1962". NobelPrize.org . Consultado el 1 de octubre de 2022 .
- ^ Jaskolski M, Dauter Z, Wlodawer A (septiembre de 2014). "Una breve historia de la cristalografía macromolecular, ilustrada por un árbol genealógico y sus frutos Nobel". The FEBS Journal . 281 (18): 3985–4009. doi :10.1111/febs.12796. PMC 6309182 . PMID 24698025.
- ^ Kendrew JC, Bodo G, Dintzis HM, Parrish RG, Wyckoff H, Phillips DC (marzo de 1958). "Un modelo tridimensional de la molécula de mioglobina obtenida por análisis de rayos X". Nature . 181 (4610): 662–666. Bibcode :1958Natur.181..662K. doi :10.1038/181662a0. PMID 13517261. S2CID 4162786.
- ^ Garman EF (marzo de 2014). "Avances en la determinación de la estructura cristalográfica de rayos X de macromoléculas biológicas". Science . 343 (6175): 1102–1108. Bibcode :2014Sci...343.1102G. doi :10.1126/science.1247829. PMID 24604194. S2CID 21067016.
- ^ "Acerca de CCP4". legacy.ccp4.ac.uk . Consultado el 2 de abril de 2021 .
- ^ Waldrop MM (enero de 2014). «Ciencia de rayos X: los peces gordos». Nature . 505 (7485): 604–606. Bibcode :2014Natur.505..604W. doi : 10.1038/505604a . PMID 24476872.
- ^ Kiel C, Serrano L (noviembre de 2012). "Datos estructurales en enfoques de biología sintética para estudiar principios generales de diseño de redes de señalización celular". Structure . 20 (11): 1806–1813. doi : 10.1016/j.str.2012.10.002 . hdl : 10230/23121 . PMID 23141693.
- ^ Wüthrich K (noviembre de 2001). "El camino hacia las estructuras de proteínas mediante RMN". Nature Structural Biology . 8 (11): 923–925. doi :10.1038/nsb1101-923. PMID 11685234. S2CID 26153265.
- ^ Henderson R, Baldwin JM, Ceska TA, Zemlin F, Beckmann E, Downing KH (junio de 1990). "Modelo para la estructura de la bacteriorodopsina basado en criomicroscopía electrónica de alta resolución". Journal of Molecular Biology . 213 (4): 899–929. doi :10.1016/S0022-2836(05)80271-2. PMID 2359127.
- ^ Callaway, Ewen (10 de febrero de 2020). "La crio-EM revolucionaria se está apoderando de la biología estructural". Nature . 578 (7794): 201. Bibcode :2020Natur.578..201C. doi : 10.1038/d41586-020-00341-9 . PMID 32047310. S2CID 211081167.
- ^ Levitt M, Warshel A (febrero de 1975). "Simulación por ordenador del plegamiento de proteínas". Nature . 253 (5494): 694–698. Bibcode :1975Natur.253..694L. doi :10.1038/253694a0. PMID 1167625. S2CID 4211714.
- ^ Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, et al. (agosto de 2021). "Predicción de la estructura de proteínas de alta precisión con AlphaFold". Nature . 596 (7873): 583–589. Bibcode :2021Natur.596..583J. doi :10.1038/s41586-021-03819-2. PMC 8371605 . PMID 34265844.
- ^ Nussinov, Ruth; Tsai, Chung-Jung; Shehu, Amarda; Jang, Hyunbum (12 de febrero de 2019). "Biología estructural computacional: éxitos, direcciones futuras y desafíos". Molecules (Basilea, Suiza) . 24 (3): E637. doi : 10.3390/molecules24030637 . ISSN: 1420-3049. PMC: 6384756. PMID : 30759724.
- ^ Iadanza MG, Jackson MP, Hewitt EW, Ranson NA, Radford SE (diciembre de 2018). "Una nueva era para comprender las estructuras amiloides y la enfermedad". Nature Reviews. Molecular Cell Biology . 19 (12): 755–773. doi :10.1038/s41580-018-0060-8. PMID 30237470. S2CID 52307956.
- ^ Fitzpatrick, Anthony WP; Falcón, Benjamín; Él, Shaoda; Murzin, Alexey G.; Murshudov, Garib; Garringer, Holly J.; Crowther, R. Antonio; Ghetti, Bernardino; Goedert, Michel; Scheres, Sjors HW (13 de julio de 2017). "Estructuras crio-EM de filamentos de tau de la enfermedad de Alzheimer". Naturaleza . 547 (7662): 185–190. Código Bib :2017Natur.547..185F. doi : 10.1038/naturaleza23002. ISSN 1476-4687. PMC 5552202 . PMID 28678775.
- ^ Engelman, Alan; Cherepanov, Peter (16 de marzo de 2012). "La biología estructural del VIH-1: perspectivas mecanicistas y terapéuticas". Nature Reviews Microbiology . 10 (4): 279–290. doi : 10.1038/nrmicro2747 . ISSN 1740-1526. PMC 3588166 . PMID 22421880. S2CID 14088805.
- ^ abc Thomas SE, Mendes V, Kim SY, Malhotra S, Ochoa-Montaño B, Blaszczyk M, Blundell TL (agosto de 2017). "Biología estructural y diseño de nuevas terapias: desde el VIH y el cáncer hasta las infecciones por micobacterias: un artículo dedicado a John Kendrew". Journal of Molecular Biology . Edición especial del centenario de John Kendrew. 429 (17): 2677–2693. doi : 10.1016/j.jmb.2017.06.014 . PMID 28648615.
- ^ Wendt KU, Weiss MS, Cramer P, Heinz DW (febrero de 2008). "Estructuras y enfermedades". Nature Structural & Molecular Biology . 15 (2): 117–120. doi :10.1038/nsmb0208-117. PMC 7097323 . PMID 18250627.
Enlaces externos
 Medios relacionados con Biología estructural en Wikimedia Commons
Medios relacionados con Biología estructural en Wikimedia Commons- Sitio web de la revista Nature: Structural & Molecular Biology
- Revista de Biología Estructural
- Biología estructural - La biblioteca virtual de bioquímica, biología molecular y biología celular
- Biología estructural en Europa
- Aprendiendo Cristalografía



