Acil-CoA tioesterasa 9
| ACOT9 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Alias | ACOT9 , ACATE2, MT-ACT48, MTACT48, CGI-16, acil-CoA tioesterasa 9 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificaciones externas | OMIM : 300862; MGI : 1928939; HomoloGene : 8206; Tarjetas genéticas : ACOT9; OMA :ACOT9 - ortólogos | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidatos | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
La acil-CoA tioesterasa 9 es una proteína codificada por el gen humano ACOT9 . Es miembro de la superfamilia de las acil-CoA tioesterasas , que es un grupo de enzimas que hidrolizan los ésteres de la coenzima A. No se conoce su función, pero se ha demostrado que actúa como una tioesterasa de cadena larga en concentraciones bajas y como una tioesterasa de cadena corta en concentraciones altas. [5]
Gene

Lugar
El gen ACOT9 está ubicado en p22.11 en el cromosoma X. Ubicado en la cadena negativa del cromosoma, el inicio está en 23.721.777 pb y el final está en 23.761.407 pb, lo que supone una extensión de 39.631 pares de bases . [6]

Alias
El gen ACOT9 es conocido principalmente por codificar la proteína acil-CoA tioesterasa 9. Otros nombres menos utilizados para el gen son ACATE2, [7] y MT-ACT48. [8]
Función
La proteína codificada por el gen ACOT9 forma parte de una familia de tioesterasas de acil-CoA , que catalizan la hidrólisis de diversos ésteres de coenzima A de diversas moléculas hasta el ácido libre más CoA. Estas enzimas también se han denominado en la literatura como acil-CoA hidrolasas, acil-CoA tioéster hidrolasas e palmitoil-CoA hidrolasas. La reacción que llevan a cabo estas enzimas es la siguiente:
Éster de CoA + H2O → ácido libre + coenzima A
Estas enzimas utilizan los mismos sustratos que las acil-CoA sintetasas de cadena larga, pero tienen un propósito único en el sentido de que generan el ácido libre y CoA, a diferencia de las acil-CoA sintetasas de cadena larga, que ligan los ácidos grasos a CoA, para producir el éster de CoA. [9] El papel de la familia de enzimas ACOT no se entiende bien; sin embargo, se ha sugerido que desempeñan un papel crucial en la regulación de los niveles intracelulares de ésteres de CoA, coenzima A y ácidos grasos libres. Estudios recientes han demostrado que los ésteres de acil-CoA tienen muchas más funciones que simplemente una fuente de energía. Estas funciones incluyen la regulación alostérica de enzimas como la acetil-CoA carboxilasa , [10] la hexoquinasa IV, [11] y la enzima condensadora de citrato. Las acil-CoA de cadena larga también regulan la apertura de los canales de potasio sensibles al ATP y la activación de las ATPasas de calcio , regulando así la secreción de insulina . [12] Una serie de otros eventos celulares también son mediados por acil-CoAs, por ejemplo la transducción de señales a través de la proteína quinasa C , inhibición de la apoptosis inducida por ácido retinoico , y participación en la gemación y fusión del sistema de endomembrana . [13] [14] [15] Los acil-CoAs también median la orientación de proteínas a varias membranas y la regulación de las subunidades α de la proteína G, porque son sustratos para la acilación de proteínas. [16] En las mitocondrias , los ésteres de acil-CoA están involucrados en la acilación de las deshidrogenasas dependientes de NAD+ mitocondriales ; debido a que estas enzimas son responsables del catabolismo de aminoácidos , esta acilación hace que todo el proceso sea inactivo. Este mecanismo puede proporcionar una diafonía metabólica y actuar para regular la relación NADH /NAD+ con el fin de mantener una oxidación beta mitocondrial óptima de los ácidos grasos. [17] El papel de los ésteres de CoA en el metabolismo de los lípidos y otros numerosos procesos intracelulares está bien definido, y por lo tanto se plantea la hipótesis de que las enzimas ACOT desempeñan un papel en la modulación de los procesos en los que participan estos metabolitos. [18]
Homología/Evolución

Ortólogos
Existen muchos ortólogos de ACOT9, siendo el ratón doméstico ( Mus musculus ) uno de los más similares, donde el gen ACOT9 se encuentra a 72,38 cM en el cromosoma X. [19] La gama de ortólogos se extiende a mamíferos, aves, anfibios, hongos anamórficos y otros. [ cita requerida ]
| Número de secuencia | Género y especie | Nombre común | Fecha de divergencia (MYA) | Número de acceso | Longitud de la secuencia | Identidad de secuencia | Similitud de secuencia | Notas |
|---|---|---|---|---|---|---|---|---|
| 1 | Homo sapiens | Humano | 0 | NP_001028755.2 | 439 | 100% | 100% | Humano |
| 2 | Músculo musculoso | Ratón doméstico | 91 | NP_062710.2 | 439 | 83% | 90% | Roedor |
| 3 | Pteropus alecto | Zorro volador negro | 97.4 | XP_006911668.1 | 480 | 81% | 91% | Murciélago |
| 4 | Gallo gallo | Pollo | 324,5 | NP_001012841.1 | 425 | 69% | 87% | Pájaro |
| 5 | Pseudopodoces humilis | Carbonero terrestre | 324,5 | XP_005516751.1 | 417 | 68% | 85% | Pájaro |
| 6 | Columba livia | Paloma bravía | 324,5 | XP_005503782.1 | 402 | 67% | 86% | Pájaro |
| 7 | Geospiza fortis | Pinzón terrestre mediano | 324,5 | XP_005424946.1 | 417 | 67% | 85% | Pájaro |
| 8 | Pelodiscus sinensis | Tortuga china de caparazón blando | 324,5 | XP_006112565.1 | 439 | 67% | 85% | Reptil |
| 9 | Xenopus tropicalis | Rana de garras occidental | 361.2 | AAI61600.1 | 418 | 65% | 82% | Anfibio |
| 10 | Danio rerio | Pez cebra | 454.6 | AAI59216.1 | 434 | 60% | 80% | Pez |
| 11 | Ceratitis capitata | Mosca de la fruta del Mediterráneo | 910 | JAB97119.1 | 433 | 32% | 58% | Insecto |
| 12 | Glarea lozoyensis 74030 | Hongo anamórfico | 1368 | EHL00310.1 | 350 | 24% | 47% | Hongo |

Parálogos
En ratones, que es uno de los ortólogos más cercanos, ACOT10 es un parálogo conocido del gen ACOT9. [20]
Expresión
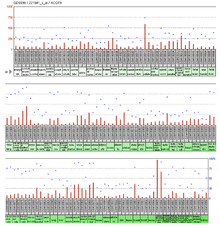
La expresión de ACOT9 es ubicua en todos los tejidos de los seres humanos. Los tejidos con un valor de más de 500 en el análisis a gran escala del transcriptoma humano fueron el globo pálido y el adenocarcinoma colorrectal. [21] El perfil de abundancia de la etiqueta de secuencia expresada (o EST) también muestra una expresión ubicua /casi ubicua en todos los tejidos humanos. [22]
Factores de transcripción
Existen numerosos factores de transcripción en toda la secuencia promotora de ACOT9. Algunos de los factores más destacados son los factores de choque térmico y los elementos de reconocimiento del factor de transcripción II B (TFIIB). [ cita requerida ]
| Factor de transcripción | Comenzar | Fin | Hebra | Secuencia |
|---|---|---|---|---|
| Elemento promotor central 1 del gen X | 683 | 693 | - | ggGCGGgaccg |
| Factor de transcripción 1 relacionado con Doublesex y mab-3 | 81 | 101 | + | ttttttgagacaTTGTctcc |
| Proteína 1 de unión al elemento sensible al AMPc | 491 | 511 | - | agggcgTGACgtcgagaagag |
| Factor de transcripción Sp4 | 660 | 676 | - | ccaggggGGCGtggccgc |
| Proteína estimulante 1, factor de transcripción de dedo de zinc ubicuo | 682 | 698 | - | tccggGGGCgggaccgc |
| Factor de choque térmico 1 | 24 | 48 | + | caggactaaactAGAAtctccagcc |
| Factor de transcripción E2F 2 | 808 | 824 | + | ccatcGCGCgcacggca |
| Factor nuclear de células T activadas 5 | 380 | 398 | + | ttGGAAagttgcccagga |
| Dominio POZ ZF5, dedo de zinc, proteína de dedo de zinc 161 (preferencia de unión al ADN secundario) | 811 | 825 | + | tcgCGCGcacggcag |
| Proteína activadora específica de células B | 678 | 706 | - | cagcggtgtccgggGGCGggaccgcggcg |
| Sitio de unión del dominio emparejado Pax-6 | 54 | 72 | + | gtctcAAGCatcagttttt |
| Dominio POZ ZF5, dedo de zinc, proteína de dedo de zinc 161 (preferencia de unión al ADN secundario) | 651 | 665 | - | ggcCGCGctgtgccg |
| Sitio de unión del dominio emparejado Pax-6 | 758 | 776 | + | ttttaTCGCctcagtttcc |
| Caja TATA LTR tipo C de mamífero | 751 | 767 | - | ggcgaTAAAagacgcac |
| Factor nuclear Y (factor de unión a la caja Y) | 624 | 638 | + | cccgCCAAtgaacgg |
| Elemento de reconocimiento del factor de transcripción II B (TFIIB) | 356 | 362 | + | ccgCGCC |
| Elemento de reconocimiento del factor de transcripción II B (TFIIB) | 440 | 446 | - | ccgCGCC |
| Elemento de reconocimiento del factor de transcripción II B (TFIIB) | 734 | 740 | - | ccgCGCC |
| Factor nuclear Y (factor de unión a la caja Y) | 581 | 595 | - | ccacTCAAtcagttg |
| Proteína de unión al potenciador/CCAAT alfa | 529 | 543 | - | tcggttgaGTAAacg |
Estructura secundaria
Hay dos regiones en la secuencia del gen ACOT9 que están etiquetadas como regiones BFIT (Brown Fat Inducible Thioesterase) y BACH (Brain Acyl CoA Hydrolase). Estas regiones son parte de una superfamilia de pliegues HotDog , que se ha descubierto que se utiliza en una variedad de funciones celulares. [23] Las predicciones muestran que hay varias hélices alfa en toda la estructura, [24] lo que sugiere que es una proteína transmembrana .
Interacciones
Se puede encontrar un sitio de escisión mitocondrial en el aminoácido 30 en la secuencia ACOT9, y la probabilidad de exportación a las mitocondrias es de 0,9374. [25] Se estima que la proteína acil-CoA tioesterasa 9 es 60,9% mitocondrial, 21,7% citoplasmática , 8,7% nuclear, 4,3% en la membrana plasmática y 4,3% en el retículo endoplásmico . [26]
Se ha descubierto que la proteína ACOT9 interactúa con las siguientes proteínas ya sea experimentalmente o mediante coexpresión: [27]
- C1orf151
- MCTS1
- C1GALT1C1
- FBXW12
- Límite de remanente
- RPS6KA3
- RAB9A
- Universidad de Columbia Británica
- CXorf26
- HCCS
Referencias
- ^ abc GRCh38: Lanzamiento de Ensembl 89: ENSG00000123130 – Ensembl , mayo de 2017
- ^ abc GRCm38: Lanzamiento de Ensembl 89: ENSMUSG00000025287 – Ensembl , mayo de 2017
- ^ "Referencia de PubMed humana:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ "Referencia de PubMed sobre ratón". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ Tillander V, Arvidsson Nordström E, Reilly J, Strozyk M, Van Veldhoven PP, Hunt MC, Alexson SE (marzo de 2014). "La acil-CoA tioesterasa 9 (ACOT9) en ratones puede proporcionar un nuevo vínculo entre el metabolismo de los ácidos grasos y los aminoácidos en las mitocondrias". Ciencias de la vida celular y molecular . 71 (5): 933–48. doi :10.1007/s00018-013-1422-1. hdl : 10616/41794 . PMID 23864032. S2CID 18767370.
- ^ Kent WJ, Sugnet CW, Furey TS, Roskin KM, Pringle TH, Zahler AM, Haussler D (12 de junio de 2002). "Human Feb. 2009 (GRCh37/hg19) Assembly". El explorador del genoma humano en UCSC . UCSC Genome Bioinformatics . Consultado el 12 de marzo de 2014 .
- ^ Gu J, MacHugh DE, McGivney BA, Park SD, Katz LM, Hill EW (noviembre de 2010). "Asociación de variantes de secuencia en los genes CKM (creatina quinasa, músculo) y COX4I2 (citocromo c oxidasa, subunidad 4, isoforma 2) con el rendimiento en carreras de caballos pura sangre". Revista veterinaria equina. Suplemento . 42 (38): 569–75. doi : 10.1111/j.2042-3306.2010.00181.x . PMID 21059062.
- ^ Poupon V, Bègue B, Gagnon J, Dautry-Varsat A, Cerf-Bensussan N, Benmerah A (julio de 1999). "Clonación molecular y caracterización de MT-ACT48, una nueva acil-CoA tioesterasa mitocondrial". The Journal of Biological Chemistry . 274 (27): 19188–94. doi : 10.1074/jbc.274.27.19188 . PMID 10383425.
- ^ Mashek DG, Bornfeldt KE, Coleman RA, Berger J, Bernlohr DA, Black P, DiRusso CC, Farber SA, Guo W, Hashimoto N, Khodiyar V, Kuypers FA, Maltais LJ, Nebert DW, Renieri A, Schaffer JE, Stahl A, Watkins PA, Vasiliou V, Yamamoto TT (octubre de 2004). "Nomenclatura revisada para la familia de genes de acil-CoA sintetasa de cadena larga de mamíferos". Revista de investigación de lípidos . 45 (10): 1958–61. doi : 10.1194/jlr.e400002-jlr200 . PMID 15292367.
- ^ Ogiwara H, Tanabe T, Nikawa J, Numa S (agosto de 1978). "Inhibición de la acetil-coenzima-A carboxilasa de hígado de rata por palmitoil-coenzima A. Formación de un complejo enzima-inhibidor equimolar". Revista Europea de Bioquímica . 89 (1): 33–41. doi :10.1111/j.1432-1033.1978.tb20893.x. PMID 29756.
- ^ Srere PA (diciembre de 1965). "Inhibición de la enzima condensadora de citrato por palmitil-coenzima A". Biochimica et Biophysica Acta (BBA) - Lípidos y metabolismo lipídico . 106 (3): 445–55. doi :10.1016/0005-2760(65)90061-5. PMID 5881327.
- ^ Gribble FM, Proks P, Corkey BE, Ashcroft FM (octubre de 1998). "Mecanismo de activación del canal de potasio sensible a ATP clonado por oleoil-CoA". The Journal of Biological Chemistry . 273 (41): 26383–7. doi : 10.1074/jbc.273.41.26383 . PMID 9756869.
- ^ Nishizuka Y (abril de 1995). "Proteína quinasa C y señalización lipídica para respuestas celulares sostenidas". FASEB Journal . 9 (7): 484–96. doi : 10.1096/fasebj.9.7.7737456 . PMID 7737456. S2CID 31065063.
- ^ Glick BS, Rothman JE (1987). "Posible función de la acil-coenzima A grasa en el transporte intracelular de proteínas". Nature . 326 (6110): 309–12. Bibcode :1987Natur.326..309G. doi :10.1038/326309a0. PMID 3821906. S2CID 4306469.
- ^ Wan YJ, Cai Y, Cowan C, Magee TR (junio de 2000). "Los acil-CoA grasos inhiben la apoptosis inducida por ácido retinoico en células Hep3B". Cancer Letters . 154 (1): 19–27. doi :10.1016/s0304-3835(00)00341-4. PMID 10799735.
- ^ Duncan JA, Gilman AG (junio de 1998). "Una tioesterasa de acil-proteína citoplasmática que elimina el palmitato de las subunidades alfa de la proteína G y p21(RAS)". The Journal of Biological Chemistry . 273 (25): 15830–7. doi : 10.1074/jbc.273.25.15830 . PMID 9624183.
- ^ Berthiaume L, Deichaite I, Peseckis S, Resh MD (marzo de 1994). "Regulación de la actividad enzimática mediante la acilación de ácidos grasos del sitio activo. Un nuevo papel para la acilación de proteínas con ácidos grasos de cadena larga". The Journal of Biological Chemistry . 269 (9): 6498–505. doi : 10.1016/S0021-9258(17)37399-4 . PMID 8120000.
- ^ Hunt MC, Alexson SE (marzo de 2002). "El papel que desempeñan las tioesterasas de acil-CoA en la mediación del metabolismo lipídico intracelular". Progress in Lipid Research . 41 (2): 99–130. doi :10.1016/s0163-7827(01)00017-0. PMID 11755680.
- ^ "Detalle del gen ACOT9". Base de datos del genoma del ratón . Consultado el 19 de junio de 2014 .
- ^ "Gene: Acot9". Lanzamiento de Ensembl 75.
- ^ "Análisis a gran escala del transcriptoma humano (HG-U133A)". Centro Nacional de Información Biotecnológica . Consultado el 10 de mayo de 2014 .
- ^ "EST Profile Hs.298885 - ACOT9: Acyl-CoA tioesterase 9" (Perfil EST Hs.298885 - ACOT9: Acil-CoA tioesterasa 9) . Consultado el 10 de mayo de 2014 .
- ^ Dillon SC, Bateman A (agosto de 2004). "El pliegue Hotdog: envolviendo una superfamilia de tioesterasas y deshidratasas". BMC Bioinformatics . 5 : 109. doi : 10.1186/1471-2105-5-109 . PMC 516016 . PMID 15307895.
- ^ "Programa Pele del Banco de trabajo de biología SDSC 3.2". [ enlace muerto ]
- ^ Claros MG, Vincens P (noviembre de 1996). "Método computacional para predecir proteínas importadas mitocondrialmente y sus secuencias de destino". Revista Europea de Bioquímica . 241 (3): 779–86. doi : 10.1111/j.1432-1033.1996.00779.x . PMID 8944766.
- ^ "Herramienta de predicción PSORTII".[ verificación necesaria ]
- ^ Jensen LJ, Kuhn M, Stark M, Chaffron S, Creevey C, Muller J, Doerks T, Julien P, Roth A, Simonovic M, Bork P, von Mering C (enero de 2009). "STRING 8: una visión global de las proteínas y sus interacciones funcionales en 630 organismos". Nucleic Acids Research . 37 (número de la base de datos): D412–6. doi :10.1093/nar/gkn760. PMC 2686466 . PMID 18940858.
Enlaces externos
- Página de detalles del gen ACOT9 y ubicación del genoma humano ACOT9 en el navegador de genoma de la UCSC .
Lectura adicional
- Mulkearns EE, Cooper JA (abril de 2012). "El dominio FCH-2 organiza estructuras recubiertas de clatrina e interactúa con Disabled-2 para la endocitosis del receptor de lipoproteína de baja densidad". Biología molecular de la célula . 23 (7): 1330–42. doi :10.1091/mbc.E11-09-0812. PMC 3315808 . PMID 22323290.
- Hunt MC, Yamada J, Maltais LJ, Wright MW, Podesta EJ, Alexson SE (septiembre de 2005). "Una nomenclatura revisada para acil-CoA tioesterasas / hidrolasas de mamíferos". Revista de investigación de lípidos . 46 (9): 2029–32. doi : 10.1194/jlr.E500003-JLR200 . PMID 16103133.
- Alkhaja AK, Jans DC, Nikolov M, Vukotic M, Lytovchenko O, Ludewig F, Schliebs W, Riedel D, Urlaub H, Jakobs S, Deckers M (enero de 2012). "MINOS1 es un componente conservado de los complejos de mitofilina y es necesario para la función mitocondrial y la organización de las crestas" (PDF) . Biología Molecular de la Célula . 23 (2): 247–57. doi :10.1091/mbc.E11-09-0774. PMC 3258170 . PMID 22114354.
- Poupon V, Bègue B, Gagnon J, Dautry-Varsat A, Cerf-Bensussan N, Benmerah A (julio de 1999). "Clonación molecular y caracterización de MT-ACT48, una nueva acil-CoA tioesterasa mitocondrial". The Journal of Biological Chemistry . 274 (27): 19188–94. doi : 10.1074/jbc.274.27.19188 . PMID 10383425.
Este artículo incorpora texto de la Biblioteca Nacional de Medicina de los Estados Unidos , que se encuentra en el dominio público .





