Tiorredoxina reductasa
| Tiorredoxina reductasa | |
|---|---|
| Identificadores | |
| Símbolo | ? |
| Interprofesional | IPR005982 |
| PROSITIO | PS00573 |
| SCOP2 | 1zof / ALCANCE / SUPFAM |
Las tiorredoxinas reductasas ( TR , TrxR ) ( EC 1.8.1.9) son enzimas que reducen la tiorredoxina (Trx). [1] Se han identificado dos clases de tiorredoxina reductasa: una clase en bacterias y algunos eucariotas y otra en animales. En bacterias, TrxR también cataliza la reducción de proteínas similares a glutaredoxina conocidas como NrdH. [2] [3] [4] Ambas clases son flavoproteínas que funcionan como homodímeros. Cada monómero contiene un grupo prostético FAD , un dominio de unión a NADPH y un sitio activo que contiene un enlace disulfuro redox-activo . [5]
Papel celular
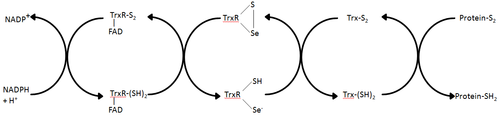
Las tiorredoxinas reductasas son enzimas que catalizan la reducción de la tiorredoxina [1] y, por lo tanto, son un componente central en el sistema de la tiorredoxina. Junto con la tiorredoxina (Trx) y el NADPH, la descripción más general de este sistema es como un sistema para reducir los enlaces disulfuro en las células. Los electrones se toman del NADPH a través de TrxR y se transfieren al sitio activo de Trx, que continúa reduciendo los disulfuros de proteínas u otros sustratos. [6] El sistema Trx existe en todas las células vivas y tiene una historia evolutiva vinculada al ADN como material genético, la defensa contra el daño oxidativo debido al metabolismo del oxígeno y la señalización redox utilizando moléculas como el peróxido de hidrógeno y el óxido nítrico. [7] [8]
Diversidad
Han evolucionado independientemente dos clases de tiorredoxina reductasa:
- Se ha identificado un tipo de TxR de alto peso molecular (PM = ~55.000) que contiene un residuo de selenocisteína en su sitio activo en eucariotas superiores, incluidos los humanos. Este TxR está relacionado con la glutatión reductasa , la tripanotiona reductasa , la reductasa mercúrica y la lipoamida deshidrogenasa . [5]
- Se ha identificado un tipo de bajo peso molecular (PM = ~ 35.000) en arqueas, bacterias y otros eucariotas. [5]
Estas dos clases de TrxR tienen solo un ~20% de identidad de secuencia en la sección de la secuencia primaria donde pueden alinearse de manera confiable. [5] La reacción neta de ambas clases de TrxR es idéntica, pero el mecanismo de acción de cada una es distinto. [9]
Los humanos expresan tres isoenzimas de tiorredoxina reductasa: tiorredoxina reductasa 1 (TrxR1, citosólica), tiorredoxina reductasa 2 (TrxR2, mitocondrial) y tiorredoxina reductasa 3 (TrxR3, específica de los testículos). [10] Cada isoenzima está codificada por un gen independiente:
|
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Estructura
E. coli
En E. coli ThxR hay dos dominios de unión, uno para FAD y otro para NADPH . La conexión entre estos dos dominios es una hoja β antiparalela de dos cadenas . [11] Cada dominio individualmente es muy similar a los dominios análogos en glutatión reductasa y lipoamida deshidrogenasa pero la orientación relativa de estos dominios en ThxR está rotada 66 grados. [11] Esto se vuelve significativo en el mecanismo de acción de la enzima que se describe a continuación. ThxR homodimeriza con la interfaz entre los dos monómeros formada por tres hélices alfa y dos bucles. [11] Cada monómero puede unirse por separado a una molécula de tiorredoxina .
- Estructura del dímero ThxR de E. coli unido a la tiorredoxina
- Estructura de ThxR de E. coli con grupos prostéticos FAD y NADPH marcados
Mamífero
La estructura del TrxR de mamíferos es similar a la de E. coli . Contiene un dominio de unión de FAD y NADPH , y una interfaz entre dos subunidades monoméricas. En el ThxR de mamíferos hay una inserción en el dominio de unión de FAD entre dos hélices alfa que forma un pequeño par de cadenas beta. [12] El disulfuro activo en la enzima se encuentra en una de estas hélices y, por lo tanto, el enlace disulfuro activo se encuentra en el dominio FAD y no en el dominio NADPH como en E. coli y otros procariotas . [12]
- Estructura de los grupos prostéticos ThxR FAD y NADPH humanos
Mecanismo

E. coli
En E. coli ThxR, la orientación espacial de los dominios FAD y NADPH es tal que los anillos redox-activos de FAD y NADPH no están muy cerca uno del otro. [1] Cuando el dominio FAD de E. coli se gira 66 grados y el dominio NADPH permanece fijo, los dos grupos prostéticos se mueven a un estrecho contacto, lo que permite que los electrones pasen del NADPH al FAD y luego al enlace disulfuro del sitio activo. [1] [15] Los residuos del sitio activo conservados en E. coli son -Cys-Ala-Thr-Cys-. [1]
Mamífero
Los TrxR de mamíferos tienen una homología de secuencia mucho mayor con la glutatión reductasa que E. coli . [1] Los residuos de Cys del sitio activo en el dominio FAD y el dominio NADPH unido están muy próximos, lo que elimina la necesidad de una rotación de 66 grados para la transferencia de electrones que se encuentra en E. coli . Una característica adicional del mecanismo de los mamíferos es la presencia de un residuo de selenocisteína en el extremo C-terminal de la proteína que es necesario para la actividad catalítica. Los residuos conservados en el sitio activo de los mamíferos son -Cys-Val-Asn-Val-Gly-Cys-. [1]
Métodos de detección
La tiorredoxina reductasa se puede cuantificar mediante varios métodos, como el ensayo DTNB utilizando el reactivo de Ellman . La serie TRFS de sondas fluorescentes basadas en disulfuro ha demostrado la detección selectiva de TrxR. [16] [17] [18] [19] Mafireyi sintetizó la primera sonda de diseleniuro que se aplicó en la detección de TrxR. [20] [21] Otros métodos de detección incluyen técnicas inmunológicas y el ensayo de selenocistina-tiorredoxina reductasa (ensayo SC-TR).
Importancia clínica
Tratamiento del cáncer
Dado que la actividad de esta enzima es esencial para el crecimiento y la supervivencia celular, es un buen objetivo para la terapia antitumoral. Además, la enzima se regula positivamente en varios tipos de cáncer, incluido el mesotelioma maligno . [22] [23] Por ejemplo, la motexafina gadolinio (MGd) es un nuevo agente quimioterapéutico que se dirige selectivamente a las células tumorales, lo que conduce a la muerte celular y la apoptosis a través de la inhibición de la tiorredoxina reductasa y la ribonucleótido reductasa .
Miocardiopatía
La miocardiopatía dilatada ( MCD ) es un diagnóstico común en casos de insuficiencia cardíaca congestiva . Las tiorredoxinas reductasas son proteínas esenciales para regular el equilibrio redox celular y mitigar el daño causado por las especies reactivas de oxígeno generadas a través de la fosforilación oxidativa en las mitocondrias . La inactivación de TrxR2 mitocondrial en ratones da como resultado un adelgazamiento de las paredes del corazón ventricular y la muerte neonatal. [10] Además, se encuentran dos mutaciones en el gen TrxR2 en pacientes diagnosticados con MCD y no en una población de control. Se plantea la hipótesis de que el impacto patológico de estas mutaciones es una capacidad deteriorada para controlar el daño oxidativo en los miocitos cardíacos . [24]
Antibiótico
Recientemente se han realizado algunas investigaciones que demuestran que la tiorredoxina reductasa de bajo peso molecular podría ser un objetivo para nuevos antibióticos (como auranofina o Ebselen. [25] ) Esto es especialmente cierto para Mycobacterium Haemophilum y podría usarse para bacterias resistentes a los antibióticos. [26]
Referencias
- ^ abcdefg Mustacich D, Powis G (febrero de 2000). "Tioredoxina reductasa". La revista bioquímica . 346 Parte 1 (1): 1–8. doi :10.1042/0264-6021:3460001. PMC 1220815 . PMID 10657232.
- ^ Jordan A, Aslund F, Pontis E, Reichard P, Holmgren A (julio de 1997). "Caracterización de NrdH de Escherichia coli. Una proteína similar a la glutaredoxina con un perfil de actividad similar a la tiorredoxina". The Journal of Biological Chemistry . 272 (29): 18044–50. doi : 10.1074/jbc.272.29.18044 . PMID 9218434.
- ^ Phulera S, Mande SC (junio de 2013). "La estructura cristalina de NrdH de Mycobacterium tuberculosis a 0,87 Å sugiere un posible modo de su actividad". Bioquímica . 52 (23): 4056–65. doi :10.1021/bi400191z. PMID 23675692.
- ^ Phulera S, Akif M, Sardesai AA, Mande SC (1 de enero de 2014). "Proteínas redox de Mycobacterium tuberculosis". Revista del Instituto Indio de Ciencias . 94 (1): 127–138. ISSN 0970-4140.
- ^ abcd Hirt RP, Müller S, Embley TM, Coombs GH (julio de 2002). "La diversidad y evolución de la tiorredoxina reductasa: nuevas perspectivas". Tendencias en parasitología . 18 (7): 302–8. doi :10.1016/S1471-4922(02)02293-6. PMID 12379950.
- ^ ab Holmgren A, Lu J (mayo de 2010). "Tiorredoxina y tiorredoxina reductasa: investigación actual con especial referencia a las enfermedades humanas". Comunicaciones de investigación bioquímica y biofísica . 396 (1): 120–4. doi :10.1016/j.bbrc.2010.03.083. PMID 20494123.
- ^ Meyer Y, Buchanan BB, Vignols F, Reichheld JP (2009). "Tiorredoxinas y glutaredoxinas: elementos unificadores en biología redox". Revisión anual de genética . 43 : 335–67. doi :10.1146/annurev-genet-102108-134201. PMID 19691428.
- ^ Lillig CH, Holmgren A (enero de 2007). "Tiorredoxina y moléculas relacionadas: de la biología a la salud y la enfermedad". Antioxidantes y señalización redox . 9 (1): 25–47. doi :10.1089/ars.2007.9.25. PMID 17115886.
- ^ Arscott LD, Gromer S, Schirmer RH, Becker K, Williams CH (abril de 1997). "El mecanismo de la tiorredoxina reductasa de la placenta humana es similar a los mecanismos de la lipoamida deshidrogenasa y la glutatión reductasa y es distinto del mecanismo de la tiorredoxina reductasa de Escherichia coli". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 94 (8): 3621–6. Bibcode :1997PNAS...94.3621A. doi : 10.1073/pnas.94.8.3621 . PMC 20490 . PMID 9108027.
- ^ ab Conrad M, Jakupoglu C, Moreno SG, Lippl S, Banjac A, Schneider M, Beck H, Hatzopoulos AK, Just U, Sinowatz F, Schmahl W, Chien KR, Wurst W, Bornkamm GW, Brielmeier M (noviembre de 2004). "Función esencial de la tiorredoxina reductasa mitocondrial en la hematopoyesis, el desarrollo del corazón y la función cardíaca". Biología molecular y celular . 24 (21): 9414–23. doi :10.1128/MCB.24.21.9414-9423.2004. PMC 522221 . PMID 15485910.
- ^ abc Williams CH (octubre de 1995). "Mecanismo y estructura de la tiorredoxina reductasa de Escherichia coli". FASEB Journal . 9 (13): 1267–76. doi : 10.1096/fasebj.9.13.7557016 . hdl : 2027.42/154540 . PMID 7557016. S2CID 26055087.
- ^ ab Sandalova T, Zhong L, Lindqvist Y, Holmgren A, Schneider G (agosto de 2001). "Estructura tridimensional de una tiorredoxina reductasa de mamíferos: implicaciones para el mecanismo y la evolución de una enzima dependiente de selenocisteína". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 98 (17): 9533–8. Bibcode :2001PNAS...98.9533S. doi : 10.1073/pnas.171178698 . PMC 55487 . PMID 11481439.
- ^ Zhong L, Arnér ES, Holmgren A (mayo de 2000). "Estructura y mecanismo de la tiorredoxina reductasa de mamíferos: el sitio activo es un selenoltiol/seleniolsulfuro redox-activo formado a partir de la secuencia cisteína-selenocisteína conservada". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 97 (11): 5854–9. Bibcode :2000PNAS...97.5854Z. doi : 10.1073/pnas.100114897 . PMC 18523 . PMID 10801974.
- ^ Becker K, Herold-Mende C, Park JJ, Lowe G, Schirmer RH (agosto de 2001). "La tiorredoxina reductasa humana es inhibida eficazmente por complejos de (2,2':6',2' '-terpiridina)platino(II). Posibles implicaciones para una nueva estrategia antitumoral". Journal of Medicinal Chemistry . 44 (17): 2784–92. doi :10.1021/jm001014i. PMID 11495589.
- ^ Lennon BW, Williams CH (agosto de 1997). "Semirreacción reductora de la tiorredoxina reductasa de Escherichia coli". Bioquímica . 36 (31): 9464–77. doi :10.1021/bi970307j. PMID 9235991.
- ^ Li X, Zhang B, Yan C, Li J, Wang S, Wei X, et al. (junio de 2019). "Una sonda fluorescente rápida y específica para la tiorredoxina reductasa que funciona a través de la escisión del enlace disulfuro". Nature Communications . 10 (1): 2745. Bibcode :2019NatCo..10.2745L. doi : 10.1038/s41467-019-10807-8 . PMC 6588570 . PMID 31227705.
- ^ Ma H, Zhang J, Zhang Z, Liu Y, Fang J (octubre de 2016). "Una sonda de respuesta rápida y emisión roja para la tiorredoxina reductasa de mamíferos". Chemical Communications . 52 (81): 12060–12063. doi :10.1039/C6CC04984B. PMID 27709154.
- ^ Zhao J, Qu Y, Gao H, Zhong M, Li X, Zhang F, et al. (noviembre de 2020). "Pérdida de la función de la tiorredoxina reductasa en un modelo de accidente cerebrovascular en ratones revelada por una sonda fluorescente de dos fotones". Chemical Communications . 56 (90): 14075–14078. doi :10.1039/D0CC05900E. PMID 33107534. S2CID 225082279.
- ^ Liu Y, Ma H, Zhang L, Cui Y, Liu X, Fang J (febrero de 2016). "Una sonda de molécula pequeña revela una disminución de la actividad de la tiorredoxina reductasa mitocondrial en un modelo de enfermedad de Parkinson". Chemical Communications . 52 (11): 2296–9. doi :10.1039/c5cc09998f. PMID 26725656.
- ^ Mafireyi TJ, Laws M, Bassett JW, Cassidy PB, Escobedo JO, Strongin RM (agosto de 2020). "Una sonda fluorescente de activación de diselenida para la detección de tiorredoxina reductasa". Angewandte Chemie . 59 (35): 15147–15151. Bibcode :2020AngCh.13215259M. doi :10.1002/ange.202004094. PMC 9438933 . PMID 32449244. S2CID 229142596.
- ^ Mafireyi TJ, Escobedo JO, Strongin RM (29 de marzo de 2021). "Sondas fluorogénicas para la actividad de la tiorredoxina reductasa". Resultados en Química . 3 : 100127. doi : 10.1016/j.rechem.2021.100127 . ISSN 2211-7156.
- ^ Nilsonne G, Sun X, Nyström C, Rundlöf AK, Potamitou Fernandes A, Björnstedt M, Dobra K (septiembre de 2006). "La selenita induce apoptosis en células de mesotelioma maligno sarcomatoide a través del estrés oxidativo". Biología y medicina de radicales libres . 41 (6): 874–85. doi :10.1016/j.freeradbiomed.2006.04.031. hdl : 10616/47514 . PMID 16934670.
- ^ Kahlos K, Soini Y, Säily M, Koistinen P, Kakko S, Pääkkö P, Holmgren A, Kinnula VL (mayo de 2001). "Regulación positiva de tiorredoxina y tiorredoxina reductasa en el mesotelioma pleural maligno humano". Revista Internacional de Cáncer . 95 (3): 198–204. doi : 10.1002/1097-0215(20010520)95:3<198::AID-IJC1034>3.0.CO;2-F . PMID 11307155.
- ^ Sibbing D, Pfeufer A, Perisic T, Mannes AM, Fritz-Wolf K, Unwin S, Sinner MF, Gieger C, Gloeckner CJ, Wichmann HE, Kremmer E, Schäfer Z, Walch A, Hinterseer M, Näbauer M, Kääb S , Kastrati A, Schömig A, Meitinger T, Bornkamm GW, Conrad M, von Beckerath N (mayo de 2011). "Las mutaciones en el gen de la tioredoxina reductasa mitocondrial TXNRD2 causan miocardiopatía dilatada". Revista europea del corazón . 32 (9): 1121–33. doi : 10.1093/eurheartj/ehq507 . hdl : 11858/00-001M-0000-0024-1F10-3 . Número de modelo: PMID21247928.
- ^ Marshall AC, Kidd SE, Lamont-Friedrich SJ, Arentz G, Hoffmann P, Coad BR, Bruning JB (marzo de 2019). "Tiorredoxina reductasa de Aspergillus fumigatus". Agentes antimicrobianos y quimioterapia . 63 (3). doi :10.1128/AAC.02281-18. PMC 6395915. PMID 30642940 .
- ^ Harbut MB, Vilchèze C, Luo X, Hensler ME, Guo H, Yang B, et al. (abril de 2015). "Auranofin ejerce actividades bactericidas de amplio espectro al dirigirse a la homeostasis redox-tiol". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 112 (14): 4453–8. Bibcode :2015PNAS..112.4453H. doi : 10.1073/pnas.1504022112 . PMC 4394260 . PMID 25831516.
Enlaces externos
- Tiorredoxina+reductasa+(NADPH) en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.