Espectroscopia de resonancia magnética nuclear

La espectroscopia de resonancia magnética nuclear , más comúnmente conocida como espectroscopia de RMN o espectroscopia de resonancia magnética ( MRS ), es una técnica espectroscópica basada en la reorientación de núcleos atómicos con espines nucleares distintos de cero en un campo magnético externo. Esta reorientación se produce con la absorción de radiación electromagnética en la región de radiofrecuencia de aproximadamente 4 a 900 MHz, que depende de la naturaleza isotópica del núcleo y aumenta proporcionalmente a la fuerza del campo magnético externo. [1] En particular, la frecuencia de resonancia de cada núcleo activo por RMN depende de su entorno químico. Como resultado, los espectros de RMN proporcionan información sobre los grupos funcionales individuales presentes en la muestra, así como sobre las conexiones entre los núcleos cercanos en la misma molécula. Como los espectros de RMN son únicos o muy característicos de compuestos individuales y grupos funcionales , la espectroscopia de RMN es uno de los métodos más importantes para identificar estructuras moleculares, particularmente de compuestos orgánicos .
El principio de RMN generalmente implica tres pasos secuenciales:
- La alineación (polarización) de los espines nucleares magnéticos en un campo magnético constante aplicado B 0 .
- La perturbación de esta alineación de los espines nucleares por un campo magnético oscilante débil, normalmente denominado pulso de radiofrecuencia (RF).
- Detección y análisis de las ondas electromagnéticas emitidas por los núcleos de la muestra como resultado de esta perturbación.
De manera similar, los bioquímicos utilizan la RMN para identificar proteínas y otras moléculas complejas. Además de la identificación, la espectroscopia de RMN proporciona información detallada sobre la estructura, la dinámica, el estado de reacción y el entorno químico de las moléculas. Los tipos más comunes de RMN son la espectroscopia de RMN de protones y carbono-13 , pero es aplicable a cualquier tipo de muestra que contenga núcleos que posean espín .
Los espectros de RMN son únicos, tienen una buena resolución, son analíticamente manejables y, a menudo, muy predecibles para moléculas pequeñas . Obviamente, se pueden distinguir diferentes grupos funcionales y los grupos funcionales idénticos con sustituyentes vecinos diferentes aún dan señales distinguibles. La RMN ha reemplazado en gran medida las pruebas de química húmeda tradicionales , como los reactivos de color o la cromatografía típica para la identificación.
El inconveniente más importante de la espectroscopia RMN es su baja sensibilidad (en comparación con otros métodos analíticos, como la espectrometría de masas ). Normalmente se necesitan entre 2 y 50 mg de una sustancia para registrar un espectro RMN de calidad decente. El método RMN no es destructivo, por lo que la sustancia puede recuperarse. Para obtener espectros RMN de alta resolución, normalmente se disuelven sustancias sólidas para formar soluciones líquidas, aunque también es posible la espectroscopia RMN de estado sólido .
La escala temporal de la RMN es relativamente larga y, por lo tanto, no es adecuada para observar fenómenos rápidos, ya que produce solo un espectro promediado. Aunque se pueden observar grandes cantidades de impurezas en un espectro de RMN, existen mejores métodos para detectarlas, ya que la RMN no es inherentemente muy sensible, aunque a frecuencias más altas, la sensibilidad es mayor.
La espectroscopia de correlación es un desarrollo de la RMN ordinaria. En la RMN bidimensional , la emisión se centra alrededor de una sola frecuencia y se observan resonancias correlacionadas. Esto permite identificar los sustituyentes vecinos del grupo funcional observado, lo que permite la identificación inequívoca de las resonancias. También existen métodos 3D y 4D más complejos y una variedad de métodos diseñados para suprimir o amplificar tipos particulares de resonancias. En la espectroscopia de efecto Overhauser nuclear (NOE), se observa la relajación de las resonancias. Como el NOE depende de la proximidad de los núcleos, cuantificar el NOE para cada núcleo permite la construcción de un modelo tridimensional de la molécula.

Los espectrómetros de RMN son relativamente caros; las universidades suelen tenerlos, pero son menos comunes en las empresas privadas. Entre 2000 y 2015, un espectrómetro de RMN costaba alrededor de 0,5 a 5 millones de dólares . [3] [4] Los espectrómetros de RMN modernos tienen un imán superconductor refrigerado por helio líquido muy potente, grande y costoso , porque la resolución depende directamente de la intensidad del campo magnético. Un campo magnético más alto también mejora la sensibilidad de la espectroscopia de RMN, que depende de la diferencia de población entre los dos niveles nucleares, que aumenta exponencialmente con la intensidad del campo magnético.
También hay disponibles máquinas menos costosas que utilizan imanes permanentes y una resolución más baja, que aún brindan un rendimiento suficiente para ciertas aplicaciones, como el monitoreo de reacciones y la verificación rápida de muestras. Incluso hay espectrómetros de resonancia magnética nuclear de sobremesa . Los espectros de RMN de protones ( núcleos de 1 H) se pueden observar incluso en el campo magnético de la Tierra . La RMN de baja resolución produce picos más amplios, que pueden superponerse fácilmente entre sí, lo que causa problemas en la resolución de estructuras complejas. El uso de campos magnéticos de mayor intensidad da como resultado una mejor sensibilidad y una mayor resolución de los picos, y es preferible para fines de investigación. [5]
Historia
El descubrimiento de la RMN se debe a Isidor Isaac Rabi , quien recibió el Premio Nobel de Física en 1944. [6] El grupo Purcell de la Universidad de Harvard y el grupo Bloch de la Universidad de Stanford desarrollaron independientemente la espectroscopia de RMN a fines de la década de 1940 y principios de la de 1950. Edward Mills Purcell y Felix Bloch compartieron el Premio Nobel de Física de 1952 por sus inventos. [7]
Criterios de RMN activa
El determinante clave de la actividad de RMN en los núcleos atómicos es el número cuántico de espín nuclear ( I ). Esta propiedad cuántica intrínseca, similar al " espín " de un átomo, caracteriza el momento angular del núcleo. Para ser activo en RMN, un núcleo debe tener un espín nuclear distinto de cero ( I ≠ 0). [8] Es este espín distinto de cero lo que permite a los núcleos interactuar con campos magnéticos externos y mostrar señales en RMN. Los átomos con una suma impar de protones y neutrones exhiben valores semienteros para el número cuántico de espín nuclear ( I = 1/2, 3/2, 5/2, etc.). Estos átomos son activos en RMN porque poseen un espín nuclear distinto de cero. Los átomos con una suma par pero un número impar de protones y un número impar de neutrones exhiben espines nucleares enteros ( I = 1, 2, 3, etc.). Por el contrario, los átomos con un número par de protones y neutrones tienen un número cuántico de espín nuclear de cero ( I = 0) y, por lo tanto, no son activos en RMN. [9] Los núcleos activos en RMN, en particular aquellos con un número cuántico de espín de 1/2, son de gran importancia en la espectroscopia de RMN. Algunos ejemplos incluyen 1 H, 13 C, 15 N y 31 P. [10]
Aspectos principales de las técnicas de RMN
Frecuencia resonante
Cuando se colocan en un campo magnético, los núcleos activos de RMN (como 1 H o 13 C) absorben la radiación electromagnética a una frecuencia característica del isótopo . [11] La frecuencia de resonancia, la energía de la radiación absorbida y la intensidad de la señal son proporcionales a la fuerza del campo magnético. Por ejemplo, en un campo magnético de 21 teslas , los núcleos de hidrógeno ( protones ) resuenan a 900 MHz. Es común referirse a un imán de 21 T como un imán de 900 MHz , ya que el hidrógeno es el núcleo más común detectado. Sin embargo, diferentes núcleos resonarán a diferentes frecuencias en esta intensidad de campo en proporción a sus momentos magnéticos nucleares . [ cita requerida ]
Manipulación de muestras

Un espectrómetro de RMN consta típicamente de un portamuestras giratorio dentro de un imán muy potente, un emisor de radiofrecuencia y un receptor con una sonda (un conjunto de antena) que va dentro del imán para rodear la muestra, opcionalmente bobinas de gradiente para mediciones de difusión y electrónica para controlar el sistema. Hacer girar la muestra suele ser necesario para promediar el movimiento de difusión, sin embargo, algunos experimentos requieren una muestra estacionaria cuando el movimiento de la solución es una variable importante. Por ejemplo, las mediciones de las constantes de difusión ( espectroscopia ordenada por difusión o DOSY) [12] [13] se realizan utilizando una muestra estacionaria con centrifugación, y las celdas de flujo se pueden utilizar para el análisis en línea de los flujos de proceso.
Disolventes deuterados
La gran mayoría de las moléculas en una solución son moléculas de disolvente, y la mayoría de los disolventes regulares son hidrocarburos y, por lo tanto, contienen núcleos de hidrógeno-1 activos en RMN. Para evitar que las señales de los átomos de hidrógeno del disolvente abrumen el experimento e interfieran en el análisis del analito disuelto, se utilizan disolventes deuterados en los que >99% de los protones se reemplazan con deuterio (hidrógeno-2). [14] El disolvente deuterado más utilizado es el deuterocloroformo (CDCl 3 ), aunque se pueden utilizar otros disolventes por diversas razones, como la solubilidad de una muestra, el deseo de controlar los enlaces de hidrógeno o los puntos de fusión o ebullición. Los desplazamientos químicos de una molécula cambian ligeramente entre disolventes y, por lo tanto, el disolvente utilizado casi siempre se informa con desplazamientos químicos. [ cita requerida ] Los espectros de RMN de protones a menudo se calibran contra el pico de protón residual del disolvente conocido [15] como un estándar interno en lugar de agregar tetrametilsilano (TMS), que se define convencionalmente como que tiene un desplazamiento químico de cero. [16]
Calzar y bloquear
Para detectar los cambios de frecuencia muy pequeños debidos a la resonancia magnética nuclear, el campo magnético aplicado debe ser extremadamente uniforme en todo el volumen de la muestra. Los espectrómetros de RMN de alta resolución utilizan cuñas para ajustar la homogeneidad del campo magnético a partes por billón ( ppb ) en un volumen de unos pocos centímetros cúbicos. Para detectar y compensar la falta de homogeneidad y la deriva en el campo magnético, el espectrómetro mantiene un "bloqueo" en la frecuencia del deuterio del disolvente con una unidad de bloqueo separada, que es esencialmente un transmisor adicional y un procesador de RF sintonizado con el núcleo de bloqueo (deuterio) en lugar de los núcleos de la muestra de interés. [17] En los espectrómetros de RMN modernos, la cuña se ajusta automáticamente, aunque en algunos casos el operador tiene que optimizar los parámetros de la cuña manualmente para obtener la mejor resolución posible. [17] [18]
Adquisición de espectros
Al excitar la muestra con un pulso de radiofrecuencia (60–1000 MHz), se obtiene una respuesta de resonancia magnética nuclear (una desintegración por inducción libre , FID). Es una señal muy débil y requiere receptores de radio sensibles para captarla. Se realiza una transformada de Fourier para extraer el espectro del dominio de frecuencia del FID del dominio de tiempo sin procesar. Un espectro de un solo FID tiene una baja relación señal-ruido , pero mejora fácilmente con el promedio de adquisiciones repetidas. Se pueden adquirir buenos espectros de RMN de 1 H con 16 repeticiones, lo que lleva solo minutos. Sin embargo, para elementos más pesados que el hidrógeno, el tiempo de relajación es bastante largo, por ejemplo, alrededor de 8 segundos para 13 C. Por lo tanto, la adquisición de espectros cuantitativos de elementos pesados puede llevar mucho tiempo, demorando decenas de minutos u horas. [ cita requerida ]
Después del pulso, los núcleos se excitan, en promedio, hasta un cierto ángulo en relación con el campo magnético del espectrómetro. El grado de excitación se puede controlar con el ancho del pulso, que normalmente es de 3 a 8 μs para el pulso óptimo de 90°. El ancho del pulso se puede determinar trazando la intensidad (con signo) en función del ancho del pulso. Sigue una curva sinusoidal y, en consecuencia, cambia de signo en anchos de pulso correspondientes a pulsos de 180° y 360°. [ cita requerida ]
Los tiempos de decaimiento de la excitación, que se miden típicamente en segundos, dependen de la efectividad de la relajación, que es más rápida para núcleos más ligeros y en sólidos, más lenta para núcleos más pesados y en soluciones, y puede ser muy larga en gases. Si el segundo pulso de excitación se envía prematuramente antes de que se complete la relajación, el vector de magnetización promedio no ha decaído al estado fundamental, lo que afecta la fuerza de la señal de una manera impredecible. En la práctica, las áreas de los picos no son proporcionales a la estequiometría; solo es posible discernir la presencia, pero no la cantidad de grupos funcionales. Se puede realizar un experimento de recuperación de inversión para determinar el tiempo de relajación y, por lo tanto, el retraso necesario entre pulsos. Se transmite un pulso de 180°, un retraso ajustable y un pulso de 90°. Cuando el pulso de 90° cancela exactamente la señal, el retraso corresponde al tiempo necesario para 90° de relajación. [19] La recuperación de inversión vale la pena para experimentos cuantitativos de 13 C, 2 D y otros que consumen mucho tiempo.
Interpretación espectral
Las señales de RMN se caracterizan normalmente por tres variables: desplazamiento químico, acoplamiento espín-espín y tiempo de relajación.
Desplazamiento químico

La diferencia de energía Δ E entre los estados de espín nuclear es proporcional al campo magnético ( efecto Zeeman ). Δ E también es sensible al entorno electrónico del núcleo, dando lugar a lo que se conoce como desplazamiento químico, δ. Los tipos más simples de gráficos de RMN son gráficos de los diferentes desplazamientos químicos de los núcleos que se estudian en la molécula. El valor de δ se expresa a menudo en términos de "blindaje": los núcleos protegidos tienen Δ E más alto . El rango de valores de δ se llama dispersión. Es bastante pequeño para las señales de 1 H, pero mucho mayor para otros núcleos. Las señales de RMN se informan en relación con una señal de referencia, generalmente la de TMS ( tetrametilsilano ). Además, dado que la distribución de las señales de RMN depende del campo, estas frecuencias se dividen por la frecuencia del espectrómetro. Sin embargo, dado que estamos dividiendo Hz por MHz, el número resultante sería demasiado pequeño y, por lo tanto, se multiplica por un millón. Por lo tanto, esta operación da un número localizador llamado "desplazamiento químico" con unidades de partes por millón. [20] El desplazamiento químico proporciona información estructural.
La conversión de los desplazamientos químicos (y de las J, véase más abajo) se denomina asignación del espectro. En el caso de los compuestos orgánicos diamagnéticos, las asignaciones de los espectros de RMN de 1 H y 13 C son extremadamente sofisticadas debido a las grandes bases de datos y a las sencillas herramientas computacionales. En general, los desplazamientos químicos de los protones son muy predecibles, ya que los desplazamientos están determinados principalmente por los efectos de apantallamiento (densidad electrónica). Los desplazamientos químicos de muchos núcleos más pesados están más fuertemente influenciados por otros factores, incluidos los estados excitados (contribución "paramagnética" al tensor de apantallamiento). Esta contribución paramagnética, que no está relacionada con el paramagnetismo ) no sólo altera las tendencias de los desplazamientos químicos, lo que complica las asignaciones, sino que también da lugar a rangos de desplazamiento químico muy amplios. Por ejemplo, la mayoría de las señales de RMN de 1 H para la mayoría de los compuestos orgánicos están dentro de las 15 ppm. Para la RMN de 31 P, el rango es de cientos de ppm. [21]
En la espectroscopia RMN paramagnética , las muestras son paramagnéticas, es decir, contienen electrones desapareados. El paramagnetismo da lugar a desplazamientos químicos muy diversos. En la espectroscopia RMN 1 H, el rango de desplazamiento químico puede alcanzar miles de ppm. [22]
Acoplamiento en J
| Multiplicidad | Relación de intensidad |
|---|---|
| Camiseta(s) sin mangas | 1 |
| Doblete (d) | 1:1 |
| Triplete (t) | 1:2:1 |
| Cuarteto (q) | 1:3:3:1 |
| Quinteto | 1:4:6:4:1 |
| Sexteto | 1:5:10:10:5:1 |
| Septeto | 1:6:15:20:15:6:1 |
Parte de la información más útil para la determinación de la estructura en un espectro de RMN unidimensional proviene del acoplamiento J, o acoplamiento escalar (un caso especial de acoplamiento espín-espín ), entre núcleos activos de RMN. Este acoplamiento surge de la interacción de diferentes estados de espín a través de los enlaces químicos de una molécula y da como resultado la división de las señales de RMN. Para un protón, el campo magnético local es ligeramente diferente dependiendo de si un núcleo adyacente apunta hacia o contra el campo magnético del espectrómetro, lo que da lugar a dos señales por protón en lugar de una. Estos patrones de división pueden ser complejos o simples y, del mismo modo, pueden ser directamente interpretables o engañosos. Este acoplamiento proporciona una visión detallada de la conectividad de los átomos en una molécula. [ cita requerida ]
La multiplicidad de la división es un efecto de los espines de los núcleos que están acoplados y el número de dichos núcleos involucrados en el acoplamiento. El acoplamiento a n núcleos de espín 1/2 equivalentes divide la señal en un multiplete n + 1 con relaciones de intensidad que siguen el triángulo de Pascal como se describe en la tabla. El acoplamiento a espines adicionales conduce a más divisiones de cada componente del multiplete, por ejemplo, el acoplamiento a dos núcleos de espín 1/2 diferentes con constantes de acoplamiento significativamente diferentes conduce a un doblete de dobletes (abreviatura: dd). Tenga en cuenta que el acoplamiento entre núcleos que son químicamente equivalentes (es decir, tienen el mismo desplazamiento químico) no tiene efecto en los espectros de RMN, y los acoplamientos entre núcleos que están distantes (generalmente más de 3 enlaces de distancia para protones en moléculas flexibles) generalmente son demasiado pequeños para causar divisiones observables. A menudo se pueden observar acoplamientos de largo alcance sobre más de tres enlaces en compuestos cíclicos y aromáticos , lo que conduce a patrones de división más complejos. [ cita requerida ]
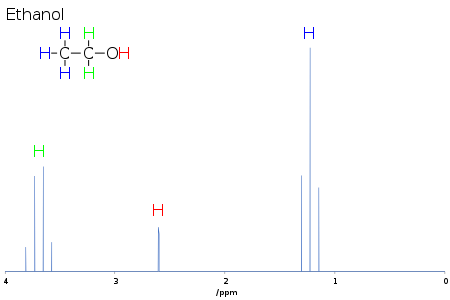
Por ejemplo, en el espectro de protones del etanol, el grupo CH 3 se divide en un triplete con una relación de intensidad de 1:2:1 por los dos protones CH 2 vecinos . De manera similar, el CH 2 se divide en un cuarteto con una relación de intensidad de 1:3:3:1 por los tres protones CH 3 vecinos . En principio, los dos protones CH 2 también se dividirían nuevamente en un doblete para formar un doblete de cuartetos por el protón hidroxilo, pero el intercambio intermolecular del protón hidroxilo ácido a menudo da como resultado una pérdida de información de acoplamiento.
El acoplamiento a cualquier núcleo de espín 1/2, como el fósforo-31 o el flúor-19, funciona de esta manera (aunque las magnitudes de las constantes de acoplamiento pueden ser muy diferentes). Pero los patrones de división difieren de los descritos anteriormente para los núcleos con espín mayor que 1/2 porque el número cuántico de espín tiene más de dos valores posibles. Por ejemplo, el acoplamiento al deuterio (un núcleo de espín 1) divide la señal en un triplete 1:1:1 porque el espín 1 tiene tres estados de espín. De manera similar, un núcleo de espín 3/2, como el 35 Cl, divide una señal en un cuarteto 1:1:1:1 y así sucesivamente.
El acoplamiento combinado con el desplazamiento químico (y la integración de los protones) nos informa no solo sobre el entorno químico de los núcleos, sino también sobre la cantidad de núcleos activos de RMN vecinos dentro de la molécula. En espectros más complejos con múltiples picos en desplazamientos químicos similares o en espectros de núcleos distintos del hidrógeno, el acoplamiento es a menudo la única forma de distinguir núcleos diferentes.
La magnitud del acoplamiento (la constante de acoplamiento J ) es un efecto de la fuerza con la que los núcleos están acoplados entre sí. En casos sencillos, se trata de un efecto de la distancia de enlace entre los núcleos, el momento magnético de los núcleos y el ángulo diedro entre ellos.
Acoplamiento de segundo orden (o fuerte)

La descripción anterior supone que la constante de acoplamiento es pequeña en comparación con la diferencia en las frecuencias de RMN entre los espines no equivalentes. Si la separación por desplazamiento disminuye (o aumenta la fuerza de acoplamiento), los patrones de intensidad del multiplete primero se distorsionan y luego se vuelven más complejos y menos fáciles de analizar (especialmente si están involucrados más de dos espines). La intensificación de algunos picos en un multiplete se logra a expensas del resto, que a veces casi desaparece en el ruido de fondo, aunque el área integrada debajo de los picos permanece constante. Sin embargo, en la mayoría de los RMN de alto campo, las distorsiones suelen ser modestas y las distorsiones características ( techo ) de hecho pueden ayudar a identificar picos relacionados.
Algunos de estos patrones pueden analizarse con el método publicado por John Pople [23] , aunque tiene un alcance limitado.
Los efectos de segundo orden disminuyen a medida que aumenta la diferencia de frecuencia entre los multipletes, de modo que los espectros de RMN de campo alto (es decir, de alta frecuencia) muestran menos distorsión que los espectros de frecuencia más baja. Los primeros espectros a 60 MHz eran más propensos a la distorsión que los espectros de máquinas posteriores que normalmente operaban a frecuencias de 200 MHz o superiores.
Además, como en la figura de la derecha, el acoplamiento J se puede utilizar para identificar la sustitución orto-meta-para de un anillo. El acoplamiento orto es el más fuerte a 15 Hz, seguido por el meta con un promedio de 2 Hz y, por último, el acoplamiento para suele ser insignificante para los estudios.
Inequivalencia magnética
Pueden producirse efectos más sutiles si los espines químicamente equivalentes (es decir, núcleos relacionados por simetría y que, por lo tanto, tienen la misma frecuencia de RMN) tienen diferentes relaciones de acoplamiento con los espines externos. Los espines que son químicamente equivalentes pero no son indistinguibles (según sus relaciones de acoplamiento) se denominan magnéticamente no equivalentes. Por ejemplo, los 4 sitios H del 1,2-diclorobenceno se dividen en dos pares químicamente equivalentes por simetría, pero un miembro individual de uno de los pares tiene diferentes acoplamientos con los espines que forman el otro par.
La inequivalencia magnética puede generar espectros muy complejos, que solo pueden analizarse mediante modelos computacionales. Estos efectos son más comunes en los espectros de RMN de sistemas aromáticos y otros sistemas no flexibles, mientras que el promedio conformacional sobre enlaces C−C en moléculas flexibles tiende a igualar los acoplamientos entre protones en carbonos adyacentes, lo que reduce los problemas de inequivalencia magnética.
Espectroscopia de correlación
La espectroscopia de correlación es uno de los varios tipos de espectroscopia de resonancia magnética nuclear (RMN) bidimensional o RMN 2D . Este tipo de experimento de RMN es más conocido por su acrónimo , COSY . Otros tipos de RMN bidimensional incluyen la espectroscopia J, la espectroscopia de intercambio (EXSY), la espectroscopia de efecto Overhauser nuclear (NOESY), la espectroscopia de correlación total (TOCSY) y los experimentos de correlación heteronuclear, como HSQC , HMQC y HMBC . En la espectroscopia de correlación, la emisión se centra en el pico de un núcleo individual; si su campo magnético está correlacionado con otro núcleo mediante acoplamiento a través del enlace (COSY, HSQC, etc.) o a través del espacio (NOE), también se puede detectar una respuesta en la frecuencia del núcleo correlacionado. Los espectros de RMN bidimensionales proporcionan más información sobre una molécula que los espectros de RMN unidimensionales y son especialmente útiles para determinar la estructura de una molécula , en particular para moléculas que son demasiado complicadas para trabajar con RMN unidimensional. El primer experimento bidimensional, COSY, fue propuesto por Jean Jeener, profesor de la Université Libre de Bruxelles, en 1971. [24] [25] Este experimento fue implementado posteriormente por Walter P. Aue, Enrico Bartholdi y Richard R. Ernst , quienes publicaron su trabajo en 1976. [26]
Resonancia magnética nuclear de estado sólido

Una variedad de circunstancias físicas no permiten estudiar las moléculas en solución y, al mismo tiempo, tampoco mediante otras técnicas espectroscópicas a nivel atómico. En medios en fase sólida, como cristales, polvos microcristalinos, geles, soluciones anisotrópicas, etc., son en particular el acoplamiento dipolar y la anisotropía por desplazamiento químico los que se vuelven dominantes para el comportamiento de los sistemas de espín nuclear. En la espectroscopia RMN en estado de solución convencional, estas interacciones adicionales conducirían a un ensanchamiento significativo de las líneas espectrales. Una variedad de técnicas permite establecer condiciones de alta resolución, que pueden, al menos para los espectros de 13 C, ser comparables a los espectros RMN en estado de solución.
Dos conceptos importantes para la espectroscopia RMN de estado sólido de alta resolución son la limitación de la posible orientación molecular mediante la orientación de la muestra y la reducción de las interacciones magnéticas nucleares anisotrópicas mediante el giro de la muestra. De este último enfoque, el giro rápido alrededor del ángulo mágico es un método muy destacado, cuando el sistema comprende núcleos de espín 1/2. Se utilizan velocidades de giro de aproximadamente 20 kHz, lo que exige un equipo especial. Actualmente [ ¿cuándo? ] se están utilizando en la espectroscopia RMN varias técnicas intermedias, con muestras de alineación parcial o movilidad reducida. [ cita requerida ]
Las aplicaciones en las que se producen efectos de RMN en estado sólido suelen estar relacionadas con investigaciones estructurales sobre proteínas de membrana, fibrillas proteicas o todo tipo de polímeros, y análisis químicos en química inorgánica, pero también incluyen aplicaciones "exóticas" como las hojas de las plantas y las pilas de combustible. Por ejemplo, Rahmani et al. estudiaron el efecto de la presión y la temperatura en el autoensamblaje de las estructuras bicelares utilizando espectroscopia de RMN de deuterio. [28]
Espectroscopia RMN biomolecular
Proteínas
Gran parte de la innovación dentro de la espectroscopia de RMN ha sido dentro del campo de la espectroscopia de RMN de proteínas , una técnica importante en biología estructural . Un objetivo común de estas investigaciones es obtener estructuras tridimensionales de alta resolución de la proteína, similar a lo que se puede lograr mediante cristalografía de rayos X. A diferencia de la cristalografía de rayos X, la espectroscopia de RMN generalmente se limita a proteínas menores de 35 kDa , aunque se han resuelto estructuras más grandes. La espectroscopia de RMN es a menudo la única forma de obtener información de alta resolución sobre proteínas parcial o totalmente intrínsecamente no estructuradas . Ahora es una herramienta común para la determinación de las relaciones de actividad de conformación donde la estructura antes y después de la interacción con, por ejemplo, un candidato a fármaco se compara con su actividad bioquímica conocida. Las proteínas son órdenes de magnitud más grandes que las pequeñas moléculas orgánicas discutidas anteriormente en este artículo, pero las técnicas básicas de RMN y algo de teoría de RMN también se aplican. Debido a que en una molécula de proteína hay un número mucho mayor de átomos que en un compuesto orgánico pequeño, los espectros 1D básicos se llenan de señales superpuestas hasta tal punto que el análisis espectral directo resulta insostenible. Por lo tanto, se han ideado experimentos multidimensionales (2, 3 o 4D) para abordar este problema. Para facilitar estos experimentos, es deseable marcar isotópicamente la proteína con 13 C y 15 N porque el isótopo predominante de origen natural 12 C no es activo en RMN y el momento cuadrupolar nuclear del isótopo predominante de origen natural 14 N impide que se obtenga información de alta resolución a partir de este isótopo de nitrógeno. El método más importante utilizado para la determinación de la estructura de las proteínas utiliza experimentos NOE para medir las distancias entre los átomos dentro de la molécula. Posteriormente, las distancias obtenidas se utilizan para generar una estructura 3D de la molécula resolviendo un problema de geometría de distancia . La RMN también se puede utilizar para obtener información sobre la dinámica y la flexibilidad conformacional de diferentes regiones de una proteína.
Ácidos nucleicos
La RMN de ácidos nucleicos es el uso de la espectroscopia de RMN para obtener información sobre la estructura y dinámica de los ácidos polinucleicos , como el ADN o el ARN . En 2003 [actualizar], casi la mitad de todas las estructuras de ARN conocidas se habían determinado mediante espectroscopia de RMN. [29]
La espectroscopia de RMN de ácidos nucleicos y proteínas es similar, pero existen diferencias. Los ácidos nucleicos tienen un porcentaje menor de átomos de hidrógeno, que son los átomos que se observan habitualmente en la espectroscopia de RMN, y debido a que las dobles hélices de los ácidos nucleicos son rígidas y aproximadamente lineales, no se pliegan sobre sí mismas para dar correlaciones de "largo alcance". [30] Los tipos de RMN que se suelen realizar con los ácidos nucleicos son la RMN de 1 H o protón , la RMN de 13 C , la RMN de 15 N y la RMN de 31 P. Casi siempre se utilizan métodos de RMN bidimensionales , como la espectroscopia de correlación (COSY) y la espectroscopia de transferencia de coherencia total (TOCSY) para detectar acoplamientos nucleares a través de enlaces, y la espectroscopia de efecto Overhauser nuclear (NOESY) para detectar acoplamientos entre núcleos que están cerca uno del otro en el espacio. [31]
Los parámetros tomados del espectro, principalmente los picos cruzados NOESY y las constantes de acoplamiento , se pueden utilizar para determinar características estructurales locales como ángulos de enlace glucosídico , ángulos diedros (usando la ecuación de Karplus ) y conformaciones de fruncido de azúcar. Para la estructura a gran escala, estos parámetros locales deben complementarse con otros supuestos o modelos estructurales, porque los errores se acumulan a medida que se recorre la doble hélice y, a diferencia de las proteínas, la doble hélice no tiene un interior compacto y no se pliega sobre sí misma. La RMN también es útil para investigar geometrías no estándar como hélices dobladas , apareamiento de bases no Watson-Crick y apilamiento coaxial . Ha sido especialmente útil para investigar la estructura de oligonucleótidos de ARN naturales, que tienden a adoptar conformaciones complejas como bucles de tallo y pseudonudos . La RMN también es útil para investigar la unión de moléculas de ácido nucleico a otras moléculas, como proteínas o fármacos, al ver qué resonancias se desplazan al unirse a la otra molécula. [31]
Carbohidratos
La espectroscopia de RMN de carbohidratos aborda cuestiones sobre la estructura y la conformación de los carbohidratos . El análisis de carbohidratos mediante RMN de 1H es un desafío debido a la variación limitada en los grupos funcionales, lo que conduce a resonancias de 1H concentradas en bandas estrechas del espectro de RMN. En otras palabras, hay una dispersión espectral deficiente. Las resonancias de protones anoméricas están segregadas de las demás debido al hecho de que los carbonos anoméricos tienen dos átomos de oxígeno. Para carbohidratos más pequeños, la dispersión de las resonancias de protones anoméricas facilita el uso de experimentos TOCSY 1D para investigar los sistemas de espín completos de residuos de carbohidratos individuales.
Descubrimiento de fármacos
El conocimiento de los mínimos de energía y las barreras de energía rotacional de las moléculas pequeñas en solución se puede obtener mediante RMN, por ejemplo, observando las preferencias conformacionales de los ligandos libres y la dinámica conformacional, respectivamente. Esto se puede utilizar para orientar las hipótesis de diseño de fármacos, ya que los valores experimentales y calculados son comparables. Por ejemplo, AstraZeneca utiliza RMN para su investigación y desarrollo en oncología. [32]
Espectroscopia RMN de alta presión
Uno de los primeros trabajos científicos dedicados al uso de la presión como parámetro variable en experimentos de RMN fue el trabajo de J. Jonas publicado en la revista Annual Review of Biophysics en 1994. [33] El uso de altas presiones en la espectroscopia de RMN fue impulsado principalmente por el deseo de estudiar sistemas bioquímicos, donde el uso de alta presión permite cambios controlados en las interacciones intermoleculares sin perturbaciones significativas.
Por supuesto, se han hecho intentos para resolver problemas científicos utilizando espectroscopia de RMN de alta presión. Sin embargo, la mayoría de ellos fueron difíciles de reproducir debido al problema del equipo para crear y mantener la alta presión. En [34] [35] [36] se dan los tipos más comunes de celdas de RMN para la realización de experimentos de RMN de alta presión. [ aclaración necesaria ]
La espectroscopia de RMN de alta presión se ha utilizado ampliamente para una variedad de aplicaciones, principalmente relacionadas con la caracterización de la estructura de las moléculas de proteínas. [37] [38] Sin embargo, en los últimos años, se han propuesto soluciones de software y diseño para caracterizar las estructuras químicas y espaciales de moléculas pequeñas en un entorno de fluido supercrítico, [39] utilizando parámetros de estado como fuerza impulsora para tales cambios. [40]
Véase también
- RMN del campo terrestre
- Espectroscopia de resonancia magnética in vivo
- Espectroscopia de resonancia magnética funcional del cerebro
- RMN de campo bajo
- Imágenes por resonancia magnética
- Cristalografía de RMN
- Base de datos de espectros de RMN
- Espectroscopia RMN de estereoisómeros
- Resonancia nuclear cuadrupolo
- Imán de campo pulsado
- Espectroscopia de inducción nuclear potenciada por protones
- Espectroscopia de resonancia magnética nuclear de triple resonancia
- RMN de campo cero
- Desacoplamiento por resonancia magnética nuclear
Métodos relacionados de espectroscopia nuclear :
Referencias
- ^ Charles P. Slichter (1963). Principios de resonancia magnética: con ejemplos de la física del estado sólido . Harper & Row. ISBN 9783540084761.
- ^ Biología estructural: aplicaciones prácticas de RMN (PDF) (2.ª ed.). Springer. 6 de septiembre de 2012. pág. 67. ISBN 978-1-4614-3964-6. Recuperado el 7 de diciembre de 2018 .
- ^ Marc S. Reisch (29 de junio de 2015). "Los aumentos de precios de los instrumentos de RMN asustan a los usuarios". CEN.
- ^ "Llevándolo a lo más alto". The Scientist. 30 de octubre de 2000.
- ^ Paudler, William (1974). Resonancia magnética nuclear . Boston: Allyn and Bacon Chemistry Series. págs. 9-11.
- ^ "Descubrimiento de la RMN".
- ^ "Página de antecedentes y teoría de la instalación de resonancia magnética nuclear". Mark Wainwright Analytical Centre - University of Southern Wales Sydney. 9 de diciembre de 2011. Archivado desde el original el 27 de enero de 2014 . Consultado el 9 de febrero de 2014 .
- ^ "4.7: Espectroscopia de RMN". Chemistry LibreTexts . 2016-07-14 . Consultado el 2023-11-25 .
- ^ Balci, Metin (2005), "Espectroscopia de RMN dinámica", Espectroscopia básica de RMN de 1H y 13C , Elsevier, págs. 213-231, doi :10.1016/b978-044451811-8.50008-5, ISBN 9780444518118, consultado el 25 de noviembre de 2023
- ^ "Núcleos activos de RMN para aplicaciones biológicas y biomédicas". Open Medscience . Consultado el 25 de noviembre de 2023 .
- ^ Shah, N.; Sattar, A.; Benanti, M.; Hollander, S.; Cheuck, L. (enero de 2006). "Espectroscopia de resonancia magnética como herramienta de diagnóstico por imágenes para el cáncer: una revisión de la literatura". The Journal of the American Osteopathic Association . 106 (1): 23–27. PMID 16428685. Archivado desde el original el 7 de abril de 2013.
- ^ Johnson Jr., CS (1999). "Espectroscopia de resonancia magnética nuclear ordenada por difusión: principios y aplicaciones". Progreso en espectroscopia de resonancia magnética nuclear . 34 (3–4): 203–256. doi :10.1016/S0079-6565(99)00003-5.
- ^ Neufeld, R.; Stalke, D. (2015). "Determinación precisa del peso molecular de moléculas pequeñas mediante DOSY-NMR utilizando curvas de calibración externas con coeficientes de difusión normalizados". Química. 6 (6): 3354–3364. doi :10.1039/C5SC00670H. PMC 5656982. PMID 29142693 .
- ^ Hanson, John E. (2013). "5. Espectroscopia de RMN en disolventes no deuterados (RMN No-D): aplicaciones en el laboratorio orgánico de pregrado". Espectroscopia de RMN en el plan de estudios de pregrado . Serie de simposios de la ACS. Vol. 1128. Sociedad Química Estadounidense. págs. 69–81. doi :10.1021/bk-2013-1128.ch005. ISBN . 9780841227941.
- ^ Gottlieb, Hugo E.; Kotlyar, Vadim; Nudelman, Abraham (1997-10-01). "Desplazamientos químicos de RMN de disolventes de laboratorio comunes como impurezas traza". The Journal of Organic Chemistry . 62 (21): 7512–7515. doi :10.1021/jo971176v. ISSN 0022-3263.
- ^ Tiers, George Van Dyke (septiembre de 1958). "Valores fiables de protección por resonancia nuclear de protones mediante "referencia interna" con tetrametil-silano". The Journal of Physical Chemistry . 62 (9): 1151–1152. doi :10.1021/j150567a041. ISSN 0022-3654.
- ^ ab "The Lock". Centro de espectroscopia de RMN . Universidad Estatal de Washington. Archivado desde el original el 28 de enero de 2020.
- ^ "Artefactos de RMN". Instalación de RMN Max T. Rogers . Universidad Estatal de Michigan. Archivado desde el original el 24 de septiembre de 2021.
- ^ Parella, Teodor. «Medición de T1 mediante recuperación de inversión» . NMRGuide3.5 . Archivado desde el original el 28 de abril de 2021.
- ^ James Keeler. "Capítulo 2: RMN y niveles de energía" (reimpreso en University of Cambridge ) . Understanding NMR Spectroscopy . University of California, Irvine . Consultado el 11 de mayo de 2007 .
- ^ Peter Atkins (1994) [1990]. Química física (5.ª ed.). Freeman.
- ^ Ott, JC; Wadepohl, H.; Enders, M.; Gade, LH (2018). "Llevando la RMN de protones en solución a su extremo: predicción y detección de una resonancia de hidruro en un complejo de hierro de espín intermedio". J. Am. Chem. Soc . 140 (50): 17413–17417. doi :10.1021/jacs.8b11330. PMID 30486649. S2CID 207195859.
- ^ Pople, JA; Bernstein, HJ; Schneider, WG (1957). "El análisis de los espectros de resonancia magnética nuclear". Can. J. Chem . 35 : 65–81. doi :10.1139/v57-143.
- ^ Aue, WP (1976). "Espectroscopia bidimensional. Aplicación a la resonancia magnética nuclear". The Journal of Chemical Physics . 64 (5): 2229. Bibcode :1976JChPh..64.2229A. doi :10.1063/1.432450.
- ^ Jeener, Jean (2007). "Jeener, Jean: Reminiscencias sobre los primeros días de la RMN 2D". Enciclopedia de resonancia magnética . doi :10.1002/9780470034590.emrhp0087. ISBN 978-0470034590.
- ^ Martin, GE; Zekter, AS, Métodos de RMN bidimensionales para establecer la conectividad molecular ; VCH Publishers, Inc: Nueva York, 1988 (p. 59)
- ^ "Instalación nacional de RMN de campo ultraalto para sólidos" . Consultado el 22 de septiembre de 2014 .
- ^ A. Rahmani, C. Knight y MR Morrow. "Respuesta a la presión hidrostática de dispersiones bicelares que contienen lípidos aniónicos: interdigitación inducida por presión". 2013, 29 (44), págs. 13481–13490, doi :10.1021/la4035694.
- ^ Furtig, Boris; Richter, cristiano; Wöhnert, Jens; Schwalbe, Harald (2003). "Espectroscopia de RMN de ARN". ChemBioChem . 4 (10): 936–62. doi :10.1002/cbic.200300700. PMID 14523911. S2CID 33523981.
- ^ Addess, Kenneth J.; Feigon, Juli (1996). "Introducción a la espectroscopia de RMN de 1 H del ADN". En Hecht, Sidney M. (ed.). Química bioorgánica: ácidos nucleicos . Nueva York: Oxford University Press. ISBN 978-0-19-508467-2.
- ^ ab Wemmer, David (2000). "Capítulo 5: Estructura y dinámica por RMN". En Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Ignacio (eds.). Ácidos nucleicos: estructuras, propiedades y funciones . Sausalito, California: University Science Books. ISBN 978-0-935702-49-1.
- ^ Balazs, Amber; Davies, Nichola; Longmire, David; Packer, Martin; Chiarparin, Elisabetta (2021). "Conformaciones de ligando libre de RMN y dinámica de resolución atómica". Discusiones sobre resonancia magnética : 1–16. doi : 10.5194/mr-2021-27 . S2CID : 233441183.
- ^ Jonas, J; Jonas, A (junio de 1994). "Espectroscopia de RMN de alta presión de proteínas y membranas". Revisión anual de biofísica y estructura biomolecular . 23 (1): 287–318. doi :10.1146/annurev.bb.23.060194.001443. ISSN 1056-8700.
- ^ Benedek, GB; Purcell, EM (1954-12-01). "Resonancia magnética nuclear en líquidos bajo alta presión". The Journal of Chemical Physics . 22 (12): 2003–2012. doi :10.1063/1.1739982. ISSN 0021-9606.
- ^ Kamatari, Y (septiembre de 2004). "Espectroscopia de RMN de alta presión para caracterizar intermediarios de plegamiento y estados desnaturalizados de proteínas". Métodos . 34 (1): 133–143. doi :10.1016/j.ymeth.2004.03.010.
- ^ Peterson, Ronald W.; Wand, A. Joshua (1 de septiembre de 2005). "Celda de alta presión autónoma, aparato y procedimiento para la preparación de proteínas encapsuladas disueltas en fluidos de baja viscosidad para espectroscopia de resonancia magnética nuclear". Review of Scientific Instruments . 76 (9): 1–7. doi :10.1063/1.2038087. ISSN 0034-6748. PMC 1343520 . PMID 16508692.
- ^ Dubois, Cécile; Herrada, Isaline; Barthe, Philippe; Roumestand, Christian (26 de noviembre de 2020). "Combinación de perturbación de alta presión con espectroscopia de RMN para una caracterización estructural y dinámica de las vías de plegamiento de proteínas". Moléculas . 25 (23): 5551. doi : 10.3390/molecules25235551 . ISSN 1420-3049. PMC 7731413 . PMID 33256081.
- ^ Gagné, Donald; Azad, Roksana; Aramini, James M.; Xu, Xingjian; Isiorho, Eta A.; Edupuganti, Uthama R.; Williams, Justin; Marcelino, Leandro Pimentel; Akasaka, Kazuyuki (26 de agosto de 2020). Uso de espectroscopia de RMN de alta presión para identificar rápidamente proteínas con vacíos internos de unión de ligandos (informe). Biofísica. doi :10.1101/2020.08.25.267195.
- ^ Jodov, IA; Belov, KV; Krestyaninov, MA; Sobornova, VV; Dyshin, AA; Kiselev, MG (agosto de 2023). "¿El DMSO afecta los cambios conformacionales de las moléculas de fármacos en medios de CO2 supercríticos?". Revista de líquidos moleculares . 384 : 122230. doi : 10.1016/j.molliq.2023.122230.
- ^ Jodov, IA; Belov, KV; Dyshin, AA; Krestyaninov, MA; Kiselev, MG (diciembre de 2022). "Efecto de la presión sobre los equilibrios conformacionales de la lidocaína en scCO2: un estudio de 2D NOESY". Revista de líquidos moleculares . 367 : 120525. doi : 10.1016/j.molliq.2022.120525.
Lectura adicional
- John D. Roberts (1959). Resonancia magnética nuclear: aplicaciones a la química orgánica. McGraw-Hill Book Company. ISBN 9781258811662.
- JAPople ; WGSchneider ; HJBernstein (1959). Resonancia magnética nuclear de alta resolución . McGraw-Hill Book Company.
- A. Abragam (1961). Los principios del magnetismo nuclear . Clarendon Press. ISBN 9780198520146.
- Charles P. Slichter (1963). Principios de resonancia magnética: con ejemplos de la física del estado sólido . Harper & Row. ISBN 9783540084761.
- John Emsley ; James Feeney; Leslie Howard Sutcliffe (1965). Espectroscopia de resonancia magnética nuclear de alta resolución . Pergamon. ISBN 9781483184081.
Enlaces externos
- James Keeler. "Understanding NMR Spectroscopy" (reimpreso en la Universidad de Cambridge ) . Universidad de California, Irvine . Consultado el 11 de mayo de 2007 .
- Conceptos básicos de RMN: una descripción general no técnica de la teoría, los equipos y las técnicas de RMN a cargo del Dr. Joseph Hornak, profesor de química en el RIT
- Bibliotecas GAMMA y PyGAMMA: GAMMA es una biblioteca C++ de código abierto escrita para la simulación de experimentos de espectroscopia de resonancia magnética nuclear. PyGAMMA es un contenedor Python para GAMMA.
- relax Software para el análisis de la dinámica de RMN
- Vespa - VeSPA (Versatile Simulation, Pulses and Analysis) es un paquete de software gratuito compuesto por tres aplicaciones Python. Estas herramientas basadas en GUI sirven para la simulación espectral de resonancia magnética (MR), el diseño de pulsos de RF y el procesamiento y análisis espectral de datos de RM.
