Recombinación V(D)J
La recombinación V(D)J (reordenamiento de unión de diversidad variable) es el mecanismo de recombinación somática que ocurre solo en los linfocitos en desarrollo durante las primeras etapas de la maduración de las células T y B. Da lugar al repertorio altamente diverso de anticuerpos/inmunoglobulinas y receptores de células T (TCR) que se encuentran en las células B y las células T , respectivamente. El proceso es una característica definitoria del sistema inmunitario adaptativo .
La recombinación V(D)J en mamíferos ocurre en los órganos linfoides primarios ( médula ósea para las células B y timo para las células T) y de una manera casi aleatoria reorganiza los segmentos de genes variables (V), de unión (J) y, en algunos casos, de diversidad (D). El proceso finalmente da como resultado nuevas secuencias de aminoácidos en las regiones de unión de antígenos de las inmunoglobulinas y los TCR que permiten el reconocimiento de antígenos de casi todos los patógenos, incluidas bacterias , virus , parásitos y gusanos , así como "células propias alteradas" como se ve en el cáncer . El reconocimiento también puede ser de naturaleza alérgica ( por ejemplo , al polen u otros alérgenos ) o puede coincidir con los tejidos del huésped y conducir a la autoinmunidad .
En 1987, Susumu Tonegawa recibió el Premio Nobel de Fisiología o Medicina "por su descubrimiento del principio genético para la generación de diversidad de anticuerpos". [1]
Fondo
Las moléculas de anticuerpos humanos (incluidos los receptores de células B ) están compuestas de cadenas pesadas y ligeras, cada una de las cuales contiene regiones constantes (C) y variables (V), codificadas genéticamente en tres loci :
- El locus pesado de inmunoglobulina ( IGH@ ) en el cromosoma 14, que contiene los segmentos genéticos de la cadena pesada de inmunoglobulina .
- El locus de inmunoglobulina kappa (κ) ( IGK@ ) en el cromosoma 2, que contiene los segmentos genéticos de un tipo (κ) de cadena ligera de inmunoglobulina .
- El locus lambda (λ) de inmunoglobulina ( IGL@ ) en el cromosoma 22, que contiene los segmentos genéticos de otro tipo (λ) de cadena ligera de inmunoglobulina.
Cada gen de cadena pesada o cadena ligera contiene múltiples copias de tres tipos diferentes de segmentos génicos para las regiones variables de las proteínas de anticuerpos. Por ejemplo, la región de la cadena pesada de inmunoglobulina humana contiene 2 segmentos génicos constantes (Cμ y Cδ) y 44 segmentos génicos variables (V), más 27 segmentos génicos de diversidad (D) y 6 segmentos génicos de unión (J). [2] Los genes de la cadena ligera poseen un solo segmento génico constante (Cκ) o cuatro (Cλ) con numerosos segmentos génicos V y J, pero no tienen segmentos génicos D. [3] La reorganización del ADN hace que una copia de cada tipo de segmento génico entre en cualquier linfocito dado, lo que genera un enorme repertorio de anticuerpos; son posibles aproximadamente 3×10 11 combinaciones, aunque algunas se eliminan debido a la autorreactividad.
La mayoría de los receptores de células T están compuestos por una cadena alfa variable y una cadena beta. Los genes de los receptores de células T son similares a los genes de inmunoglobulina en que también contienen múltiples segmentos génicos V, D y J en sus cadenas beta (y segmentos génicos V y J en sus cadenas alfa) que se reorganizan durante el desarrollo del linfocito para proporcionar a esa célula un receptor de antígeno único. El receptor de células T en este sentido es el equivalente topológico de un fragmento de unión al antígeno del anticuerpo, siendo ambos parte de la superfamilia de las inmunoglobulinas.
La respuesta autoinmune se previene eliminando las células que reaccionan por sí mismas. Esto ocurre en el timo al probar la célula contra una serie de antígenos propios expresados a través de la función del regulador autoinmune (AIRE). El locus de la cadena ligera lambda de inmunoglobulina contiene genes codificadores de proteínas que pueden perderse con su reordenamiento. Esto se basa en un mecanismo fisiológico y no es patogénico para las leucemias o los linfomas. Una célula persiste si crea un producto exitoso que no reacciona por sí sola; de lo contrario, se elimina por apoptosis .
Inmunoglobulinas
Cadena pesada
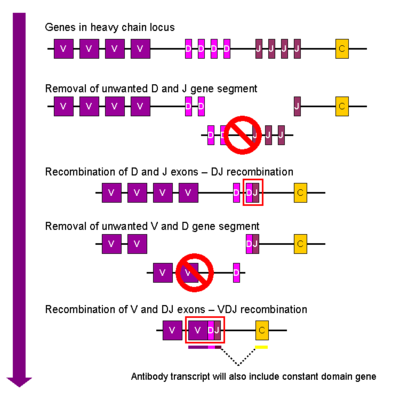
En la célula B en desarrollo , el primer evento de recombinación que ocurre es entre un segmento génico D y uno J del locus de la cadena pesada. Cualquier ADN entre estos dos segmentos génicos se elimina. Esta recombinación DJ es seguida por la unión de un segmento génico V, de una región aguas arriba del complejo DJ recién formado, formando un segmento génico VDJ reorganizado. Todos los demás segmentos génicos entre los segmentos V y D ahora se eliminan del genoma de la célula. Se genera una transcripción primaria (ARN no empalmado) que contiene la región VDJ de la cadena pesada y las cadenas constantes mu y delta (C μ y C δ ). (es decir, la transcripción primaria contiene los segmentos: VDJC μ -C δ ). El ARN primario se procesa para agregar una cola poliadenilada (poli-A) después de la cadena C μ y para eliminar la secuencia entre el segmento VDJ y este segmento génico constante. La traducción de este ARNm conduce a la producción de la proteína de cadena pesada IgM .
Cadena ligera
Las cadenas kappa (κ) y lambda (λ) de los loci de la cadena ligera de inmunoglobulina se reorganizan de una manera muy similar, excepto que las cadenas ligeras carecen de un segmento D. En otras palabras, el primer paso de la recombinación de las cadenas ligeras implica la unión de las cadenas V y J para dar un complejo VJ antes de la adición del gen de la cadena constante durante la transcripción primaria. La traducción del ARNm empalmado para las cadenas kappa o lambda da como resultado la formación de la proteína de cadena ligera Ig κ o Ig λ.
El ensamblaje de la cadena pesada de Ig μ y una de las cadenas ligeras da como resultado la formación de una forma unida a la membrana de la inmunoglobulina IgM que se expresa en la superficie de la célula B inmadura.
Receptores de células T
Durante el desarrollo de los timocitos , las cadenas del receptor de células T (TCR) experimentan esencialmente la misma secuencia de eventos de recombinación ordenada que la descrita para las inmunoglobulinas. La recombinación D a J ocurre primero en la cadena β del TCR. Este proceso puede implicar la unión del segmento génico D β 1 a uno de los seis segmentos J β 1 o la unión del segmento génico D β 2 a uno de los seis segmentos J β 2. [3] La recombinación DJ es seguida (como se mencionó anteriormente) por reordenamientos V β a D β J β . Todos los segmentos génicos entre los segmentos génicos V β -D β -J β en el complejo recién formado se eliminan y se sintetiza la transcripción primaria que incorpora el gen de dominio constante (V β -D β -J β -C β ). La transcripción del ARNm elimina cualquier secuencia intermedia y permite la traducción de la proteína de longitud completa para la cadena β del TCR.
La reorganización de la cadena alfa (α) del TCR sigue a la reorganización de la cadena β y se asemeja a la reorganización de V a J descrita para las cadenas ligeras de Ig (ver arriba). El ensamblaje de las cadenas β y α da como resultado la formación del αβ-TCR que se expresa en la mayoría de las células T.
Mecanismo
Enzimas y componentes clave
El proceso de recombinación V(D)J está mediado por la recombinasa VDJ, que es una colección diversa de enzimas. Las enzimas clave involucradas son los genes activadores de la recombinación 1 y 2 (RAG), la desoxinucleotidil transferasa terminal (TdT) y la nucleasa Artemis , un miembro de la vía ubicua de unión de extremos no homólogos (NHEJ) para la reparación del ADN. [4] Se sabe que varias otras enzimas están involucradas en el proceso e incluyen la proteína quinasa dependiente del ADN (DNA-PK), la proteína 4 de complementación cruzada de reparación de rayos X (XRCC4), la ADN ligasa IV , el factor 1 de unión de extremos no homólogos (NHEJ1; también conocido como Cernunnos o factor similar a XRCC4 [XLF]), el recientemente descubierto Paralog de XRCC4 y XLF (PAXX) y las ADN polimerasas λ y μ. [5] Algunas enzimas involucradas son específicas de los linfocitos ( por ejemplo , RAG, TdT), mientras que otras se encuentran en otros tipos de células e incluso de manera ubicua ( por ejemplo , componentes NHEJ).
Para mantener la especificidad de la recombinación, la recombinasa V(D)J reconoce y se une a las secuencias de señal de recombinación (RSS) que flanquean los segmentos de los genes variable (V), de diversidad (D) y de unión (J). Las RSS están compuestas por tres elementos: un heptámero de siete nucleótidos conservados, una región espaciadora de 12 o 23 pares de bases de longitud y un nonámero de nueve nucleótidos conservados. Si bien la mayoría de las RSS varían en secuencia, las secuencias de consenso del heptámero y el nonámero son CACAGTG y ACAAAAACC, respectivamente; y aunque la secuencia de la región espaciadora está poco conservada, la longitud está muy conservada. [6] [7] La longitud de la región espaciadora corresponde aproximadamente a una (12 pares de bases) o dos vueltas (23 pares de bases) de la hélice de ADN. Siguiendo lo que se conoce como la regla 12/23, los segmentos genéticos que se van a recombinar suelen estar adyacentes a RSS de diferentes longitudes de espaciador ( es decir , uno tiene un "12RSS" y otro tiene un "23RSS"). [8] Esta es una característica importante en la regulación de la recombinación V(D)J. [9]
Proceso
La recombinación V(D)J comienza cuando la recombinasa V(D)J (a través de la actividad de RAG1) se une a un RSS que flanquea un segmento de gen codificante (V, D o J) y crea un corte de cadena sencilla en el ADN entre la primera base del RSS (justo antes del heptámero) y el segmento codificante. Esto es esencialmente neutral desde el punto de vista energético (no necesita hidrólisis de ATP ) y da como resultado la formación de un grupo hidroxilo 3' libre y un grupo fosfato 5' en la misma cadena. La recombinasa coloca el grupo hidroxilo reactivo para atacar el enlace fosfodiéster de la cadena opuesta, formando dos extremos de ADN: una horquilla (tallo-bucle) en el segmento codificante y un extremo romo en el segmento señal. [10] El modelo actual es que el corte del ADN y la formación de la horquilla ocurren en ambas cadenas simultáneamente (o casi) en un complejo conocido como centro de recombinación . [11] [12] [13] [14]
Los extremos romos de la señal se unen para formar una pieza circular de ADN que contiene todas las secuencias intermedias entre los segmentos codificantes, conocida como unión de señal (aunque de naturaleza circular, no debe confundirse con un plásmido ). Si bien en un principio se pensaba que se perdían durante las divisiones celulares sucesivas, hay evidencia de que las uniones de señal pueden volver a ingresar al genoma y provocar patologías al activar oncogenes o interrumpir la función de los genes supresores de tumores [Ref].
Los extremos codificantes se procesan aún más antes de su ligadura mediante varios eventos que finalmente conducen a la diversidad de unión. [15] El procesamiento comienza cuando la DNA-PK se une a cada extremo roto del ADN y recluta a varias otras proteínas, incluidas Artemis, XRCC4, ADN ligasa IV, Cernunnos y varias ADN polimerasas. [16] La DNA-PK forma un complejo que conduce a su autofosforilación , lo que resulta en la activación de Artemis. Las horquillas del extremo codificante se abren por la actividad de Artemis. [17] Si se abren en el centro, resultará un extremo de ADN romo; sin embargo, en muchos casos, la apertura está "descentrada" y da como resultado bases adicionales que permanecen en una hebra (un saliente). Estos se conocen como nucleótidos palindrómicos (P) debido a la naturaleza palindrómica de la secuencia producida cuando las enzimas de reparación del ADN resuelven el saliente. [18] El proceso de apertura de la horquilla por Artemis es un paso crucial de la recombinación V(D)J y es defectuoso en el modelo de ratón con inmunodeficiencia combinada grave (scid) .
A continuación, XRCC4, Cernunnos y DNA-PK alinean los extremos del ADN y reclutan a la desoxinucleotidil transferasa terminal (TdT), una ADN polimerasa independiente de la plantilla que añade nucleótidos no moldeados (N) al extremo codificante. La adición es mayoritariamente aleatoria, pero la TdT muestra una preferencia por los nucleótidos G/C. [19] Como ocurre con todas las ADN polimerasas conocidas, la TdT añade nucleótidos a una hebra en una dirección de 5' a 3'. [20]
Por último, las exonucleasas pueden eliminar bases de los extremos codificantes (incluidos los nucleótidos P o N que se hayan podido formar). Las ADN polimerasas λ y μ luego insertan nucleótidos adicionales según sea necesario para que los dos extremos sean compatibles para la unión. Este es un proceso estocástico, por lo tanto, puede ocurrir cualquier combinación de adición de nucleótidos P y N y eliminación exonucleolítica (o ninguna en absoluto). Finalmente, los extremos codificantes procesados son ligados entre sí por la ADN ligasa IV. [21]
Todos estos eventos de procesamiento dan como resultado un paratopo que es altamente variable, incluso cuando se recombinan los mismos segmentos genéticos. La recombinación V(D)J permite la generación de inmunoglobulinas y receptores de células T para antígenos que ni el organismo ni su(s) ancestro(s) necesitan haber encontrado previamente, lo que permite una respuesta inmune adaptativa a nuevos patógenos que se desarrollan o a aquellos que cambian con frecuencia ( por ejemplo , la gripe estacional ). Sin embargo, una advertencia importante para este proceso es que la secuencia de ADN debe permanecer en marco para mantener la secuencia de aminoácidos correcta en el producto proteico final. Si la secuencia resultante está fuera de marco, el desarrollo de la célula se detendrá y la célula no sobrevivirá hasta la madurez. La recombinación V(D)J es, por lo tanto, un proceso muy costoso que debe ser (y es) estrictamente regulado y controlado.
Véase también
- Receptor de células B
- Receptor de células T
- Instituto de Inmunología de Basilea
- Charles M. Steinberg
- Célula NKT
- Gen activador de la recombinación
Referencias
- ^ «El Premio Nobel de Fisiología o Medicina 1987». nobelprize.org . Archivado desde el original el 13 de febrero de 2021. Consultado el 26 de diciembre de 2014 .
- ^ Li A, Rue M, Zhou J, et al. (junio de 2004). "Utilización de segmentos de genes variables, de diversidad y de unión de la cadena pesada de Ig en niños con leucemia linfoblástica aguda de linaje B: implicaciones para los mecanismos de recombinación VDJ y para la patogénesis". Blood . 103 (12): 4602–9. doi : 10.1182/blood-2003-11-3857 . PMID 15010366.
- ^ ab Abbas, Abul K. (2018). "Desarrollo de linfocitos y reordenamiento de genes de receptores de antígenos". Inmunología celular y molecular (novena edición). Filadelfia, PA: Elsevier. ISBN 978-0-323-47978-3.
- ^ Ma, Yunmei; Lu, Haihui; Schwarz, Klaus; Lieber, Michael (septiembre de 2005). "Reparación de roturas de ADN de doble cadena mediante la vía de unión de extremos de ADN no homólogo humano: el modelo de procesamiento iterativo". Ciclo celular . 4 (9): 1193–1200. doi : 10.4161/cc.4.9.1977 . PMID 16082219.
- ^ Malu, Shruti; Malshetty, Vidyasagar; Francisco, Dailia; Cortés, Patricia (2012). "Papel de la unión de extremos no homólogos en la recombinación V (D) J". Investigación inmunológica . 54 (1–3): 233–246. doi :10.1007/s12026-012-8329-z. PMID 22569912. S2CID 45771818.
- ^ Ramsden, Dale; Baetz, Kristin; Wu, Gillian (1994). "Conservación de la secuencia en espaciadores de secuencias de señales de recombinación". Investigación de ácidos nucleicos . 22 (10): 1785–1796. doi :10.1093/nar/22.10.1785. PMC 308075 . PMID 8208601.
- ^ Cowell, Lindsay; Davila, Marco; Ramsden, Dale; Kelsoe, Garnett (2004). "Herramientas computacionales para comprender la variabilidad de secuencias en señales de recombinación". Reseñas inmunológicas . 200 : 57–69. doi :10.1111/j.0105-2896.2004.00171.x. PMID 15242396. S2CID 40771963.
- ^ van Gent, Dik; Ramsden, Dale; Gellert, Martin (1996). "Las proteínas RAG1 y RAG2 establecen la regla 12/23 en la recombinación V(D)J". Cell . 85 (1): 107–13. doi : 10.1016/s0092-8674(00)81086-7 . PMID 8620529.
- ^ Hiom, Kevin; Gellert, Martin (1998). "Ensamblaje de un complejo de señal emparejado 12/23: un punto de control crítico en la recombinación V(D)J". Molecular Cell . 1 (7): 1011–1019. doi : 10.1016/s1097-2765(00)80101-x . PMID 9651584.
- ^ Schatz, David; Swanson, Patrick (2011). "Recombinación V(D)J: mecanismos de iniciación". Revisión anual de genética . 45 : 167–202. doi :10.1146/annurev-genet-110410-132552. PMID 21854230.
- ^ Schatz, David; Ji, Yanhong (2011). "Centros de recombinación y la orquestación de la recombinación V(D)J". Nature Reviews Immunology . 11 (4): 251–263. doi :10.1038/nri2941. PMID 21394103. S2CID 33489235.
- ^ Curry, John; Geier, Jamie; Schlissel, Mark (2005). "Mella de la secuencia de señal de recombinación monocatenaria in vivo: evidencia de un modelo de captura de sinapsis". Nature Immunology . 6 (12): 1272–1279. doi :10.1038/ni1270. PMID 16286921. S2CID 10975251.
- ^ Agrawal, Alka; Schatz, David (1997). "RAG1 y RAG2 forman un complejo sináptico estable después de la escisión con ADN que contiene extremos señal en la recombinación V(D)J". Cell . 89 (1): 43–53. doi : 10.1016/s0092-8674(00)80181-6 . PMID 9094713.
- ^ Fugmann, Sebastian; Lee, AIfred; Shockett, Penny; Villey, Isabelle; Schatz, David (2000). "Las proteínas RAG y la recombinación V(D)J: complejos, extremos y transposición". Revisión anual de inmunología . 18 : 495–527. doi :10.1146/annurev.immunol.18.1.495. PMID 10837067.
- ^ Lewis, Susanna (1994). "El mecanismo de unión V(D)J: lecciones de análisis moleculares, inmunológicos y comparativos". Advances in Immunology Volumen 56. Vol. 56. págs. 27–150. doi :10.1016/s0065-2776(08)60450-2. ISBN 9780120224562. Número de identificación personal 8073949.
- ^ Helmink, Beth; Sleckman, Barry (2012). "La respuesta y la reparación de las roturas de doble cadena de ADN mediadas por RAG". Revisión anual de inmunología . 30 : 175–202. doi :10.1146/annurev-immunol-030409-101320. PMC 4038028 . PMID 22224778.
- ^ Ma, Yunmei; Schwarz, Klaus; Lieber, Michael (2005). "La endonucleasa Artemis:DNA-PKcs corta bucles, solapas y huecos del ADN". Reparación del ADN . 4 (7): 845–851. doi :10.1016/j.dnarep.2005.04.013. PMID 15936993.
- ^ Lu, Haihui; Schwarz, Klaus; Lieber, Michael (2007). "Hasta qué punto la apertura de la horquilla por el complejo Artemis:DNA-PKcs puede contribuir a la diversidad de la unión en la recombinación V(D)J". Nucleic Acids Research . 35 (20): 6917–6923. doi :10.1093/nar/gkm823. PMC 2175297 . PMID 17932067.
- ^ Gauss, George; Lieber, Michael (1996). "Restricciones mecanicistas sobre la diversidad en la recombinación V(D)J humana". Biología molecular y celular . 16 (1): 258–269. doi :10.1128/MCB.16.1.258. PMC 230999 . PMID 8524303.
- ^ Benedict, Cindy; Gilfillan, Susan; Thai, To-Ha; Kearney, John (2000). "Desoxinucleotidil transferasa terminal y desarrollo del repertorio". Reseñas inmunológicas . 175 : 150–157. doi :10.1111/j.1600-065x.2000.imr017518.x. PMID 10933600. S2CID 20634390.
- ^ van Gent, DC; van der Burg, M (10 de diciembre de 2007). "Unión de extremos no homólogos, un asunto complicado". Oncogene . 26 (56): 7731–40. doi :10.1038/sj.onc.1210871. PMID 18066085.
Lectura adicional
- Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC (2000). Capítulo 24, Evolución a nivel molecular. En: Genetics . Nueva York: McGraw-Hill. pp. 805–807. ISBN 978-0-07-299587-9.
- Recombinación V(D)J. Serie: Avances en medicina y biología experimental, vol. 650 Ferrier, Pierre (Ed.) Landes Bioscience 2009, XII, 199 págs. ISBN 978-1-4419-0295-5