Aminoácido proteinogénico

Los aminoácidos proteinogénicos son aminoácidos que se incorporan biosintéticamente a las proteínas durante la traducción . La palabra "proteinógeno" significa "creador de proteínas". A lo largo de la vida conocida , hay 22 aminoácidos codificados genéticamente (proteinogénicos), 20 en el código genético estándar y otros 2 ( selenocisteína y pirrolisina ) que pueden incorporarse mediante mecanismos especiales de traducción. [1]
Por el contrario, los aminoácidos no proteinogénicos son aquellos que no se incorporan a las proteínas (como el GABA , la L -DOPA o la triyodotironina ), se incorporan incorrectamente en lugar de un aminoácido codificado genéticamente o no se producen directamente y de forma aislada por la maquinaria celular estándar (como la hidroxiprolina ). Esto último suele ser el resultado de la modificación postraduccional de las proteínas. Algunos aminoácidos no proteinogénicos se incorporan a péptidos no ribosómicos que son sintetizados por sintetasas de péptidos no ribosómicos.
Tanto los eucariotas como los procariotas pueden incorporar selenocisteína a sus proteínas a través de una secuencia de nucleótidos conocida como elemento SECIS , que indica a la célula que traduzca un codón UGA cercano como selenocisteína (UGA normalmente es un codón de terminación ). En algunos procariotas metanogénicos , el codón UAG (normalmente un codón de terminación) también puede traducirse a pirrolisina . [2]
En los eucariotas, sólo hay 21 aminoácidos proteinogénicos, los 20 del código genético estándar, más la selenocisteína . Los humanos pueden sintetizar 12 de estos a partir de otros o de otras moléculas del metabolismo intermediario. Los otros nueve deben ser consumidos (normalmente como sus derivados proteicos), por lo que se denominan aminoácidos esenciales . Los aminoácidos esenciales son histidina , isoleucina , leucina , lisina , metionina , fenilalanina , treonina , triptófano y valina (es decir, H, I, L, K, M, F, T, W, V). [3]
Se ha descubierto que los aminoácidos proteinogénicos están relacionados con el conjunto de aminoácidos que pueden ser reconocidos por los sistemas de autoaminoacilación de ribozimas . [4] Por lo tanto, los aminoácidos no proteinogénicos habrían sido excluidos por el éxito evolutivo contingente de las formas de vida basadas en nucleótidos. Se han ofrecido otras razones para explicar por qué ciertos aminoácidos no proteinogénicos específicos generalmente no se incorporan a las proteínas; por ejemplo, la ornitina y la homoserina se ciclan contra la cadena principal del péptido y fragmentan la proteína con vidas medias relativamente cortas , mientras que otros son tóxicos porque pueden incorporarse por error a las proteínas, como el análogo de arginina canavanina .
Se ha sugerido que la selección evolutiva de ciertos aminoácidos proteinogénicos de la sopa primordial se debe a su mejor incorporación a una cadena polipeptídica en oposición a los aminoácidos no proteinogénicos. [5]
Estructuras
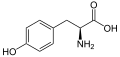
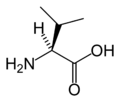
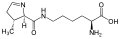
A continuación se muestran las estructuras y abreviaturas de los 21 aminoácidos que están codificados directamente para la síntesis de proteínas por el código genético de los eucariotas. Las estructuras que se indican a continuación son estructuras químicas estándar, no las formas típicas de zwitteriones que existen en soluciones acuosas.

- L -alanina
(Ala/A) - L -Arginina
(Arg/R) - L -Asparagina
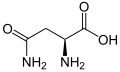
(Asn/N) - Ácido L
-aspártico (Asp/D) - L -Cisteína
(Cys/C) - Ácido L
-glutámico (Glu/E) - L -Glutamina
(Gln/Q) - Glicina
(Gly/G) - L- Histidina
(His/H) - L -Isoleucina
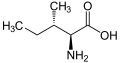
(Ile/I) - L -leucina
(Leu/L) - L -Lisina
(Lys/K) - L -Metionina
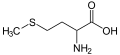
(Met/M) - L -Fenilalanina
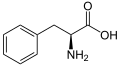
(Phe/F) - L -Prolina
(Pro/P) - L -Serina
(Ser/S) - L -treonina
(Thr/T) - L -Triptófano
(Trp/W) - L -tirosina
(Tyr/Y) - L -Valina
(Val/V)
La IUPAC / IUBMB ahora también recomienda abreviaturas estándar para los siguientes dos aminoácidos:
- L -Selenocisteína
(Sec/U) - L -pirrolizina
(Pyl/O)
Propiedades químicas
A continuación se muestra una tabla que enumera los símbolos de una letra, los símbolos de tres letras y las propiedades químicas de las cadenas laterales de los aminoácidos estándar. Las masas enumeradas se basan en promedios ponderados de los isótopos elementales en sus abundancias naturales . La formación de un enlace peptídico da como resultado la eliminación de una molécula de agua . Por lo tanto, la masa de la proteína es igual a la masa de aminoácidos que la componen menos 18,01524 Da por enlace peptídico.
Propiedades químicas generales
| Aminoácido | Corto | Abreviatura | Masa media ( Da ) | pi | pK 1 (α-COO - ) | pK2 (α - NH3 + ) |
|---|---|---|---|---|---|---|
| Alanina | A | Ala | 89.09404 | 6.01 | 2.35 | 9.87 |
| Cisteína | do | Cis | 121.15404 | 5.05 | 1,92 | 10,70 |
| Ácido aspártico | D | Áspid | 133.10384 | 2,85 | 1,99 | 9,90 |
| Ácido glutámico | mi | pegamento | 147.13074 | 3.15 | 2.10 | 9.47 |
| Fenilalanina | F | fen | 165.19184 | 5.49 | 2.20 | 9.31 |
| Glicina | GRAMO | Gly | 75.06714 | 6.06 | 2.35 | 9,78 |
| Histidina | yo | Su | 155.15634 | 7.60 | 1,80 | 9.33 |
| Isoleucina | I | Isla | 131.17464 | 6.05 | 2.32 | 9,76 |
| Lisina | K | Lis | 146.18934 | 9.60 | 2.16 | 9.06 |
| Leucina | yo | Leu | 131.17464 | 6.01 | 2.33 | 9,74 |
| Metionina | METRO | Conocí | 149.20784 | 5.74 | 2.13 | 9.28 |
| Asparagina | norte | ASN | 132.11904 | 5.41 | 2.14 | 8.72 |
| Pirrolisina | Oh | Pyl | 255.31 | ? | ? | ? |
| Prolina | PAG | Pro | 115.13194 | 6.30 | 1,95 | 10.64 |
| Glutamina | Q | Gln | 146.14594 | 5.65 | 2.17 | 9.13 |
| Arginina | R | Argento | 174.20274 | 10,76 | 1.82 | 8,99 |
| Serina | S | Ser | 105.09344 | 5.68 | 2.19 | 9.21 |
| Treonina | yo | El | 119.12034 | 5.60 | 2.09 | 9.10 |
| Selenocisteína | tú | Segundo | 168.053 | 5.47 | 1.91 | 10 |
| Valina | V | Val | 117.14784 | 6.00 | 2.39 | 9,74 |
| Triptófano | Yo | Trp | 204.22844 | 5.89 | 2.46 | 9.41 |
| Tirosina | Y | Tiro | 181.19124 | 5.64 | 2.20 | 9.21 |
Propiedades de la cadena lateral
| Aminoácido | Corto | Abreviatura | Cadena lateral | Hidrofóbico | pKa § | Polar | pH | Pequeño | Diminuto | Aromático o alifático | Volumen de van der Waals (Å 3 ) |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Alanina | A | Ala | -Canal 3 |  | - |  | - |  |  | Alifático | 67 |
| Cisteína | do | Cis | -CH2SH |  | 8.55 |  | ácido |  |  | - | 86 |
| Ácido aspártico | D | Áspid | -CH2COOH |  | 3.67 |  | ácido |  |  | - | 91 |
| Ácido glutámico | mi | pegamento | -CH2CH2COOH |  | 4.25 |  | ácido |  |  | - | 109 |
| Fenilalanina | F | fen | -CH2C6H5 |  | - |  | - |  |  | Aromático | 135 |
| Glicina | GRAMO | Gly | -H |  | - |  | - |  |  | - | 48 |
| Histidina | yo | Su | -CH2 - C3H3N2 |  | 6.54 |  | básico débil |  |  | Aromático | 118 |
| Isoleucina | I | Isla | -CH ( CH3 ) CH2CH3 |  | - |  | - |  |  | Alifático | 124 |
| Lisina | K | Lis | - ( CH2 ) 4NH2 |  | 10.40 |  | básico |  |  | - | 135 |
| Leucina | yo | Leu | -CH2CH ( CH3 ) 2 |  | - |  | - |  |  | Alifático | 124 |
| Metionina | METRO | Conocí | -CH2CH2SCH3 |  | - |  | - |  |  | Alifático | 124 |
| Asparagina | norte | ASN | -CH2CONH2 |  | - |  | - |  |  | - | 96 |
| Pirrolisina | Oh | Pyl | -(CH 2 ) 4 NHCO C 4 H 5 N CH 3 |  | DAKOTA DEL NORTE |  | básico débil |  |  | - | ? |
| Prolina | PAG | Pro | -CH2CH2CH2 - |  | - |  | - |  |  | - | 90 |
| Glutamina | Q | Gln | -CH2CH2CONH2 |  | - |  | - |  |  | - | 114 |
| Arginina | R | Argento | -(CH2 ) 3NH - C(NH) NH2 |  | 12.3 |  | fuertemente básico |  |  | - | 148 |
| Serina | S | Ser | -CH2OH |  | - |  | - |  |  | - | 73 |
| Treonina | yo | El | -CH(OH) CH3 |  | - |  | - |  |  | - | 93 |
| Selenocisteína | tú | Segundo | -CH2SeH |  | 5.43 |  | ácido |  |  | - | ? |
| Valina | V | Val | -CH( CH3 ) 2 |  | - |  | - |  |  | Alifático | 105 |
| Triptófano | Yo | Trp | -CH2C8H6N |  | - |  | - |  |  | Aromático | 163 |
| Tirosina | Y | Tiro | -CH2 - C6H4OH |  | 9.84 |  | débil ácido |  |  | Aromático | 141 |
§: Los valores de Asp, Cys, Glu, His, Lys y Tyr se determinaron utilizando el residuo de aminoácido ubicado centralmente en un pentapéptido de alanina. [6] El valor de Arg es de Pace et al. (2009). [7] El valor de Sec es de Byun & Kang (2011). [8]
ND: No se ha informado del valor pKa de la pirrolisina.
Nota: El valor de pKa de un residuo de aminoácido en un péptido pequeño suele ser ligeramente diferente cuando se encuentra dentro de una proteína. En ocasiones, se utilizan los cálculos de pKa de proteínas para calcular el cambio en el valor de pKa de un residuo de aminoácido en esta situación.
Expresión genética y bioquímica
| Aminoácido | Corto | Abreviatura | Codón (es) | Aparición | Esencial‡ en humanos | |||
|---|---|---|---|---|---|---|---|---|
| en proteínas arqueanas (%)& | en proteínas bacterianas (%)& | en proteínas eucariotas (%)& | en proteínas humanas (%)& | |||||
| Alanina | A | Ala | GCU, CCG, CAG, GCG | 8.2 | 10.06 | 7.63 | 7.01 | No |
| Cisteína | do | Cis | Universidad Gubernamental, Universidad de Georgia | 0,98 | 0,94 | 1,76 | 2.3 | Condicionalmente |
| Ácido aspártico | D | Áspid | GAU, CAG | 6.21 | 5.59 | 5.4 | 4.73 | No |
| Ácido glutámico | mi | pegamento | GAA, MORDAZA | 7,69 | 6.15 | 6.42 | 7.09 | Condicionalmente |
| Fenilalanina | F | fen | UUUU, UUC | 3.86 | 3.89 | 3.87 | 3,65 | Sí |
| Glicina | GRAMO | Gly | GGU, GGC, GGA, GGG | 7.58 | 7,76 | 6.33 | 6.58 | Condicionalmente |
| Histidina | yo | Su | CAU, CAC | 1,77 | 2.06 | 2.44 | 2.63 | Sí |
| Isoleucina | I | Isla | AUU, AUC, AUA | 7.03 | 5.89 | 5.1 | 4.33 | Sí |
| Lisina | K | Lis | AAA, AAG | 5.27 | 4.68 | 5.64 | 5.72 | Sí |
| Leucina | yo | Leu | UUA, UUG, CUU, CUC, CUA, CUG | 9.31 | 10.09 | 9.29 | 9,97 | Sí |
| Metionina | METRO | Conocí | AGO | 2.35 | 2.38 | 2.25 | 2.13 | Sí |
| Asparagina | norte | ASN | AAU, AAC | 3.68 | 3.58 | 4.28 | 3.58 | No |
| Pirrolisina | Oh | Pyl | *UAG | 0 | 0 | 0 | 0 | No |
| Prolina | PAG | Pro | CCU, CCC, CCA, CCG | 4.26 | 4.61 | 5.41 | 6.31 | No |
| Glutamina | Q | Gln | CAA, CAG | 2.38 | 3.58 | 4.21 | 4.77 | No |
| Arginina | R | Argento | CGU, CGC, CGA, CGG, AGA, AGG | 5.51 | 5.88 | 5.71 | 5.64 | Condicionalmente |
| Serina | S | Ser | UCU, UCC, UCA, UCG, AGU, AGC | 6.17 | 5,85 | 8.34 | 8.33 | No |
| Treonina | yo | El | ACU, ACC, ACA, ACG | 5.44 | 5.52 | 5.56 | 5.36 | Sí |
| Selenocisteína | tú | Segundo | Universidad de Georgia** | 0 | 0 | 0 | >0 | No |
| Valina | V | Val | GUU, GUC, GUA, GUG | 7.8 | 7.27 | 6.2 | 5,96 | Sí |
| Triptófano | Yo | Trp | UGG | 1.03 | 1.27 | 1.24 | 1.22 | Sí |
| Tirosina | Y | Tiro | UAU, UAC | 3.35 | 2,94 | 2.87 | 2.66 | Condicionalmente |
| Codón de terminación† | - | Término | UAA, UAG, UGA†† | ? | ? | ? | — | — |
* UAG es normalmente el codón de terminación ámbar , pero en organismos que contienen la maquinaria biológica codificada por el grupo de genes pylTSBCD se incorporará el aminoácido pirrolisina. [9]
** UGA es normalmente el codón de terminación ópalo (o umber), pero codifica selenocisteína si está presente un elemento SECIS .
† El codón de terminación no es un aminoácido, pero se incluye para completar.
†† UAG y UGA no siempre actúan como codones de terminación (ver arriba).
‡ Un aminoácido esencial no puede ser sintetizado en humanos y, por lo tanto, debe ser suministrado en la dieta. Los aminoácidos condicionalmente esenciales normalmente no son necesarios en la dieta, pero deben ser suministrados exógenamente a poblaciones específicas que no los sintetizan en cantidades adecuadas.
& La ocurrencia de aminoácidos se basa en 135 Archaea, 3775 Bacteria, 614 proteomas Eukaryota y proteoma humano (21 006 proteínas) respectivamente. [10]
Espectrometría de masas
En la espectrometría de masas de péptidos y proteínas, es útil conocer las masas de los residuos. La masa del péptido o proteína es la suma de las masas de los residuos más la masa del agua ( masa monoisotópica = 18,01056 Da; masa media = 18,0153 Da). Las masas de los residuos se calculan a partir de las fórmulas químicas tabuladas y los pesos atómicos. [11] En la espectrometría de masas , los iones también pueden incluir uno o más protones ( masa monoisotópica = 1,00728 Da; masa media* = 1,0074 Da). *Los protones no pueden tener una masa media, esto infiere confusamente que los deuterones son un isótopo válido, pero deberían ser una especie diferente (véase Hydron (química) ).
| Aminoácido | Corto | Abreviatura | Fórmula | Misa del lunes§ ( Da ) | Masa media ( Da ) |
|---|---|---|---|---|---|
| Alanina | A | Ala | C3H5NO | 71.03711 | 71.0779 |
| Cisteína | do | Cis | C3H5NOS | 103.00919 | 103.1429 |
| Ácido aspártico | D | Áspid | C4H5NO3 | 115.02694 | 115.0874 |
| Ácido glutámico | mi | pegamento | C5H7NO3 | 129.04259 | 129.1140 |
| Fenilalanina | F | fen | C9H9NO | 147.06841 | 147.1739 |
| Glicina | GRAMO | Gly | C2H3NO | 57.02146 | 57.0513 |
| Histidina | yo | Su | C6H7N3O | 137.05891 | 137.1393 |
| Isoleucina | I | Isla | C6H11NO | 113.08406 | 113.1576 |
| Lisina | K | Lis | C6H12N2O | 128.09496 | 128.1723 |
| Leucina | yo | Leu | C6H11NO | 113.08406 | 113.1576 |
| Metionina | METRO | Conocí | C 5 H 9 N.º | 131.04049 | 131.1961 |
| Asparagina | norte | ASN | C4H6N2O2 | 114.04293 | 114.1026 |
| Pirrolisina | Oh | Pyl | C12H19N3O2 | 237.14773 | 237.2982 |
| Prolina | PAG | Pro | C5H7NO | 97.05276 | 97.1152 |
| Glutamina | Q | Gln | C5H8N2O2 | 128.05858 | 128.1292 |
| Arginina | R | Argento | C6H12N4O | 156.10111 | 156.1857 |
| Serina | S | Ser | C3H5NO2 | 87.03203 | 87.0773 |
| Treonina | yo | El | C4H7NO2 | 101.04768 | 101.1039 |
| Selenocisteína | tú | Segundo | C 3 H 5 NARIZ | 150.95364 | 150.0489 |
| Valina | V | Val | C5H9NO | 99.06841 | 99.1311 |
| Triptófano | Yo | Trp | C11H10N2O | 186.07931 | 186.2099 |
| Tirosina | Y | Tiro | C9H9NO2 | 163.06333 | 163.1733 |
Estequiometría y coste metabólico en la célula
La siguiente tabla muestra la abundancia de aminoácidos en las células de E. coli y el costo metabólico (ATP) para la síntesis de los aminoácidos. Los números negativos indican que los procesos metabólicos son favorables en términos de energía y no cuestan ATP neto de la célula. [12] La abundancia de aminoácidos incluye aminoácidos en forma libre y en forma de polimerización (proteínas).
| Aminoácido | Corto | Abreviatura | Abundancia (número de moléculas (×10 8 ) por célula de E. coli ) | Coste del ATP en síntesis | |
|---|---|---|---|---|---|
Condiciones aeróbicas | Condiciones anaeróbicas | ||||
| Alanina | A | Ala | 2.9 | -1 | 1 |
| Cisteína | do | Cis | 0,52 | 11 | 15 |
| Ácido aspártico | D | Áspid | 1.4 | 0 | 2 |
| Ácido glutámico | mi | pegamento | 1.5 | -7 | -1 |
| Fenilalanina | F | fen | 1.1 | -6 | 2 |
| Glicina | GRAMO | Gly | 3.5 | -2 | 2 |
| Histidina | yo | Su | 0,54 | 1 | 7 |
| Isoleucina | I | Isla | 1.7 | 7 | 11 |
| Lisina | K | Lis | 2.0 | 5 | 9 |
| Leucina | yo | Leu | 2.6 | -9 | 1 |
| Metionina | METRO | Conocí | 0,88 | 21 | 23 |
| Asparagina | norte | ASN | 1.4 | 3 | 5 |
| Pirrolisina | Oh | Pyl | - | - | - |
| Prolina | PAG | Pro | 1.3 | -2 | 4 |
| Glutamina | Q | Gln | 1.5 | -6 | 0 |
| Arginina | R | Argento | 1.7 | 5 | 13 |
| Serina | S | Ser | 1.2 | -2 | 2 |
| Treonina | yo | El | 1.5 | 6 | 8 |
| Selenocisteína | tú | Segundo | - | - | - |
| Valina | V | Val | 2.4 | -2 | 2 |
| Triptófano | Yo | Trp | 0,33 | -7 | 7 |
| Tirosina | Y | Tiro | 0,79 | -8 | 2 |
Observaciones
| Aminoácido | Abreviatura | Observaciones | |
|---|---|---|---|
| Alanina | A | Ala | Muy abundante y muy versátil, es más rígido que la glicina, pero lo suficientemente pequeño como para plantear sólo pequeños límites estéricos para la conformación de la proteína. Se comporta de forma bastante neutra y puede localizarse tanto en las regiones hidrófilas del exterior de la proteína como en las zonas hidrófobas del interior. |
| Asparagina o ácido aspártico | B | Asc | Un marcador de posición cuando cualquier aminoácido puede ocupar una posición |
| Cisteína | do | Cis | El átomo de azufre se une fácilmente a los iones de metales pesados . En condiciones oxidantes, dos cisteínas pueden unirse en un enlace disulfuro para formar el aminoácido cistina . Cuando las cistinas son parte de una proteína, por ejemplo la insulina , la estructura terciaria se estabiliza, lo que hace que la proteína sea más resistente a la desnaturalización ; por lo tanto, los enlaces disulfuro son comunes en proteínas que tienen que funcionar en entornos hostiles, incluidas las enzimas digestivas (p. ej., pepsina y quimotripsina ) y las proteínas estructurales (p. ej., queratina ). Los disulfuros también se encuentran en péptidos demasiado pequeños para mantener una forma estable por sí solos (p. ej., insulina ). |
| Ácido aspártico | D | Áspid | El aspartato y el glutamato se comportan de manera similar al ácido glutámico y tienen un grupo ácido hidrófilo con una fuerte carga negativa. Por lo general, se encuentran en la superficie externa de la proteína, lo que la hace soluble en agua. Se unen a moléculas e iones con carga positiva y se utilizan a menudo en enzimas para fijar el ión metálico. Cuando se encuentran dentro de la proteína, el aspartato y el glutamato suelen estar emparejados con arginina y lisina. |
| Ácido glutámico | mi | pegamento | El glu se comporta de manera similar al ácido aspártico y tiene una cadena lateral más larga y ligeramente más flexible. |
| Fenilalanina | F | fen | La fenilalanina, la tirosina y el triptófano son esenciales para los seres humanos y contienen un grupo aromático grande y rígido en la cadena lateral. Son los aminoácidos más grandes. Al igual que la isoleucina, la leucina y la valina, son hidrófobos y tienden a orientarse hacia el interior de la molécula de proteína plegada. La fenilalanina se puede convertir en tirosina. |
| Glicina | GRAMO | Gly | Debido a los dos átomos de hidrógeno en el carbono α, la glicina no es ópticamente activa . Es el aminoácido más pequeño, gira fácilmente y agrega flexibilidad a la cadena proteica. Es capaz de encajar en los espacios más estrechos, por ejemplo, la triple hélice del colágeno . Como no suele desearse demasiada flexibilidad, como componente estructural, es menos común que la alanina. |
| Histidina | yo | Su | La histidina es esencial para los seres humanos. Incluso en condiciones ligeramente ácidas, se produce la protonación del nitrógeno, lo que modifica las propiedades de la histidina y del polipéptido en su conjunto. Muchas proteínas la utilizan como mecanismo regulador, modificando la conformación y el comportamiento del polipéptido en regiones ácidas como el endosoma tardío o el lisosoma , lo que obliga a cambiar la conformación de las enzimas. Sin embargo, para ello solo se necesitan unas pocas histidinas, por lo que es comparativamente escasa. |
| Isoleucina | I | Isla | La isoleucina, la leucina y la valina son esenciales para el ser humano. La isoleucina, la leucina y la valina tienen grandes cadenas laterales alifáticas hidrofóbicas. Sus moléculas son rígidas y sus interacciones hidrofóbicas mutuas son importantes para el correcto plegamiento de las proteínas, ya que estas cadenas tienden a estar ubicadas dentro de la molécula de proteína. |
| Leucina o isoleucina | Yo | Xle | Un marcador de posición cuando cualquier aminoácido puede ocupar una posición |
| Lisina | K | Lis | La lisina es esencial para los seres humanos y se comporta de manera similar a la arginina. Contiene una cadena lateral larga y flexible con un extremo cargado positivamente. La flexibilidad de la cadena hace que la lisina y la arginina sean adecuadas para unirse a moléculas con muchas cargas negativas en sus superficies. Por ejemplo, las proteínas que se unen al ADN tienen sus regiones activas ricas en arginina y lisina. La fuerte carga hace que estos dos aminoácidos sean propensos a ubicarse en las superficies hidrófilas externas de las proteínas; cuando se encuentran en el interior, generalmente se combinan con un aminoácido correspondiente con carga negativa, por ejemplo, aspartato o glutamato. |
| Leucina | yo | Leu | La leucina es esencial para los humanos y se comporta de manera similar a la isoleucina y la valina. |
| Metionina | METRO | Conocí | El metilo es esencial para los seres humanos. Siempre es el primer aminoácido que se incorpora a una proteína y, a veces, se elimina después de la traducción. Al igual que la cisteína, contiene azufre, pero con un grupo metilo en lugar de hidrógeno. Este grupo metilo se puede activar y se utiliza en muchas reacciones en las que se añade un nuevo átomo de carbono a otra molécula. |
| Asparagina | norte | ASN | Similar al ácido aspártico, Asn contiene un grupo amida donde Asp tiene un carboxilo . |
| Pirrolisina | Oh | Pyl | Similar a la lisina , pero tiene un anillo de pirrolina unido. |
| Prolina | PAG | Pro | Pro contiene un anillo inusual en el grupo amino del extremo N, que fuerza a la secuencia de amida CO-NH a adoptar una conformación fija. Puede alterar las estructuras de plegamiento de proteínas, como la hélice α o la lámina β , y forzar la torcedura deseada en la cadena proteica. Es común en el colágeno y a menudo sufre una modificación postraduccional a hidroxiprolina . |
| Glutamina | Q | Gln | Al igual que el ácido glutámico, la Gln contiene un grupo amida , mientras que la Glu tiene un grupo carboxilo . Se utiliza en proteínas y como depósito de amoníaco ; es el aminoácido más abundante en el organismo. |
| Arginina | R | Argento | Funcionalmente similar a la lisina. |
| Serina | S | Ser | La serina y la treonina tienen un grupo corto que termina en un grupo hidroxilo. Su hidrógeno es fácil de eliminar, por lo que la serina y la treonina suelen actuar como donantes de hidrógeno en las enzimas. Ambas son muy hidrófilas, por lo que las regiones externas de las proteínas solubles tienden a ser ricas en ellas. |
| Treonina | yo | El | Esencial para los humanos, Thr se comporta de manera similar a la serina. |
| Selenocisteína | tú | Segundo | El análogo de selenio de la cisteína, en el que el selenio reemplaza al átomo de azufre . |
| Valina | V | Val | Esencial para los humanos, Val se comporta de manera similar a la isoleucina y la leucina. |
| Triptófano | Yo | Trp | Esencial para los humanos, el Trp se comporta de manera similar a la fenilalanina y la tirosina. Es un precursor de la serotonina y es naturalmente fluorescente . |
| Desconocido | incógnita | Xaa | Marcador de posición cuando el aminoácido es desconocido o no es importante. |
| Tirosina | Y | Tiro | La tirosina se comporta de manera similar a la fenilalanina (precursora de la tirosina) y al triptófano, y es precursora de la melanina , la epinefrina y las hormonas tiroideas . Naturalmente fluorescente , su fluorescencia suele extinguirse mediante la transferencia de energía a los triptófanos. |
| Ácido glutámico o glutamina | O | Gracias | Un marcador de posición cuando cualquier aminoácido puede ocupar una posición |

Catabolismo
Los aminoácidos se pueden clasificar según las propiedades de sus productos principales: [13]
- Glucogénico, cuyos productos tienen la capacidad de formar glucosa por gluconeogénesis.
- Cetogénico, en el que los productos no tienen la capacidad de formar glucosa: estos productos aún pueden usarse para la cetogénesis o la síntesis de lípidos .
- Aminoácidos catabolizados en productos glucogénicos y cetogénicos.
Véase también
Referencias
- ^ Ambrogelly A, Palioura S, Söll D (enero de 2007). "Expansión natural del código genético". Nature Chemical Biology . 3 (1): 29–35. doi :10.1038/nchembio847. PMID 17173027.
- ^ Lobanov AV, Turanov AA, Hatfield DL, Gladyshev VN (agosto de 2010). "Funciones duales de los codones en el código genético". Critical Reviews in Biochemistry and Molecular Biology . 45 (4): 257–65. doi :10.3109/10409231003786094. PMC 3311535 . PMID 20446809.
- ^ Young VR (agosto de 1994). "Requerimientos de aminoácidos en adultos: argumentos a favor de una revisión importante de las recomendaciones actuales" (PDF) . The Journal of Nutrition . 124 (8 Suppl): 1517S–1523S. doi :10.1093/jn/124.suppl_8.1517S. PMID 8064412.
- ^ Erives A (agosto de 2011). "Un modelo de enzimas de ARN proto-anticodón que requieren homoquiralidad de L-aminoácidos". Journal of Molecular Evolution . 73 (1–2): 10–22. Bibcode :2011JMolE..73...10E. doi :10.1007/s00239-011-9453-4. PMC 3223571 . PMID 21779963.
- ^ Frenkel-Pinter, Moran; Haynes, Jay W.; C, Martin; Petrov, Anton S.; Burcar, Bradley T.; Krishnamurthy, Ramanarayanan; Hud, Nicholas V.; Leman, Luke J.; Williams, Loren Dean (13 de agosto de 2019). "Incorporación selectiva de aminoácidos catiónicos proteínicos sobre no proteínicos en reacciones de oligomerización prebiótica modelo". Actas de la Academia Nacional de Ciencias . 116 (33): 16338–16346. Bibcode :2019PNAS..11616338F. doi : 10.1073/pnas.1904849116 . ISSN 0027-8424. PMC 6697887 . PMID 31358633.
- ^ Thurlkill RL, Grimsley GR, Scholtz JM, Pace CN (mayo de 2006). "Valores pK de los grupos ionizables de proteínas". Protein Science . 15 (5): 1214–8. doi :10.1110/ps.051840806. PMC 2242523 . PMID 16597822.
- ^ Pace CN, Grimsley GR, Scholtz JM (mayo de 2009). "Grupos ionizables de proteínas: valores de pKa y su contribución a la estabilidad y solubilidad de las proteínas". The Journal of Biological Chemistry . 284 (20): 13285–9. doi : 10.1074/jbc.R800080200 . PMC 2679426 . PMID 19164280.
- ^ Byun BJ, Kang YK (mayo de 2011). "Preferencias conformacionales y valor pK(a) del residuo de selenocisteína". Biopolímeros . 95 (5): 345–53. doi :10.1002/bip.21581. PMID 21213257. S2CID 11002236.
- ^ Rother M, Krzycki JA (agosto de 2010). "Selenocisteína, pirrolisina y el metabolismo energético único de las arqueas metanogénicas". Archaea . 2010 : 1–14. doi : 10.1155/2010/453642 . PMC 2933860 . PMID 20847933.
- ^ Kozlowski LP (enero de 2017). "Proteome-pI: base de datos de puntos isoeléctricos del proteoma". Nucleic Acids Research . 45 (D1): D1112–D1116. doi :10.1093/nar/gkw978. PMC 5210655 . PMID 27789699.
- ^ "Pesos atómicos y composiciones isotópicas de todos los elementos". NIST . Consultado el 12 de diciembre de 2016 .
- ^ Phillips R, Kondev J, Theriot J, Garcia HG, Orme N (2013). Biología física de la célula (Segunda edición). Garland Science. pág. 178. ISBN 978-0-8153-4450-6.
- ^ Ferrier DR (2005). "Capítulo 20: Degradación y síntesis de aminoácidos". En Champe PC, Harvey RA, Ferrier DR (eds.). Reseñas ilustradas de Lippincott: bioquímica (Reseñas ilustradas de Lippincott) . Hagerstwon, MD: Lippincott Williams & Wilkins. ISBN 978-0-7817-2265-0.
Referencias generales
- Nelson, David L.; Cox, Michael M. (2000). Principios de bioquímica de Lehninger (3.ª ed.). Worth Publishers. ISBN 978-1-57259-153-0.
- Kyte J, Doolittle RF (mayo de 1982). "Un método simple para mostrar el carácter hidropático de una proteína". Journal of Molecular Biology . 157 (1): 105–32. CiteSeerX 10.1.1.458.454 . doi :10.1016/0022-2836(82)90515-0. PMID 7108955.
- Meierhenrich, Uwe J. (2008). Aminoácidos y asimetría de la vida (1.ª ed.). Springer. ISBN 978-3-540-76885-2.
- Bioquímica, Harpers (2015). Harpers Illustrated Biochemistry (30.ª ed.). Lange. ISBN 978-0-07-182534-4.
Enlaces externos
- El origen del código de una sola letra para los aminoácidos