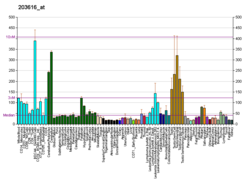
ADN polimerasa beta
| POLB | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificadores | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Alias | POLB , ADN polimerasa beta | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificaciones externas | OMIM : 174760; MGI : 97740; HomoloGene : 2013; Tarjetas genéticas : POLB; OMA :POLB - ortólogos | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidatos | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Elemento regulador del bucle de tallo II en POLB | |
|---|---|
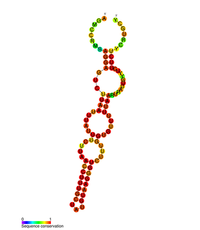 Estructura secundaria prevista del elemento regulador del bucle de tallo II (M2) en POLB | |
| Identificadores | |
| Símbolo | POLB |
| RFAM | RF01455 |
| Gen NCBI | 5423 |
| HGNC | 9174 |
| OMI | 174760 |
| Secuencia de referencia | NM_002690 |
| Otros datos | |
| Tipo de ARN | Cis-reg |
| Dominio(s) | Mamíferos |
| Lugar | Cap. 8 pág. 11.2 |
| Estructuras del PDB | PDBe |
La ADN polimerasa beta , también conocida como POLB , es una enzima presente en eucariotas . En los humanos, está codificada por el gen POLB . [4]
Función
En las células eucariotas , la ADN polimerasa beta (POLB) realiza la reparación por escisión de bases (BER) necesaria para el mantenimiento del ADN , la replicación , la recombinación y la resistencia a los fármacos. [4]
El ADN mitocondrial de las células de mamíferos está constantemente bajo ataque de los radicales de oxígeno liberados durante la producción de ATP . Las mitocondrias de las células de mamíferos contienen un sistema eficiente de reparación por escisión de bases que emplea POLB y que elimina algunos de los frecuentes daños oxidativos del ADN . [5] Por lo tanto, POLB tiene un papel clave en el mantenimiento de la estabilidad del genoma mitocondrial . [5]
Un análisis de la fidelidad de la replicación del ADN por la polimerasa beta en las neuronas de ratones jóvenes y muy viejos indicó que el envejecimiento no tiene un efecto significativo en la fidelidad de la síntesis del ADN por la polimerasa beta. [6] Se consideró que este hallazgo proporcionaba evidencia contra la teoría de la catástrofe de errores del envejecimiento. [6] [7]
Reparación por escisión de base
Cabelof et al. midieron la capacidad de reparar el daño del ADN por la vía BER en tejidos de ratones jóvenes (de 4 meses) y viejos (de 24 meses). [8] En todos los tejidos examinados ( cerebro , hígado , bazo y testículos ), la capacidad de reparar el daño del ADN disminuyó significativamente con la edad, y la reducción en la capacidad de reparación se correlacionó con niveles disminuidos de ADN polimerasa beta tanto a nivel de proteína como de ARN mensajero . Numerosos investigadores han informado una acumulación de daño del ADN con la edad, especialmente en el cerebro y el hígado. [9] Cabelof et al. [8] sugirieron que la incapacidad de la vía BER para reparar daños a lo largo del tiempo puede proporcionar una explicación mecanicista para las frecuentes observaciones de acumulación de daño en el ADN con la edad.
Regulación de la expresión
La ADN polimerasa beta mantiene la integridad del genoma al participar en la reparación por escisión de bases . La sobreexpresión del ARNm de POLB se ha correlacionado con varios tipos de cáncer, mientras que las deficiencias en POLB resultan en hipersensibilidad a los agentes alquilantes , apoptosis inducida y ruptura cromosómica. Por lo tanto, es esencial que la expresión de POLB esté estrictamente regulada. [10] [11] [12] [13]
El gen POLB se regula positivamente por la unión del factor de transcripción CREB1 al elemento de respuesta a AMPc (CRE) presente en el promotor del gen POLB en respuesta a la exposición a agentes alquilantes. [14] [15] La expresión del gen POLB también se regula a nivel postranscripcional, ya que se ha demostrado que el 3' UTR del ARNm de POLB contiene tres estructuras de tallo y bucle que influyen en la expresión génica. [16] Estas estructuras de tres tallos y bucles se conocen como M1, M2 y M3, donde M2 y M3 tienen un papel clave en la regulación génica. M3 contribuye a la expresión génica, ya que contiene la señal de poliadenilación seguida del sitio de escisión y poliadenilación, contribuyendo así al procesamiento del pre-ARNm . Se ha demostrado que M2 se conserva evolutivamente y, a través de la mutagénesis, se demostró que esta estructura de tallo y bucle actúa como un elemento desestabilizador del ARN.
Además de estos elementos reguladores cis presentes en el 3'UTR, se cree que una proteína transactiva , HAX1 , contribuye a la regulación de la expresión génica. Los ensayos de tres híbridos en levadura han demostrado que esta proteína se une a los bucles de tallo dentro del 3'UTR del ARNm de POLB, sin embargo, el mecanismo exacto por el cual esta proteína regula la expresión génica aún está por determinar.
Interacciones
Se ha demostrado que la ADN polimerasa beta interactúa con PNKP [17] y XRCC1 . [18] [19] [20] [21]
Véase también
Referencias
- ^ abc GRCm38: Lanzamiento de Ensembl 89: ENSMUSG00000031536 – Ensembl , mayo de 2017
- ^ "Referencia de PubMed humana:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ "Referencia de PubMed sobre ratón". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ ab "NCBI Gene: ADN polimerasa beta".
- ^ ab Prasad R, Çağlayan M, Dai DP, Nadalutti CA, Zhao ML, Gassman NR, et al. (Diciembre de 2017). "ADN polimerasa β: un eslabón perdido de la maquinaria de reparación por escisión de bases en las mitocondrias de los mamíferos". Reparación del ADN . 60 : 77–88. doi :10.1016/j.dnarep.2017.10.011. PMC 5919216 . PMID 29100041.
- ^ ab Subba Rao K, Martin GM, Loeb LA (octubre de 1985). "Fidelidad de la ADN polimerasa beta en neuronas de ratones jóvenes y muy viejos". Journal of Neurochemistry . 45 (4): 1273–8. doi :10.1111/j.1471-4159.1985.tb05553.x. PMID 3161998. S2CID 84448241.
- ^ Orgel LE (junio de 1973). "Envejecimiento de clones de células de mamíferos". Nature . 243 (5408): 441–5. Bibcode :1973Natur.243..441O. doi :10.1038/243441a0. PMID 4591306. S2CID 4153800.
- ^ ab Cabelof DC, Raffoul JJ, Yanamadala S, Ganir C, Guo Z, Heydari AR (marzo de 2002). "Atenuación de la reparación por escisión de bases dependiente de la ADN polimerasa beta y aumento de la mutagenicidad inducida por DMS en ratones viejos". Mutation Research . 500 (1–2): 135–45. doi :10.1016/s0027-5107(02)00003-9. PMC 3339152 . PMID 11890943.
- ^ Bernstein C, Bernstein H (1991). Envejecimiento, sexo y reparación del ADN . San Diego: Academic Press. págs. 46-60. ISBN 0-12-092860-4.
- ^ Canitrot Y, Cazaux C, Fréchet M, Bouayadi K, Lesca C, Salles B, Hoffmann JS (octubre de 1998). "La sobreexpresión de la ADN polimerasa beta en células produce un fenotipo mutador y una menor sensibilidad a los fármacos contra el cáncer". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 95 (21): 12586–90. Bibcode :1998PNAS...9512586C. doi : 10.1073/pnas.95.21.12586 . PMC 22874 . PMID 9770529.
- ^ Bergoglio V, Pillaire MJ, Lacroix-Triki M, Raynaud-Messina B, Canitrot Y, Bieth A, et al. (junio de 2002). "La ADN polimerasa beta desregulada induce inestabilidad cromosómica y tumorogénesis". Cancer Research . 62 (12): 3511–4. PMID 12067997.
- ^ Bergoglio V, Canitrot Y, Hogarth L, Minto L, Howell SB, Cazaux C, Hoffmann JS (septiembre de 2001). "Expresión y actividad mejoradas de la ADN polimerasa beta en células tumorales de ovario humano: impacto en la sensibilidad hacia agentes antitumorales". Oncogene . 20 (43): 6181–7. doi :10.1038/sj.onc.1204743. PMID 11593426. S2CID 30039904.
- ^ Srivastava DK, Husain I, Arteaga CL, Wilson SH (junio de 1999). "Diferencias en la expresión de la ADN polimerasa beta en tumores humanos seleccionados y líneas celulares". Carcinogénesis . 20 (6): 1049–54. doi : 10.1093/carcin/20.6.1049 . PMID 10357787.
- ^ He F, Yang XP, Srivastava DK, Wilson SH (enero de 2003). "Expresión del gen de la ADN polimerasa beta: el activador del promotor CREB-1 se regula positivamente en células de ovario de hámster chino por estrés inducido por agentes alquilantes de ADN". Química biológica . 384 (1): 19–23. doi :10.1515/BC.2003.003. PMID 12674496. S2CID 33798724.
- ^ Narayan S, He F, Wilson SH (agosto de 1996). "Activación del promotor de la ADN polimerasa beta humana por un agente alquilante de ADN a través de la fosforilación inducida de la proteína 1 de unión al elemento de respuesta al AMPc". The Journal of Biological Chemistry . 271 (31): 18508–13. doi : 10.1074/jbc.271.31.18508 . PMID 8702497.
- ^ Sarnowska E, Grzybowska EA, Sobczak K, Konopinski R, Wilczynska A, Szwarc M, et al. (2007). "La estructura de horquilla dentro del 3'UTR del ARNm de la ADN polimerasa beta actúa como un elemento regulador postranscripcional e interactúa con Hax-1". Nucleic Acids Research . 35 (16): 5499–510. doi :10.1093/nar/gkm502. PMC 2018635 . PMID 17704138.
- ^ Whitehouse CJ, Taylor RM, Thistlethwaite A, Zhang H, Karimi-Busheri F, Lasko DD, et al. (enero de 2001). "XRCC1 estimula la actividad de la polinucleótido quinasa humana en los extremos del ADN dañado y acelera la reparación de roturas de cadena sencilla del ADN". Cell . 104 (1): 107–17. doi : 10.1016/S0092-8674(01)00195-7 . PMID 11163244. S2CID 1487128.
- ^ Wang L, Bhattacharyya N, Chelsea DM, Escobar PF, Banerjee S (noviembre de 2004). "Una nueva proteína nuclear, MGC5306, interactúa con la ADN polimerasa beta y tiene un papel potencial en el fenotipo celular". Cancer Research . 64 (21): 7673–7. doi :10.1158/0008-5472.CAN-04-2801. PMID 15520167. S2CID 35569215.
- ^ Fan J, Otterlei M, Wong HK, Tomkinson AE, Wilson DM (2004). "XRCC1 co-localiza e interactúa físicamente con PCNA". Investigación de ácidos nucleicos . 32 (7): 2193–201. doi :10.1093/nar/gkh556. PMC 407833 . PMID 15107487.
- ^ Kubota Y, Nash RA, Klungland A, Schär P, Barnes DE, Lindahl T (diciembre de 1996). "Reconstitución de la reparación por escisión de bases de ADN con proteínas humanas purificadas: interacción entre la ADN polimerasa beta y la proteína XRCC1". The EMBO Journal . 15 (23): 6662–70. doi :10.1002/j.1460-2075.1996.tb01056.x. PMC 452490 . PMID 8978692.
- ^ Bhattacharyya N, Banerjee S (julio de 2001). "Un nuevo papel de XRCC1 en las funciones de una variante beta de la ADN polimerasa". Bioquímica . 40 (30): 9005–13. doi :10.1021/bi0028789. PMID 11467963.
Lectura adicional
- Date T, Tanihara K, Yamamoto S, Nomura N, Matsukage A (septiembre de 1992). "Dos regiones del ARNm de la ADN polimerasa beta humana suprimen la traducción en Escherichia coli". Nucleic Acids Research . 20 (18): 4859–64. doi :10.1093/nar/20.18.4859. PMC 334243 . PMID 1408801.
- Wang L, Patel U, Ghosh L, Banerjee S (septiembre de 1992). "Mutaciones de la ADN polimerasa beta en el cáncer colorrectal humano". Cancer Research . 52 (17): 4824–7. PMID 1511447.
- Tokui T, Inagaki M, Nishizawa K, Yatani R, Kusagawa M, Ajiro K, et al. (junio de 1991). "Inactivación de la ADN polimerasa beta mediante fosforilación in vitro con proteína quinasa C". The Journal of Biological Chemistry . 266 (17): 10820–4. doi : 10.1016/S0021-9258(18)99092-7 . PMID 2040602.
- SenGupta DN, Zmudzka BZ, Kumar P, Cobianchi F, Skowronski J, Wilson SH (abril de 1986). "Secuencia del ARNm de la ADN polimerasa beta humana obtenida mediante clonación de ADNc". Comunicaciones de investigación bioquímica y biofísica . 136 (1): 341–7. doi :10.1016/0006-291X(86)90916-2. PMID 2423078.
- Zmudzka BZ, Fornace A, Collins J, Wilson SH (octubre de 1988). "Caracterización del ARNm de la ADN polimerasa beta: ciclo celular y respuesta de crecimiento en células humanas cultivadas". Nucleic Acids Research . 16 (20): 9587–96. doi :10.1093/nar/16.20.9587. PMC 338765 . PMID 2460824.
- Widen SG, Kedar P, Wilson SH (noviembre de 1988). "Gen de la beta-polimerasa humana. Estructura de la región flanqueante 5' y promotor activo". The Journal of Biological Chemistry . 263 (32): 16992–8. doi : 10.1016/S0021-9258(18)37488-X . PMID 3182828.
- Abbotts J, SenGupta DN, Zmudzka B, Widen SG, Notario V, Wilson SH (febrero de 1988). "Expresión de la ADN polimerasa beta humana en Escherichia coli y caracterización de la enzima recombinante". Bioquímica . 27 (3): 901–9. doi :10.1021/bi00403a010. PMID 3284575.
- Dobashi Y, Kubota Y, Shuin T, Torigoe S, Yao M, Hosaka M (abril de 1995). "Polimorfismos en el gen de la ADN polimerasa beta humana". Human Genetics . 95 (4): 389–90. doi :10.1007/bf00208961. PMID 7705833. S2CID 22658070.
- Chyan YJ, Ackerman S, Shepherd NS, McBride OW, Widen SG, Wilson SH, Wood TG (julio de 1994). "La estructura del gen de la ADN polimerasa beta humana. Evidencia de empalme alternativo en la expresión génica". Nucleic Acids Research . 22 (14): 2719–25. doi :10.1093/nar/22.14.2719. PMC 308239 . PMID 7914364.
- Maruyama K, Sugano S (enero de 1994). "Oligo-capping: un método simple para reemplazar la estructura de capuchón de los ARNm eucariotas con oligorribonucleótidos". Gene . 138 (1–2): 171–4. doi :10.1016/0378-1119(94)90802-8. PMID 8125298.
- Chang M, Burmer GC, Sweasy J, Loeb LA, Edelhoff S, Disteche CM, et al. (mayo de 1994). "Evidencia contra la ADN polimerasa beta como gen candidato para el síndrome de Werner". Human Genetics . 93 (5): 507–12. doi :10.1007/bf00202813. PMID 8168825. S2CID 34824853.
- Chyan YJ, Strauss PR, Wood TG, Wilson SH (agosto de 1996). "Identificación de nuevas isoformas de ARNm para la ADN polimerasa beta humana". ADN y biología celular . 15 (8): 653–9. doi :10.1089/dna.1996.15.653. PMID 8769567.
- Pelletier H, Sawaya MR, Wolfle W, Wilson SH, Kraut J (octubre de 1996). "Estructuras cristalinas de la ADN polimerasa beta humana complejada con ADN: implicaciones para el mecanismo catalítico, la procesividad y la fidelidad". Biochemistry . 35 (39): 12742–61. doi :10.1021/bi952955d. PMID 8841118.
- Pelletier H, Sawaya MR, Wolfle W, Wilson SH, Kraut J (octubre de 1996). "Una base estructural para la mutagenicidad de iones metálicos y la selectividad de nucleótidos en la ADN polimerasa beta humana". Biochemistry . 35 (39): 12762–77. doi :10.1021/bi9529566. PMID 8841119.
- Pelletier H, Sawaya MR (octubre de 1996). "Caracterización de los motivos de unión de iones metálicos en hélice-horquilla-hélice en la ADN polimerasa beta humana mediante análisis estructural con rayos X". Biochemistry . 35 (39): 12778–87. doi :10.1021/bi960790i. PMID 8841120.
- Kubota Y, Nash RA, Klungland A, Schär P, Barnes DE, Lindahl T (diciembre de 1996). "Reconstitución de la reparación por escisión de bases de ADN con proteínas humanas purificadas: interacción entre la ADN polimerasa beta y la proteína XRCC1". The EMBO Journal . 15 (23): 6662–70. doi :10.1002/j.1460-2075.1996.tb01056.x. PMC 452490 . PMID 8978692.
- Bennett RA, Wilson DM, Wong D, Demple B (julio de 1997). "Interacción de la endonucleasa apurínica humana y la ADN polimerasa beta en la vía de reparación por escisión de bases". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 94 (14): 7166–9. Bibcode :1997PNAS...94.7166B. doi : 10.1073/pnas.94.14.7166 . PMC 23779 . PMID 9207062.
- Sawaya MR, Prasad R, Wilson SH, Kraut J, Pelletier H (septiembre de 1997). "Estructuras cristalinas de la ADN polimerasa beta humana complejada con ADN con huecos y muescas: evidencia de un mecanismo de ajuste inducido". Biochemistry . 36 (37): 11205–15. doi :10.1021/bi9703812. PMID 9287163.
- Bhattacharyya N, Banerjee S (septiembre de 1997). "Una variante de la ADN polimerasa beta actúa como un mutante negativo dominante". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 94 (19): 10324–9. Bibcode :1997PNAS...9410324B. doi : 10.1073/pnas.94.19.10324 . PMC 23361 . PMID 9294209.
- Suzuki Y, Yoshitomo-Nakagawa K, Maruyama K, Suyama A, Sugano S (octubre de 1997). "Construcción y caracterización de una biblioteca de ADNc enriquecida en longitud completa y enriquecida en el extremo 5'". Gene . 200 (1–2): 149–56. doi :10.1016/S0378-1119(97)00411-3. PMID 9373149.
- Tan XH, Zhao M, Pan KF, Dong Y, Dong B, Feng GJ, et al. (marzo de 2005). "Mutación frecuente relacionada con la sobreexpresión de la ADN polimerasa beta en tumores primarios y lesiones precancerosas del estómago humano". Cancer Letters . 220 (1): 101–14. doi :10.1016/j.canlet.2004.07.049. PMID 15737693.
Enlaces externos
- Entrada Rfam para el elemento regulador del bucle de tallo II (M2) en POLB