Nucleosoma

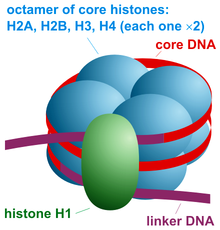
Un nucleosoma es la unidad estructural básica del empaquetamiento del ADN en eucariotas . La estructura de un nucleosoma consiste en un segmento de ADN enrollado alrededor de ocho proteínas histonas [1] y se asemeja a un hilo enrollado alrededor de un carrete. El nucleosoma es la subunidad fundamental de la cromatina . Cada nucleosoma está compuesto por un poco menos de dos vueltas de ADN enrolladas alrededor de un conjunto de ocho proteínas llamadas histonas, que se conocen como octámero de histonas . Cada octámero de histonas está compuesto por dos copias de cada una de las proteínas histonas H2A , H2B , H3 y H4 .
El ADN debe compactarse en nucleosomas para caber dentro del núcleo celular . [2] Además de la envoltura de los nucleosomas, la cromatina eucariota se compacta aún más al plegarse en una serie de estructuras más complejas, formando finalmente un cromosoma . Cada célula humana contiene alrededor de 30 millones de nucleosomas. [3]
Se cree que los nucleosomas portan información heredada epigenéticamente en forma de modificaciones covalentes de sus histonas centrales . Las posiciones de los nucleosomas en el genoma no son aleatorias, y es importante saber dónde se ubica cada nucleosoma porque esto determina la accesibilidad del ADN a las proteínas reguladoras . [4]
Los nucleosomas fueron observados por primera vez como partículas en el microscopio electrónico por Don y Ada Olins en 1974, [5] y su existencia y estructura (como octámeros de histonas rodeados por aproximadamente 200 pares de bases de ADN) fueron propuestas por Roger Kornberg . [6] [7] El papel del nucleosoma como regulador de la transcripción fue demostrado por Lorch et al. in vitro [8] en 1987 y por Han y Grunstein [9] y Clark-Adams et al. [10] in vivo en 1988.
La partícula central del nucleosoma consta de aproximadamente 146 pares de bases (pb) de ADN [11] envueltos en 1,67 vueltas superhelicoidales levógiras alrededor de un octámero de histonas , que consta de 2 copias de cada una de las histonas centrales H2A , H2B , H3 y H4 . [12] Las partículas centrales están conectadas por tramos de ADN de enlace , que pueden tener una longitud de hasta aproximadamente 80 pb. Técnicamente, un nucleosoma se define como la partícula central más una de estas regiones de enlace; sin embargo, la palabra a menudo es sinónimo de la partícula central. [13] Ahora hay mapas de posicionamiento de nucleosomas de todo el genoma disponibles para muchos organismos modelo y células humanas. [14]
Las histonas de enlace, como H1 y sus isoformas, participan en la compactación de la cromatina y se ubican en la base del nucleosoma cerca de la entrada y salida del ADN, uniéndose a la región de enlace del ADN. [15] Los nucleosomas no condensados sin la histona de enlace se asemejan a "cuentas en una cadena de ADN" bajo un microscopio electrónico . [16]
A diferencia de la mayoría de las células eucariotas, los espermatozoides maduros utilizan en gran medida protaminas para empaquetar su ADN genómico, probablemente para lograr una proporción de empaquetamiento aún mayor. [17] También se han encontrado equivalentes de histonas y una estructura de cromatina simplificada en Archaea , [18] lo que sugiere que los eucariotas no son los únicos organismos que usan nucleosomas.
Estructura
Estructura de la partícula central

Descripción general
Los estudios estructurales pioneros realizados en la década de 1980 por el grupo de Aaron Klug proporcionaron la primera evidencia de que un octámero de proteínas histonas envuelve el ADN alrededor de sí mismo en aproximadamente 1,7 vueltas de una superhélice levógira. [19] En 1997, el grupo de Richmond resolvió la primera estructura cristalina del nucleosoma con resolución casi atómica, mostrando los detalles más importantes de la partícula. El ADN palindrómico del satélite alfa humano , crítico para lograr la estructura cristalina del nucleosoma de 1997, fue desarrollado por el grupo de Bunick en el Laboratorio Nacional de Oak Ridge en Tennessee. [20] [21] [22] [23] [24] Hasta la fecha se han resuelto las estructuras de más de 20 partículas de núcleo de nucleosoma diferentes, [25] incluidas las que contienen variantes de histonas e histonas de diferentes especies. La estructura de la partícula central del nucleosoma está notablemente conservada, e incluso un cambio de más de 100 residuos entre las histonas de rana y de levadura da como resultado mapas de densidad electrónica con una desviación cuadrática media general de solo 1,6Å. [26]
La partícula central del nucleosoma (NCP)
La partícula central del nucleosoma (mostrada en la figura) consta de aproximadamente 146 pares de bases de ADN [11] envueltos en 1,67 vueltas superhelicoidales levógiras alrededor del octámero de histonas , que consta de 2 copias de cada una de las histonas centrales H2A , H2B , H3 y H4 . Los nucleosomas adyacentes están unidos por un tramo de ADN libre denominado ADN de enlace (que varía de 10 a 80 pb de longitud según la especie y el tipo de tejido [18] ). Toda la estructura genera un cilindro de 11 nm de diámetro y 5,5 nm de altura.


Las partículas del núcleo del nucleosoma se observan cuando se trata la cromatina en interfase para hacer que se despliegue parcialmente. La imagen resultante, a través de un microscopio electrónico, es "cuentas en una cuerda". La cuerda es el ADN, mientras que cada cuenta en el nucleosoma es una partícula del núcleo. La partícula del núcleo del nucleosoma está compuesta de ADN y proteínas histonas. [29]
La digestión parcial de la cromatina por la ADNasa revela su estructura nucleosomal. Debido a que las porciones de ADN de las partículas centrales del nucleosoma son menos accesibles para la ADNasa que las secciones de enlace, el ADN se digiere en fragmentos de longitudes iguales a la multiplicidad de distancias entre nucleosomas (180, 360, 540 pares de bases, etc.). Por lo tanto, durante la electroforesis en gel de ese ADN se observa un patrón muy característico similar a una escalera . [27] Dicha digestión también puede ocurrir en condiciones naturales durante la apoptosis ("suicidio celular" o muerte celular programada), porque la autodestrucción del ADN es típicamente su función. [30]
Interacciones de proteínas dentro del nucleosoma
Las proteínas histonas centrales contienen un motivo estructural característico denominado "pliegue de histonas", que consta de tres hélices alfa (α1-3) separadas por dos bucles (L1-2). En solución, las histonas forman heterodímeros H2A-H2B y heterotetrámeros H3-H4. Las histonas dimerizan alrededor de sus largas hélices α2 en una orientación antiparalela y, en el caso de H3 y H4, dos de estos dímeros forman un haz de 4 hélices estabilizado por una extensa interacción H3-H3'. El dímero H2A/H2B se une al tetrámero H3/H4 debido a las interacciones entre H4 y H2B, que incluyen la formación de un grupo hidrofóbico. [12] El octámero de histonas está formado por un tetrámero central H3/H4 intercalado entre dos dímeros H2A/H2B. Debido a la carga altamente básica de las cuatro histonas centrales, el octámero de histonas es estable solo en presencia de ADN o concentraciones de sal muy altas.
Interacciones entre histonas y ADN
El nucleosoma contiene más de 120 interacciones directas proteína-ADN y varios cientos de interacciones mediadas por agua. [31] Las interacciones directas proteína-ADN no se distribuyen uniformemente sobre la superficie del octámero, sino que se ubican en sitios discretos. Esto se debe a la formación de dos tipos de sitios de unión al ADN dentro del octámero: el sitio α1α1, que utiliza la hélice α1 de dos histonas adyacentes, y el sitio L1L2 formado por los bucles L1 y L2. Los enlaces salinos y los enlaces de hidrógeno entre los grupos básicos e hidroxilo de la cadena lateral y las amidas de la cadena principal con los fosfatos de la cadena principal del ADN forman la mayor parte de las interacciones con el ADN. Esto es importante, dado que la distribución ubicua de los nucleosomas a lo largo de los genomas requiere que sea un factor de unión al ADN no específico de la secuencia. Aunque los nucleosomas tienden a preferir algunas secuencias de ADN sobre otras, [32] son capaces de unirse prácticamente a cualquier secuencia, lo que se cree que se debe a la flexibilidad en la formación de estas interacciones mediadas por agua. Además, se realizan interacciones no polares entre las cadenas laterales de las proteínas y los grupos desoxirribosa, y una cadena lateral de arginina se intercala en el surco menor del ADN en los 14 sitios donde se enfrenta a la superficie del octámero. La distribución y la fuerza de los sitios de unión del ADN sobre la superficie del octámero distorsionan el ADN dentro del núcleo del nucleosoma. El ADN no está doblado de manera uniforme y también contiene defectos de torsión. La torsión del ADN en forma B libre en solución es de 10,5 pb por vuelta. Sin embargo, la torsión total del ADN nucleosómico es de solo 10,2 pb por vuelta, variando de un valor de 9,4 a 10,9 pb por vuelta.
Dominios de cola de histonas
Las extensiones de la cola de las histonas constituyen hasta el 30% en masa de las histonas, pero no son visibles en las estructuras cristalinas de los nucleosomas debido a su alta flexibilidad intrínseca, y se ha pensado que están en gran parte desestructuradas. [33] Las colas N-terminales de las histonas H3 y H2B pasan a través de un canal formado por los surcos menores de las dos hebras de ADN, que sobresalen del ADN cada 20 pb. La cola N-terminal de la histona H4, por otro lado, tiene una región de aminoácidos altamente básicos (16-25), que, en la estructura cristalina, forma una interacción con la región de superficie altamente ácida de un dímero H2A-H2B de otro nucleosoma, siendo potencialmente relevante para la estructura de orden superior de los nucleosomas. Se piensa que esta interacción ocurre también en condiciones fisiológicas, y sugiere que la acetilación de la cola H4 distorsiona la estructura de orden superior de la cromatina. [ cita requerida ]
Estructura de orden superior

La organización del ADN que se logra mediante el nucleosoma no puede explicar completamente el empaquetamiento del ADN observado en el núcleo celular. Es necesaria una mayor compactación de la cromatina en el núcleo celular, pero aún no se comprende bien. El conocimiento actual [25] es que los nucleosomas repetidos con ADN "enlazador" intermedio forman una fibra de 10 nm , descrita como "cuentas en una cuerda", y tienen una relación de empaquetamiento de aproximadamente cinco a diez. [18] Una cadena de nucleosomas se puede organizar en una fibra de 30 nm , una estructura compactada con una relación de empaquetamiento de ~50 [18] y cuya formación depende de la presencia de la histona H1 .
Se ha presentado una estructura cristalina de un tetranucleosoma y se ha utilizado para construir una estructura propuesta de la fibra de 30 nm como una hélice de dos inicios. [34] Todavía hay una cierta cantidad de controversia con respecto a este modelo, ya que es incompatible con los datos recientes de microscopía electrónica . [35] Más allá de esto, la estructura de la cromatina es poco entendida, pero se sugiere clásicamente que la fibra de 30 nm está dispuesta en bucles a lo largo de un andamiaje proteico central para formar eucromatina transcripcionalmente activa . Una mayor compactación conduce a heterocromatina transcripcionalmente inactiva .
Dinámica
Aunque el nucleosoma es un complejo proteína-ADN muy estable, no es estático y se ha demostrado que sufre una serie de reordenamientos estructurales diferentes, incluido el deslizamiento del nucleosoma y la exposición del sitio de ADN. Dependiendo del contexto, los nucleosomas pueden inhibir o facilitar la unión de factores de transcripción. Las posiciones de los nucleosomas están controladas por tres contribuciones principales: primero, la afinidad de unión intrínseca del octámero de histona depende de la secuencia de ADN. Segundo, el nucleosoma puede ser desplazado o reclutado por la unión competitiva o cooperativa de otros factores proteicos. Tercero, el nucleosoma puede ser translocado activamente por complejos de remodelación dependientes de ATP. [36]
Deslizamiento del nucleosoma
El trabajo realizado en el laboratorio de Bradbury mostró que los nucleosomas reconstituidos en la secuencia de posicionamiento de ADN 5S fueron capaces de reposicionarse traduccionalmente en secuencias adyacentes cuando se incubaron térmicamente. [37] Trabajos posteriores mostraron que este reposicionamiento no requirió la interrupción del octámero de histonas, pero era consistente con los nucleosomas que pueden "deslizarse" a lo largo del ADN en cis . En 2008, se reveló además que los sitios de unión de CTCF actúan como anclas de posicionamiento de nucleosomas de modo que, cuando se utilizan para alinear varias señales genómicas, se pueden identificar fácilmente múltiples nucleosomas flanqueantes. [38] Aunque los nucleosomas son intrínsecamente móviles, los eucariotas han desarrollado una gran familia de enzimas de remodelación de cromatina dependientes de ATP para alterar la estructura de la cromatina, muchas de las cuales lo hacen a través del deslizamiento de nucleosomas. En 2012, el laboratorio de Beena Pillai ha demostrado que el deslizamiento de nucleosomas es uno de los posibles mecanismos para la expresión de genes específicos de tejido a gran escala. El trabajo muestra que el sitio de inicio de la transcripción de los genes expresados en un tejido particular, están desprovistos de nucleosomas, mientras que el mismo conjunto de genes en otro tejido donde no se expresan, están unidos a nucleosomas. [39]
Exposición del sitio de ADN
El trabajo del laboratorio Widom ha demostrado que el ADN nucleosómico se encuentra en equilibrio entre un estado envuelto y desenrollado. Las mediciones de estas tasas utilizando FRET con resolución temporal revelaron que el ADN dentro del nucleosoma permanece completamente envuelto durante solo 250 ms antes de desenrollarse durante 10-50 ms y luego volver a enrollarse rápidamente. [40] Esto implica que el ADN no necesita disociarse activamente del nucleosoma, sino que hay una fracción significativa de tiempo durante la cual es completamente accesible. De hecho, esto se puede extender a la observación de que la introducción de una secuencia de unión al ADN dentro del nucleosoma aumenta la accesibilidad de las regiones adyacentes del ADN cuando está unido. [41] Esta propensión del ADN dentro del nucleosoma a "respirar" tiene importantes consecuencias funcionales para todas las proteínas de unión al ADN que operan en un entorno de cromatina. [40] En particular, la respiración dinámica de los nucleosomas desempeña un papel importante en la restricción del avance de la ARN polimerasa II durante la elongación de la transcripción. [42]
Región libre del nucleosoma
Los promotores de genes activos tienen regiones libres de nucleosomas (NFR). Esto permite que el ADN promotor sea accesible a varias proteínas, como los factores de transcripción. La región libre de nucleosomas normalmente abarca 200 nucleótidos en S. cerevisiae [43] . Los nucleosomas bien posicionados forman límites de NFR. Estos nucleosomas se denominan +1-nucleosoma y −1-nucleosoma y se encuentran a distancias canónicas aguas abajo y aguas arriba, respectivamente, del sitio de inicio de la transcripción. [44] El +1-nucleosoma y varios nucleosomas aguas abajo también tienden a incorporar la variante de histona H2A.Z. [44]
Modulación de la estructura del nucleosoma
Los genomas eucariotas están asociados de forma ubicua a la cromatina; sin embargo, las células deben regular espacial y temporalmente loci específicos independientemente de la cromatina en masa. Para lograr el alto nivel de control necesario para coordinar procesos nucleares como la replicación, reparación y transcripción del ADN, las células han desarrollado una variedad de medios para modular local y específicamente la estructura y función de la cromatina. Esto puede implicar la modificación covalente de histonas, la incorporación de variantes de histonas y la remodelación no covalente mediante enzimas de remodelación dependientes de ATP.
Modificaciones postraduccionales de las histonas

Desde que se descubrieron a mediados de la década de 1960, se ha predicho que las modificaciones de las histonas afectan la transcripción. [45] El hecho de que la mayoría de las primeras modificaciones postraduccionales encontradas se concentraran dentro de las extensiones de la cola que sobresalen del núcleo del nucleosoma condujo a dos teorías principales con respecto al mecanismo de modificación de las histonas. La primera de las teorías sugirió que pueden afectar las interacciones electrostáticas entre las colas de las histonas y el ADN para "aflojar" la estructura de la cromatina. Más tarde se propuso que las combinaciones de estas modificaciones pueden crear epítopos de unión con los que reclutar otras proteínas. [46] Recientemente, dado que se han encontrado más modificaciones en las regiones estructuradas de las histonas, se ha propuesto que estas modificaciones pueden afectar las interacciones histona-ADN [47] e histona-histona [48] dentro del núcleo del nucleosoma. Se predice que las modificaciones (como la acetilación o la fosforilación) que reducen la carga del núcleo globular de la histona "aflojan" la asociación núcleo-ADN; La fuerza del efecto depende de la ubicación de la modificación dentro del núcleo. [49] Se ha demostrado que algunas modificaciones están correlacionadas con el silenciamiento génico; otras parecen estar correlacionadas con la activación génica. Las modificaciones comunes incluyen acetilación , metilación o ubiquitinación de lisina ; metilación de arginina ; y fosforilación de serina . La información almacenada de esta manera se considera epigenética , ya que no está codificada en el ADN pero aún así se hereda a las células hijas. El mantenimiento de un estado reprimido o activado de un gen a menudo es necesario para la diferenciación celular . [18]
Variantes de histonas
Aunque las histonas se conservan notablemente a lo largo de la evolución, se han identificado varias formas variantes. Esta diversificación de la función de las histonas se limita a H2A y H3, siendo H2B y H4 en su mayoría invariantes. H2A puede ser reemplazada por H2AZ (que conduce a una estabilidad reducida del nucleosoma) o H2AX (que está asociada con la reparación del ADN y la diferenciación de células T ), mientras que los cromosomas X inactivos en los mamíferos están enriquecidos en macroH2A. H3 puede ser reemplazado por H3.3 (que se correlaciona con genes activos y elementos reguladores) y en los centrómeros H3 es reemplazado por CENPA . [18]
Remodelación de nucleosomas dependiente de ATP
Una serie de reacciones distintas se asocian con el término remodelación de la cromatina dependiente de ATP . Se ha demostrado que las enzimas de remodelación deslizan los nucleosomas a lo largo del ADN, [50] alteran los contactos histona-ADN hasta el punto de desestabilizar el dímero H2A/H2B [51] [52] y generan torsión superhelicoidal negativa en el ADN y la cromatina. [53] Recientemente, se ha demostrado que la enzima de remodelación Swr1 introduce la variante de histona H2A.Z en los nucleosomas. [54] En la actualidad, no está claro si todas estas representan reacciones distintas o simplemente resultados alternativos de un mecanismo común. Lo que comparten todas, y de hecho el sello distintivo de la remodelación de la cromatina dependiente de ATP, es que todas dan como resultado una accesibilidad alterada del ADN.
Los estudios que analizan la activación genética in vivo [55] y, lo que es más sorprendente, la remodelación in vitro [56] han revelado que los eventos de remodelación de la cromatina y la unión de factores de transcripción son de naturaleza cíclica y periódica. Si bien no se conocen las consecuencias de esto para el mecanismo de reacción de la remodelación de la cromatina, la naturaleza dinámica del sistema puede permitirle responder más rápidamente a los estímulos externos. Un estudio reciente indica que las posiciones de los nucleosomas cambian significativamente durante el desarrollo de células madre embrionarias de ratón, y estos cambios están relacionados con la unión de factores de transcripción del desarrollo. [57]
Remodelación dinámica de nucleosomas en el genoma de la levadura
Estudios en 2007 han catalogado las posiciones de los nucleosomas en levaduras y han demostrado que los nucleosomas están agotados en las regiones promotoras y los orígenes de replicación . [58] [59] [60] Alrededor del 80% del genoma de la levadura parece estar cubierto por nucleosomas [61] y el patrón de posicionamiento de los nucleosomas se relaciona claramente con las regiones de ADN que regulan la transcripción , las regiones que se transcriben y las regiones que inician la replicación del ADN. [62] Más recientemente, un nuevo estudio examinó los cambios dinámicos en el reposicionamiento de los nucleosomas durante un evento de reprogramación transcripcional global para dilucidar los efectos sobre el desplazamiento de los nucleosomas durante los cambios transcripcionales de todo el genoma en levaduras ( Saccharomyces cerevisiae ). [63] Los resultados sugirieron que los nucleosomas que se localizaron en las regiones promotoras se desplazan en respuesta al estrés (como el choque térmico ). Además, la eliminación de nucleosomas generalmente correspondió a la activación transcripcional y el reemplazo de nucleosomas generalmente correspondió a la represión transcripcional, presumiblemente porque los sitios de unión de factores de transcripción se volvieron más o menos accesibles, respectivamente. En general, solo uno o dos nucleosomas se reposicionaron en el promotor para efectuar estos cambios transcripcionales. Sin embargo, incluso en regiones cromosómicas que no estaban asociadas con cambios transcripcionales, se observó reposicionamiento de nucleosomas, lo que sugiere que el cubrimiento y descubrimiento del ADN transcripcional no necesariamente produce un evento transcripcional. Después de la transcripción, la región de ADNr tiene que protegerse de cualquier daño, lo que sugiere que las proteínas HMGB juegan un papel importante en la protección de la región libre de nucleosomas. [64] [65]
Defectos de torsión del ADN
Los defectos de torsión del ADN se producen cuando la adición de uno o unos pocos pares de bases de un segmento de ADN se transfieren al siguiente segmento, lo que da como resultado un cambio de la torsión del ADN. Esto no solo cambiará la torsión del ADN, sino que también cambiará la longitud. [66] Este defecto de torsión eventualmente se mueve alrededor del nucleosoma a través de la transferencia del par de bases, esto significa que las torsiones del ADN pueden causar el deslizamiento del nucleosoma. [67] Las estructuras cristalinas del nucleosoma han demostrado que las ubicaciones de superhélice 2 y 5 en el nucleosoma se encuentran comúnmente como donde ocurren los defectos de torsión del ADN, ya que estos son sitios de unión de remodeladores comunes. [68] Hay una variedad de remodeladores de cromatina, pero todos comparten la existencia de un motor ATPasa que facilita el deslizamiento de la cromatina en el ADN a través de la unión e hidrólisis de ATP. [69] La ATPasa tiene un estado abierto y cerrado. Cuando el motor ATPasa cambia de estados abierto y cerrado, el dúplex de ADN cambia de geometría y exhibe inclinación de pares de bases. [68] La iniciación de los defectos de torsión a través del motor ATPasa hace que se acumule tensión alrededor del sitio de remodelación. La tensión se libera cuando se completa el deslizamiento del ADN a lo largo del nucleosoma a través de la propagación de dos defectos de torsión (uno en cada hebra) en direcciones opuestas. [69]
Ensamblaje de nucleosomasin vitro

Los nucleosomas se pueden ensamblar in vitro utilizando histonas nativas purificadas o recombinantes. [70] [71] Una técnica estándar para cargar el ADN alrededor de las histonas implica el uso de diálisis salina . Una reacción que consiste en los octámeros de histonas y una plantilla de ADN desnudo se puede incubar juntos a una concentración de sal de 2 M. Al disminuir de manera constante la concentración de sal, el ADN se equilibrará en una posición en la que se envuelva alrededor de los octámeros de histonas, formando nucleosomas. En condiciones apropiadas, este proceso de reconstitución permite mapear experimentalmente la afinidad de posicionamiento de los nucleosomas de una secuencia dada. [72]
Partículas de núcleo de nucleosoma reticuladas por disulfuro
Un avance reciente en la producción de partículas de núcleo de nucleosoma con estabilidad mejorada implica enlaces cruzados de disulfuro específicos del sitio . [73] Se pueden introducir dos enlaces cruzados diferentes en la partícula de núcleo de nucleosoma. Un primero reticula las dos copias de H2A a través de una cisteína introducida (N38C) dando como resultado un octámero de histona que es estable frente a la pérdida del dímero H2A/H2B durante la reconstitución del nucleosoma. Se puede introducir un segundo enlace cruzado entre la cola de histona N-terminal H3 y los extremos de ADN del nucleosoma a través de un nucleótido convertible incorporado. [74] El enlace cruzado ADN-octámero de histona estabiliza la partícula de núcleo de nucleosoma frente a la disociación de ADN a concentraciones de partículas muy bajas y a concentraciones elevadas de sal.
Ensamblaje de nucleosomasen vivo

Los nucleosomas son la unidad básica de empaquetamiento del ADN genómico, formada por proteínas histonas alrededor de las cuales se enrolla el ADN. Actúan como andamio para la formación de una estructura de cromatina de orden superior, así como para una capa de control regulador de la expresión génica. Los nucleosomas se ensamblan rápidamente sobre el ADN recién sintetizado detrás de la horquilla de replicación.
H3 y H4
Las histonas H3 y H4 de los nucleosomas viejos desensamblados se mantienen en la vecindad y se distribuyen aleatoriamente en el ADN recién sintetizado. [75] Se ensamblan mediante el complejo del factor de ensamblaje de cromatina 1 (CAF-1), que consta de tres subunidades (p150, p60 y p48). [76] Las H3 y H4 recién sintetizadas se ensamblan mediante el factor de ensamblaje de acoplamiento de replicación (RCAF). El RCAF contiene la subunidad Asf1, que se une a las proteínas H3 y H4 recién sintetizadas. [77] Las proteínas H3 y H4 antiguas conservan sus modificaciones químicas, lo que contribuye a la transmisión de la firma epigenética. Las proteínas H3 y H4 recién sintetizadas se acetilan gradualmente en diferentes residuos de lisina como parte del proceso de maduración de la cromatina. [78] También se cree que las proteínas H3 y H4 antiguas en los nuevos nucleosomas reclutan enzimas modificadoras de histonas que marcan las nuevas histonas, lo que contribuye a la memoria epigenética.
H2A y H2B
A diferencia de las antiguas H3 y H4, las antiguas proteínas histonas H2A y H2B se liberan y degradan; por lo tanto, las proteínas H2A y H2B recién ensambladas se incorporan a los nuevos nucleosomas. [79] H2A y H2B se ensamblan en dímeros que luego se cargan en los nucleosomas por la proteína de ensamblaje de nucleosomas-1 (NAP-1), que también ayuda con el deslizamiento de los nucleosomas. [80] Los nucleosomas también están espaciados por complejos de remodelación de nucleosomas dependientes de ATP que contienen enzimas como Isw1 Ino80 y Chd1, y posteriormente se ensamblan en una estructura de orden superior. [81] [82]
Galería
Estructura cristalina de la partícula central del nucleosoma ( PDB : 1EQZ [ 28] ): diferentes vistas que muestran detalles del plegamiento y la organización de las histonas. Las histonas H2A , H2B , H3 , H4 y el ADN están coloreados.
Véase también
Referencias
- ^ Reece J, Campbell N (2006). Biología . San Francisco: Benjamin Cummings. ISBN 978-0-8053-6624-2.
- ^ Alberts B (2002). "ADN cromosómico y su empaquetamiento en la fibra de cromatina". Biología molecular de la célula (4.ª ed.). Nueva York: Garland Science. pág. 207. ISBN 978-0-8153-4072-0.
- ^ lberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). ADN cromosómico y su empaquetamiento en la fibra de cromatina. Garland Science.
- ^ Teif VB, Clarkson CT (2019). "Posicionamiento de nucleosomas". Enciclopedia de bioinformática y biología computacional . 2 : 308–317. doi :10.1016/B978-0-12-809633-8.20242-2. ISBN 9780128114322. Número de identificación del sujeto 43929234.
- ^ Olins AL, Olins DE (enero de 1974). "Unidades de cromatina esferoide (cuerpos v)". Science . 183 (4122): 330–332. Bibcode :1974Sci...183..330O. doi :10.1126/science.183.4122.330. PMID 4128918. S2CID 83480762.
- ^ McDonald D (diciembre de 2005). «Milestone 9, (1973-1974) The nucleosome theory: An alternative string theory» (Historias fundamentales de la naturaleza: expresión génica ) . doi :10.1038/nrm1798.
- ^ Kornberg RD (mayo de 1974). "Estructura de la cromatina: una unidad repetitiva de histonas y ADN". Science . 184 (4139): 868–871. Bibcode :1974Sci...184..868K. doi :10.1126/science.184.4139.868. PMID 4825889.
- ^ Lorch Y, LaPointe JW, Kornberg RD (abril de 1987). "Los nucleosomas inhiben la iniciación de la transcripción pero permiten la elongación de la cadena con el desplazamiento de las histonas". Cell . 49 (2): 203–210. doi :10.1016/0092-8674(87)90561-7. PMID 3568125. S2CID 21270171.
- ^ Han M, Grunstein M (diciembre de 1988). "La pérdida de nucleosomas activa los promotores descendentes de la levadura in vivo". Cell . 55 (6): 1137–1145. doi :10.1016/0092-8674(88)90258-9. PMID 2849508. S2CID 41520634.
- ^ Clark-Adams CD, Norris D, Osley MA, Fassler JS, Winston F (febrero de 1988). "Los cambios en la dosis de genes de histonas alteran la transcripción en levadura". Genes & Development . 2 (2): 150–159. doi : 10.1101/gad.2.2.150 . PMID 2834270.
- ^ ab En diferentes cristales se observaron valores de 146 y 147 pares de bases.
- ^ ab Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (septiembre de 1997). "Estructura cristalina de la partícula del núcleo del nucleosoma a una resolución de 2,8 A". Nature . 389 (6648): 251–260. Bibcode :1997Natur.389..251L. doi :10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ Alberts B (2007). Biología molecular de la célula (5.ª ed.). Nueva York: Garland Science. pág. 211. ISBN 978-0-8153-4106-2.
- ^ Shtumpf M, Piroeva KV, Agrawal SP, Jacob DR, Teif VB (junio de 2022). "NucPosDB: una base de datos de posicionamiento de nucleosomas in vivo y nucleosómica de ADN libre de células". Chromosoma . 131 (1–2): 19–28. doi :10.1007/s00412-021-00766-9. PMC 8776978 . PMID 35061087.
- ^ Zhou YB, Gerchman SE, Ramakrishnan V, Travers A, Muyldermans S (septiembre de 1998). "Posición y orientación del dominio globular de la histona H5 en el nucleosoma". Nature . 395 (6700): 402–405. Bibcode :1998Natur.395..402Z. doi : 10.1038/26521 . PMID 9759733. S2CID 204997317.
- ^ Thoma F, Koller T, Klug A (noviembre de 1979). "Participación de la histona H1 en la organización del nucleosoma y de las superestructuras dependientes de la sal de la cromatina". The Journal of Cell Biology . 83 (2 Pt 1): 403–427. doi :10.1083/jcb.83.2.403. PMC 2111545 . PMID 387806.
- ^ Clarke HJ (1992). "Composición nuclear y de cromatina de gametos de mamíferos y embriones tempranos". Bioquímica y biología celular . 70 (10–11): 856–866. doi :10.1139/o92-134. PMID 1297351.
- ^ abcdef Felsenfeld G, Groudine M (enero de 2003). "Control de la doble hélice". Nature . 421 (6921): 448–453. Bibcode :2003Natur.421..448F. doi : 10.1038/nature01411 . PMID 12540921.
- ^ Richmond TJ, Finch JT, Rushton B, Rhodes D, Klug A (1984). "Estructura de la partícula del núcleo del nucleosoma a una resolución de 7 A". Nature . 311 (5986): 532–7. Bibcode :1984Natur.311..532R. doi :10.1038/311532a0. PMID 6482966. S2CID 4355982.
- ^ Harp JM, Palmer EL, York MH, Gewiess A, Davis M, Bunick GJ (octubre de 1995). "Separación preparativa de partículas del núcleo del nucleosoma que contienen ADN de secuencia definida en múltiples fases de traducción". Electroforesis . 16 (10): 1861–1864. doi :10.1002/elps.11501601305. PMID 8586054. S2CID 20178479.
- ^ Palmer EL, Gewiess A, Harp JM, York MH, Bunick GJ (octubre de 1995). "Producción a gran escala de fragmentos de ADN palíndromo". Analytical Biochemistry . 231 (1): 109–114. doi :10.1006/abio.1995.1509. PMID 8678288.
- ^ Harp JM, Uberbacher EC, Roberson AE, Palmer EL, Gewiess A, Bunick GJ (marzo de 1996). "Análisis de difracción de rayos X de cristales que contienen partículas de núcleo de nucleosoma doblemente simétricas". Acta Crystallographica. Sección D, Cristalografía biológica . 52 (Pt 2): 283–288. Bibcode :1996AcCrD..52..283H. doi : 10.1107/S0907444995009139 . PMID 15299701.
- ^ Harp JM, Hanson BL, Timm DE, Bunick GJ (diciembre de 2000). "Asimetrías en la partícula del núcleo del nucleosoma a una resolución de 2,5 A". Acta Crystallographica. Sección D, Cristalografía biológica . 56 (Pt 12): 1513–1534. doi :10.1107/s0907444900011847. PMID 11092917.
- ^ Hanson BL, Alexander C, Harp JM, Bunick GJ (2004). "Preparación y cristalización de partículas del núcleo del nucleosoma". Cromatina y enzimas de remodelación de la cromatina, parte A. Métodos en enzimología. Vol. 375. págs. 44–62. doi :10.1016/s0076-6879(03)75003-4. ISBN 9780121827793. Número de identificación personal 14870658.
- ^ ab Chakravarthy S, Park YJ, Chodaparambil J, Edayathumangalam RS, Luger K (febrero de 2005). "Estructura y propiedades dinámicas de partículas del núcleo del nucleosoma". FEBS Letters . 579 (4): 895–898. Bibcode :2005FEBSL.579..895C. doi : 10.1016/j.febslet.2004.11.030 . PMID 15680970. S2CID 41706403.
- ^ White CL, Suto RK, Luger K (septiembre de 2001). "La estructura de la partícula central del nucleosoma de la levadura revela cambios fundamentales en las interacciones entre nucleosomas". The EMBO Journal . 20 (18): 5207–5218. doi :10.1093/emboj/20.18.5207. PMC 125637 . PMID 11566884.
- ^ ab Stryer L (1995). Bioquímica (cuarta edición). Nueva York - Basingstoke: WH Freeman and Company. ISBN 978-0716720096.
- ^ ab Harp JM, Hanson BL, Timm DE, Bunick GJ (diciembre de 2000). "Asimetrías en la partícula del núcleo del nucleosoma a una resolución de 2,5 A". Acta Crystallographica. Sección D, Cristalografía biológica . 56 (Pt 12): 1513–1534. doi :10.1107/S0907444900011847. PMID 11092917. PDB ID: 1EQZ.
- ^ Alberts B (2009). Biología celular esencial (2.ª ed.). Nueva York: Garland Science.
- ^ Allen, Paul D.; Newland, Adrian C. (1 de junio de 1998). "Análisis electroforético de ADN para la detección de apoptosis". Biotecnología molecular . 9 (3): 247–251. doi :10.1007/BF02915798. ISSN 1559-0305. PMID 9718585.
- ^ Davey CA, Sargent DF, Luger K, Maeder AW, Richmond TJ (junio de 2002). "Interacciones mediadas por solventes en la estructura de la partícula del núcleo del nucleosoma a una resolución de 1,9 a". Journal of Molecular Biology . 319 (5): 1097–1113. doi :10.1016/S0022-2836(02)00386-8. PMID 12079350.
- ^ Segal E, Fondufe-Mittendorf Y, Chen L, Thåström A, Field Y, Moore IK, et al. (agosto de 2006). "Un código genómico para el posicionamiento de nucleosomas". Nature . 442 (7104): 772–778. Bibcode :2006Natur.442..772S. doi :10.1038/nature04979. PMC 2623244 . PMID 16862119.
- ^ Zheng C, Hayes JJ (abril de 2003). "Estructuras e interacciones de los dominios de cola de histonas centrales". Biopolímeros . 68 (4): 539–546. doi :10.1002/bip.10303. PMID 12666178.
- ^ Schalch T, Duda S, Sargent DF, Richmond TJ (julio de 2005). "Estructura de rayos X de un tetranucleosoma y sus implicaciones para la fibra de cromatina". Nature . 436 (7047): 138–141. Bibcode :2005Natur.436..138S. doi :10.1038/nature03686. PMID 16001076. S2CID 4387396.
- ^ Robinson PJ, Fairall L, Huynh VA, Rhodes D (abril de 2006). "Las mediciones EM definen las dimensiones de la fibra de cromatina de "30 nm": evidencia de una estructura compacta e interdigitada". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 103 (17): 6506–6511. Bibcode :2006PNAS..103.6506R. doi : 10.1073/pnas.0601212103 . PMC 1436021 . PMID 16617109.
- ^ Teif VB, Rippe K (septiembre de 2009). "Predicción de las posiciones de los nucleosomas en el ADN: combinación de preferencias de secuencia intrínsecas y actividades de remodelación". Nucleic Acids Research . 37 (17): 5641–5655. doi :10.1093/nar/gkp610. PMC 2761276 . PMID 19625488.
- ^ Pennings S, Muyldermans S, Meersseman G, Wyns L (mayo de 1989). "Formación, estabilidad y posicionamiento de histonas centrales de nucleosomas reensamblados en ADN derivado de nucleosomas y otros". Journal of Molecular Biology . 207 (1): 183–192. doi :10.1016/0022-2836(89)90449-X. PMID 2738923.
- ^ Fu Y, Sinha M, Peterson CL, Weng Z (julio de 2008). Van Steensel B (ed.). "La proteína de unión al aislador CTCF posiciona 20 nucleosomas alrededor de sus sitios de unión en todo el genoma humano". PLOS Genetics . 4 (7): e1000138. doi : 10.1371/journal.pgen.1000138 . PMC 2453330 . PMID 18654629.
- ^ Bargaje R, Alam MP, Patowary A, Sarkar M, Ali T, Gupta S, et al. (octubre de 2012). "La proximidad del nucleosoma que contiene H2A.Z al sitio de inicio de la transcripción influye en los niveles de expresión génica en el hígado y el cerebro de los mamíferos". Nucleic Acids Research . 40 (18): 8965–8978. doi :10.1093/nar/gks665. PMC 3467062 . PMID 22821566.
- ^ ab Li G, Levitus M, Bustamante C, Widom J (enero de 2005). "Accesibilidad rápida y espontánea del ADN nucleosómico". Nature Structural & Molecular Biology . 12 (1): 46–53. doi :10.1038/nsmb869. PMID 15580276. S2CID 14540078.
- ^ Li G, Widom J (agosto de 2004). "Los nucleosomas facilitan su propia invasión". Nature Structural & Molecular Biology . 11 (8): 763–769. doi :10.1038/nsmb801. PMID 15258568. S2CID 11299024.
- ^ Hodges C, Bintu L, Lubkowska L, Kashlev M, Bustamante C (julio de 2009). "Las fluctuaciones nucleosomales gobiernan la dinámica de la transcripción de la ARN polimerasa II". Science . 325 (5940): 626–628. Bibcode :2009Sci...325..626H. doi :10.1126/science.1172926. PMC 2775800 . PMID 19644123.
- ^ Yuan GC, Liu YJ, Dion MF, Slack MD, Wu LF, Altschuler SJ, Rando OJ (julio de 2005). "Identificación a escala genómica de las posiciones de los nucleosomas en S. cerevisiae". Science . 309 (5734): 626–630. Bibcode :2005Sci...309..626Y. doi : 10.1126/science.1112178 . PMID 15961632. S2CID 43625066.
- ^ ab Lai WK, Pugh BF (septiembre de 2017). "Comprensión de la dinámica de los nucleosomas y sus vínculos con la expresión génica y la replicación del ADN". Nature Reviews. Molecular Cell Biology . 18 (9): 548–562. doi :10.1038/nrm.2017.47. PMC 5831138 . PMID 28537572.
- ^ Allfrey VG, Faulkner R, Mirsky AE (mayo de 1964). "Acetilación y metilación de histonas y su posible papel en la regulación de la síntesis de ARN". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 51 (5): 786–794. Bibcode :1964PNAS...51..786A. doi : 10.1073/pnas.51.5.786 . PMC 300163 . PMID 14172992.
- ^ Strahl BD, Allis CD (enero de 2000). "El lenguaje de las modificaciones covalentes de las histonas". Nature . 403 (6765): 41–45. Bibcode :2000Natur.403...41S. doi :10.1038/47412. PMID 10638745. S2CID 4418993.
- ^ Cosgrove MS, Boeke JD, Wolberger C (noviembre de 2004). "Movilidad regulada de nucleosomas y el código de histonas". Nature Structural & Molecular Biology . 11 (11): 1037–1043. doi :10.1038/nsmb851. PMID 15523479. S2CID 34704745.
- ^ Ye J, Ai X, Eugeni EE, Zhang L, Carpenter LR, Jelinek MA, et al. (abril de 2005). "La acetilación de la lisina 91 de la histona H4 es una modificación del dominio central asociada con el ensamblaje de la cromatina". Molecular Cell . 18 (1): 123–130. doi :10.1016/j.molcel.2005.02.031. PMC 2855496 . PMID 15808514.
- ^ Fenley AT, Adams DA, Onufriev AV (septiembre de 2010). "El estado de carga del núcleo globular de histonas controla la estabilidad del nucleosoma". Biophysical Journal . 99 (5): 1577–1585. Bibcode :2010BpJ....99.1577F. doi :10.1016/j.bpj.2010.06.046. PMC 2931741 . PMID 20816070.
- ^ Whitehouse I, Flaus A, Cairns BR, White MF, Workman JL, Owen-Hughes T (agosto de 1999). "Movilización de nucleosomas catalizada por el complejo SWI/SNF de levadura". Nature . 400 (6746): 784–787. Bibcode :1999Natur.400..784W. doi :10.1038/23506. PMID 10466730. S2CID 2841873.
- ^ Kassabov SR, Zhang B, Persinger J, Bartholomew B (febrero de 2003). "SWI/SNF desenrolla, desliza y vuelve a envolver el nucleosoma". Molecular Cell . 11 (2): 391–403. doi : 10.1016/S1097-2765(03)00039-X . PMID 12620227.
- ^ Bruno M, Flaus A, Stockdale C, Rencurel C, Ferreira H, Owen-Hughes T (diciembre de 2003). "Intercambio de dímeros de histona H2A/H2B mediante actividades de remodelación de la cromatina dependientes de ATP". Molecular Cell . 12 (6): 1599–1606. doi :10.1016/S1097-2765(03)00499-4. PMC 3428624 . PMID 14690611.
- ^ Havas K, Flaus A, Phelan M, Kingston R, Wade PA, Lilley DM, Owen-Hughes T (diciembre de 2000). "Generación de torsión superhelicoidal mediante actividades de remodelación de la cromatina dependientes de ATP". Cell . 103 (7): 1133–1142. doi : 10.1016/S0092-8674(00)00215-4 . PMID 11163188. S2CID 7911590.
- ^ Mizuguchi G, Shen X, Landry J, Wu WH, Sen S, Wu C (enero de 2004). "Intercambio impulsado por ATP de la variante de la histona H2AZ catalizado por el complejo de remodelación de cromatina SWR1". Science . 303 (5656): 343–348. Bibcode :2004Sci...303..343M. doi : 10.1126/science.1090701 . PMID 14645854. S2CID 9881829.
- ^ Métivier R, Penot G, Hübner MR, Reid G, Brand H, Kos M, Gannon F (diciembre de 2003). "El receptor de estrógeno alfa dirige el reclutamiento ordenado, cíclico y combinatorio de cofactores en un promotor objetivo natural". Cell . 115 (6): 751–763. doi : 10.1016/S0092-8674(03)00934-6 . PMID 14675539. S2CID 145525.
- ^ Nagaich AK, Walker DA, Wolford R, Hager GL (abril de 2004). "Unión periódica rápida y desplazamiento del receptor de glucocorticoides durante la remodelación de la cromatina". Molecular Cell . 14 (2): 163–174. doi : 10.1016/S1097-2765(04)00178-9 . PMID 15099516.
- ^ Teif VB, Vainshtein Y, Caudron-Herger M, Mallm JP, Marth C, Höfer T, Rippe K (noviembre de 2012). "Posicionamiento de nucleosomas en todo el genoma durante el desarrollo de células madre embrionarias". Nature Structural & Molecular Biology . 19 (11): 1185–1192. doi :10.1038/nsmb.2419. PMID 23085715. S2CID 34509771.
- ^ Albert I, Mavrich TN, Tomsho LP, Qi J, Zanton SJ, Schuster SC, Pugh BF (marzo de 2007). "Configuraciones translacionales y rotacionales de los nucleosomas H2A.Z en el genoma de Saccharomyces cerevisiae". Nature . 446 (7135): 572–576. Bibcode :2007Natur.446..572A. doi :10.1038/nature05632. PMID 17392789. S2CID 4416890.
- ^ Li B, Carey M, Workman JL (febrero de 2007). "El papel de la cromatina durante la transcripción". Cell . 128 (4): 707–719. doi : 10.1016/j.cell.2007.01.015 . PMID 17320508. S2CID 1773333.
- ^ Whitehouse I, Rando OJ, Delrow J, Tsukiyama T (diciembre de 2007). "La remodelación de la cromatina en los promotores suprime la transcripción antisentido". Nature . 450 (7172): 1031–1035. Bibcode :2007Natur.450.1031W. doi :10.1038/nature06391. PMID 18075583. S2CID 4305576.
- ^ Lee W, Tillo D, Bray N, Morse RH, Davis RW, Hughes TR, Nislow C (octubre de 2007). "Atlas de alta resolución de la ocupación de nucleosomas en levaduras". Nature Genetics . 39 (10): 1235–1244. doi :10.1038/ng2117. PMID 17873876. S2CID 12816925.
- ^ Eaton ML, Galani K, Kang S, Bell SP, MacAlpine DM (abril de 2010). "La posición conservada de los nucleosomas define los orígenes de la replicación". Genes & Development . 24 (8): 748–753. doi :10.1101/gad.1913210. PMC 2854390 . PMID 20351051.
- ^ Shivaswamy S, Bhinge A, Zhao Y, Jones S, Hirst M, Iyer VR (marzo de 2008). "Remodelación dinámica de nucleosomas individuales en un genoma eucariota en respuesta a perturbaciones transcripcionales". PLOS Biology . 6 (3): e65. doi : 10.1371/journal.pbio.0060065 . PMC 2267817 . PMID 18351804.
- ^ Murugesapillai D, McCauley MJ, Huo R, Nelson Holte MH, Stepanyants A, Maher LJ, et al. (agosto de 2014). "La formación de puentes y bucles de ADN por HMO1 proporciona un mecanismo para estabilizar la cromatina libre de nucleosomas". Nucleic Acids Research . 42 (14): 8996–9004. doi :10.1093/nar/gku635. PMC 4132745 . PMID 25063301.
- ^ Murugesapillai D, McCauley MJ, Maher LJ, Williams MC (febrero de 2017). "Estudios de moléculas individuales de proteínas de flexión de ADN arquitectural del grupo B de alta movilidad". Biophysical Reviews . 9 (1): 17–40. doi :10.1007/s12551-016-0236-4. PMC 5331113 . PMID 28303166.
- ^ Winger J, Nodelman IM, Levendosky RF, Bowman GD (mayo de 2018). "Un mecanismo de defecto de torsión para la translocación dependiente de ATP del ADN nucleosómico". eLife . 7 : e34100. doi : 10.7554/eLife.34100 . PMC 6031429 . PMID 29809147.
- ^ Bowman GD (agosto de 2019). "Descubrimiento de un nuevo paso en el deslizamiento de nucleosomas". Tendencias en ciencias bioquímicas . 44 (8): 643–645. doi :10.1016/j.tibs.2019.05.001. PMC 7092708 . PMID 31171402.
- ^ ab Nodelman IM, Bowman GD (mayo de 2021). "Biofísica de la remodelación de la cromatina". Revisión anual de biofísica . 50 (1): 73–93. doi :10.1146/annurev-biophys-082520-080201. PMC 8428145 . PMID 33395550.
- ^ ab Brandani GB, Takada S (noviembre de 2018). Onufriev A (ed.). "Los remodeladores de cromatina combinan el movimiento de la oruga con la formación de defectos de torsión para deslizar el ADN nucleosómico". PLOS Computational Biology . 14 (11): e1006512. Bibcode :2018PLSCB..14E6512B. doi : 10.1371/journal.pcbi.1006512 . PMC 6237416 . PMID 30395604.
- ^ Hayes JJ, Lee KM (mayo de 1997). "Reconstitución y análisis in vitro de mononucleosomas que contienen ADN y proteínas definidos". Métodos . 12 (1): 2–9. doi : 10.1006/meth.1997.0441 . PMID 9169189.
- ^ Dyer PN, Edayathumangalam RS, White CL, Bao Y, Chakravarthy S, Muthurajan UM, Luger K (2004). "Reconstitución de partículas del núcleo del nucleosoma a partir de histonas recombinantes y ADN". Cromatina y enzimas de remodelación de la cromatina, parte A. Métodos en enzimología. Vol. 375. págs. 23–44. doi :10.1016/s0076-6879(03)75002-2. ISBN 9780121827793. Número de identificación personal 14870657.
- ^ Yenidunya A, Davey C, Clark D, Felsenfeld G, Allan J (abril de 1994). "Posicionamiento de nucleosomas en promotores de genes de globina humana y de pollo in vitro. Nuevas técnicas de mapeo". Journal of Molecular Biology . 237 (4): 401–414. doi :10.1006/jmbi.1994.1243. PMID 8151701.
- ^ Frouws TD, Barth PD, Richmond TJ (enero de 2018). "Nucleosomas reticulados por disulfuro específicos del sitio con estabilidad mejorada". Journal of Molecular Biology . 430 (1): 45–57. doi :10.1016/j.jmb.2017.10.029. PMC 5757783 . PMID 29113904.
- ^ Ferentz AE, Verdine GL (1994). "El enfoque de los nucleósidos convertibles: ingeniería estructural de los ácidos nucleicos mediante enlaces cruzados disulfuro". En Eckstein F, Lilley DM (eds.). Nucleic Acids and Molecular Biology . Vol. 8. págs. 14–40. doi :10.1007/978-3-642-78666-2_2. ISBN 978-3-642-78668-6.
- ^ Yamasu K, Senshu T (enero de 1990). "Segregación conservadora de unidades tetraméricas de histonas H3 y H4 durante la replicación de nucleosomas". Journal of Biochemistry . 107 (1): 15–20. doi :10.1093/oxfordjournals.jbchem.a122999. PMID 2332416.
- ^ Kaufman PD, Kobayashi R, Kessler N, Stillman B (junio de 1995). "Las subunidades p150 y p60 del factor de ensamblaje de cromatina I: un vínculo molecular entre las histonas recién sintetizadas y la replicación del ADN". Cell . 81 (7): 1105–1114. doi : 10.1016/S0092-8674(05)80015-7 . PMID 7600578. S2CID 13502921.
- ^ Tyler JK, Adams CR, Chen SR, Kobayashi R, Kamakaka RT, Kadonaga JT (diciembre de 1999). "El complejo RCAF media el ensamblaje de la cromatina durante la replicación y reparación del ADN". Nature . 402 (6761): 555–560. Bibcode :1999Natur.402..555T. doi :10.1038/990147. PMID 10591219. S2CID 205097512.
- ^ Benson LJ, Gu Y, Yakovleva T, Tong K, Barrows C, Strack CL, et al. (abril de 2006). "Modificaciones de H3 y H4 durante la replicación de la cromatina, el ensamblaje de nucleosomas y el intercambio de histonas". The Journal of Biological Chemistry . 281 (14): 9287–9296. doi : 10.1074/jbc.M512956200 . PMID 16464854.
- ^ Louters L, Chalkley R (junio de 1985). "Intercambio de histonas H1, H2A y H2B in vivo". Bioquímica . 24 (13): 3080–3085. doi :10.1021/bi00334a002. PMID 4027229.
- ^ Park YJ, Chodaparambil JV, Bao Y, McBryant SJ, Luger K (enero de 2005). "La proteína de ensamblaje de nucleosomas 1 intercambia dímeros de histona H2A-H2B y ayuda al deslizamiento del nucleosoma". The Journal of Biological Chemistry . 280 (3): 1817–1825. doi : 10.1074/jbc.M411347200 . PMID 15516689.
- ^ Vincent JA, Kwong TJ, Tsukiyama T (mayo de 2008). "La remodelación de la cromatina dependiente de ATP moldea el panorama de replicación del ADN". Nature Structural & Molecular Biology . 15 (5): 477–484. doi :10.1038/nsmb.1419. PMC 2678716 . PMID 18408730.
- ^ Yadav T, Whitehouse I (abril de 2016). "Ensamblaje y posicionamiento de nucleosomas acoplados a replicación por enzimas de remodelación de cromatina dependientes de ATP". Cell Reports . 15 (4): 715–723. doi :10.1016/J.CELREP.2016.03.059. PMC 5063657 . PMID 27149855.
Enlaces externos
- MBInfo - ¿Qué son los nucleosomas?
- Nucleosomas en el sitio web de Richmond Lab
- Nucleosomas de la proteopedia
- Nucleosoma en el PDB
- Remodelación dinámica de nucleosomas individuales en un genoma eucariota en respuesta a perturbaciones transcripcionales
- Datos y herramientas de posicionamiento de nucleosomas en línea (lista anotada, actualizada constantemente)
- Estructura de la proteína histona
- HistoneDB 2.0 - Base de datos de histonas y variantes en NCBI


