Haplogrupo de ADN mitocondrial humano

En genética humana , un haplogrupo de ADN mitocondrial humano es un haplogrupo definido por diferencias en el ADN mitocondrial humano . Los haplogrupos se utilizan para representar los puntos de ramificación principales en el árbol filogenético mitocondrial. Comprender la trayectoria evolutiva del linaje femenino ha ayudado a los genetistas de poblaciones a rastrear la herencia matrilineal de los humanos modernos hasta sus orígenes en África y su posterior expansión por todo el mundo.
Los nombres de las letras de los haplogrupos (no solo los haplogrupos de ADN mitocondrial) van de la A a la Z. Como los haplogrupos fueron nombrados en el orden de su descubrimiento, el orden alfabético no tiene ningún significado en términos de relaciones genéticas reales.
La mujer hipotética que se encuentra en la raíz de todos estos grupos (es decir, solo los haplogrupos de ADN mitocondrial) es el antepasado común más reciente (ARM) matrilineal de todos los seres humanos que viven actualmente . Se la suele llamar Eva mitocondrial .
La velocidad a la que muta el ADN mitocondrial se conoce como reloj molecular mitocondrial . Es un área de investigación en curso, y un estudio informa que se produce una mutación cada 8000 años. [2]
Filogenia
| Parte de una serie sobre |
| Genealogía genética |
|---|
| Conceptos |
| Temas relacionados |

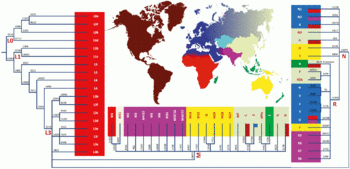
Este árbol filogenético se basa en Van Oven (2009). [4] En junio de 2022, se sugirió una filogenia alternativa para el haplogrupo L [5]
- L ( Eva mitocondrial )
Principales haplogrupos de ADNmt

Macrohaplogrupo L
El macrohaplogrupo L es el más basal de los haplogrupos de ADNmt humano, del que descienden todos los demás haplogrupos (en concreto, el haplogrupo L3). Se encuentra principalmente en África.
- Haplogrupo L0
- L1-7
- Haplogrupo L1
- L2-7
- Largo 3'4'6
- 5'7 de largo
Macrohaplogrupo M
El macrohaplogrupo M se encuentra principalmente en Asia y América. Sus descendientes son el haplogrupo M , el haplogrupo C , el haplogrupo Z , el haplogrupo D , el haplogrupo E , el haplogrupo G y el haplogrupo Q.
Macrohaplogrupo N
El macrohaplogrupo N se encuentra principalmente en Australia, América y partes de Asia. Sus descendientes son el haplogrupo N , el haplogrupo O , el haplogrupo A , el haplogrupo S , el haplogrupo I , el haplogrupo W , el haplogrupo X y el haplogrupo Y , así como el macrohaplogrupo R.
Macrohaplogrupo R
El macrohaplogrupo R se encuentra principalmente en Europa, el norte de África, el Pacífico y partes de Asia y América. Sus descendientes son el haplogrupo R , el haplogrupo B , el haplogrupo F , el haplogrupo H , el haplogrupo V , el haplogrupo J , el haplogrupo T , el haplogrupo U y el haplogrupo K.
Cronología
| Haplogrupo | Tiempo estimado de origen ( kya ) [6] | Posible lugar de origen | Frecuencias más altas |
|---|---|---|---|
| yo | 200 | África | |
| L1-6 | 170 | África Oriental | |
| L2-6 | 150 | África Oriental | |
| L0 | 150 | África Oriental | |
| L1 | 140 | África central | |
| L3-6 | 130 | ||
| L5 | 120 | ||
| L2 | 90 | ||
| Nivel 3 | 70 | África Oriental | |
| norte | 70 | África Oriental o Asia Occidental | |
| METRO | 60 | África Oriental, Asia Occidental o Asia Meridional | |
| R | 60 | Asia meridional o sudeste asiático | |
| tú | 55 | África del Noreste o India (Asia Meridional) | |
| RT'JT | 55 | Oriente Medio | |
| JT | 50 | Oriente Medio | |
| U8 | 50 | Asia occidental | |
| R9 | 47 | ||
| B4 | 44 | ||
| F | 43 | ||
| U4'9 | 42 | Asia central | |
| U5 | 35 | Asia occidental | |
| U6 | 35 | África del Norte | |
| Yo | 35 | ||
| incógnita | 30 | ||
| K | 30 | ||
| U5a | 27 | ||
| Alto voltaje | 27 | Próximo Oriente | |
| J1a | 27 | Próximo Oriente | |
| yo | 27 | Mesopotamia | |
| K1 | 27 | ||
| I | 26 | ||
| J1 | 24 | Próximo Oriente | |
| Yo | 20 | ||
| U4 | 20 | Asia central | |
| X2 | 20 | ||
| yo | 20 | Asia occidental | |
| U5a1 | 18 | Europa | |
| J1b | 11 | ||
| V | 14 | ||
| X2a | 13 | América del norte | |
| H1 | 12 | ||
| H3 | 12 | ||
| X1 | 10 |
Distribución geográfica
Un artículo de 2004 sugirió que los haplogrupos más comunes en las poblaciones modernas de Asia occidental, el norte de África y Europa eran: H, J, K, N1, T, U4, U5, V, X y W. [7]
Haplogrupos africanos: L0, L1, L2, L3, L4, L5, L6, T, U5a
Haplogrupos australianos: M42a, M42c, M14, M15, Q, S, O, N, P. (Referencias 1, 2, 3, 4, 5, 6)
Haplogrupos asiáticos: F, C, W, M, D, N, K, U, T, A, B, C, Z, U muchas variantes numéricas para cada sección
Software de investigación
Asignación
- mtHap: la herramienta de James Lick (múltiples formatos de entrada).
- Buscador de clados YSEQ mt: herramienta de haplogrupos basada en FASTA. Reemplazó a HAPLOFIND. [8]
- HaploGrep: herramienta basada en VCF. [9] [10]
- Haplocheck: la herramienta de Mitoverse tanto para WGS como para WES. [11]
- Haplotracker: una herramienta para fragmentos cortos. [12]
- Buscador de haplogrupos: la herramienta de conversión de SNP a haplogrupos de Ian Logan.
Tener una cita
- Modelo computacional de mutación de ADNmt: calculadora de mutaciones de Ian Logan.
Filogenia
- Haploárbol de ADNmt de FTDNA: el árbol de ADNmt más grande.
- MTree de YFull: un árbol de ADN mitocondrial. Carga más rápida (más simple).
- PhyloTreemt: un árbol de ADNmt tradicional (última actualización el 18 de febrero de 2016). [13]
- HIP: Árboles de haplogrupos.
Mapas
Antiguo
- Haplomap HIP: Mapa uniparental antiguo.
- ADN AH: el antiguo mapa uniparental de Nicky Rosenblatt. [14]
- ADN antiguo: un mapa de ADNa realizado con Map Maker .
Moderno
- HRAS mtDNA: un generador de mapas de calor.
Bases de datos
Antiguo
- AmtDB: una base de datos de muestras de ADNmt antiguas. [15]
- Todo el conjunto de datos de ADN antiguo: base de datos uniparental de Carlos Quiles (con BAM).
Moderno
- Comprobador de secuencias de ADNmt de GenBank: herramienta de comparación de accesiones basada en GenBank de Ian Logan.
- mitoYDNA: Una base de datos uniparental.
- MITOMAP: Una base de datos del genoma del ADNmt. [16]
Véase también
- Genética mitocondrial humana
- Genealogía genética
- Matrilinealidad
- Haplogrupos de ADN del cromosoma Y humano
- Genética de poblaciones
Referencias
- ^ Rishishwar L, Jordan IK (2017). "Implicaciones de la evolución humana y la mezcla para la terapia de reemplazo mitocondrial". BMC Genomics . 18 (1): 140. doi : 10.1186/s12864-017-3539-3 . PMC 5299762 . PMID 28178941.
- ^ Loogvali, Eva-Liis; Kivisild, Toomas; Margus, Tõnu; Villems, Richard (2009), O'Rourke, Dennis (ed.), "Explicando la imperfección del reloj molecular de las mitocondrias de los homínidos", PLOS ONE , 4 (12): e8260, Bibcode :2009PLoSO...4.8260L, doi : 10.1371/journal.pone.0008260 , PMC 2794369 , PMID 20041137
- ^ Kivisild T (2015). "Ancestro materno e historia de la población a partir de genomas mitocondriales completos". Investig Genet . 6 : 3. doi : 10.1186/s13323-015-0022-2 . PMC 4367903 . PMID 25798216.
- ^ ab van Oven M, Kayser M (febrero de 2009). "Árbol filogenético completo actualizado de la variación global del ADN mitocondrial humano". Human Mutation . 30 (2): E386–94. doi : 10.1002/humu.20921 . PMID 18853457. S2CID 27566749.
- ^ Maier P, Runfeldt G, Estes R, Vilar M (2022). "Haplogrupo mitocondrial africano L7: un linaje humano materno de 100.000 años descubierto mediante una reevaluación y una nueva secuenciación". Nature . 12 (1): 10747. Bibcode :2022NatSR..1210747M. doi : 10.1038/s41598-022-13856-0 . PMC 9232647 . PMID 35750688. S2CID 250021505.
- ^ "Corrección para la selección purificadora: un suplemento mejorado del reloj molecular mitocondrial humano" (PDF) . Cell : 82–83 [89]. 2009. Archivado desde el original (PDF) el 29 de diciembre de 2009.
- ^ Villems, Richard; Usanga, Esien; Mikerezi, Ilia; Gölge, Mukaddes; Claustres, Mireille; Michalodimitrakis, Emmanuel N.; Pappa, Kalliopi I.; Anagnou, Nicolás P.; Chaventré, André; Moisan, Jean-Paul; Richard, Christelle; Grechanina, Elena; Balanovska, Elena V.; Rudán, Pavao; Puzyrev, Valéry; Stepanov, Vadim; Khusnutdinova, Elsa K.; Gusar, Vladislava; Balanovsky, Oleg P.; Pericic, Marijana; Barać, Lovorka; Golubenko, María; Lunkina, Arina; Laos, Sirle; Pennarun, Erwan; Parik, Juri; Tolk, Helle-Viivi; Reidla, Maere; Tambets, Kristiina; Metspalu, Ene; Kivisild, Toomas; Derenko, Miroslava V.; Malyarchuk, Boris A.; Roostalu, Urmas; Loogväli, Eva-Liis (1 de noviembre de 2004). "Uniformidad desunida: un lienzo cladístico de varios colores del haplogrupo H del ADNmt en Eurasia". Biología Molecular y Evolución . 21 (11): 2012-2021. doi : 10.1093/molbev/msh209 . PMID 15254257 - a través de academic.oup.com.
- ^ Capri, Miriam; Castellani, Gastone; Franceschi, Claudio; Lomartire, Laura; Sevini, Federica; Vianello, Darío (12 de junio de 2013). "HAPLOFIND: un nuevo método para la asignación de haplogrupos de ADNmt de alto rendimiento". Mutación humana . 34 (9): 1189-1194. doi :10.1002/humu.22356. eISSN 1098-1004.
- ^ Binna, Robert; Kloss-Brandstätter, Anita; Kronenberg, Florián; Pacher, Dominic; Schönherr, Sebastián; Specht, Günther; Weissensteiner, Hansi (19 de octubre de 2010). "HaploGrep: un algoritmo rápido y fiable para la clasificación automática de haplogrupos de ADN mitocondrial". Mutatón humano: variación, informática y enfermedad . 32 (1): 25–32. doi :10.1002/humu.21382. eISSN 1098-1004.
- ^ Kronenberg, Florian; Forer, Lukas; Schönherr, Sebastian; Weissensteiner, Hansi (23 de abril de 2023). "Haplogrep 3: una plataforma interactiva de clasificación y análisis de haplogrupos". Investigación de ácidos nucleicos . 51 (1): 263–268. doi :10.1093/nar/gkad284. eISSN 1362-4962. PMID 37070190.
- ^ García-Olivares, Victor; et al. (15 de octubre de 2021) [recibido el 4 de agosto de 2021]. "Evaluación comparativa de los clasificadores de haplogrupos del ADN mitocondrial humano a partir de datos de secuencias de todo el genoma y todo el exoma". Scientific Reports . 11 (20510): 20510. doi :10.1038/s41598-021-99895-5. eISSN 2045-2322. PMC 8519921 . PMID 34654896.
- ^ Kim, Dong-han; Kim, Kijeong; Kim, Kyung-yong; Kim, Yoonyeong; Kwon, Chulhwan (23 de abril de 2020). "Haplotracker: una aplicación web para la haploagrupación mitocondrial sencilla y precisa utilizando fragmentos cortos de ADN". bioRxiv 10.1101/2020.04.23.057646v1 .
- ^ Kayser, Manfred; van Oven, Mannis (13 de octubre de 2008). "Árbol filogenético completo actualizado de la variación global del ADN mitocondrial humano". Human Mutation . 30 (2): 386–394. doi : 10.1002/humu.20921 . eISSN 1098-1004. PMID 18853457.
- ^ Varios (30 de mayo de 2017). "El mapa del ADN antiguo de Rosenblatt". Anthrogenica .
- ^ Chyleński, Maciej; Ehler, Edvard; Juras, Anna; Moravčík, Ondřej; Novotný, Jiří; Pačes, enero (24 de septiembre de 2018). "AmtDB: una base de datos de genomas mitocondriales humanos antiguos". Investigación de ácidos nucleicos . 47 (D1): 29–32. doi : 10.1093/nar/gky843. eISSN 1362-4962.
- ^ Brown, Michael D.; Kogelnik, Andreas M.; Lott, Marie T.; Navathe, Shamkant B.; Wallace, Douglas C. (1 de enero de 1996). "MITOMAP: una base de datos del genoma mitocondrial humano". Nucleic Acids Research . 24 (1): 177–179. doi :10.1093/nar/24.1.177. eISSN 1362-4962.
Enlaces externos
- Lista de proyectos de haplogrupos de ADNmt en ISOGG.
Árbol filogenético de los haplogrupos del ADN mitocondrial (ADNmt) humano | |||||||||||||||||||||||||||||||||||||||
| Eva mitocondrial ( L ) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1–6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | Nivel 3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| METRO | norte | ||||||||||||||||||||||||||||||||||||||
| República Checa | D | mi | GRAMO | Q | Oh | A | S | R | I | Yo | incógnita | Y | |||||||||||||||||||||||||||
| do | O | B | F | R0 | Pre-JT | PAG | tú | ||||||||||||||||||||||||||||||||
| Alto voltaje | JT | K | |||||||||||||||||||||||||||||||||||||
| yo | V | Yo | yo | ||||||||||||||||||||||||||||||||||||
