Logotipo de secuencia

En bioinformática , un logotipo de secuencia es una representación gráfica de la conservación de la secuencia de nucleótidos (en una cadena de ADN / ARN ) o aminoácidos (en secuencias de proteínas ). [1] Un logotipo de secuencia se crea a partir de una colección de secuencias alineadas y representa la secuencia de consenso y la diversidad de las secuencias. Los logotipos de secuencia se utilizan con frecuencia para representar características de secuencia, como sitios de unión de proteínas en el ADN o unidades funcionales en proteínas.
Descripción general
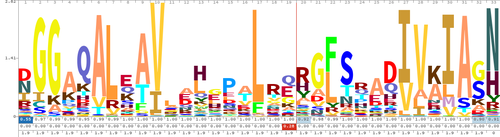
Un logotipo de secuencia consta de una pila de letras en cada posición. Los tamaños relativos de las letras indican su frecuencia en las secuencias. La altura total de las letras representa el contenido de información de la posición, en bits.
Creación de logotipos
Para crear logotipos de secuencias, se alinean secuencias de ADN, ARN o proteínas relacionadas, o secuencias de ADN que tienen sitios de unión conservados comunes, de modo que las partes más conservadas creen buenas alineaciones. Luego, se puede crear un logotipo de secuencia a partir de la alineación de secuencias múltiples conservadas . El logotipo de secuencia mostrará qué tan bien se conservan los residuos en cada posición: cuanto mayor sea el número de residuos, más altas serán las letras, porque mejor es la conservación en esa posición. Los diferentes residuos en la misma posición se escalan de acuerdo con su frecuencia. La altura de toda la pila de residuos es la información medida en bits . Los logotipos de secuencias se pueden usar para representar sitios de unión de ADN conservados , donde se unen los factores de transcripción .
El contenido de información (eje y) de la posición viene dado por: [2]
- Para los aminoácidos,
- para los ácidos nucleicos,
¿Dónde está la incertidumbre (a veces llamada entropía de Shannon ) de la posición?
Aquí, es la frecuencia relativa de la base o aminoácido en la posición , y es la corrección de muestra pequeña para una alineación de letras. [2] [3] La altura de la letra en la columna está dada por
La aproximación para la corrección de muestra pequeña, , viene dada por:
donde es 4 para nucleótidos, 20 para aminoácidos y es el número de secuencias en la alineación.
Logotipo de consenso
Un logotipo de consenso es una variación simplificada de un logotipo de secuencia que se puede incrustar en formato de texto. Al igual que un logotipo de secuencia, un logotipo de consenso se crea a partir de una colección de secuencias de proteínas o ADN/ARN alineadas y transmite información sobre la conservación de cada posición de un motivo de secuencia o alineación de secuencia [1] [4] . Sin embargo, un logotipo de consenso muestra solo información de conservación, y no explícitamente la información de frecuencia de cada nucleótido o aminoácido en cada posición. En lugar de una pila formada por varios caracteres, que denota la frecuencia relativa de cada carácter, el logotipo de consenso representa el grado de conservación de cada posición utilizando la altura del carácter de consenso en esa posición.


Ventajas y desventajas
La principal y obvia ventaja de los logotipos de consenso sobre los logotipos de secuencia es su capacidad de ser incrustados como texto en cualquier editor/visor compatible con Rich Text Format y, por lo tanto, en manuscritos científicos. Como se describió anteriormente, el logotipo de consenso es un cruce entre logotipos de secuencia y secuencias de consenso . Como resultado, en comparación con un logotipo de secuencia, el logotipo de consenso omite información (la contribución relativa de cada carácter a la conservación de esa posición en el motivo/alineamiento). Por lo tanto, se debe utilizar preferentemente un logotipo de secuencia siempre que sea posible. Dicho esto, la necesidad de incluir figuras gráficas para mostrar logotipos de secuencia ha perpetuado el uso de secuencias de consenso en manuscritos científicos, aunque no transmitan información sobre la conservación y la frecuencia. [5] Los logotipos de consenso representan, por lo tanto, una mejora con respecto a las secuencias de consenso siempre que la información del motivo/alineamiento se deba restringir al texto.
Extensiones
Los modelos ocultos de Markov (HMM) no solo consideran el contenido de información de las posiciones alineadas en una alineación, sino también de las inserciones y eliminaciones. En un logotipo de secuencia HMM utilizado por Pfam , se agregan tres filas para indicar las frecuencias de ocupación (presencia) e inserción, así como la longitud de inserción esperada. [6]
Véase también
Referencias
- ^ ab Schneider TD; Stephens RM (1990). "Logotipos de secuencias: una nueva forma de mostrar secuencias de consenso". Nucleic Acids Res . 18 (20): 6097–6100. doi :10.1093/nar/18.20.6097. PMC 332411 . PMID 2172928.
- ^ ab Schneider TD; Stormo GD (1986). "Contenido de información de los sitios de unión en secuencias de nucleótidos" (PDF) . Journal of Molecular Biology . 188 (3): 415–431. doi :10.1016/0022-2836(86)90165-8. PMID 3525846.
- ^ Basharin GP (1959). "Sobre una estimación estadística de la entropía de una secuencia de variables aleatorias independientes". Teoría de la probabilidad y sus aplicaciones . 4 (3): 333–336. doi :10.1137/1104033.
- ^ Anzaldi LJ; Muñoz-Fernández D; Erill I. (2012). "BioWord: una suite de manipulación de secuencias para Microsoft Word". BMC Bioinformatics . 13 (124): 124. doi : 10.1186/1471-2105-13-124 . PMC 3546851 . PMID 22676326.
- ^ Schneider TD (2002). "Secuencia de consenso Zen". Appl Bioinform . 1 (3): 111–119. PMC 1852464 . PMID 15130839.
- ^ Wheeler, Travis J; Clements, Jody; Finn, Robert D (13 de enero de 2014). "Skylign: una herramienta para crear logotipos informativos e interactivos que representan alineaciones de secuencias y perfiles de modelos ocultos de Markov". BMC Bioinformatics . 15 (1): 7. doi : 10.1186/1471-2105-15-7 . PMC 3893531 . PMID 24410852.
Enlaces externos
- Cómo leer logotipos de secuencias.
- Recomendaciones para realizar logotipos secuenciales.
- Erill, I., "Una introducción suave al contenido de información en los sitios de unión de factores de transcripción", Eprint
- ¿Qué es (en) un logotipo de secuencia?











