Isoforma de proteína
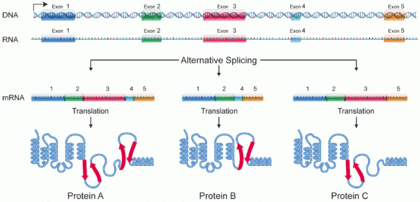
Una isoforma de proteína , o " variante de proteína ", [1] es un miembro de un conjunto de proteínas altamente similares que se originan a partir de un solo gen y son el resultado de diferencias genéticas. [2] Si bien muchas desempeñan funciones biológicas iguales o similares, algunas isoformas tienen funciones únicas. Un conjunto de isoformas de proteína puede formarse a partir de empalmes alternativos , uso variable del promotor u otras modificaciones postranscripcionales de un solo gen; las modificaciones postranscripcionales generalmente no se consideran. (Para eso, consulte Proteoformas ). A través de los mecanismos de empalme de ARN , el ARNm tiene la capacidad de seleccionar diferentes segmentos codificadores de proteínas ( exones ) de un gen, o incluso diferentes partes de exones del ARN para formar diferentes secuencias de ARNm. Cada secuencia única produce una forma específica de una proteína.
El descubrimiento de isoformas podría explicar la discrepancia entre el pequeño número de regiones codificantes de proteínas de los genes revelados por el proyecto genoma humano y la gran diversidad de proteínas observadas en un organismo: diferentes proteínas codificadas por el mismo gen podrían aumentar la diversidad del proteoma . Las isoformas a nivel de ARN se caracterizan fácilmente mediante estudios de transcripción de ADNc . Muchos genes humanos poseen isoformas de empalme alternativo confirmadas . Se ha estimado que se pueden identificar ~100.000 etiquetas de secuencia expresada ( EST ) en humanos. [1] Las isoformas a nivel de proteína pueden manifestarse en la eliminación de dominios completos o bucles más cortos, generalmente ubicados en la superficie de la proteína. [3]
Definición
Un solo gen tiene la capacidad de producir múltiples proteínas que difieren tanto en estructura como en composición; [4] [5] este proceso está regulado por el empalme alternativo del ARNm, aunque no está claro en qué medida dicho proceso afecta la diversidad del proteoma humano, ya que la abundancia de isoformas de transcripción del ARNm no se correlaciona necesariamente con la abundancia de isoformas de proteína. [6] Las comparaciones de la estructura tridimensional de la proteína se pueden utilizar para ayudar a determinar qué isoformas, si las hay, representan productos proteicos funcionales, y la estructura de la mayoría de las isoformas en el proteoma humano ha sido predicha por AlphaFold y publicada públicamente en isoform.io. [7] La especificidad de las isoformas traducidas se deriva de la estructura/función de la proteína, así como del tipo de célula y la etapa de desarrollo durante la cual se producen. [4] [5] Determinar la especificidad se vuelve más complicado cuando una proteína tiene múltiples subunidades y cada subunidad tiene múltiples isoformas.
Por ejemplo, la proteína quinasa activada por AMP 5' (AMPK), una enzima que desempeña diferentes funciones en las células humanas, tiene 3 subunidades: [8]
- α, dominio catalítico, tiene dos isoformas: α1 y α2 que están codificadas a partir de PRKAA1 y PRKAA2
- β, dominio regulador, tiene dos isoformas: β1 y β2 que están codificadas a partir de PRKAB1 y PRKAB2
- γ, dominio regulador, tiene tres isoformas: γ1, γ2 y γ3 que están codificadas a partir de PRKAG1 , PRKAG2 y PRKAG3.
En el músculo esquelético humano, la forma preferida es α2β2γ1. [8] Pero en el hígado humano, la forma más abundante es α1β2γ1. [8]
Mecanismo

Los mecanismos primarios que producen isoformas de proteínas son el empalme alternativo y el uso variable del promotor, aunque las modificaciones debidas a cambios genéticos, como mutaciones y polimorfismos , a veces también se consideran isoformas distintas. [9]
El empalme alternativo es el principal proceso de modificación postranscripcional que produce isoformas de transcripción del ARNm y es un mecanismo molecular importante que puede contribuir a la diversidad de proteínas. [5] El espliceosoma , una gran ribonucleoproteína , es la máquina molecular dentro del núcleo responsable de la escisión y ligadura del ARN , eliminando segmentos no codificantes de proteínas ( intrones ). [10]
Debido a que el empalme es un proceso que ocurre entre la transcripción y la traducción , sus efectos primarios se han estudiado principalmente a través de técnicas genómicas ; por ejemplo, se han utilizado análisis de microarrays y secuenciación de ARN para identificar transcripciones empalmadas alternativamente y medir sus abundancias. [9] La abundancia de transcripciones se utiliza a menudo como un indicador de la abundancia de isoformas de proteínas, aunque los experimentos de proteómica que utilizan electroforesis en gel y espectrometría de masas han demostrado que la correlación entre las transcripciones y los recuentos de proteínas suele ser baja, y que una isoforma de proteína suele ser dominante. [11] Un estudio de 2015 afirma que la causa de esta discrepancia probablemente ocurre después de la traducción, aunque el mecanismo es esencialmente desconocido. [12] En consecuencia, aunque el empalme alternativo se ha implicado como un vínculo importante entre la variación y la enfermedad, no hay evidencia concluyente de que actúe principalmente produciendo nuevas isoformas de proteínas. [11]
El empalme alternativo generalmente describe un proceso estrictamente regulado en el que las transcripciones alternativas son generadas intencionalmente por la maquinaria de empalme. Sin embargo, dichas transcripciones también son producidas por errores de empalme en un proceso llamado "empalme ruidoso", y también son potencialmente traducidas en isoformas de proteínas. Aunque se cree que aproximadamente el 95% de los genes multiexónicos son empalmados alternativamente, un estudio sobre empalme ruidoso observó que la mayoría de las diferentes transcripciones de baja abundancia son ruido, y predice que la mayoría de las transcripciones alternativas e isoformas de proteínas presentes en una célula no son funcionalmente relevantes. [13]
Otros pasos reguladores transcripcionales y postranscripcionales también pueden producir diferentes isoformas de proteínas. [14] El uso variable del promotor ocurre cuando la maquinaria transcripcional de una célula ( ARN polimerasa , factores de transcripción y otras enzimas ) comienza la transcripción en diferentes promotores (la región de ADN cerca de un gen que sirve como sitio de unión inicial), lo que da como resultado transcripciones e isoformas de proteínas ligeramente modificadas.
Características
En general, una isoforma de proteína se etiqueta como la secuencia canónica en función de criterios como su prevalencia y similitud con secuencias ortólogas (o funcionalmente análogas) en otras especies. [15] Se supone que las isoformas tienen propiedades funcionales similares, ya que la mayoría tienen secuencias similares y comparten algunos o la mayoría de los exones con la secuencia canónica. Sin embargo, algunas isoformas muestran una divergencia mucho mayor (por ejemplo, a través del trans-splicing ) y pueden compartir pocos o ningún exón con la secuencia canónica. Además, pueden tener diferentes efectos biológicos (por ejemplo, en un caso extremo, la función de una isoforma puede promover la supervivencia celular, mientras que otra promueve la muerte celular) o pueden tener funciones básicas similares pero diferir en su localización subcelular. [16] Sin embargo, un estudio de 2016 caracterizó funcionalmente todas las isoformas de 1492 genes y determinó que la mayoría de las isoformas se comportan como "aloformas funcionales". Los autores llegaron a la conclusión de que las isoformas se comportan como proteínas distintas después de observar que las funciones de la mayoría de las isoformas no se superponían. [17] Debido a que el estudio se realizó en células in vitro , no se sabe si las isoformas en el proteoma humano expresado comparten estas características. Además, debido a que la función de cada isoforma generalmente debe determinarse por separado, la mayoría de las isoformas identificadas y predichas aún tienen funciones desconocidas.
Conceptos relacionados
Glicoforma
Una glicoforma es una isoforma de una proteína que difiere solo con respecto al número o tipo de glicano unido . Las glicoproteínas a menudo consisten en varias glicoformas diferentes, con alteraciones en el sacárido u oligosacárido unido . Estas modificaciones pueden resultar de diferencias en la biosíntesis durante el proceso de glicosilación , o debido a la acción de glicosidasas o glicosiltransferasas . Las glicoformas se pueden detectar a través de un análisis químico detallado de glicoformas separadas, pero se detectan más convenientemente a través de una reacción diferencial con lectinas , como en la cromatografía de afinidad de lectina y la electroforesis de afinidad de lectina . Ejemplos típicos de glicoproteínas que consisten en glicoformas son las proteínas sanguíneas como orosomucoide , antitripsina y haptoglobina . Una variación inusual de glicoforma se observa en la molécula de adhesión celular neuronal, NCAM que involucra ácidos polisiálicos, PSA .
Ejemplos
- G-actina : a pesar de su naturaleza conservada, tiene un número variable de isoformas (al menos seis en los mamíferos).
- La creatina quinasa , cuya presencia en la sangre puede utilizarse como ayuda en el diagnóstico del infarto de miocardio , existe en tres isoformas.
- La hialuronano sintasa , la enzima responsable de la producción de hialuronano, tiene tres isoformas en las células de mamíferos.
- La UDP-glucuronosiltransferasa , una superfamilia de enzimas responsable de la vía de desintoxicación de muchos fármacos, contaminantes ambientales y compuestos endógenos tóxicos, tiene 16 isoformas conocidas codificadas en el genoma humano. [18]
- G6PDA: la proporción normal de isoformas activas en las células de cualquier tejido es de 1:1 compartida con G6PDG. Esta es precisamente la proporción normal de isoformas en la hiperplasia. Solo una de estas isoformas se encuentra durante la neoplasia. [19]
La monoaminooxidasa , una familia de enzimas que catalizan la oxidación de monoaminas, existe en dos isoformas, MAO-A y MAO-B.
Véase también
Referencias
- ^ ab Brett D, Pospisil H, Valcárcel J, Reich J, Bork P (enero de 2002). "Empalme alternativo y complejidad del genoma". Genética de la Naturaleza . 30 (1): 29–30. doi :10.1038/ng803. PMID 11743582. S2CID 2724843.
- ^ Schlüter H, Apweiler R, Holzhütter HG, Jungblut PR (septiembre de 2009). "Encontrar el camino en proteómica: una nomenclatura de especies de proteínas". Chemistry Central Journal . 3 : 11. doi : 10.1186/1752-153X-3-11 . PMC 2758878 . PMID 19740416.
- ^ Kozlowski, L.; Orlowski, J.; Bujnicki, JM (2012). "Predicción de la estructura para proteínas empalmadas alternativamente". Empalme alternativo de pre-ARNm . p. 582. doi :10.1002/9783527636778.ch54. ISBN 9783527636778.
- ^ ab Andreadis A, Gallego ME, Nadal-Ginard B (1987-01-01). "Generación de diversidad de isoformas proteicas mediante splicing alternativo: implicaciones mecanicistas y biológicas". Revisión anual de biología celular . 3 (1): 207–42. doi :10.1146/annurev.cb.03.110187.001231. PMID 2891362.
- ^ abc Breitbart RE, Andreadis A, Nadal-Ginard B (1 de enero de 1987). "Empalme alternativo: un mecanismo ubicuo para la generación de múltiples isoformas proteicas a partir de genes individuales". Revisión anual de bioquímica . 56 (1): 467–95. doi :10.1146/annurev.bi.56.070187.002343. PMID 3304142.
- ^ Liu Y, Beyer A, Aebersold R (abril de 2016). "Sobre la dependencia de los niveles de proteína celular en la abundancia de ARNm". Cell . 165 (3): 535–50. doi : 10.1016/j.cell.2016.03.014 . hdl : 20.500.11850/116226 . PMID 27104977.
- ^ Sommer, Markus J.; Cha, Sooyoung; Varabyou, Ales; Rincon, Natalia; Park, Sukhwan; Minkin, Ilia; Pertea, Mihaela; Steinegger, Martin; Salzberg, Steven L. (15 de diciembre de 2022). "Identificación de isoformas guiada por la estructura para el transcriptoma humano". eLife . 11 : e82556. doi : 10.7554/eLife.82556 . PMC 9812405 . PMID 36519529.
- ^ abc Dasgupta B, Chhipa RR (marzo de 2016). "Lecciones en evolución sobre el papel complejo de la AMPK en la fisiología normal y el cáncer". Tendencias en ciencias farmacológicas . 37 (3): 192–206. doi :10.1016/j.tips.2015.11.007. PMC 4764394 . PMID 26711141.
- ^ ab Kornblihtt AR, Schor IE, Alló M, Dujardin G, Petrillo E, Muñoz MJ (marzo de 2013). "Empalme alternativo: un paso fundamental entre la transcripción y la traducción eucariotas". Nature Reviews Molecular Cell Biology . 14 (3): 153–65. doi :10.1038/nrm3525. hdl : 11336/21049 . PMID 23385723. S2CID 54560052.
- ^ Lee Y, Rio DC (1 de enero de 2015). "Mecanismos y regulación del empalme alternativo de pre-ARNm". Revisión anual de bioquímica . 84 (1): 291–323. doi :10.1146/annurev-biochem-060614-034316. PMC 4526142 . PMID 25784052.
- ^ ab Tress ML, Abascal F, Valencia A (febrero de 2017). "El empalme alternativo puede no ser la clave de la complejidad del proteoma". Tendencias en ciencias bioquímicas . 42 (2): 98–110. doi :10.1016/j.tibs.2016.08.008. PMC 6526280 . PMID 27712956.
- ^ Battle A, Khan Z, Wang SH, Mitrano A, Ford MJ, Pritchard JK, Gilad Y (febrero de 2015). "Variación genómica. Impacto de la variación regulatoria del ARN a la proteína". Science . 347 (6222): 664–7. doi :10.1126/science.1260793. PMC 4507520 . PMID 25657249.
- ^ Pickrell JK, Pai AA, Gilad Y, Pritchard JK (diciembre de 2010). "El empalme ruidoso impulsa la diversidad de isoformas del ARNm en células humanas". PLOS Genetics . 6 (12): e1001236. doi : 10.1371/journal.pgen.1001236 . PMC 3000347 . PMID 21151575.
- ^ Smith LM, Kelleher NL (marzo de 2013). "Proteoforma: un término único que describe la complejidad de las proteínas". Nature Methods . 10 (3): 186–7. doi :10.1038/nmeth.2369. PMC 4114032 . PMID 23443629.
- ^ Li HD, Menon R, Omenn GS, Guan Y (diciembre de 2014). "Revisitando la identificación de isoformas de empalme canónico a través de la integración de evidencia proteómica y genómica funcional" (PDF) . Proteomics . 14 (23–24): 2709–18. doi :10.1002/pmic.201400170. PMC 4372202 . PMID 25265570.
- ^ Sundvall M, Veikkolainen V, Kurppa K, Salah Z, Tvorogov D, van Zoelen EJ, Aqeilan R, Elenius K (diciembre de 2010). "Muerte o supervivencia celular promovida por isoformas alternativas de ErbB4". Biología Molecular de la Célula . 21 (23): 4275–86. doi :10.1091/mbc.E10-04-0332. PMC 2993754 . PMID 20943952.
- ^ Yang X, Coulombe-Huntington J, Kang S, Sheynkman GM, Hao T, Richardson A, et al. (febrero de 2016). "Expansión generalizada de las capacidades de interacción de proteínas mediante empalme alternativo". Cell . 164 (4): 805–17. doi :10.1016/j.cell.2016.01.029. PMC 4882190 . PMID 26871637.
- ^ Barre L, Fournel-Gigleux S, Finel M, Netter P, Magdalou J, Ouzzine M (marzo de 2007). "Especificidad de sustrato de la UDP-glucuronosiltransferasa humana UGT2B4 y UGT2B7. Identificación de un residuo crítico de aminoácido aromático en la posición 33". The FEBS Journal . 274 (5): 1256–64. doi : 10.1111/j.1742-4658.2007.05670.x . PMID 17263731.
- ^ Pathoma, Fundamentos de patología
Enlaces externos
- Isoformas de proteínas de entrada MeSH
- Definiciones Isoforma
