Taxonomía evolutiva
This article may be too technical for most readers to understand. (July 2018) |
La taxonomía evolutiva , sistemática evolutiva o clasificación darwiniana es una rama de la clasificación biológica que busca clasificar los organismos utilizando una combinación de relación filogenética (descendencia compartida), relación progenitor-descendiente (descendencia serial) y grado de cambio evolutivo . Este tipo de taxonomía puede considerar taxones completos en lugar de especies individuales , de modo que se puede inferir que grupos de especies dan origen a nuevos grupos. [1] El concepto encontró su forma más conocida en la síntesis evolutiva moderna de principios de la década de 1940.
La taxonomía evolutiva difiere de la taxonomía linneana estricta predarwinista (que produce sólo listas ordenadas) en que construye árboles evolutivos. Mientras que en la nomenclatura filogenética cada taxón debe constar de un único nodo ancestral y todos sus descendientes, la taxonomía evolutiva permite excluir grupos de sus taxones progenitores (por ejemplo, no se considera que los dinosaurios incluyan a las aves, sino que las hayan originado ), lo que permite taxones parafiléticos . [2] [3]
Origen de la taxonomía evolutiva

La taxonomía evolutiva surgió como resultado de la influencia de la teoría de la evolución en la taxonomía de Linneo . La idea de traducir la taxonomía de Linneo en una especie de dendrograma de los reinos animal y vegetal fue formulada hacia fines del siglo XVIII, mucho antes de que se publicara el libro de Charles Darwin El origen de las especies . [4] El primero en sugerir que los organismos tenían descendencia común fue Pierre-Louis Moreau de Maupertuis en su Essai de Cosmologie de 1751 , [5] [6] La transmutación de especies entró en círculos científicos más amplios con Zoönomia de Erasmus Darwin de 1796 y Philosophie Zoologique de Jean-Baptiste Lamarck de 1809. [7] La idea se popularizó en el mundo angloparlante con la especulativa pero ampliamente leída Vestiges of the Natural History of Creation , publicada anónimamente por Robert Chambers en 1844. [8]
Tras la aparición de El origen de las especies , las representaciones del árbol de la vida se hicieron populares en las obras científicas. En El origen de las especies , el ancestro siguió siendo en gran medida una especie hipotética; Darwin se ocupó principalmente de demostrar el principio, absteniéndose cuidadosamente de especular sobre las relaciones entre organismos vivos o fósiles y utilizando solo ejemplos teóricos. [7] En contraste, Chambers había propuesto hipótesis específicas, por ejemplo, la evolución de los mamíferos placentarios a partir de los marsupiales. [9]
Tras la publicación de Darwin, Thomas Henry Huxley utilizó los fósiles de Archaeopteryx y Hesperornis para argumentar que las aves son descendientes de los dinosaurios. [10] De este modo, un grupo de animales actuales podría vincularse a un grupo fósil. La descripción resultante, la de que los dinosaurios "dieron origen" a las aves o fueron "los ancestros de ellas", exhibe el sello distintivo esencial del pensamiento taxonómico evolutivo.
En las últimas tres décadas se ha producido un aumento espectacular del uso de secuencias de ADN para reconstruir la filogenia y un cambio paralelo en el énfasis de la taxonomía evolutiva hacia la «sistemática filogenética» de Hennig. [7]
Hoy, con la llegada de la genómica moderna , los científicos de todas las ramas de la biología utilizan la filogenia molecular para orientar sus investigaciones. Un método común es el alineamiento de secuencias múltiples . [ cita requerida ]
Thomas Cavalier-Smith , [7] GG Simpson y Ernst Mayr [11] son algunos taxónomos evolutivos representativos.
Nuevos métodos en la sistemática evolutiva moderna
This section may rely excessively on sources too closely associated with the subject, potentially preventing the article from being verifiable and neutral. (July 2018) |
Recientemente han surgido intentos de combinar métodos modernos de cladística, filogenética y análisis de ADN con puntos de vista clásicos de la taxonomía. Algunos autores han descubierto que el análisis filogenético es aceptable científicamente siempre que se permita la parafilia, al menos para ciertos grupos. Esta postura es promovida en artículos de Tod F. Stuessy [12] y otros. Richard H. Zander ha presentado una forma particularmente estricta de sistemática evolutiva en varios artículos, pero la resume en su "Marco para la sistemática postfilogenética". [13]
En resumen, la sistemática pluralista de Zander se basa en la incompletitud de cada una de las teorías: un método que no puede refutar una hipótesis es tan poco científico como una hipótesis que no puede ser refutada. La cladística genera sólo árboles de ascendencia compartida, no de ascendencia serial. La evolución de taxones en serie no puede abordarse analizando la ascendencia compartida con métodos cladísticos. Hipótesis como la radiación adaptativa de un único taxón ancestral no pueden ser refutadas con la cladística. La cladística ofrece una manera de agrupar por transformaciones de rasgos, pero ningún árbol evolutivo puede ser completamente dicotómico. La filogenética postula taxones ancestrales compartidos como agentes causales de dicotomías, pero no hay evidencia de la existencia de tales taxones. La sistemática molecular utiliza datos de secuencias de ADN para rastrear cambios evolutivos, por lo que la parafilia y, a veces, la polifilia filogenética señalan transformaciones ancestro-descendiente a nivel de taxón, pero por lo demás la filogenética molecular no prevé la parafilia extinta. Se necesita un análisis transformacional adicional para inferir la descendencia serial.

El cactus o comagrama de Bessey es el mejor árbol evolutivo para mostrar tanto la ascendencia compartida como la serial. Primero, se genera un cladograma o clave natural. Se identifican los taxones ancestrales generalizados y se anotan los taxones descendientes especializados que se desprenden del linaje con una línea de un color que representa al progenitor a través del tiempo. Luego se diseña un cactus o comagrama de Bessey que representa tanto la ascendencia compartida como la serial. Los taxones progenitores pueden tener uno o más taxones descendientes. Se pueden proporcionar medidas de apoyo en términos de factores de Bayes, siguiendo el método de análisis transformacional de Zander utilizando decibanes.
El análisis cladístico agrupa a los taxones en función de sus rasgos compartidos, pero incorpora un modelo de ramificación dicotómica tomado de la fenética. Es esencialmente una clave natural dicotómica simplificada, aunque se toleran las inversiones. El problema, por supuesto, es que la evolución no es necesariamente dicotómica. Un taxón ancestral que genera dos o más descendientes requiere un árbol más largo y menos parsimonioso. Un nodo de cladograma resume todos los rasgos distales a él, no de ningún taxón en particular, y la continuidad en un cladograma es de nodo a nodo, no de taxón a taxón. Este no es un modelo de evolución, sino una variante del análisis de conglomerados jerárquico (cambios de rasgos y ramas no ultramétricas). Por eso, un árbol basado únicamente en rasgos compartidos no se llama árbol evolutivo, sino simplemente árbol cladístico. Este árbol refleja en gran medida las relaciones evolutivas a través de las transformaciones de los rasgos, pero ignora las relaciones creadas por la transformación a nivel de especie de los taxones existentes.
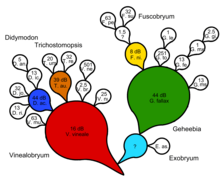
La filogenética intenta introducir un elemento serial postulando ancestros compartidos ad hoc, no demostrables, en cada nodo de un árbol cladístico. Para un cladograma completamente dicotómico, hay un ancestro compartido invisible menos que el número de taxones terminales. Obtenemos, en efecto, una clave natural dicotómica con un ancestro compartido invisible que genera cada par. Esto no puede implicar una explicación basada en procesos sin la justificación de la dicotomía y la suposición de los ancestros compartidos como causas. La forma cladística de análisis de las relaciones evolutivas no puede refutar ningún escenario evolutivo genuino que incorpore la transformación serial, según Zander. [14]
Zander ha detallado métodos para generar medidas de soporte para la descendencia serial molecular [15] y para la descendencia serial morfológica utilizando factores de Bayes y análisis Bayes secuencial a través del deciban de Turing o la adición de bits informativos de Shannon. [16] [17] [18] [19]
El árbol de la vida

A finales del siglo XIX y principios del XX, a medida que se fueron descubriendo y reconociendo más y más grupos de fósiles, los paleontólogos trabajaron para comprender la historia de los animales a lo largo de los siglos vinculando los grupos conocidos. [20] El árbol de la vida se fue trazando lentamente y los grupos de fósiles fueron ocupando su lugar en el árbol a medida que aumentaba la comprensión. [ cita requerida ]
Estos grupos aún conservaron sus rangos taxonómicos linneanos formales . Algunos de ellos son parafiléticos en el sentido de que, aunque cada organismo del grupo está vinculado a un ancestro común por una cadena ininterrumpida de ancestros intermedios dentro del grupo, algunos otros descendientes de ese ancestro se encuentran fuera del grupo. La evolución y distribución de los diversos taxones a través del tiempo se muestra comúnmente como un diagrama de husos (a menudo llamado Romerograma en honor al paleontólogo estadounidense Alfred Romer ) donde varios husos se ramifican entre sí, y cada huso representa un taxón. El ancho de los husos pretende implicar la abundancia (a menudo el número de familias) representada en función del tiempo. [21]
A finales del siglo XIX , la paleontología de vertebrados había trazado la secuencia evolutiva de los vertebrados tal como se la conoce actualmente, y a principios del siglo XX se llegó a una comprensión razonable de la secuencia evolutiva del reino vegetal . La unión de los distintos árboles en un gran Árbol de la Vida sólo fue posible gracias a los avances en microbiología y bioquímica en el período entre las dos guerras mundiales. [ cita requerida ]
Diferencia terminológica
Los dos enfoques, la taxonomía evolutiva y la sistemática filogenética derivada de Willi Hennig , difieren en el uso de la palabra " monofilético ". Para los sistemáticos evolucionistas, "monofilético" significa solo que un grupo se deriva de un único ancestro común. En la nomenclatura filogenética , hay una advertencia adicional de que las especies ancestrales y todos los descendientes deben incluirse en el grupo. [2] El término " holofilético " se ha propuesto para el último significado. Como ejemplo, los anfibios son monofiléticos bajo la taxonomía evolutiva, ya que han surgido de los peces solo una vez. Bajo la taxonomía filogenética, los anfibios no constituyen un grupo monofilético en el sentido de que los amniotas ( reptiles , aves y mamíferos ) han evolucionado a partir de un ancestro anfibio y, sin embargo, no se consideran anfibios. Dichos grupos parafiléticos son rechazados en la nomenclatura filogenética, pero los taxónomos evolucionistas los consideran una señal de descendencia serial. [22]
Referencias
- ^ Mayr, Ernst y Bock, WJ (2002), "Clasificaciones y otros sistemas de ordenación" (PDF) , J. Zool. Syst. Evol. Research , 40 (4): 169–94, doi :10.1046/j.1439-0469.2002.00211.x
- ^ ab Grant, V. (2003), "Incongruencia entre los sistemas cladísticos y taxonómicos", American Journal of Botany , 90 (9): 1263–70, doi :10.3732/ajb.90.9.1263, PMID 21659226
- ^ Aubert, D (2015). "Un análisis formal de la terminología filogenética: hacia una reconsideración del paradigma actual en sistemática". Phytoneuron . 2015–66: 1–54.
- ^ Archibald, J. David (2009). "El 'árbol de la vida' predarwinista (1840) de Edward Hitchcock" (PDF) . Revista de Historia de la Biología . 42 (3): 561–592. CiteSeerX 10.1.1.688.7842 . doi :10.1007/s10739-008-9163-y. PMID 20027787. S2CID 16634677.
- ^ JS Bromley, La nueva historia moderna de Cambridge: El ascenso de Gran Bretaña y Rusia, 1688-1715/25, Archivo CUP, 1970, ISBN 978-0-521-07524-4 , págs. 62-63.
- ^ Geoffrey Russell Richards Treasure, La creación de la Europa moderna, 1648-1780, Taylor & Francis, 1985, ISBN 978-0-416-72370-0 , pág. 142
- ^ abcd Cavalier-Smith, Thomas (2010). "Filogenia profunda, grupos ancestrales y las cuatro edades de la vida" (PDF) . Philosophical Transactions of the Royal Society B . 365 (1537): 111–132. doi :10.1098/rstb.2009.0161. PMC 2842702 . PMID 20008390.
- ^ Secord, James A. (2000), Sensación victoriana: la extraordinaria publicación, recepción y autoría secreta de vestigios de la historia natural de la creación, Chicago: University of Chicago Press, ISBN 978-0-852-3-5 978-0-226-74410-0, archivado desde el original el 16 de mayo de 2008 , consultado el 17 de enero de 2011
- ^ Chambers, Robert (1844). Vestiges of the Natural History of Creation. Londres: John Churchill. p. 122. Consultado el 10 de mayo de 2013 .
- ^ Huxley, TH (1876): Lecciones sobre la evolución. New York Tribune . Extra. no 36. En Collected Essays IV: pp 46-138 texto original con figuras
- ^ Mahner, Martín; Bunge, Mario. Fundamentos de la Biofilosofía . Springer, pág. 251.
- ^ Stuessy, Tod F. (2010). "Parafilia y origen y clasificación de las angiospermas". Taxon . 59 (3): 689–693. doi :10.1002/tax.593001.
- ^ Zander, Richard (2013). Marco para la sistemática postfilogenética . St. Louis: Zetetic Publications, Amazon CreateSpace.
- ^ Zander, Richard (2014). "Respuesta a una crítica particularmente desagradable en la revista Cladistics". Phytoneuron . 2014 (110): 1–4.
- ^ Zander, Richard (2014). "Medidas de apoyo para transformaciones macroevolutivas caulísticas en árboles evolutivos". Anales del Jardín Botánico de Missouri . 100 (1–2): 100–107. doi :10.3417/2012090. S2CID 86754093.
- ^ Zander, Richard (2014). «Determinación clásica de la monofilia ejemplificada con Didymodon s. lat. (Bryophyta). Parte 1 de 3, sinopsis y conceptos simplificados» (PDF) . Phytoneuron . 2014 (78): 1–7 . Consultado el 4 de enero de 2015 .
- ^ Zander, Richard (2014). «Determinación clásica de la monofilia, ejemplificada con Didymodon s. lat. (Bryophyta). Parte 2 de 3, conceptos» (PDF) . Phytoneuron . 2014 (79): 1–23 . Consultado el 4 de enero de 2015 .
- ^ Zander, Richard (2014). «Determinación clásica de monofilia ejemplificada con Didymodon s. lat. (Bryophyta). Parte 3 de 3, análisis» (PDF) . Phytoneuron . 2014 (80): 1–19 . Consultado el 4 de enero de 2015 .
- ^ Zander RH 2018. Sistemática macroevolutiva de Streptotrichaceae de Bryophyta y aplicación a la estabilidad termodinámica de los ecosistemas. Edición 2. Zetetic Publications, CreateSpace Independent Publishing, Amazon, St. Louis.
- ^ Rudwick, MJS (1985), El significado de los fósiles: episodios en la historia de la paleontología , University of Chicago Press , pág. 24, ISBN 978-0-226-73103-2
- ^ White, T.; Kazlev, MA "Filogenia y sistemática: glosario - Romerograma". palaeos.com . Consultado el 21 de enero de 2013 .
- ^ Zander, RH (2008). "Inferencias evolutivas a partir de la no monofilia en árboles moleculares". Taxon . 57 (4): 1182–1188. doi :10.1002/tax.574011.

