Replicación viral
Este artículo necesita citas adicionales para su verificación . ( abril de 2020 ) |
| Ciclo de vida del virus de la influenza |
|---|
 |
|
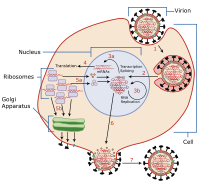
La replicación viral es la formación de virus biológicos durante el proceso de infección en las células huésped diana. Los virus deben primero entrar en la célula para que pueda producirse la replicación viral. Mediante la generación de abundantes copias de su genoma y el empaquetamiento de estas copias, el virus continúa infectando nuevos huéspedes. La replicación entre virus es muy variada y depende del tipo de genes implicados en ellos. La mayoría de los virus ADN se ensamblan en el núcleo, mientras que la mayoría de los virus ARN se desarrollan únicamente en el citoplasma. [1]
Producción/replicación viral
Los virus se multiplican únicamente en células vivas. La célula huésped debe proporcionar la energía , la maquinaria sintética y los precursores de bajo peso molecular para la síntesis de proteínas y ácidos nucleicos virales. [2]
La replicación del virus ocurre en siete etapas:
- Adjunto
- Entrada (penetración)
- Sin recubrimiento
- Replicación
- Asamblea
- Maduración
- Liberación (Etapa de Liberación).
Adjunto
Es el primer paso de la replicación viral. Algunos virus se adhieren a la membrana celular de la célula huésped e inyectan su ADN o ARN en el huésped para iniciar la infección. La adhesión a una célula huésped a menudo se logra mediante una proteína de adhesión del virus que se extiende desde la cubierta proteica, también conocida como cápside , de un virus. Esta proteína es responsable de la unión a un receptor de superficie en la membrana plasmática (o carbohidratos de membrana) de una célula huésped. Los virus pueden explotar las funciones normales del receptor celular para permitir que se produzca la adhesión imitando las moléculas que se unen a los receptores de la célula huésped. Por ejemplo, el rinovirus utiliza su proteína de adhesión del virus para unirse al receptor ICAM-1 en las células huésped que normalmente se utiliza para facilitar la adhesión entre otras células huésped. [3]
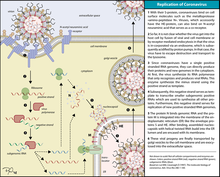
Entrada
La entrada, o penetración, es el segundo paso en la replicación viral. Este paso se caracteriza por el paso del virus a través de la membrana plasmática de la célula huésped. La forma más común en que un virus logra entrar en la célula huésped es mediante endocitosis mediada por receptores , que no tiene ningún costo energético para el virus, solo para la célula huésped. La endocitosis mediada por receptores ocurre cuando una molécula (en este caso un virus) se une a un receptor en la membrana de la célula. Una serie de señales químicas de esta unión hace que la célula envuelva al virus adherido en la membrana plasmática a su alrededor formando una vesícula que contiene el virus dentro de la célula. [3]
Los virus entran en las células huésped mediante una variedad de mecanismos, incluidas las rutas endocíticas y no endocíticas. [4] También pueden fusionarse en la membrana plasmática y pueden propagarse dentro del huésped a través de la fusión o fusión célula-célula. [5] Los virus se adhieren a las proteínas en la superficie de la célula huésped conocidas como receptores celulares o factores de adhesión para ayudar a la entrada. [6] La evidencia muestra que los virus utilizan canales iónicos en las células huésped durante la entrada viral. Fusión: Las proteínas virales externas promueven la fusión del virión con la membrana plasmática. [7] Esto forma un poro en la membrana del huésped, y después de la entrada, el virión queda sin cubierta y su material genómico luego se transfiere al citoplasma. [8] Fusión de célula a célula: Algunos virus estimulan la expresión de proteínas específicas en las superficies de las células infectadas para atraer células no infectadas. [9] Esta interacción hace que la célula no infectada se fusione con la célula infectada a niveles de pH más bajos para formar una célula multinuclear conocida como sincitio. [10] Vías endocíticas: proceso por el cual se forma una vesícula intracelular por invaginación de la membrana, lo que da como resultado la ingestión de componentes extracelulares y unidos a la membrana, en este contexto, un virus. [11] Vías no endocíticas: proceso por el cual las partículas virales se liberan en la célula por fusión de la envoltura viral extracelular y la membrana de la célula huésped. [4]
Sin recubrimiento
El desprendimiento de la envoltura es el tercer paso en la replicación viral. El desprendimiento de la envoltura se define como la eliminación de la "envoltura" proteica del virión y la liberación de su material genético. Este paso ocurre en la misma área en la que ocurre la transcripción viral. Diferentes virus tienen diversos mecanismos para el desprendimiento de la envoltura. Algunos virus de ARN, como los rinovirus, utilizan el pH bajo en los endosomas de una célula huésped para activar su mecanismo de desprendimiento de la envoltura. Esto implica que el rinovirus libera una proteína que crea agujeros en el endosoma y permite que el virus libere su genoma a través de los agujeros. Muchos virus de ADN viajan al núcleo de la célula huésped y liberan su material genético a través de los poros nucleares. [3]
Replicación
El cuarto paso del ciclo viral es la replicación, que se define por la rápida producción del genoma viral. La forma en que un virus experimenta la replicación depende del tipo de material genético que posee el virus. En función de su material genético, los virus secuestrarán la maquinaria celular correspondiente para dicho material genético. Los virus que contienen ADN de doble cadena (dsDNA) comparten el mismo tipo de material genético que todos los organismos y, por lo tanto, pueden utilizar las enzimas de replicación en el núcleo de la célula huésped para replicar el genoma viral. Muchos virus de ARN normalmente se replican en el citosol y pueden acceder directamente a los ribosomas de la célula huésped para fabricar proteínas virales una vez que el ARN está en forma replicativa.
Los virus pueden atravesar dos tipos de ciclos de vida: el ciclo lítico y el ciclo lisogénico. En el ciclo lítico, el virus introduce su genoma en una célula huésped e inicia la replicación secuestrando la maquinaria celular del huésped para hacer nuevas copias del virus. [12] En el ciclo de vida lisogénico, el genoma viral se incorpora al genoma del huésped. El genoma del huésped atravesará su ciclo de vida normal, replicándose y dividiéndose, replicando el genoma viral junto con el suyo propio. [13] El genoma viral puede ser activado para comenzar la producción viral a través de estimulantes químicos y ambientales. [14] Una vez que un virus lisogénico entra en el ciclo de vida lítico, continuará en las vías de producción viral y procederá con la transcripción / producción de ARNm. (p. ej.: herpes labial, virus del herpes simple (HSV)-1, bacteriófagos lisogénicos, etc.)
Asamblea
El ensamblaje es el proceso en el que las proteínas y genomas virales recién fabricados se reúnen y se juntan para formar virus inmaduros. Al igual que en los otros pasos, la forma en que se ensambla un virus en particular depende del tipo de virus que sea. El ensamblaje puede ocurrir en la membrana plasmática, el citosol, el núcleo, el aparato de Golgi y otras ubicaciones dentro de la célula huésped. Algunos virus solo insertan su genoma en una cápside una vez que esta se completa, mientras que en otros virus la cápside envolverá al genoma a medida que se copia. [2]
Maduración
Este es el paso final antes de que se forme un virus competente. Esto generalmente implica modificaciones de la cápside que se proporcionan a las enzimas (codificadas por el huésped o por el virus). [3]
Liberación (etapa de liberación)
El paso final de la replicación viral es la liberación, que es cuando los virus recién ensamblados y maduros abandonan la célula huésped. La forma en que un virus se libera de la célula huésped depende del tipo de virus que sea. Un tipo común de liberación es la gemación. Esto ocurre cuando los virus que forman su envoltura a partir de la membrana plasmática del huésped doblan la membrana alrededor de la cápside. A medida que el virus dobla la membrana plasmática, comienza a envolver toda la cápside hasta que el virus ya no está adherido a la célula huésped. Otra forma común en que los virus abandonan la célula huésped es a través de la lisis celular , donde los virus lisan la célula haciendo que estalle, lo que libera los virus maduros que estaban en la célula huésped. [3]
Sistema de clasificación de Baltimore
Los virus se clasifican en siete tipos de genes, cada uno de los cuales tiene sus propias familias de virus, que a su vez tienen diferentes estrategias de replicación. [15] David Baltimore , un biólogo ganador del Premio Nobel , ideó un sistema llamado Sistema de Clasificación de Baltimore para clasificar diferentes virus en función de su estrategia de replicación única. Hay siete estrategias de replicación diferentes basadas en este sistema (Clase Baltimore I, II, III, IV, V, VI, VII). Las siete clases de virus se enumeran aquí brevemente y en generalidades. [16]
Clase 1: Virus de ADN bicatenario
Este tipo de virus normalmente debe entrar en el núcleo del huésped antes de poder replicarse. Algunos de estos virus requieren polimerasas de la célula huésped para replicar su genoma , mientras que otros, como los adenovirus o los virus del herpes, codifican sus propios factores de replicación. Sin embargo, en ambos casos, la replicación del genoma viral depende en gran medida de un estado celular permisivo para la replicación del ADN y, por lo tanto, del ciclo celular . El virus puede inducir a la célula a sufrir una división celular forzada , lo que puede conducir a la transformación de la célula y, en última instancia, al cáncer . Un ejemplo de una familia dentro de esta clasificación es Adenoviridae .
Sólo existe un ejemplo bien estudiado en el que una familia de virus de clase 1 no se replica dentro del núcleo. Se trata de la familia Poxvirus , que comprende virus altamente patógenos que infectan a los vertebrados .
Clase 2: Virus de ADN monocatenario
Los virus que entran en esta categoría incluyen aquellos que no han sido tan estudiados, pero que aún así son muy comunes en los vertebrados. Dos ejemplos son los Circoviridae y los Parvoviridae . Se replican dentro del núcleo y forman un intermediario de ADN de doble cadena durante la replicación. Un Anellovirus humano llamado TTV se incluye dentro de esta clasificación y se encuentra en casi todos los humanos, infectándolos asintomáticamente en casi todos los órganos principales .
Virus ARN: La polimerasa de los virus ARN carece de las funciones de corrección de errores que se encuentran en la polimerasa de los virus ADN. Esto contribuyó a que los virus ARN tuvieran una fidelidad replicativa menor en comparación con los virus ADN, lo que provocó que los virus ARN fueran altamente mutagénicos, lo que puede aumentar su tasa de supervivencia general. [17] Los virus ARN carecen de la capacidad de identificar y reparar nucleótidos mal emparejados o dañados y, por lo tanto, los genomas de ARN son propensos a mutaciones introducidas por mecanismos intrínsecos y extrínsecos a la replicación viral. [18] Los virus ARN presentan un arma terapéutica de doble filo: los virus ARN pueden resistir el desafío de los medicamentos antivirales, causar epidemias e infectar múltiples especies hospedadoras debido a su naturaleza mutagénica, lo que los hace difíciles de tratar. Sin embargo, la proteína transcriptasa inversa que a menudo viene con el virus ARN se puede utilizar como un objetivo indirecto para los virus ARN, lo que evita la transcripción y la síntesis de partículas virales. [19] (Esta es la base de los medicamentos contra el SIDA y el VIH [20] )
Clase 3: Virus de ARN bicatenario
Al igual que la mayoría de los virus con genomas de ARN , los virus de ARN bicatenario no dependen de las polimerasas del huésped para replicarse en la medida en que lo hacen los virus con genomas de ADN . Los virus de ARN bicatenario no se han estudiado tan bien como otras clases. Esta clase incluye dos familias principales, Reoviridae y Birnaviridae . La replicación es monocistrónica e incluye genomas individuales y segmentados, lo que significa que cada uno de los genes codifica solo una proteína, a diferencia de otros virus, que presentan una traducción más compleja.
Clases 4 y 5: Virus de ARN monocatenario
Estos virus se dividen en dos tipos, pero ambos comparten el hecho de que la replicación se produce principalmente en el citoplasma y no depende tanto del ciclo celular como en el caso de los virus de ADN. Esta clase de virus es también uno de los más estudiados, junto con los virus de ADN de doble cadena.
Clase 4: Virus de ARN monocatenario - sentido positivo
Los virus ARN de sentido positivo y, de hecho, todos los genes definidos como de sentido positivo pueden ser accedidos directamente por los ribosomas del huésped para formar inmediatamente proteínas. Estos pueden dividirse en dos grupos, los cuales se replican en el citoplasma:
- Virus con ARNm policistrónico , en los que el ARN genómico forma el ARNm y se traduce en un producto poliproteico que posteriormente se escinde para formar las proteínas maduras. Esto significa que el gen puede utilizar varios métodos para producir proteínas a partir de la misma cadena de ARN, lo que reduce el tamaño de su genoma.
- Virus con transcripción compleja, para lo cual se pueden utilizar ARNm subgenómicos , desplazamiento del marco ribosómico y procesamiento proteolítico de poliproteínas, todos ellos mecanismos diferentes para producir proteínas a partir de la misma hebra de ARN.
Ejemplos de esta clase incluyen las familias Coronaviridae , Flaviviridae y Picornaviridae .
Clase 5: Virus de ARN monocatenario - sentido negativo
Los virus de ARN de sentido negativo y, de hecho, todos los genes definidos como de sentido negativo no pueden ser accedidos directamente por los ribosomas del huésped para formar proteínas de inmediato. En cambio, deben ser transcritos por las polimerasas virales en el sentido positivo complementario "legible". Estos también pueden dividirse en dos grupos:
- Virus que contienen genomas no segmentados , cuyo primer paso en la replicación es la transcripción del genoma de cadena negativa por la ARN polimerasa dependiente del ARN viral para producir ARNm monocistrónicos que codifican las diversas proteínas virales. Luego se produce una copia del genoma de sentido positivo que sirve como plantilla para la producción del genoma de cadena negativa. La replicación se realiza dentro del citoplasma.
- Virus con genomas segmentados cuya replicación ocurre en el citoplasma y para los cuales la ARN polimerasa dependiente del ARN viral produce ARNm monocistrónicos a partir de cada segmento del genoma.
Los ejemplos de esta clase incluyen las familias Orthomyxoviridae , Paramyxoviridae , Bunyaviridae , Filoviridae y Rhabdoviridae (que incluye la rabia ).
Clase 6: Virus de ARN monocatenario de sentido positivo que se replican a través de un intermediario de ADN
Una familia muy estudiada de esta clase de virus son los retrovirus . Una característica definitoria es el uso de la transcriptasa inversa para convertir el ARN de sentido positivo en ADN. En lugar de utilizar el ARN como plantilla de proteínas, utilizan el ADN para crear las plantillas, que se empalma en el genoma del huésped mediante la integrasa . La replicación puede entonces comenzar con la ayuda de las polimerasas de la célula huésped.
Clase 7: Virus de ADN bicatenario que se replican a través de un ARN intermediario monocatenario
Este pequeño grupo de virus, ejemplificado por el virus de la hepatitis B , tiene un genoma bicatenario con huecos que posteriormente se rellena para formar un círculo cerrado covalentemente ( ADNccc ) que sirve como plantilla para la producción de ARNm virales y un ARN subgenómico . El ARN pregenómico sirve como plantilla para la transcriptasa inversa viral y para la producción del genoma de ADN.
Referencias
- ^ Roberts, RJ (2001). Patología de los peces (3.ª ed.). Elsevier Health Sciences.
- ^ ab Brooks, Geo.; Carroll, Karen C.; Butel, Janet; Morse, Stephen (21 de diciembre de 2012). Microbiología médica de Jawetz Melnick y Adelberg (26.ª edición). McGraw Hill Professional. ISBN 978-0-07-181578-9.
- ^ abcde Louten, Jennifer (2016). "Replicación de virus". Virología humana esencial . Elsevier. págs. 49–70. doi :10.1016/b978-0-12-800947-5.00004-1. ISBN 978-0-12-800947-5. Número de pieza 7149683 .
- ^ ab Dimitrov, Dimiter S. (febrero de 2004). "Entrada de virus: mecanismos moleculares y aplicaciones biomédicas". Nature Reviews Microbiology . 2 (2): 109–122. doi :10.1038/nrmicro817. PMC 7097642 . PMID 15043007.
- ^ Sobhy, Haitham (2017). "Una revisión comparativa de la entrada y la adhesión viral durante infecciones por virus dsDNA grandes y gigantes". Archivos de Virología . 162 (12): 3567–3585. doi :10.1007/s00705-017-3497-8. PMC 5671522 . PMID 28866775.
- ^ Marsh, M (2006). "Entrada de virus: ábrete sésamo". Cell . 124 (4). Cell Press: 729–740. doi :10.1016/j.cell.2006.02.007. PMC 7112260 . PMID 16497584.
- ^ Grove, Joe (2011). "La biología celular de la entrada de virus mediada por receptores". The Journal of Cell Biology . 195 (7). Journal of Cell Biology: 1071–1082. doi :10.1083/jcb.201108131. PMC 3246895 . PMID 22123832.
- ^ Kielian, Margaret (2006). "Proteínas de fusión de membranas virales: más de una forma de hacer una horquilla". Nature Reviews Microbiology . 4 (1). Nature: 67–76. doi :10.1038/nrmicro1326. PMC 7097298 . PMID 16357862.
- ^ Sattentau, Quentin (2008). "Cómo evitar el vacío: propagación de virus humanos de célula a célula". Nature Reviews Microbiology . 6 (11): 815–826. doi :10.1038/nrmicro1972. PMID 18923409.
- ^ Zhong, Peng (febrero de 2013). "Transmisión de virus de célula a célula". Current Opinion in Virology . Entrada de virus / Virología ambiental. 3 (1): 44–50. doi :10.1016/j.coviro.2012.11.004. PMC 3587356 . PMID 23219376.
- ^ Barrow, Eric (2013). "Perspectivas multiescala de la entrada del virus por endocitosis". Virology Journal . 10 : 177. doi : 10.1186/1743-422X-10-177 . PMC 3679726 . PMID 23734580.
- ^ Campbell, Allan (2003). "El futuro de la biología de los bacteriófagos". Nature Reviews Genetics . 4 (6): 471–477. doi :10.1038/nrg1089. PMC 7097159 . PMID 12776216.
- ^ Cristina, Howard-Varona (2017). "Lisogenia en la naturaleza: mecanismos, impacto y ecología de los fagos templados". The ISME Journal . 11 (7): 1511–1520. Bibcode :2017ISMEJ..11.1511H. doi :10.1038/ismej.2017.16. PMC 5520141 . PMID 28291233.
- ^ Zhang, Menghui (2022). "Las transiciones del ciclo de vida de los fagos templados: factores reguladores y posibles implicaciones ecológicas". Virus . 14 (9). MDPI: 1904. doi : 10.3390/v14091904 . PMC 9502458 . PMID 36146712.
- ^ Girdhar, Khyati; Powis, Amaya; Raisingani, Amol; Chrudinová, Martina; Huang, Ruixu; Tran, Tu; Sevgi, Kaan; Dogus Dogru, Yusuf; Altindis, Emrah (29 de septiembre de 2021). "Virus y metabolismo: los efectos de las infecciones virales y las insulinas virales en el metabolismo del huésped". Revista Anual de Virología . 8 (1): 373–391. doi : 10.1146/annurev-virology-091919-102416 . ISSN 2327-056X. PMC 9175272 . PMID 34586876.
- ^ Dimmock, Nigel; et al. (2007). Introducción a la virología moderna (6.ª ed.). Blackwell Publishing.
- ^ Hofer, Ursula (2013). "Engañando a la maquinaria de corrección de pruebas del coronavirus". Nature Reviews Microbiology . 11 (10): 662–663. doi :10.1038/nrmicro3125. PMC 7097040 . PMID 24018385.
- ^ Everett, Clinton Smith (2017). "La maleabilidad no tan infinita de los virus de ARN: determinantes virales y celulares de las tasas de mutación de los virus de ARN". PLOS Pathogens . 13 (4): e1006254. doi : 10.1371/journal.ppat.1006254 . PMC 5407569 . PMID 28448634.
- ^ Barr, JN (2016). "Inestabilidad genética de los virus ARN". Estabilidad del genoma . Elsevier: 21–35. doi :10.1016/B978-0-12-803309-8.00002-1. ISBN 978-0-12-803309-8. Número de pieza 7149711 .
- ^ "Tratamiento del VIH: aspectos básicos". hivinfo.nih.gov . NIH.
