Proteólisis paralela rápida
La proteólisis paralela rápida ( FASTpp ) es un método para determinar la termoestabilidad de las proteínas midiendo qué fracción de proteína resiste la digestión proteolítica rápida. [1]
Historia y antecedentes
La proteólisis se utiliza ampliamente en bioquímica y biología celular para investigar la estructura de las proteínas . [2] [3] En la "proteólisis limitada con tripsina", pequeñas cantidades de proteasa digieren tanto las proteínas plegadas como las desplegadas, pero a velocidades muy diferentes: las proteínas no estructuradas se cortan más rápidamente, mientras que las proteínas estructuradas se cortan a una velocidad más lenta (a veces en órdenes de magnitud). Recientemente, se han propuesto varios otros ensayos de estabilidad de proteínas basados en la proteólisis, que explotan otras proteasas con alta especificidad para escindir proteínas desplegadas. Estos incluyen la proteólisis de pulsos, [4] la calorimetría de barrido proteolítico [5] y FASTpp.
Cómo funciona
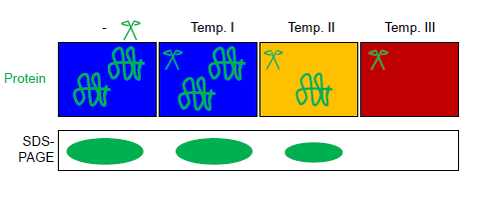
FASTpp mide la cantidad de proteína que resiste la digestión bajo diversas condiciones. Para ello, se utiliza una proteasa termoestable, que corta específicamente en residuos hidrofóbicos expuestos. El ensayo FASTpp combina el desdoblamiento térmico, la especificidad de una proteasa termoestable para la fracción desdoblada con el poder de separación de SDS-PAGE . [6] Debido a esta combinación, FASTpp puede detectar cambios en la fracción plegada en un amplio rango fisicoquímico de condiciones que incluyen temperaturas de hasta 85 °C, pH 6–9, presencia o ausencia de todo el proteoma . Las aplicaciones van desde la biotecnología hasta el estudio de mutaciones puntuales y ensayos de unión de ligandos .
Aplicaciones
FASTpp se ha utilizado para investigar: [1]
- Efecto del lisado sobre la estabilidad de las proteínas
- Estabilidad térmica del proteoma [7]
- Plegado y encuadernado acoplados [8]
- Efectos del ligando sobre la fracción plegada y la estabilidad [9]
- Efectos de las mutaciones sobre la fracción plegada y la estabilidad (por ejemplo, mutaciones puntuales / mutaciones sin sentido [9] [10] )
- Estabilidad cinética de las proteínas [11]
Tecnología
En primer lugar, se genera un lisado celular mediante el batido de perlas de vidrio, la homogeneización a presión o métodos de lisis química o física que no desnaturalizan la(s) proteína(s) de interés. (Opcionalmente, para un análisis dirigido), se purifica una proteína de interés a partir de este lisado mediante métodos de afinidad basados en etiquetas intrínsecamente desordenadas [12] u otras estrategias de purificación adecuadas, que a menudo implican varios pasos cromatográficos ortogonales.
Esta solución de proteína (total o purificada) se divide en alícuotas en varios tubos de una tira de PCR. Todas las alícuotas se exponen en paralelo en un ciclador de PCR de gradiente térmico a diferentes temperaturas máximas en presencia de la proteasa termoestable termolisina (ver figura). El control automático de la temperatura se logra en un ciclador de gradiente térmico (comúnmente utilizado para PCR ). Los productos de reacción se pueden separar mediante SDS-PAGE o transferencia Western . [6] La proteasa termolisina se puede inactivar completamente con EDTA . Esta característica de la termolisina hace que FASTpp sea compatible con la digestión posterior con tripsina , por ejemplo, para espectrometría de masas . [13] [14] [7]
Referencias
- ^ ab Minde, DP; Maurice, MM; Rüdiger, SGD (2012). Uversky, Vladimir N (ed.). "Determinación de la estabilidad biofísica de proteínas en lisados mediante un ensayo de proteólisis rápida, FASTpp". PLOS ONE . 7 (10): e46147. Bibcode :2012PLoSO...746147M. doi : 10.1371/journal.pone.0046147 . PMC 3463568 . PMID 23056252.
- ^ Johnson, DE; Xue, B.; Sickmeier, MD; Meng, J.; Cortese, MS; Oldfield, CJ; Le Gall, T.; Dunker, AK; Uversky, VN (2012). "Caracterización de alto rendimiento del desorden intrínseco en proteínas de la Protein Structure Initiative". Revista de biología estructural . 180 (1): 201–215. doi :10.1016/j.jsb.2012.05.013. PMC 3578346 . PMID 22651963.
- ^ Hoelen, H.; Kleizen, B.; Schmidt, A.; Richardson, J.; Charitou, P.; Thomas, PJ; Braakman, I. (2010). Uversky, Vladimir N (ed.). "El defecto de plegamiento primario y el rescate de ΔF508 CFTR emergen durante la traducción del dominio mutante". PLOS ONE . 5 (11): e15458. Bibcode :2010PLoSO...515458H. doi : 10.1371/journal.pone.0015458 . PMC 2994901 . PMID 21152102.
- ^ Park, C.; Marqusee, S. (2005). "Proteólisis pulsátil: un método simple para la determinación cuantitativa de la estabilidad de las proteínas y la unión de ligandos". Nature Methods . 2 (3): 207–212. doi :10.1038/nmeth740. PMID 15782190. S2CID 21364478.
- ^ Tur-Arlandis, G.; Rodríguez-Larrea, D.; Ibarra-Molero, B.; Sánchez-Ruiz, JM (2010). "Calorimetría de barrido proteolítico: una nueva metodología que investiga las características fundamentales de la estabilidad cinética de las proteínas". Revista Biofísica . 98 (6): L12-L14. Código Bib : 2010BpJ....98L..12T. doi :10.1016/j.bpj.2009.11.028. PMC 2849053 . PMID 20303845.
- ^ ab Laemmli, Reino Unido (1970). "Escisión de proteínas estructurales durante el ensamblaje de la cabeza del bacteriófago T4". Nature . 227 (5259): 680–685. Bibcode :1970Natur.227..680L. doi :10.1038/227680a0. PMID 5432063. S2CID 3105149.
- ^ ab Leuenberger, P; Ganscha, S; Kahraman, A (2017). "El análisis de la termoestabilidad de proteínas en toda la célula revela los determinantes de la termoestabilidad". Science . 355 (6327): eaai7825. doi :10.1126/science.aai7825. PMID 28232526. S2CID 8432125.
- ^ Demarest, SJ; Martinez-Yamout, M.; Chung, J.; Chen, H.; Xu, W.; Dyson, HJ ; Evans, RM; Wright, PE (2002). "Plegamiento sinérgico mutuo en el reclutamiento de CBP/p300 por coactivadores del receptor nuclear p160". Nature . 415 (6871): 549–553. doi :10.1038/415549a. PMID 11823864. S2CID 4423920.
- ^ ab Robaszkiewicz, K.; Ostrowska, Z.; Cyranka-Czaja, A.; Moraczewska, J. (2015). "Las interacciones tropomiosina-troponina deterioradas reducen la activación del filamento delgado de actina". Biochimica et Biophysica Acta (BBA) - Proteínas y proteómica . 1854 (5): 381–390. doi :10.1016/j.bbapap.2015.01.004. PMID 25603119.
- ^ Minde, DP; Anvarian, Z.; Rüdiger, SG; Maurice, MM (2011). "Desorden desordenado: ¿Cómo las mutaciones sin sentido en la proteína supresora de tumores APC conducen al cáncer?". Molecular Cancer . 10 : 101. doi : 10.1186/1476-4598-10-101 . PMC 3170638 . PMID 21859464.
- ^ Tur-Arlandis, G.; Rodríguez-Larrea, D.; Ibarra-Molero, B.; Sánchez-Ruiz, JM (2010). "Calorimetría de barrido proteolítico: una nueva metodología que investiga las características fundamentales de la estabilidad cinética de las proteínas". Revista Biofísica . 98 (6): L12-L14. Código Bib : 2010BpJ....98L..12T. doi :10.1016/j.bpj.2009.11.028. PMC 2849053 . PMID 20303845.
- ^ Minde, DP; Halff, EF; Tans, SJ (2013). "Diseñando el desorden: cuentos de colas inesperadas". Proteínas intrínsecamente desordenadas . 1 (1): e26790. doi :10.4161/idp.26790. PMC 5424805 . PMID 28516025.
- ^ Chang, Y; Schlebach, JP; Verheul, RA; Park, C (2012). "Enfoque proteómico simplificado para descubrir interacciones proteína-ligando". Protein Science . 21 (9): 1280–7. doi :10.1002/pro.2112. PMC 3631357 . PMID 22733688.
- ^ Park, C; Marqusee, S (2005). "Proteólisis pulsátil: un método simple para la determinación cuantitativa de la estabilidad de las proteínas y la unión de ligandos". Nature Methods . 2 (3): 207–12. doi :10.1038/nmeth740. PMID 15782190. S2CID 21364478.