Aconitasa
| aconitato hidratasa | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Ilustración de la aconitasa de cerdo en un complejo con el grupo [Fe 4 S 4 ]. La proteína está coloreada por la estructura secundaria y los átomos de hierro son azules y los de azufre, rojos. [1] | |||||||||
| Identificadores | |||||||||
| N.º CE | 4.2.1.3 | ||||||||
| N.º CAS | 9024-25-3 | ||||||||
| Bases de datos | |||||||||
| IntEnz | Vista de IntEnz | ||||||||
| BRENDA | Entrada de BRENDA | ||||||||
| Expasí | Vista de NiceZyme | ||||||||
| BARRIL | Entrada de KEGG | ||||||||
| MetaCiclo | vía metabólica | ||||||||
| PRIAMO | perfil | ||||||||
| Estructuras del PDB | RCSB AP APBE APSUMA | ||||||||
| Ontología genética | AmiGO / QuickGO | ||||||||
| |||||||||
| Familia de la aconitasa (aconitato hidratasa) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estructura de la aconitasa. [2] | |||||||||
| Identificadores | |||||||||
| Símbolo | Aconitasa | ||||||||
| Pfam | PF00330 | ||||||||
| Interprofesional | IPR001030 | ||||||||
| PROSITIO | PDOC00423 | ||||||||
| SCOP2 | 1aco / ALCANCE / SUPFAM | ||||||||
| |||||||||
La aconitasa (aconitato hidratasa; EC 4.2.1.3) es una enzima que cataliza la isomerización estereoespecífica de citrato a isocitrato a través de cis - aconitato en el ciclo del ácido tricarboxílico , un proceso no redox -activo. [3] [4] [5]
Estructura
La aconitasa, que se muestra en las estructuras en el margen derecho de esta página, tiene dos estructuras ligeramente diferentes, dependiendo de si está activada o inactivada. [6] [7] En la forma inactiva, su estructura se divide en cuatro dominios. [6] Contando desde el extremo N , solo los primeros tres de estos dominios están involucrados en interacciones cercanas con el grupo [3Fe-4S], pero el sitio activo consta de residuos de los cuatro dominios, incluido el dominio C-terminal más grande . [6] El grupo Fe-S y un SO2−
4El anión también reside en el sitio activo. [6] Cuando la enzima se activa, gana un átomo de hierro adicional, creando un grupo [4Fe-4S]. [7] [8] Sin embargo, la estructura del resto de la enzima casi no cambia; los átomos conservados entre las dos formas están esencialmente en las mismas posiciones, hasta una diferencia de 0,1 angstroms. [7]
Función
A diferencia de la mayoría de las proteínas de hierro-azufre que funcionan como transportadores de electrones, el grupo de hierro-azufre de la aconitasa reacciona directamente con un sustrato enzimático. La aconitasa tiene un grupo activo [Fe 4 S 4 ] 2+ , que puede convertirse en una forma inactiva [Fe 3 S 4 ] + . Se ha demostrado que tres residuos de cisteína (Cys) son ligandos del centro [Fe 4 S 4 ]. En el estado activo, el ion de hierro lábil del grupo [Fe 4 S 4 ] no está coordinado por Cys sino por moléculas de agua.
La proteína de unión al elemento sensible al hierro (IRE-BP) y la 3-isopropilmalato deshidratasa (α-isopropilmalato isomerasa; EC 4.2.1.33), una enzima que cataliza el segundo paso en la biosíntesis de leucina , son homólogos conocidos de la aconitasa. Los elementos reguladores del hierro (IRE) constituyen una familia de estructuras de tallo-bucle no codificantes de 28 nucleótidos que regulan el almacenamiento de hierro, la síntesis del hemo y la captación de hierro. También participan en la unión a los ribosomas y controlan el recambio (degradación) del ARNm . La proteína reguladora específica, la IRE-BP, se une a los IRE en las regiones 5' y 3', pero solo al ARN en forma apo, sin el grupo Fe-S. La expresión de IRE-BP en cultivos celulares ha revelado que la proteína funciona como una aconitasa activa, cuando las células están repletas de hierro, o como una proteína de unión al ARN activa, cuando las células están desprovistas de hierro. Las IRE-BP mutantes, en las que cualquiera o todos los tres residuos de Cys involucrados en la formación de Fe-S son reemplazados por serina , no tienen actividad de aconitasa, pero conservan las propiedades de unión al ARN.
La aconitasa es inhibida por el fluoroacetato , por lo tanto, el fluoroacetato es venenoso. El fluoroacetato, en el ciclo del ácido cítrico, se convierte en fluorocitrato por acción de la citrato sintasa. El fluorocitrato inhibe competitivamente a la aconitasa, deteniendo el ciclo del ácido cítrico. [9] El grupo de azufre de hierro es muy sensible a la oxidación por superóxido . [10]
Mecanismo
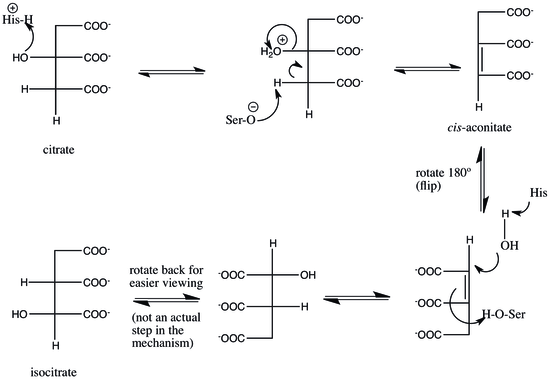

La aconitasa emplea un mecanismo de deshidratación-hidratación. [11] Los residuos catalíticos involucrados son His-101 y Ser-642. [11] His-101 protona el grupo hidroxilo en C3 del citrato, lo que le permite salir como agua, y Ser-642 abstrae simultáneamente el protón en C2, creando un doble enlace entre C2 y C3, y formando el llamado intermedio cis -aconitato (los dos grupos carboxilo en el doble enlace son cis ). [11] [14] El átomo de carbono del que se elimina el hidrógeno es el que proviene del oxaloacetato en el paso anterior del ciclo del ácido cítrico, no el que proviene del acetil CoA , aunque estos dos carbonos son equivalentes excepto que uno es " pro -R" y el otro " pro -S" (ver Proquiralidad ). [15] : 393 En este punto, el intermedio se gira 180°. [11] Esta rotación se denomina "giro". [12] Debido a este giro, se dice que el intermedio pasa de un "modo citrato" a un "modo isocitrato". [16]
La forma exacta en que se produce este cambio es discutible. Una teoría es que, en el paso limitante de la velocidad del mecanismo, el cis -aconitato se libera de la enzima y luego se vuelve a unir en el modo isocitrato para completar la reacción. [16] Este paso limitante de la velocidad garantiza que se forme la estereoquímica correcta , específicamente (2R,3S), en el producto final. [16] [17] Otra hipótesis es que el cis -aconitato permanece unido a la enzima mientras cambia del modo citrato al modo isocitrato. [11]
En cualquier caso, la inversión del cis -aconitato permite que los pasos de deshidratación e hidratación ocurran en caras opuestas del intermedio. [11] La aconitasa cataliza la eliminación/adición trans de agua, y la inversión garantiza que se forme la estereoquímica correcta en el producto. [11] [12] Para completar la reacción, los residuos de serina e histidina invierten sus acciones catalíticas originales: la histidina, ahora básica, abstrae un protón del agua, preparándola como nucleófilo para atacar en C2, y la serina protonada es desprotonada por el doble enlace cis -aconitato para completar la hidratación, produciendo isocitrato. [11]

Miembros de la familia
Las aconitasas se expresan en bacterias y en humanos. Los humanos expresan las siguientes dos isoenzimas de aconitasa :
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Mapa interactivo de rutas
Haga clic en los genes, proteínas y metabolitos que aparecen a continuación para acceder a los artículos correspondientes. [§ 1]
- ^ El mapa interactivo de la ruta se puede editar en WikiPathways: "TCACycle_WP78".
Referencias
- ^ PDB : 7ACN ; Lauble H, Kennedy MC, Beinert H, Stout CD (1992). "Estructuras cristalinas de la aconitasa con enlaces isocitrato y nitroisocitrato". Bioquímica . 31 (10): 2735–48. doi :10.1021/bi00125a014. PMID 1547214.
- ^ PDB : 1ACO ; Lauble H, Kennedy MC, Beinert H, Stout CD (1994). "Estructuras cristalinas de la aconitasa con enlaces trans-aconitato y nitrocitrato". Revista de biología molecular . 237 (4): 437–51. doi :10.1006/jmbi.1994.1246. PMID 8151704.
- ^ Beinert H, Kennedy MC (diciembre de 1993). "Aconitasa, una proteína de dos caras: enzima y factor regulador del hierro". FASEB Journal . 7 (15): 1442–9. doi : 10.1096/fasebj.7.15.8262329 . PMID 8262329. S2CID 1107246.
- ^ Flint DH, Allen RM (1996). "Proteínas de hierro y azufre con funciones no redox". Chemical Reviews . 96 (7): 2315–34. doi :10.1021/cr950041r. PMID 11848829.
- ^ Beinert H, Kennedy MC, Stout CD (noviembre de 1996). "La aconitasa como proteína, enzima y proteína reguladora del hierro con signo negativo de hierro". Chemical Reviews . 96 (7): 2335–2374. doi :10.1021/cr950040z. PMID 11848830.
- ^ abcd Robbins AH, Stout CD (1989). "La estructura de la aconitasa". Proteínas . 5 (4): 289–312. doi :10.1002/prot.340050406. PMID 2798408. S2CID 36219029.
- ^ abc Robbins AH, Stout CD (mayo de 1989). "Estructura de la aconitasa activada: formación del grupo [4Fe-4S] en el cristal". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 86 (10): 3639–43. Bibcode :1989PNAS...86.3639R. doi : 10.1073/pnas.86.10.3639 . PMC 287193 . PMID 2726740.
- ^ Lauble H, Kennedy MC, Beinert H, Stout CD (marzo de 1992). "Estructuras cristalinas de la aconitasa con isocitrato y nitroisocitrato unidos". Bioquímica . 31 (10): 2735–48. doi :10.1021/bi00125a014. PMID 1547214.
- ^ Morrison JF, Peters RA (noviembre de 1954). "Bioquímica del envenenamiento por fluoroacetato: el efecto del fluorocitrato sobre la aconitasa purificada". Biochem. J . 58 (3): 473–9. doi :10.1042/bj0580473. PMC 1269923 . PMID 13208639.
- ^ Gardner PR (2002). "Aconitasa: objetivo sensible y medida del superóxido". Superóxido dismutasa . Métodos en enzimología. Vol. 349. págs. 9-23. doi :10.1016/S0076-6879(02)49317-2. ISBN. 978-0-12-182252-1. Número de identificación personal 11912933.
- ^ abcdefghi Takusagawa F. "Capítulo 16: Ciclo del ácido cítrico" (PDF) . Nota de Takusagawa . Universidad de Kansas. Archivado desde el original (PDF) el 24 de marzo de 2012 . Consultado el 10 de julio de 2011 .
- ^ abc Beinert H, Kennedy MC, Stout CD (noviembre de 1996). "Aconitasa como proteína, enzima y proteína reguladora del hierro con signo menos hierro" (PDF) . Chemical Reviews . 96 (7): 2335–2374. doi :10.1021/cr950040z. PMID 11848830. Archivado desde el original (PDF) el 2011-08-11 . Consultado el 2011-05-16 .
- ^ ab PDB : 1C96 ; Lloyd SJ, Lauble H, Prasad GS, Stout CD (diciembre de 1999). "El mecanismo de la aconitasa: 1.8 Una resolución de la estructura cristalina del complejo S642a:citrato". Protein Sci . 8 (12): 2655–62. doi :10.1110/ps.8.12.2655. PMC 2144235 . PMID 10631981.
- ^ Han D, Canali R, Garcia J, Aguilera R, Gallaher TK, Cadenas E (septiembre de 2005). "Sitios y mecanismos de inactivación de la aconitasa por peroxinitrito: modulación por citrato y glutatión". Bioquímica . 44 (36): 11986–96. doi :10.1021/bi0509393. PMID 16142896.
- ^ Lubert Stryer (1981). Bioquímica (2.ª ed.). Págs. 295-296.
- ^ abc Lauble H, Stout CD (mayo de 1995). "Características estéricas y conformacionales del mecanismo de la aconitasa". Proteins . 22 (1): 1–11. doi :10.1002/prot.340220102. PMID 7675781. S2CID 43006515.
- ^ "Familia de la aconitasa". Base de datos de grupos prostéticos e iones metálicos en sitios activos de proteínas, versión 2.0 . Universidad de Leeds. 2 de febrero de 1999. Archivado desde el original el 8 de junio de 2011. Consultado el 10 de julio de 2011 .
Lectura adicional
Enlaces externos
- Aconitasa en los encabezados de materias médicas (MeSH) de la Biblioteca Nacional de Medicina de EE. UU.
- Proteopedia Aconitasa : la estructura de la aconitasa en 3D interactivo



