Reportero fluorescente sensible al glutamato
Una proteína fluorescente diseñada genéticamente que cambia su fluorescencia cuando se une al neurotransmisor glutamato . [1] Los reporteros fluorescentes sensibles al glutamato (iGluSnFR, pronunciado coloquialmente ' olfateador de pegamento ') se utilizan para monitorear la actividad de las terminales presinápticas mediante microscopía de fluorescencia . Los GluSnFR son una clase de sensores optogenéticos utilizados en la investigación en neurociencia . [2] En el tejido cerebral, la microscopía de dos fotones se utiliza típicamente para monitorear la fluorescencia de GluSnFR.
Diseño
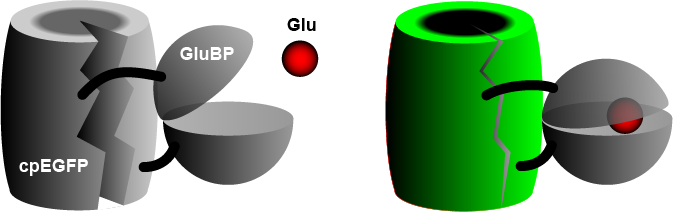
El iGluSnFR, ampliamente utilizado, consiste en una proteína fluorescente verde mejorada permutada circularmente (cpEGFP) fusionada a una proteína de unión al glutamato (GluBP) de una bacteria . [3] Cuando GluBP se une a una molécula de glutamato , cambia su forma, juntando el barril de EGFP , lo que aumenta la fluorescencia. Se incluye un segmento peptídico específico ( PDGFR ) para llevar el sensor al exterior de la membrana celular . [4] En la versión más reciente de Aggarwal et al. (2022), [1] los investigadores introdujeron iGluSnFR en dos dominios de anclaje adicionales, un ancla de glicosilfostidilinositol (GPI) y una forma modificada del dominio citosólico terminal de Stargazin con un ligando PDZ.
Historia
Los primeros sensores de glutamato fluorescentes codificados genéticamente (FLIPE, GluSnFR y SuperGluSnFR) se construyeron uniendo la proteína fluorescente cian (CFP) y la proteína fluorescente amarilla (YFP) a una proteína de unión al glutamato bacteriano (GluBP). [5] [6] La unión del glutamato cambió la distancia entre CFP e YFP, cambiando la eficiencia de la transferencia de energía ( FRET ) entre los dos fluoróforos . [7] [8] Se logró un gran avance en la visualización de la liberación de glutamato con iGluSnFR, un sensor de glutamato de un solo fluoróforo basado en EGFP que produce un aumento de ~5 veces en la fluorescencia. [3] Para medir la transmisión sináptica a altas frecuencias, recientemente se han desarrollado nuevas variantes de iGluSnFR con cinética acelerada. [9] [10]
Referencias
- ^ ab Aggarwal, Abhi; Liu, Rui; Chen, Yang; Ralowicz, Amelia J.; Bergerson, Samuel J.; Tomaska, Filip; Hanson, Timothy L.; Hasseman, Jeremy P.; Reep, Daniel; Tsegaye, Getahun; Yao, Pantong; Ji, Xiang; Kloos, Marino; Walpita, Deepika; Patel, Ronak (15 de febrero de 2022). "Indicadores de glutamato con cinética de activación y localización mejoradas para la transmisión sináptica de imágenes": 2022.02.13.480251. doi :10.1101/2022.02.13.480251. hdl : 20.500.11850/613938 .
{{cite journal}}: Requiere citar revista|journal=( ayuda ) - ^ Hefendehl, JK; LeDue, J.; Ko, RWY; Mahler, J.; Murphy, TH; MacVicar, BA (11 de noviembre de 2016). "Mapeo de la disfunción del transportador de glutamato sináptico in vivo en regiones que rodean las placas Aβ mediante imágenes de dos fotones iGluSnFR". Nature Communications . 7 : 13441. Bibcode :2016NatCo...713441H. doi :10.1038/ncomms13441. PMC 5114608 . PMID 27834383.
- ^ ab Marvin, Jonathan S; Borghuis, Bart G; Tian, Lin; Cichon, Joseph; Harnett, Mark T; Akerboom, Jasper; Gordus, Andrew; Renninger, Sabine L; Chen, Tsai-Wen (2013). "Una sonda fluorescente optimizada para visualizar la neurotransmisión del glutamato". Nature Methods . 10 (2): 162–170. doi :10.1038/nmeth.2333. ISSN 1548-7105. PMC 4469972 . PMID 23314171.
- ^ Marvin, Jonathan S.; Schreiter, Eric R.; Echevarría, Ileabett M.; Looger, Loren L. (1 de noviembre de 2011). "Un sensor de maltosa con alta relación señal-ruido y codificado genéticamente". Proteínas: estructura, función y bioinformática . 79 (11): 3025–3036. doi :10.1002/prot.23118. ISSN 1097-0134. PMC 3265398 . PMID 21989929.
- ^ Hu, Yonglin; Fan, Cheng-Peng; Fu, Guangsen; Zhu, Deyu; Jin, Qi; Wang, Da-Cheng (2008). "Estructura cristalina de una proteína de unión a glutamato/aspartato complejada con una molécula de glutamato: base estructural de la especificidad del ligando a resolución atómica". Journal of Molecular Biology . 382 (1): 99–111. doi :10.1016/j.jmb.2008.06.091. PMID 18640128.
- ^ De Lorimier, Robert M.; Smith, J. Jeff; Dwyer, Mary A.; Looger, Loren L.; Sali, Kevin M.; Paavola, Chad D.; Rizk, Shahir S.; Sadigov, Shamil; Conrad, David W. (1 de noviembre de 2002). "Construcción de una familia de biosensores fluorescentes". Protein Science . 11 (11): 2655–2675. doi :10.1110/ps.021860. ISSN 1469-896X. PMC 2373719 . PMID 12381848.
- ^ Okumoto, Sakiko; Looger, Loren L.; Micheva, Kristina D.; Reimer, Richard J.; Smith, Stephen J.; Frommer, Wolf B. (14 de junio de 2005). "Detección de la liberación de glutamato de las neuronas mediante nanosensores FRET de superficie codificados genéticamente". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 102 (24): 8740–8745. Bibcode :2005PNAS..102.8740O. doi : 10.1073/pnas.0503274102 . ISSN 0027-8424. PMC 1143584 . PMID 15939876.
- ^ Hires, Samuel Andrew; Zhu, Yongling; Tsien, Roger Y. (18 de marzo de 2008). "Medición óptica del derrame y la recaptación sináptica de glutamato mediante reporteros fluorescentes sensibles al glutamato optimizados por el enlazador". Actas de la Academia Nacional de Ciencias . 105 (11): 4411–4416. Bibcode :2008PNAS..105.4411H. doi : 10.1073/pnas.0712008105 . ISSN 0027-8424. PMC 2393813 . PMID 18332427.
- ^ Helassa, Nordine; Dürst, Céline D.; Coates, Catherine; Kerruth, Silke; Arif, Urwa; Schulze, Christian; Wiegert, J. Simon; Geeves, Michael; Oertner, Thomas G.; Török, Katalin (22 de mayo de 2018). "Sensores de glutamato ultrarrápidos resuelven la liberación de alta frecuencia en las sinapsis colaterales de Schaffer". Actas de la Academia Nacional de Ciencias . 115 (21): 5594–5599. doi : 10.1073/pnas.1720648115 . PMC 6003469 . PMID 29735711.
- ^ Marvin, Jonathan S.; Scholl, Benjamin; Wilson, Daniel E.; Podgorski, Kaspar; Kazemipour, Abbas; Müller, Johannes Alexander; Schoch, Susanne; Quiroz, Francisco José Urra; Rebola, Nelson; Bao, Huan; Little, Justin P. (noviembre de 2018). "Estabilidad, afinidad y variantes cromáticas del sensor de glutamato iGluSnFR". Nature Methods . 15 (11): 936–939. doi :10.1038/s41592-018-0171-3. ISSN 1548-7105. PMC 6394230 . PMID 30377363.