Genética del envejecimiento
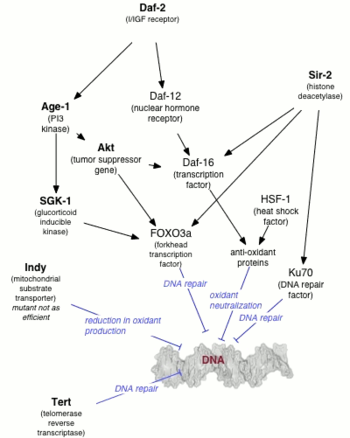
La genética del envejecimiento se ocupa generalmente de la prolongación de la vida asociada a alteraciones genéticas, más que de enfermedades que aceleran el envejecimiento y conducen a una reducción de la esperanza de vida.
La primera mutación que se descubrió que aumentaba la longevidad en un animal fue el gen age-1 en Caenorhabditis elegans . Michael Klass descubrió que la esperanza de vida de C. elegans podía verse alterada por mutaciones, pero Klass creía que el efecto se debía a la reducción del consumo de alimentos ( restricción calórica ). [1] Thomas Johnson demostró más tarde que la prolongación de la vida de hasta un 65% se debía a la mutación en sí misma y no a la restricción calórica, [2] y nombró al gen age-1 con la expectativa de que se encontrarían otros genes que controlan el envejecimiento. El gen age-1 codifica la subunidad catalítica de la fosfatidilinositol 3-quinasa de clase I (PI3K).
Una década después del descubrimiento de Johnson, Cynthia Kenyon demostró que daf-2 , uno de los dos genes esenciales para la formación de larvas de dauer [3] , duplicaba la expectativa de vida de C. elegans . [4] Kenyon demostró que los mutantes daf-2 , que formarían dauer por encima de los 25 °C (77 °F), evitarían el estado dauer por debajo de los 20 °C (68 °F) con una duplicación de la expectativa de vida. [4] Antes del estudio de Kenyon, se creía comúnmente que la expectativa de vida solo podía aumentarse a costa de una pérdida de capacidad reproductiva, pero los nematodos de Kenyon mantuvieron la capacidad reproductiva juvenil, así como la juventud en general. Se demostró que la modificación genética posterior (mutación PI3K-null) a C. elegans extendió la expectativa de vida máxima diez veces. [5] [6]
Se demostró que los mutantes de larga vida de C. elegans ( age-1 y daf-2 ) son resistentes al estrés oxidativo y a la luz ultravioleta . [7] Estos mutantes de larga vida tenían una mayor capacidad de reparación del ADN que el C. elegans de tipo salvaje . [7] La eliminación del gen de reparación por escisión de nucleótidos Xpa-1 aumentó la sensibilidad a los rayos ultravioleta y redujo la esperanza de vida de los mutantes de larga vida. Estos hallazgos respaldan la hipótesis de que el daño del ADN tiene un papel significativo en el proceso de envejecimiento . [7]
Las modificaciones genéticas en otras especies no han logrado una extensión de la expectativa de vida tan grande como la observada en C. elegans . La expectativa de vida de Drosophila melanogaster se ha duplicado. [8] Las mutaciones genéticas en ratones pueden aumentar la expectativa de vida máxima a 1,5 veces lo normal, y hasta 1,7 veces lo normal cuando se combinan con restricción calórica . [9]
En la levadura, la histona desacetilasa dependiente de NAD + Sir2 es necesaria para el silenciamiento genómico en tres loci: los loci de apareamiento de la levadura , los telómeros y el ADN ribosómico (ADNr). En algunas especies de levadura, el envejecimiento replicativo puede ser causado parcialmente por la recombinación homóloga entre repeticiones de ADNr; la escisión de repeticiones de ADNr da como resultado la formación de círculos de ADNr extracromosómicos (ERC). Estos ERC se replican y segregan preferentemente a la célula madre durante la división celular, y se cree que dan lugar a la senescencia celular al titular (competir por) factores nucleares esenciales . No se han observado ERC en otras especies (ni siquiera en todas las cepas de la misma especie de levadura) de levadura (que también muestran senescencia replicativa), y no se cree que los ERC contribuyan al envejecimiento en organismos superiores como los humanos (no se ha demostrado que se acumulen en mamíferos de manera similar a la levadura). Se ha encontrado ADN circular extracromosómico (eccDNA) en gusanos, moscas y seres humanos. Se desconoce el origen y el papel del eccDNA en el envejecimiento, si es que tiene alguno.
A pesar de la falta de una conexión entre el ADN circular y el envejecimiento en organismos superiores, copias adicionales de Sir2 son capaces de extender la vida útil tanto de gusanos como de moscas (aunque, en moscas, este hallazgo no ha sido replicado por otros investigadores, y el activador de Sir2 resveratrol no aumenta de manera reproducible la vida útil en ninguna de las especies. [10] ) No está claro si los homólogos de Sir2 en organismos superiores tienen algún papel en la vida útil, pero se ha demostrado que la proteína SIRT1 humana desacetila p53 , Ku70 y la familia forkhead de factores de transcripción . SIRT1 también puede regular acetilatos como CBP/p300 , y se ha demostrado que desacetila residuos de histonas específicos.
RAS1 y RAS2 también afectan el envejecimiento en la levadura y tienen un homólogo humano. Se ha demostrado que la sobreexpresión de RAS2 prolonga la vida útil en la levadura.
Otros genes regulan el envejecimiento de la levadura aumentando la resistencia al estrés oxidativo . La superóxido dismutasa , una proteína que protege contra los efectos de los radicales libres mitocondriales , puede prolongar la vida útil de la levadura en fase estacionaria cuando se sobreexpresa.
En organismos superiores, es probable que el envejecimiento esté regulado en parte a través de la vía insulina/IGF-1. Las mutaciones que afectan la señalización similar a la insulina en gusanos, moscas y el eje hormona de crecimiento/IGF1 en ratones están asociadas con una mayor esperanza de vida. En levadura, la actividad de Sir2 está regulada por la nicotinamidasa PNC1. La PNC1 se regula positivamente a nivel transcripcional en condiciones estresantes como la restricción calórica , el choque térmico y el choque osmótico . Al convertir la nicotinamida en niacina , se elimina la nicotinamida, inhibiendo la actividad de Sir2. Una nicotinamidasa que se encuentra en humanos, conocida como PBEF, puede cumplir una función similar, y una forma secretada de PBEF conocida como visfatina puede ayudar a regular los niveles séricos de insulina . Sin embargo, no se sabe si estos mecanismos también existen en humanos, ya que existen diferencias obvias en la biología entre humanos y organismos modelo.
Se ha demostrado que la actividad de Sir2 aumenta con la restricción calórica. Debido a la falta de glucosa disponible en las células, hay más NAD+ disponible y puede activar Sir2. Se informó que el resveratrol , un estilbenoide que se encuentra en la piel de las uvas rojas , prolonga la vida de las levaduras, los gusanos y las moscas (la prolongación de la vida en moscas y gusanos ha demostrado ser irreproducible por investigadores independientes [10] ). Se ha demostrado que activa Sir2 y, por lo tanto, imita los efectos de la restricción calórica, si se acepta que la restricción calórica depende de hecho de Sir2.
Según la base de datos GenAge de genes relacionados con el envejecimiento, hay más de 1.800 genes que alteran la esperanza de vida en organismos modelo : 838 en la lombriz intestinal ( Caenorhabditis elegans ), 883 en la levadura de panadería ( Saccharomyces cerevisiae ), 170 en la mosca de la fruta ( Drosophila melanogaster ) y 126 en el ratón ( Mus musculus ). [11]
La siguiente es una lista de genes relacionados con la longevidad a través de investigaciones [11] en organismos modelo :
| Podospora | Saccharomyces | Caenorhabditis | Drosophila | Música |
|---|---|---|---|---|
| grisea | LAG1 | daf-2 | césped1 | Proposición 1 |
| LAC1 | edad-1 /daf-23 | gato 1 | p66shc (no verificado de forma independiente) | |
| hoyo-1 | Ghr | |||
| RAS1 | daf-18 | mes | mckl1 | |
| RAS2 | akt-1/akt-2 | |||
| PHB1 | daf-16 | |||
| PHB2 | daf-12 | |||
| CDC7 | control-1 | |||
| BUD1 | viejo-1 | |||
| RTG2 | spe-26 | |||
| RPD3 | reloj-1 | |||
| HDA1 | mev-1 | |||
| Sir2 | ||||
| aak-2 | ||||
| Sir4-42 | ||||
| UTH4 | ||||
| YGL023 | ||||
| SGS1 | ||||
| RAD52 | ||||
| FOB1 |

En julio de 2020, los científicos, utilizando datos biológicos públicos sobre 1,75 millones de personas con una esperanza de vida conocida en general, identificaron 10 loci genómicos que parecen influir intrínsecamente en la esperanza de vida , la esperanza de vida y la longevidad (de los cuales la mitad no se había informado anteriormente con importancia a nivel de todo el genoma y la mayoría se asocian con enfermedades cardiovasculares ) e identificaron el metabolismo del hemo como un candidato prometedor para futuras investigaciones en este campo. Su estudio sugiere que los altos niveles de hierro en la sangre probablemente reducen, y los genes involucrados en el metabolismo del hierro probablemente aumentan, los años de vida saludable en los seres humanos. [13] [12]
Ned Sharpless y sus colaboradores demostraron el primer vínculo in vivo entre la expresión de p16 y la esperanza de vida. [14] Encontraron una expresión reducida de p16 en algunos tejidos de ratones con mutaciones que prolongan la esperanza de vida, así como en ratones que habían prolongado su esperanza de vida mediante la restricción alimentaria. Jan van Deursen y Darren Baker, en colaboración con Andre Terzic de la Clínica Mayo en Rochester, Minnesota, proporcionaron la primera evidencia in vivo de un vínculo causal entre la senescencia celular y el envejecimiento al prevenir la acumulación de células senescentes en ratones progeroides BubR1 . [15] En ausencia de células senescentes, los tejidos de los ratones mostraron una importante mejora en la carga habitual de trastornos relacionados con la edad. No desarrollaron cataratas , evitaron el desgaste muscular habitual con la edad. Conservaron las capas de grasa en la piel que normalmente se adelgazan con la edad y, en las personas, causan arrugas. Un segundo estudio dirigido por Jan van Deursen en colaboración con un equipo de colaboradores de la Clínica Mayo y la Universidad de Groningen, proporcionó la primera evidencia directa in vivo de que la senescencia celular causa signos de envejecimiento al eliminar células senescentes de ratones progeroides mediante la introducción de un gen suicida inducible por fármacos y luego tratar a los ratones con el fármaco para matar las células senescentes de forma selectiva, en lugar de disminuir la p16 de todo el cuerpo. [16] Otro estudio de Mayo dirigido por James Kirkland en colaboración con Scripps y otros grupos demostró que los senolíticos, fármacos que se dirigen a las células senescentes, mejoran la función cardíaca y mejoran la reactividad vascular en ratones viejos, alivian la alteración de la marcha causada por la radiación en ratones y retrasan la fragilidad, la disfunción neurológica y la osteoporosis en ratones progeroides. El descubrimiento de los fármacos senolíticos se basó en un enfoque impulsado por hipótesis: los investigadores aprovecharon la observación de que las células senescentes son resistentes a la apoptosis para descubrir que las vías pro-supervivencia están reguladas al alza en estas células. Demostraron que estas vías de supervivencia son el "talón de Aquiles" de las células senescentes utilizando métodos de interferencia de ARN , incluidas las vías relacionadas con Bcl-2, AKT, p21 y tirosina quinasa. Luego utilizaron medicamentos que se sabe que actúan sobre las vías identificadas y demostraron que estos medicamentos matan las células senescentes por apoptosis en cultivo y disminuyen la carga de células senescentes en múltiples tejidos in vivo. Es importante destacar que estos medicamentos tuvieron efectos a largo plazo después de una dosis única, consistente con la eliminación de células senescentes, en lugar de un efecto temporal que requiere la presencia continua de los medicamentos. Este fue el primer estudio que mostró que la eliminación de células senescentes mejora la función en ratones cronológicamente envejecidos. [17]
Capacidad de reparación del ADN y envejecimiento determinados genéticamente
La capacidad genéticamente determinada para reparar los daños en el ADN parece ser un factor clave del envejecimiento en comparaciones entre varias especies de aves y animales. Cuando se comparó la tasa de acumulación de daños en el ADN (roturas de doble cadena) en los leucocitos de delfines, cabras, renos, flamencos americanos y buitres leonados con la longevidad de individuos de estas diferentes especies, se encontró que las especies con vidas más largas tienen una acumulación más lenta de daños en el ADN. [18] La actividad de la enzima PARP1, empleada en varios procesos de reparación del ADN, se comparó en trece especies diferentes de mamíferos y se encontró que su actividad se correlacionaba con la esperanza de vida máxima de la especie. [19] En los humanos, la capacidad de reparación del ADN determinada genéticamente parece influir en la esperanza de vida. Se descubrió que las líneas de células linfoblastoides establecidas a partir de muestras de sangre de humanos que vivieron más de 100 años (centenarios) tenían una actividad significativamente mayor de la proteína de reparación del ADN poli (ADP-ribosa) polimerasa ( PARP ) que las líneas celulares de individuos más jóvenes (de 20 a 70 años). [20]
Rebaños para aumentar la longevidad
Efecto de la edad paterna
Un artículo de 2008 encontró una asociación en forma de U entre la edad paterna y la tasa de mortalidad general en los niños (es decir, la tasa de mortalidad hasta los 18 años). [21] Aunque las tasas de mortalidad relativas fueron más altas, las cifras absolutas fueron bajas, debido a la incidencia relativamente baja de anomalías genéticas. El estudio ha sido criticado por no ajustar la salud materna, que podría tener un gran efecto en la mortalidad infantil. [22] Los investigadores también encontraron una correlación entre la edad paterna y la muerte de los hijos por lesiones o envenenamiento, lo que indica la necesidad de controlar los factores de confusión sociales y conductuales. [23]
En 2012, un estudio demostró que una mayor edad en el momento de la paternidad tiende a aumentar la longitud de los telómeros en los hijos durante hasta dos generaciones. Dado que la longitud de los telómeros tiene efectos sobre la salud y la mortalidad, esto puede tener efectos sobre la salud y la tasa de envejecimiento en estos hijos. Los autores especularon que este efecto puede proporcionar un mecanismo por el cual las poblaciones tienen cierta plasticidad para adaptar la longevidad a diferentes contextos sociales y ecológicos. [24]Véase también
- Genética del autismo
- Genética de la obesidad
- Estrategias para la ingeniería de senescencia despreciable
Referencias
- ^ Klass MR (1983). "Un método para el aislamiento de mutantes de longevidad en el nematodo Caenorhabditis elegans y resultados iniciales". Mecanismos de envejecimiento y desarrollo . 22 (3–4): 279–286. doi :10.1016/0047-6374(83)90082-9. PMID 6632998. S2CID 6870538.
- ^ Friedman DB, Johnson TE (1988). "Una mutación en el gen age-1 en Caenorhabditis elegans alarga la vida y reduce la fertilidad hermafrodita" ( PDF ) . Genética . 118 (1): 75–86. doi :10.1093/genetics/118.1.75. PMC 1203268 . PMID 8608934.
- ^ Gottlieb S, Ruvkun G (1994). "daf-2, daf-16 y daf-23: genes que interactúan genéticamente y controlan la formación de Dauer en Caenorhabditis elegans". Genética . 137 (1): 107–120. doi :10.1093/genetics/137.1.107. PMC 1205929 . PMID 8056303.
- ^ ab Kenyon C, Chang J, Gensch E, Rudner A, Tabtiang R (1993). "Un mutante de C. elegans que vive el doble que el tipo salvaje". Nature . 366 (6454): 461–4. Bibcode :1993Natur.366..461K. doi :10.1038/366461a0. PMID 8247153. S2CID 4332206.
- ^ Ayyadevara S, Alla R, Thaden JJ, Shmookler Reis RJ (2008). "Notable longevidad y resistencia al estrés de mutantes nulos de PI3K en nematodos". Aging Cell . 7 (1): 13–22. doi : 10.1111/j.1474-9726.2007.00348.x . PMID 17996009. S2CID 2470599.
- ^ Shmookler Reis RJ, Bharill P, Tazearslan C, Ayyadevara S (2009). "Las mutaciones de longevidad extrema orquestan el silenciamiento de múltiples vías de señalización". Biochimica et Biophysica Acta (BBA) - Temas generales . 1790 (10): 1075–83. doi :10.1016/j.bbagen.2009.05.011. PMC 2885961 . PMID 19465083.
- ^ abc Hyun M, Lee J, Lee K, May A, Bohr VA, Ahn B (marzo de 2008). "Longevidad y resistencia al estrés correlacionadas con capacidad de reparación del ADN en Caenorhabditis elegans". Nucleic Acids Res . 36 (4): 1380–9. doi :10.1093/nar/gkm1161. PMC 2275101 . PMID 18203746.
- ^ Tatar M, Kopelman A, Epstein D, Tu MP, Yin CM, Garofalo RS (1988). "Un homólogo mutante del receptor de insulina de Drosophila que extiende la vida útil y afecta la función neuroendocrina". Science . 292 (5514): 107–110. doi :10.1126/science.1057987. PMID 11292875. S2CID 28565101.
- ^ Bartke A, Wright JC, Mattison JA, Ingram DK, Miller RA, Roth GS (2001). "Extensión de la vida útil de ratones longevos" (PDF) . Nature . 414 (6862): 412. doi : 10.1038/35106646 . hdl :2027.42/62803. PMID: 11719795. S2CID : 4379930.
- ^ ab Bass TM; Weinkove D; Houthoofd K; Gems D; Partridge L (octubre de 2007). "Efectos del resveratrol en la longevidad de Drosophila melanogaster y Caenorhabditis elegans ". Mecanismos de envejecimiento y desarrollo . 128 (10): 546–552. doi :10.1016/j.mad.2007.07.007. PMID 17875315. S2CID 1780784.
- ^ ab "Base de datos GenAge" . Consultado el 26 de febrero de 2011 .
- ^ ab Timmers, Paul RHJ; Wilson, James F.; Joshi, Peter K.; Deelen, Joris (16 de julio de 2020). "El análisis genómico multivariado implica nuevos loci y el metabolismo del hemo en el envejecimiento humano". Nature Communications . 11 (1): 3570. Bibcode :2020NatCo..11.3570T. doi : 10.1038/s41467-020-17312-3 . PMC 7366647 . PMID 32678081.
 El texto y las imágenes están disponibles bajo una licencia Creative Commons Atribución 4.0 Internacional.
El texto y las imágenes están disponibles bajo una licencia Creative Commons Atribución 4.0 Internacional. - ^ "Los niveles de hierro en sangre podrían ser clave para retrasar el envejecimiento, según un estudio genético". phys.org . Consultado el 18 de agosto de 2020 .
- ^ Krishnamurthy, J; Torrice, C; Ramsey, MR; Kovalev, GI; Al-Regaiey, K; Su, L; Sharpless, NE (2004). "La expresión de Ink4a/Arf es un biomarcador del envejecimiento". J. Clin. Invest . 114 (9): 1299–1307. doi :10.1172/JCI22475. PMC 524230. PMID 15520862 .
- ^ Panadero DJ; Dawlaty MM; Wijshake T; Jeganathan KB; Malureanu L; van Ree JH; Crespo-Díaz R; Reyes S; Seaburg L; Shapiro V; Behfar A; Terzic A; van de Sluis B; van Deursen JM (enero de 2013). "El aumento de la expresión de BubR1 protege contra la aneuploidía y el cáncer y prolonga la esperanza de vida saludable". Biol celular natural . 15 (1): 96-102. doi :10.1038/ncb2643. PMC 3707109 . PMID 23242215.
- ^ Baker, D.; Wijshake, T.; Tchkonia, T.; LeBrasseur, N.; Childs, B.; van de Sluis, B.; Kirkland, J.; van Deursen, J. (10 de noviembre de 2011). "La eliminación de células senescentes positivas para p16Ink4a retrasa los trastornos asociados al envejecimiento". Nature . 479 (7372): 232–6. Bibcode :2011Natur.479..232B. doi :10.1038/nature10600. PMC 3468323 . PMID 22048312.
- ^ Zhu, Y; Checa, T; Pirtskhalava, T; Gower, CA; Ding, H; Giorgadze, N; Palmer, Alaska; Ikeno, Y; Hubbard, GB; Lenburg, M; O'Hara, SP; LaRusso, NF; Miller, JD; Roos, CM; Verzosa, GC; LeBrasseur, NK; Reyezuelo, JD; Farr, JN; Khosla, S; Fuerte, MB; McGowan, SJ; Fuhrmann-Stroissnigg, H; Gurkar, AU; Zhao, J; Colángelo, D; Dorronsoro, A; Ling, YY; Barghouthy, AS; Navarro, DC; Sanó, T; Robbins, PD; Niedernhofer, LJ; Kirkland, JL (9 de marzo de 2015). "El talón de Aquiles de las células senescentes: del transcriptoma a los fármacos senolíticos". Envejecimiento celular . 14 (4): 644–658. doi :10.1111/acel.12344. PMC 4531078 . PMID 25754370.
- ^ Whittemore K, Martínez-Nevado E, Blasco MA (noviembre de 2019). "Tasas más lentas de acumulación de daño en el ADN en leucocitos se correlacionan con vidas más largas en varias especies de aves y mamíferos". Aging (Albany NY) . 11 (21): 9829–9845. doi :10.18632/aging.102430. PMC 6874430. PMID 31730540 . .
- ^ Bürkle A, Grube K, Küpper JH (1992). "Poli(ADP-ribosilación): su papel en la amplificación inducible del ADN y su correlación con la longevidad de las especies de mamíferos". Exp Clin Immunogenet . 9 (4): 230–40. PMID 1307244.
- ^ Muiras ML, Müller M, Schächter F, Bürkle A (abril de 1998). "Aumento de la actividad poli (ADP-ribosa) polimerasa en líneas celulares linfoblastoides de centenarios". J. Mol Med (Berl) . 76 (5): 346–54. doi :10.1007/s001090050226. PMID 9587069.
- ^ Zhu JL, Vestergaard M, Madsen KM, Olsen J (2008). "Edad paterna y mortalidad en niños". Revista Europea de Epidemiología . 23 (7): 443–7. doi :10.1007/s10654-008-9253-3. PMID 18437509. S2CID 2092996.
- ^ "En este estudio en particular, no se realizó ningún ajuste en función de la salud de la madre, y esto podría haber tenido un gran efecto en la mortalidad infantil". Servicio Nacional de Salud (Reino Unido), "Los padres mayores y la muerte de los niños" (consultado el 15 de noviembre de 2013)
- ^ Tournaye 2009, pág. 102
- ^ Eisenberg DT, Hayes MG, Kuzawa CW (junio de 2012). "La edad paterna tardía de reproducción en humanos se asocia con telómeros más largos a lo largo de dos generaciones de descendientes". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 109 (26): 10251–6. Bibcode :2012PNAS..10910251E. doi : 10.1073/pnas.1202092109 . PMC 3387085 . PMID 22689985.
Fuentes
- Tournaye, Herman (junio de 2009). "Envejecimiento reproductivo masculino". En Bewley, Susan; Ledger, William; Nikolaou, Dimitrios (eds.). Envejecimiento reproductivo . Cambridge University Press. págs. 95–104. doi :10.1017/CBO9781107784734.012. ISBN 978-1-906985-13-4.
Enlaces externos
- Recursos genómicos sobre el envejecimiento humano, una colección de bases de datos y herramientas diseñadas para ayudar a los investigadores a estudiar la genética del envejecimiento humano
- La base de datos NetAge, una base de datos en línea y herramientas de análisis de redes para la investigación biogerontológica
