Estudio de asociación de todo el genoma
En genómica , un estudio de asociación de todo el genoma ( estudio GWA o GWAS ) es un estudio observacional de un conjunto de variantes genéticas de todo el genoma en diferentes individuos para ver si alguna variante está asociada con un rasgo. Los estudios GWA generalmente se centran en las asociaciones entre polimorfismos de un solo nucleótido (SNP) y rasgos como las principales enfermedades humanas, pero también se pueden aplicar a cualquier otra variante genética y a cualquier otro organismo.
Cuando se aplican a datos humanos, los estudios GWA comparan el ADN de participantes que tienen distintos fenotipos para un rasgo o enfermedad en particular. Estos participantes pueden ser personas con una enfermedad (casos) y personas similares sin la enfermedad (controles), o pueden ser personas con diferentes fenotipos para un rasgo en particular, por ejemplo, la presión arterial. Este enfoque se conoce como fenotipo primero, en el que los participantes se clasifican primero por su(s) manifestación(es) clínica(s), a diferencia del genotipo primero . Cada persona proporciona una muestra de ADN, de la cual se leen millones de variantes genéticas utilizando matrices de SNP . Si hay evidencia estadística significativa de que un tipo de la variante (un alelo ) es más frecuente en personas con la enfermedad, se dice que la variante está asociada con la enfermedad. Luego se considera que los SNP asociados marcan una región del genoma humano que puede influir en el riesgo de enfermedad.
Los estudios GWA investigan el genoma completo, a diferencia de los métodos que prueban específicamente un pequeño número de regiones genéticas preespecificadas. Por lo tanto, los GWAS son un enfoque que no se basa en candidatos , a diferencia de los estudios basados en candidatos específicos de genes . Los estudios GWA identifican SNP y otras variantes en el ADN asociadas con una enfermedad, pero no pueden especificar por sí solos qué genes son causales. [1] [2] [3]
El primer GWAS exitoso publicado en 2002 estudió el infarto de miocardio. [4] Este diseño de estudio se implementó luego en el estudio histórico GWA 2005 que investigó a pacientes con degeneración macular relacionada con la edad , y encontró dos SNP con una frecuencia de alelos significativamente alterada en comparación con los controles sanos. [5] A partir de 2017 [actualizar], más de 3000 estudios GWA humanos han examinado más de 1800 enfermedades y rasgos, y se han encontrado miles de asociaciones de SNP. [6] Excepto en el caso de enfermedades genéticas raras , estas asociaciones son muy débiles, pero si bien cada asociación individual puede no explicar gran parte del riesgo, brindan información sobre genes y vías críticos y pueden ser importantes cuando se consideran en conjunto .
Fondo

Dos genomas humanos cualesquiera difieren en millones de formas diferentes. Hay pequeñas variaciones en los nucleótidos individuales de los genomas ( SNP ), así como muchas variaciones más grandes, como deleciones , inserciones y variaciones en el número de copias . Cualquiera de estas puede causar alteraciones en los rasgos de un individuo, o fenotipo , que puede ser cualquier cosa desde el riesgo de enfermedad hasta propiedades físicas como la altura. [8] Alrededor del año 2000, antes de la introducción de los estudios GWA, el principal método de investigación era a través de estudios de herencia de ligamiento genético en familias. Este enfoque había demostrado ser muy útil para los trastornos de un solo gen . [9] [8] [10] Sin embargo, para enfermedades comunes y complejas, los resultados de los estudios de ligamiento genético resultaron difíciles de reproducir. [8] [10] Una alternativa sugerida a los estudios de ligamiento fue el estudio de asociación genética . Este tipo de estudio pregunta si el alelo de una variante genética se encuentra con más frecuencia de lo esperado en individuos con el fenotipo de interés (por ejemplo, con la enfermedad en estudio). Los primeros cálculos sobre el poder estadístico indicaron que este enfoque podría ser mejor que los estudios de ligamiento para detectar efectos genéticos débiles. [11]
Además del marco conceptual, varios factores adicionales permitieron los estudios GWA. Uno fue la aparición de los biobancos , que son repositorios de material genético humano que redujeron en gran medida el costo y la dificultad de recolectar cantidades suficientes de especímenes biológicos para el estudio. [12] Otro fue el Proyecto Internacional HapMap , que, a partir de 2003, identificó la mayoría de los SNP comunes interrogados en un estudio GWA. [13] La estructura de haplobloques identificada por el proyecto HapMap también permitió centrarse en el subconjunto de SNP que describiría la mayor parte de la variación. También el desarrollo de los métodos para genotipar todos estos SNP utilizando matrices de genotipado fue un prerrequisito importante. [14]
Métodos


El enfoque más común de los estudios GWA es la configuración de caso-control , que compara dos grandes grupos de individuos, un grupo de control sano y un grupo de casos afectados por una enfermedad. Todos los individuos en cada grupo suelen ser genotipados en SNP conocidos comunes. El número exacto de SNP depende de la tecnología de genotipado, pero normalmente son un millón o más. [7] Para cada uno de estos SNP, luego se investiga si la frecuencia de alelos se altera significativamente entre el caso y el grupo de control. [16] En tales configuraciones, la unidad fundamental para informar los tamaños del efecto es la razón de probabilidades . La razón de probabilidades es la relación de dos probabilidades, que en el contexto de los estudios GWA son las probabilidades de caso para individuos que tienen un alelo específico y las probabilidades de caso para individuos que no tienen ese mismo alelo.
Ejemplo : supongamos que hay dos alelos, T y C. El número de individuos en el grupo de casos que tiene el alelo T está representado por 'A' y el número de individuos en el grupo de control que tiene el alelo T está representado por 'B'. De manera similar, el número de individuos en el grupo de casos que tiene el alelo C está representado por 'X' y el número de individuos en el grupo de control que tiene el alelo C está representado por 'Y'. En este caso, la razón de probabilidades para el alelo T es A:B (que significa 'A a B', en la terminología de probabilidades estándar) dividido por X:Y, que en notación matemática es simplemente (A/B)/(X/Y).
Cuando la frecuencia de alelos en el grupo de casos es mucho mayor que en el grupo de control, la razón de probabilidades es mayor que 1, y viceversa para una frecuencia de alelos menor. Además, un valor P para la significancia de la razón de probabilidades se calcula típicamente usando una prueba de chi-cuadrado simple . Encontrar razones de probabilidades que sean significativamente diferentes de 1 es el objetivo del estudio GWA porque esto demuestra que un SNP está asociado con la enfermedad. [16] Debido a que se prueban tantas variantes, es una práctica estándar requerir que el valor p sea menor que5 × 10 −8 para considerar una variante significativa.
Variaciones en el enfoque de casos y controles . Una alternativa común a los estudios GWA de casos y controles es el análisis de datos fenotípicos cuantitativos, por ejemplo, la altura o las concentraciones de biomarcadores o incluso la expresión genética . Asimismo, se pueden utilizar estadísticas alternativas diseñadas para patrones de dominancia o penetración recesiva . [16] Los cálculos se realizan normalmente utilizando software de bioinformática como SNPTEST y PLINK, que también incluyen soporte para muchas de estas estadísticas alternativas. [15] [17] Los GWAS se centran en el efecto de los SNP individuales. Sin embargo, también es posible que las interacciones complejas entre dos o más SNP ( epistasia ) puedan contribuir a enfermedades complejas. Debido al número potencialmente exponencial de interacciones, detectar interacciones estadísticamente significativas en los datos de GWAS es un desafío tanto computacional como estadístico. Esta tarea se ha abordado en publicaciones existentes que utilizan algoritmos inspirados en la minería de datos. [18] Además, los investigadores intentan integrar los datos de GWA con otros datos biológicos como la red de interacción proteína-proteína para extraer resultados más informativos. [19] [20] A pesar del desafío previamente percibido que plantea la gran cantidad de combinaciones de SNP, un estudio reciente ha revelado con éxito mapas epistáticos completos con una resolución a nivel de genes en plantas/Arabidopsis thaliana [21]
.jpg/440px-13059_2024_3202_Fig5_HTML_(1).jpg)

Un paso clave en la mayoría de los estudios de GWA es la imputación de genotipos en SNP que no están en el chip de genotipo utilizado en el estudio. [23] Este proceso aumenta en gran medida la cantidad de SNP que se pueden probar para la asociación, aumenta la potencia del estudio y facilita el metanálisis de GWAS en distintas cohortes. La imputación de genotipos se lleva a cabo mediante métodos estadísticos que imputan datos genotípicos a un conjunto de paneles de referencia de haplotipos, que normalmente han sido densamente genotipados utilizando la secuenciación del genoma completo. Estos métodos aprovechan el intercambio de haplotipos entre individuos en tramos cortos de secuencia para imputar alelos. Los paquetes de software existentes para la imputación de genotipos incluyen IMPUTE2, [24] Minimac, Beagle [25] y MaCH. [26]
Además del cálculo de la asociación, es común tener en cuenta cualquier variable que potencialmente podría confundir los resultados. El sexo, la edad y la ascendencia son ejemplos comunes de variables de confusión. Además, también se sabe que muchas variaciones genéticas están asociadas con las poblaciones geográficas e históricas en las que surgieron las mutaciones por primera vez. [27] Debido a esta asociación, los estudios deben tener en cuenta el origen geográfico y étnico de los participantes controlando lo que se llama estratificación de la población . Si no lo hicieran, los estudios podrían producir resultados falsos positivos. [28]
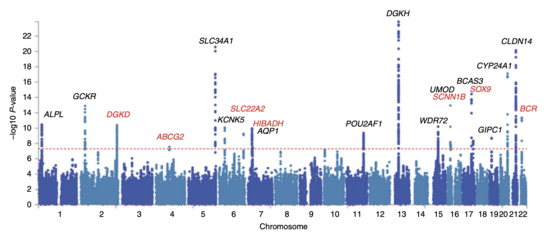
Una vez que se han calculado los odds ratios y los valores P para todos los SNP, un enfoque común es crear un gráfico de Manhattan . En el contexto de los estudios de GWA, este gráfico muestra el logaritmo negativo del valor P como función de la ubicación genómica. Por lo tanto, los SNP con la asociación más significativa se destacan en el gráfico, generalmente como pilas de puntos debido a la estructura de haplobloques. Es importante destacar que el umbral del valor P para la significación se corrige para múltiples problemas de prueba. El umbral exacto varía según el estudio, [29] pero el umbral de significación convencional para todo el genoma es5 × 10 −8 para ser significativo frente a cientos de miles a millones de SNP probados. [7] [16] [30] Los estudios GWA generalmente realizan el primer análisis en una cohorte de descubrimiento, seguido de la validación de los SNP más significativos en una cohorte de validación independiente. [31]
Resultados


Se han hecho intentos de crear catálogos completos de SNP que se han identificado a partir de estudios de GWA. [33] A partir de 2009, los SNP asociados con enfermedades se cuentan por miles. [34]
El primer estudio GWA, realizado en 2005, comparó a 96 pacientes con degeneración macular relacionada con la edad (DMAE) con 50 controles sanos. [35] Identificó dos SNP con una frecuencia de alelos significativamente alterada entre los dos grupos. Estos SNP se ubicaron en el gen que codifica el factor H del complemento , lo que fue un hallazgo inesperado en la investigación de la DMAE. Los hallazgos de estos primeros estudios GWA impulsaron posteriormente una mayor investigación funcional hacia la manipulación terapéutica del sistema del complemento en la DMAE. [36]
Otra publicación histórica en la historia de los estudios GWA fue el estudio Wellcome Trust Case Control Consortium (WTCCC), el estudio GWA más grande jamás realizado en el momento de su publicación en 2007. El WTCCC incluyó 14.000 casos de siete enfermedades comunes (~2.000 individuos para cada una de las siguientes: enfermedad cardíaca coronaria , diabetes tipo 1 , diabetes tipo 2 , artritis reumatoide , enfermedad de Crohn , trastorno bipolar e hipertensión ) y 3.000 controles compartidos. [15] Este estudio logró descubrir muchos genes asociados con estas enfermedades. [15] [37]
Desde estos primeros estudios de referencia de GWA, ha habido dos tendencias generales. [38] Una ha sido hacia tamaños de muestra cada vez más grandes. En 2018, varios estudios de asociación de todo el genoma están alcanzando un tamaño de muestra total de más de 1 millón de participantes, incluidos 1,1 millones en un estudio de todo el genoma sobre el logro educativo [39] seguido de otro en 2022 con 3 millones de individuos [40] y un estudio de insomnio que contiene 1,3 millones de individuos. [41] La razón es el impulso hacia la detección confiable de SNP de riesgo que tienen tamaños de efecto más pequeños y una frecuencia de alelos más baja. Otra tendencia ha sido hacia el uso de fenotipos definidos más estrictamente, como los lípidos en sangre , la proinsulina o biomarcadores similares. [42] [43] Estos se denominan fenotipos intermedios y sus análisis pueden ser de valor para la investigación funcional de biomarcadores. [44]
Una variante de GWAS utiliza participantes que son parientes de primer grado de personas con una enfermedad. Este tipo de estudio se ha denominado estudio de asociación de todo el genoma por poder ( GWAX ). [45]
Un punto central de debate en los estudios GWA ha sido que la mayoría de las variaciones de SNP encontradas por los estudios GWA están asociadas con solo un pequeño aumento del riesgo de la enfermedad y tienen solo un pequeño valor predictivo. La razón de probabilidades mediana es de 1,33 por SNP de riesgo, y solo unos pocos muestran razones de probabilidades superiores a 3,0. [1] [46] Estas magnitudes se consideran pequeñas porque no explican gran parte de la variación hereditaria. Esta variación hereditaria se estima a partir de estudios de heredabilidad basados en gemelos monocigóticos . [47] Por ejemplo, se sabe que el 40% de la varianza en la depresión se puede explicar por diferencias hereditarias, pero los estudios GWA solo explican una minoría de esta varianza. [47]
Aplicaciones clínicas y ejemplos
Un desafío para el futuro estudio exitoso de GWA es aplicar los hallazgos de una manera que acelere el desarrollo de medicamentos y diagnósticos, incluida una mejor integración de los estudios genéticos en el proceso de desarrollo de medicamentos y un enfoque en el papel de la variación genética en el mantenimiento de la salud como modelo para diseñar nuevos medicamentos y diagnósticos . [48] Varios estudios han analizado el uso de marcadores de SNP de riesgo como un medio para mejorar directamente la precisión del pronóstico . Algunos han encontrado que la precisión del pronóstico mejora, [49] mientras que otros informan solo beneficios menores de este uso. [50] En general, un problema con este enfoque directo es la pequeña magnitud de los efectos observados. Un efecto pequeño en última instancia se traduce en una mala separación de casos y controles y, por lo tanto, solo una pequeña mejora de la precisión del pronóstico. Por lo tanto, una aplicación alternativa es el potencial de los estudios de GWA para dilucidar la fisiopatología . [51]
Tratamiento de la hepatitis C
Uno de esos éxitos está relacionado con la identificación de la variante genética asociada con la respuesta al tratamiento contra el virus de la hepatitis C. En el caso de la hepatitis C de genotipo 1 tratada con interferón-alfa-2a pegilado o interferón-alfa-2b pegilado combinado con ribavirina , un estudio de GWA [52] ha demostrado que los SNP cerca del gen humano IL28B , que codifica el interferón lambda 3, están asociados con diferencias significativas en la respuesta al tratamiento. Un informe posterior demostró que las mismas variantes genéticas también están asociadas con la eliminación natural del virus de la hepatitis C de genotipo 1. [53] Estos importantes hallazgos facilitaron el desarrollo de la medicina personalizada y permitieron a los médicos personalizar las decisiones médicas en función del genotipo del paciente. [54]
eQTL, LDL y enfermedad cardiovascular
El objetivo de dilucidar la fisiopatología también ha llevado a un mayor interés en la asociación entre los SNP de riesgo y la expresión génica de genes cercanos, los llamados estudios de loci de rasgos cuantitativos de expresión (eQTL). [55] La razón es que los estudios GWAS identifican SNP de riesgo, pero no genes de riesgo, y la especificación de genes está un paso más cerca de objetivos farmacológicos procesables . Como resultado, los principales estudios GWA para 2011 generalmente incluían un análisis eQTL extenso. [56] [57] [58] Uno de los efectos eQTL más fuertes observados para un SNP de riesgo identificado por GWA es el locus SORT1. [42] Los estudios de seguimiento funcional de este locus utilizando ARN interferente pequeño y ratones knock-out de genes han arrojado luz sobre el metabolismo de las lipoproteínas de baja densidad , que tienen importantes implicaciones clínicas para la enfermedad cardiovascular . [42] [59] [60]
Fibrilación auricular
Por ejemplo, un metaanálisis realizado en 2018 reveló el descubrimiento de 70 nuevos loci asociados con la fibrilación auricular . Se han identificado diferentes variantes asociadas con genes codificadores de factores de transcripción , como TBX3 y TBX5 , NKX2-5 o PITX2 , que están involucrados en la regulación de la conducción cardíaca, en la modulación del canal iónico y en el desarrollo cardíaco. También se identificaron nuevos genes involucrados en la taquicardia ( CASQ2 ) o asociados con la alteración de la comunicación entre células musculares cardíacas ( PKP2 ). [61]
Esquizofrenia
Investigación que utilizó un modelo computacional de predicción de interacción de proteínas de alta precisión (HiPPIP) que descubrió 504 nuevas interacciones proteína-proteína (PPI) asociadas con genes vinculados a la esquizofrenia . [62] [63] [64] Si bien la evidencia que respalda la base genética de la esquizofrenia no es controvertida, un estudio encontró que 25 genes candidatos de esquizofrenia descubiertos a partir de GWAS tenían poca asociación con la esquizofrenia, lo que demuestra que los GWAS por sí solos pueden ser insuficientes para identificar genes candidatos. [65]
Aplicaciones de conservación
Los estudios de GWA a nivel de población pueden utilizarse para identificar genes adaptativos que ayuden a evaluar la capacidad de las especies para adaptarse a las condiciones ambientales cambiantes a medida que el clima global se vuelve más cálido . [66] Esto podría ayudar a determinar el riesgo de extinción de las especies y, por lo tanto, podría ser una herramienta importante para la planificación de la conservación . El uso de estudios de GWA para determinar genes adaptativos podría ayudar a dilucidar la relación entre la diversidad genética neutra y adaptativa .
Aplicaciones agrícolas
Etapas del crecimiento de las plantas y componentes del rendimiento
Los estudios de GWA actúan como una herramienta importante en el mejoramiento de plantas. Con grandes datos de genotipado y fenotipado, los GWAS son poderosos para analizar modos de herencia complejos de caracteres que son componentes importantes del rendimiento, como el número de granos por espiga, el peso de cada grano y la estructura de la planta. En un estudio sobre GWAS en trigo de primavera, los GWAS han revelado una fuerte correlación de la producción de grano con los datos de enraizamiento, biomasa y número de granos por espiga. [67] El estudio de GWA también es un éxito en el estudio de la arquitectura genética de caracteres complejos en el arroz. [68]
Patógenos de plantas
La aparición de patógenos vegetales ha planteado serias amenazas a la salud de las plantas y a la biodiversidad. En este sentido, la identificación de tipos silvestres que tienen resistencia natural a ciertos patógenos podría ser de vital importancia. Además, necesitamos predecir qué alelos están asociados con la resistencia. Los estudios GWA son una herramienta poderosa para detectar las relaciones entre ciertas variantes y la resistencia al patógeno vegetal , lo que resulta beneficioso para el desarrollo de nuevos cultivares resistentes a los patógenos. [69]
Pollo
El primer estudio de GWA en pollos fue realizado por Abasht y Lamont [70] en 2007. Este GWA se utilizó para estudiar el rasgo de gordura en la población F2 hallado previamente. Se encontraron SNP significativamente relacionados en 10 cromosomas (1, 2, 3, 4, 7, 8, 10, 12, 15 y 27).
Limitaciones
Los estudios GWA tienen varios problemas y limitaciones que pueden solucionarse mediante un control de calidad y una configuración de estudio adecuados. La falta de grupos de casos y controles bien definidos, el tamaño insuficiente de la muestra y el control para la estratificación de la población son problemas comunes. [2] En cuanto al problema estadístico de las pruebas múltiples, se ha observado que "el enfoque GWA puede ser problemático porque la enorme cantidad de pruebas estadísticas realizadas presenta un potencial sin precedentes de resultados falsos positivos ". [2] Es por eso que todos los GWAS modernos utilizan un umbral de valor p muy bajo. Además de problemas fácilmente corregibles como estos, han surgido algunos problemas más sutiles pero importantes. Un estudio GWA de alto perfil que investigó a individuos con vidas muy largas para identificar SNP asociados con la longevidad es un ejemplo de esto. [71] La publicación fue objeto de escrutinio debido a una discrepancia entre el tipo de matriz de genotipado en el grupo de casos y control, lo que provocó que varios SNP se resaltaran falsamente como asociados con la longevidad. [72] El estudio fue posteriormente retractado , [73] pero luego se publicó un manuscrito modificado. [74] Actualmente, muchos estudios de análisis de grupos de genoma completo controlan la matriz de genotipado. Si existen diferencias sustanciales entre los grupos en cuanto al tipo de matriz de genotipado, como sucede con cualquier factor de confusión, los estudios de análisis de grupos de genoma completo podrían dar como resultado un falso positivo. Otra consecuencia es que dichos estudios no pueden detectar la contribución de mutaciones muy raras que no están incluidas en la matriz o que no pueden imputarse. [75]
Además, los estudios GWA identifican variantes de riesgo candidatas para la población de la que se realiza su análisis, y como la mayoría de los estudios GWA provienen históricamente de bases de datos europeas, existe una falta de traducción de las variantes de riesgo identificadas a otras poblaciones no europeas. [76] Por ejemplo, los estudios GWA para enfermedades como la enfermedad de Alzheimer se han realizado principalmente en poblaciones caucásicas, lo que no proporciona una visión adecuada de otras poblaciones étnicas, incluidos los afroamericanos o los asiáticos orientales . Las estrategias alternativas sugeridas implican el análisis de ligamiento . [77] [78] Más recientemente, el precio rápidamente decreciente de la secuenciación del genoma completo también ha proporcionado una alternativa realista a los estudios GWA basados en matrices de genotipado . La secuenciación de alto rendimiento tiene el potencial de eludir algunas de las deficiencias de los GWA sin secuenciación. [79] El apareamiento selectivo entre rasgos puede inflar las estimaciones de similitud del fenotipo genético. [80]
Mapeo fino
Las matrices de genotipado diseñadas para GWAS se basan en el desequilibrio de ligamiento para proporcionar cobertura de todo el genoma mediante la genotipificación de un subconjunto de variantes. Debido a esto, es poco probable que las variantes asociadas informadas sean las variantes causales reales. Las regiones asociadas pueden contener cientos de variantes que abarcan grandes regiones y muchos genes diferentes, lo que dificulta la interpretación biológica de los loci de GWAS. El mapeo fino es un proceso para refinar estas listas de variantes asociadas a un conjunto creíble que probablemente incluya la variante causal.
El mapeo fino requiere que todas las variantes en la región asociada hayan sido genotipadas o imputadas (cobertura densa), un control de calidad muy estricto que dé como resultado genotipos de alta calidad y tamaños de muestra grandes suficientes para separar las señales altamente correlacionadas. Existen varios métodos diferentes para realizar el mapeo fino, y todos los métodos producen una probabilidad posterior de que una variante en ese locus sea causal. Debido a que los requisitos suelen ser difíciles de satisfacer, todavía hay ejemplos limitados de estos métodos que se aplican de manera más general.
Véase también
- Mapeo de asociaciones
- Estudio de asociación de todo el transcriptoma
- Epidemiología
- Diversidad genética
- Interacción gen-ambiente
- Genómica
- Desequilibrio de ligamiento
- Epidemiología molecular
- Puntuación poligénica
- Genética de poblaciones
- Epidemiología genética
- Hipótesis de enfermedad común-variante común
- Estudio de asociación del microbioma en su conjunto
- Biología de la conservación
- Herramienta WGAViewer diseñada para ayudar a interpretar los resultados generados a partir de un estudio de asociación de todo el genoma
Referencias
- ^ ab Manolio TA (julio de 2010). "Estudios de asociación de todo el genoma y evaluación del riesgo de enfermedad". The New England Journal of Medicine . 363 (2): 166–76. doi : 10.1056/NEJMra0905980 . PMID 20647212.
- ^ abc Pearson TA, Manolio TA (marzo de 2008). "Cómo interpretar un estudio de asociación de todo el genoma". JAMA . 299 (11): 1335–44. doi :10.1001/jama.299.11.1335. PMID 18349094.
- ^ "Estudios de asociación del genoma completo". Instituto Nacional de Investigación del Genoma Humano .
- ^ Ozaki K, Ohnishi Y, Iida A, Sekine A, Yamada R, Tsunoda T, et al. (diciembre de 2002). "SNP funcionales en el gen de la linfotoxina alfa que están asociados con la susceptibilidad al infarto de miocardio". Nature Genetics . 32 (4): 650–4. doi :10.1038/ng1047. PMID 12426569. S2CID 21414260.
- ^ Klein RJ, Zeiss C, Chew EY, Tsai JY, Sackler RS, Haynes C, et al. (abril de 2005). "Polimorfismo del factor H del complemento en la degeneración macular relacionada con la edad". Science . 308 (5720): 385–9. Bibcode :2005Sci...308..385K. doi :10.1126/science.1109557. PMC 1512523 . PMID 15761122.
- ^ "Catálogo GWAS: Catálogo NHGRI-EBI de estudios de asociación de todo el genoma publicados". Laboratorio Europeo de Biología Molecular . Consultado el 18 de abril de 2017 .
- ^ abc Bush WS, Moore JH (2012). Lewitter F, Kann M (eds.). "Capítulo 11: Estudios de asociación de todo el genoma". PLOS Computational Biology . 8 (12): e1002822. Bibcode :2012PLSCB...8E2822B. doi : 10.1371/journal.pcbi.1002822 . PMC 3531285 . PMID 23300413.
- ^ abc Strachan T, Read A (2011). Genética molecular humana (4.ª ed.). Garland Science. págs. 467–495. ISBN 978-0-8153-4149-9.
- ^ "Herencia mendeliana en línea en el hombre". Archivado desde el original el 5 de diciembre de 2011. Consultado el 6 de diciembre de 2011 .
- ^ ab Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M (noviembre de 2001). "Escaneos de todo el genoma de enfermedades humanas complejas: es difícil encontrar un vínculo verdadero". American Journal of Human Genetics . 69 (5): 936–50. doi :10.1086/324069. PMC 1274370 . PMID 11565063.
- ^ Risch N, Merikangas K (septiembre de 1996). "El futuro de los estudios genéticos de enfermedades humanas complejas". Science . 273 (5281): 1516–7. Bibcode :1996Sci...273.1516R. doi :10.1126/science.273.5281.1516. PMID 8801636. S2CID 5228523.
- ^ Greely HT (2007). "Los fundamentos éticos y legales inquietantes de los biobancos genómicos a gran escala". Revisión anual de genómica y genética humana . 8 : 343–64. doi : 10.1146/annurev.genom.7.080505.115721 . PMID 17550341.
- ^ El Proyecto Internacional HapMap, Gibbs RA, Belmont JW, Hardenbol P, Willis TD, Yu F, Yang H, Ch'Ang LY, Huang W (diciembre de 2003). "El Proyecto Internacional HapMap" (PDF) . Nature . 426 (6968): 789–96. Bibcode :2003Natur.426..789G. doi :10.1038/nature02168. hdl : 2027.42/62838 . PMID 14685227. S2CID 4387110.
- ^ Schena M, Shalon D, Davis RW, Brown PO (octubre de 1995). "Monitoreo cuantitativo de patrones de expresión génica con un microarreglo de ADN complementario". Science . 270 (5235): 467–70. Bibcode :1995Sci...270..467S. doi :10.1126/science.270.5235.467. PMID 7569999. S2CID 6720459.
- ^ abcd Wellcome Trust Case Control Consortium, Burton PR (junio de 2007). "Estudio de asociación de todo el genoma de 14.000 casos de siete enfermedades comunes y 3.000 controles compartidos". Nature . 447 (7145): 661–78. Bibcode :2007Natur.447..661B. doi :10.1038/nature05911. PMC 2719288 . PMID 17554300.
- ^ abcd Clarke GM, Anderson CA, Pettersson FH, Cardon LR, Morris AP, Zondervan KT (febrero de 2011). "Análisis estadístico básico en estudios genéticos de casos y controles". Nature Protocols . 6 (2): 121–33. doi :10.1038/nprot.2010.182. PMC 3154648 . PMID 21293453.
- ^ Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D, et al. (septiembre de 2007). "PLINK: un conjunto de herramientas para la asociación de todo el genoma y los análisis de ligamiento basados en la población". American Journal of Human Genetics . 81 (3): 559–75. doi :10.1086/519795. PMC 1950838 . PMID 17701901.
- ^ Llinares-López F, Grimm DG, Bodenham DA, Gieraths U, Sugiyama M, Rowan B, Borgwardt K (junio de 2015). "Detección de intervalos de heterogeneidad genética asociados a rasgos complejos en todo el genoma". Bioinformática . 31 (12): i240-9. doi :10.1093/bioinformatics/btv263. PMC 4559912 . PMID 26072488.
- ^ Ayati M, Erten S, Chance MR, Koyutürk M (diciembre de 2015). "MOBAS: identificación de subredes de proteínas asociadas a enfermedades mediante puntuación basada en modularidad". Revista EURASIP sobre bioinformática y biología de sistemas . 2015 (1): 7. doi : 10.1186/s13637-015-0025-6 . PMC 5270451. PMID 28194175 .
- ^ Ayati M, Koyutürk M (1 de enero de 2015). "Evaluación de la asociación de enfermedades colectivas de múltiples loci genómicos". Actas de la 6.ª Conferencia de la ACM sobre bioinformática, biología computacional e informática de la salud . BCB '15. Nueva York, NY, EE. UU.: ACM. págs. 376–385. doi :10.1145/2808719.2808758. ISBN . 978-1-4503-3853-0. Número de identificación del sujeto 5942777.
- ^ Carré C, Carluer JB, Chaux C, Estoup-Streiff C, Roche N, Hosy E, Mas A, Krouk G (marzo de 2024). "GWAS de próxima generación: mapas de interacción epistática 2D completos recuperan parte de la heredabilidad faltante y mejoran la predicción fenotípica". Biología del genoma. doi:10.1186/s13059-024-03202-0. PMID 38523316. S2CID 146570
- ^ ab Carré C, Carluer JB, Chaux C, Estoup-Streiff C, Roche N, Hosy E, Mas A, Krouk G (25 de marzo de 2024). "GWAS de próxima generación: mapas de interacción epistática 2D completos recuperan parte de la heredabilidad faltante y mejoran la predicción fenotípica". Biología del genoma . 25 (1): 76. doi : 10.1186/s13059-024-03202-0 . ISSN 1474-760X. PMC 10962106 . PMID 38523316.
- ^ Marchini J, Howie B (julio de 2010). "Imputación de genotipos para estudios de asociación de todo el genoma". Nature Reviews Genetics . 11 (7): 499–511. doi :10.1038/nrg2796. PMID 20517342. S2CID 1465707.
- ^ Howie B, Marchini J, Stephens M (noviembre de 2011). "Imputación de genotipos con miles de genomas". G3 . 1 (6): 457–70. doi :10.1534/g3.111.001198. PMC 3276165 . PMID 22384356.
- ^ Browning BL, Browning SR (febrero de 2009). "Un enfoque unificado para la imputación de genotipos y la inferencia de la fase del haplotipo para grandes conjuntos de datos de tríos e individuos no relacionados". American Journal of Human Genetics . 84 (2): 210–23. doi :10.1016/j.ajhg.2009.01.005. PMC 2668004 . PMID 19200528.
- ^ Li Y, Willer CJ, Ding J, Scheet P, Abecasis GR (diciembre de 2010). "MaCH: uso de datos de secuencia y genotipo para estimar haplotipos y genotipos no observados". Epidemiología genética . 34 (8): 816–34. doi :10.1002/gepi.20533. PMC 3175618 . PMID 21058334.
- ^ Novembre J, Johnson T, Bryc K, Kutalik Z, Boyko AR, Auton A, Indap A, King KS, Bergmann S, Nelson MR, Stephens M, Bustamante CD (noviembre de 2008). "Los genes reflejan la geografía en Europa". Nature . 456 (7218): 98–101. Bibcode :2008Natur.456...98N. doi :10.1038/nature07331. PMC 2735096 . PMID 18758442.
- ^ Charney E (enero de 2017). "Genes, comportamiento y genética del comportamiento". Wiley Interdisciplinary Reviews. Cognitive Science . 8 (1–2): e1405. doi :10.1002/wcs.1405. hdl : 10161/13337 . PMID 27906529.
- ^ Wittkowski KM, Sonakya V, Bigio B, Tonn MK, Shic F, Ascano M, Nasca C, Gold-Von Simson G (enero de 2014). "Un nuevo enfoque de bioestadística computacional implica una desfosforilación alterada de los receptores de factores de crecimiento asociada con la gravedad del autismo". Psiquiatría Traslacional . 4 (1): e354. doi :10.1038/tp.2013.124. PMC 3905234 . PMID 24473445.
- ^ Barsh GS, Copenhaver GP, Gibson G, Williams SM (julio de 2012). "Directrices para estudios de asociación de todo el genoma". PLOS Genetics . 8 (7): e1002812. doi : 10.1371/journal.pgen.1002812 . PMC 3390399 . PMID 22792080.
- ^ Smith SM, Douaud G, Chen W, Hanayik T, Alfaro-Almagro F, Sharp K, Elliott LT (2021). "Un conjunto ampliado de estudios de asociación de fenotipos de imágenes cerebrales en todo el genoma en el Biobanco del Reino Unido". Nat Neurosci . 24 (5): 737–745. doi : 10.1038/s41593-021-00826-4 . PMC 7610742 . PMID 33875891.
- ^ Sanna S, Li B, Mulas A, Sidore C, Kang HM, Jackson AU, et al. (julio de 2011). Gibson G (ed.). "El mapeo preciso de cinco loci asociados con el colesterol de lipoproteínas de baja densidad detecta variantes que duplican la heredabilidad explicada". PLOS Genetics . 7 (7): e1002198. doi : 10.1371/journal.pgen.1002198 . PMC 3145627 . PMID 21829380.
- ^ Hindorff LA, Sethupathy P, Junkins HA, Ramos EM, Mehta JP, Collins FS, Manolio TA (junio de 2009). "Posibles implicaciones etiológicas y funcionales de los loci de asociación de todo el genoma para enfermedades y rasgos humanos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 106 (23): 9362–7. Bibcode :2009PNAS..106.9362H. doi : 10.1073/pnas.0903103106 . PMC 2687147 . PMID 19474294.
- ^ Johnson AD, O'Donnell CJ (enero de 2009). "Una base de datos de acceso abierto de resultados de asociación de todo el genoma". BMC Medical Genetics . 10 : 6. doi : 10.1186/1471-2350-10-6 . PMC 2639349 . PMID 19161620.
- ^ Haines JL, Hauser MA, Schmidt S, Scott WK, Olson LM, Gallins P, Spencer KL, Kwan SY, Noureddine M, Gilbert JR, Schnetz-Boutaud N, Agarwal A, Postel EA, Pericak-Vance MA (abril de 2005). "La variante del factor H del complemento aumenta el riesgo de degeneración macular relacionada con la edad". Science . 308 (5720): 419–21. Bibcode :2005Sci...308..419H. doi : 10.1126/science.1110359 . PMID 15761120. S2CID 32716116.
- ^ Fridkis-Hareli M, Storek M, Mazsaroff I, Risitano AM, Lundberg AS, Horvath CJ, Holers VM (octubre de 2011). "Diseño y desarrollo de TT30, un nuevo inhibidor de la convertasa C3/C5 dirigido a C3d para el tratamiento de enfermedades mediadas por la vía alternativa del complemento humano". Blood . 118 (17): 4705–13. doi :10.1182/blood-2011-06-359646. PMC 3208285 . PMID 21860027.
- ^ "Hoy se publica el estudio más grande jamás realizado sobre la genética de enfermedades comunes" (Comunicado de prensa). Wellcome Trust Case Control Consortium. 6 de junio de 2007. Archivado desde el original el 4 de junio de 2008. Consultado el 19 de junio de 2008 .
- ^ Ioannidis JP, Thomas G, Daly MJ (mayo de 2009). "Validación, aumento y refinamiento de las señales de asociación de todo el genoma". Nature Reviews Genetics . 10 (5): 318–29. doi :10.1038/nrg2544. PMC 7877552 . PMID 19373277. S2CID 6463743.
- ^ Lee JJ, Wedow R, Okbay A, Kong E, Maghzian O, Zacher M, Nguyen-Viet TA, Bowers P, Sidorenko J, Karlsson Linnér R, et al. (julio de 2018). "Descubrimiento de genes y predicción poligénica a partir de un estudio de asociación de todo el genoma sobre el nivel educativo en 1,1 millones de individuos". Nature Genetics . 50 (8): 1112–1121. doi :10.1038/s41588-018-0147-3. PMC 6393768 . PMID 30038396.
- ^ Okbay A, Wu Y, Wang N, Jayashankar H, Bennett M, Nehzati SM, et al. (abril de 2022). "Predicción poligénica del nivel educativo dentro y entre familias a partir de análisis de asociación de todo el genoma en 3 millones de individuos". Nature Genetics . 54 (4): 437–449. doi :10.1038/s41588-022-01016-z. hdl : 11368/3026010 . PMC 9005349 . PMID 35361970.
- ^ Jansen PR, Watanabe K, Stringer S, Skene N, Bryois J, Hammerschlag AR, et al. (marzo de 2019). "El análisis de todo el genoma del insomnio en 1.331.010 individuos identifica nuevos loci de riesgo y vías funcionales". Nature Genetics . 51 (3): 394–403. bioRxiv 10.1101/214973 . doi : 10.1038/s41588-018-0333-3 . hdl : 1871.1/08af5d9e-8621-41f1-97c5-e77a1063495f . PMID 30804565.
- ^ abc Kathiresan S, Willer CJ, Peloso GM, Demissie S, Musunuru K, Schadt EE, et al. (Enero de 2009). "Las variantes comunes en 30 loci contribuyen a la dislipidemia poligénica". Genética de la Naturaleza . 41 (1): 56–65. doi :10.1038/ng.291. PMC 2881676 . PMID 19060906.
- ^ Strawbridge RJ, Dupuis J , Prokopenko I, Barker A, Ahlqvist E, Rybin D, et al. (octubre de 2011). "La asociación de todo el genoma identifica nueve variantes comunes asociadas con los niveles de proinsulina en ayunas y proporciona nuevos conocimientos sobre la fisiopatología de la diabetes tipo 2". Diabetes . 60 (10): 2624–34. doi :10.2337/db11-0415. PMC 3178302 . PMID 21873549.
- ^ Danesh J, Pepys MB (noviembre de 2009). "Proteína C reactiva y enfermedad coronaria: ¿existe un vínculo causal?". Circulation . 120 (21): 2036–9. doi : 10.1161/CIRCULATIONAHA.109.907212 . PMID 19901186.
- ^ Liu JZ, Erlich Y, Pickrell JK (marzo de 2017). "Mapeo de asociaciones de casos y controles por proxy utilizando antecedentes familiares de enfermedades". Nature Genetics . 49 (3): 325–331. doi :10.1038/ng.3766. PMID 28092683. S2CID 5598845.
- ^ Ku CS, Loy EY, Pawitan Y, Chia KS (abril de 2010). "La búsqueda de estudios de asociación de todo el genoma: ¿dónde estamos ahora?". Journal of Human Genetics . 55 (4): 195–206. doi : 10.1038/jhg.2010.19 . PMID 20300123.
- ^ ab Maher B (noviembre de 2008). "Genomas personales: el caso de la heredabilidad faltante". Nature . 456 (7218): 18–21. doi : 10.1038/456018a . PMID 18987709.
- ^ Iadonato SP, Katze MG (septiembre de 2009). "Genómica: el virus de la hepatitis C se vuelve personal". Naturaleza . 461 (7262): 357–8. Código Bib :2009Natur.461..357I. doi : 10.1038/461357a . PMID 19759611. S2CID 7602652.

- ^ Muehlschlegel JD, Liu KY, Perry TE, Fox AA, Collard CD, Shernan SK, Body SC (septiembre de 2010). "La variante del cromosoma 9p21 predice la mortalidad después de una cirugía de bypass de la arteria coronaria". Circulation . 122 (11 Suppl): S60–5. doi :10.1161/CIRCULATIONAHA.109.924233. PMC 2943860 . PMID 20837927.
- ^ Paynter NP, Chasman DI, Paré G, Buring JE, Cook NR, Miletich JP, Ridker PM (febrero de 2010). "Asociación entre una puntuación de riesgo genético basada en la literatura y eventos cardiovasculares en mujeres". JAMA . 303 (7): 631–7. doi :10.1001/jama.2010.119. PMC 2845522 . PMID 20159871.
- ^ Couzin-Frankel J (junio de 2010). "Los genes de las enfermedades cardíacas importantes resultan esquivos". Science . 328 (5983): 1220–1. Bibcode :2010Sci...328.1220C. doi :10.1126/science.328.5983.1220. PMID 20522751.

- ^ Ge D, Fellay J, Thompson AJ, Simon JS, Shianna KV, Urban TJ, Heinzen EL, Qiu P, Bertelsen AH, Muir AJ, Sulkowski M, McHutchison JG, Goldstein DB (septiembre de 2009). "La variación genética en IL28B predice la eliminación viral inducida por el tratamiento de la hepatitis C". Nature . 461 (7262): 399–401. Bibcode :2009Natur.461..399G. doi :10.1038/nature08309. PMID 19684573. S2CID 1707096.
- ^ Thomas DL, Thio CL, Martin MP, Qi Y, Ge D, O'Huigin C, Kidd J, Kidd K, Khakoo SI, Alexander G, Goedert JJ, Kirk GD, Donfield SM, Rosen HR, Tobler LH, Busch MP, McHutchison JG, Goldstein DB, Carrington M (octubre de 2009). "Variación genética en IL28B y eliminación espontánea del virus de la hepatitis C". Nature . 461 (7265): 798–801. Bibcode :2009Natur.461..798T. doi :10.1038/nature08463. PMC 3172006 . PMID 19759533.
- ^ Lu YF, Goldstein DB, Angrist M, Cavalleri G (julio de 2014). "Medicina personalizada y diversidad genética humana". Cold Spring Harbor Perspectives in Medicine . 4 (9): a008581. doi :10.1101/cshperspect.a008581. PMC 4143101 . PMID 25059740.
- ^ Folkersen L, van't Hooft F, Chernogubova E, Agardh HE, Hansson GK, Hedin U, Liska J, Syvänen AC, Paulsson-Berne G, Paulssson-Berne G, Franco-Cereceda A, Hamsten A, Gabrielsen A, Eriksson P (agosto de 2010). "La asociación de variantes de riesgo genético con la expresión de genes proximales identifica nuevos genes de susceptibilidad a la enfermedad cardiovascular". Circulación: Genética Cardiovascular . 3 (4): 365–73. doi : 10.1161/CIRCGENÉTICA.110.948935 . PMID 20562444.
- ^ Bown MJ, Jones GT, Harrison SC, Wright BJ, Bumpstead S, Baas AF, et al. (noviembre de 2011). "El aneurisma aórtico abdominal está asociado con una variante en la proteína 1 relacionada con el receptor de lipoproteína de baja densidad". American Journal of Human Genetics . 89 (5): 619–27. doi :10.1016/j.ajhg.2011.10.002. PMC 3213391 . PMID 22055160.
- ^ Consorcio de Genética de la Enfermedad de la Arteria Coronaria (C4D) (marzo de 2011). "Un estudio de asociación de todo el genoma en europeos y asiáticos del sur identifica cinco nuevos loci para la enfermedad de la arteria coronaria". Nature Genetics . 43 (4): 339–44. doi :10.1038/ng.782. PMID 21378988. S2CID 39712343.
{{cite journal}}: CS1 maint: nombres numéricos: lista de autores ( enlace )[ enlace muerto permanente ]
- ^ Johnson T, Gaunt TR, Newhouse SJ, Padmanabhan S, Tomaszewski M, Kumari M, et al. (diciembre de 2011). "Loci de presión arterial identificados con una matriz centrada en genes". American Journal of Human Genetics . 89 (6): 688–700. doi :10.1016/j.ajhg.2011.10.013. PMC 3234370 . PMID 22100073.
- ^ Dubé JB, Johansen CT, Hegele RA (junio de 2011). "Sortilina: un sospechoso inusual en el metabolismo del colesterol: desde la identificación mediante GWAS hasta los análisis bioquímicos in vivo, la sortilina ha sido identificada como un nuevo mediador del metabolismo de las lipoproteínas humanas". BioEssays . 33 (6): 430–7. doi :10.1002/bies.201100003. PMID 21462369.

- ^ Bauer RC, Stylianou IM, Rader DJ (abril de 2011). "Validación funcional de nuevas vías en el metabolismo de lipoproteínas identificadas por genética humana". Current Opinion in Lipidology . 22 (2): 123–8. doi :10.1097/MOL.0b013e32834469b3. PMID 21311327. S2CID 24020035.

- ^ Roselli C, Chafin M, Weng L (2018). "Estudio de asociación de genoma multiétnico para la fibrilación auricular". Nature Genetics . 50 (9): 1225–1233. doi :10.1038/s41588-018-0133-9. PMC 6136836 . PMID 29892015.
- ^ Ganapathiraju MK, Thahir M, Handen A, Sarkar SN, Sweet RA, Nimgaonkar VL, Loscher CE, Bauer EM, Chaparala S (27 de abril de 2016). "Interactoma de la esquizofrenia con 504 nuevas interacciones proteína-proteína". npj Schizophrenia . 2 : 16012. doi :10.1038/npjschz.2016.12. PMC 4898894 . PMID 27336055.
- ^ "Nuevo estudio sobre esquizofrenia se centra en las interacciones proteína-proteína". psychcentral.com . 3 de mayo de 2016. Archivado desde el original el 11 de enero de 2020 . Consultado el 22 de abril de 2023 .
- ^ Ganapathiraju M, Chaparala S, Lo C (abril de 2018). "F200. Elucidación del papel de los cilios en las enfermedades neuropsiquiátricas mediante el análisis del interactoma". Boletín de esquizofrenia . 44 (suppl_1): S298-9. doi :10.1093/schbul/sby017.731. PMC 5887623 .
- ^ Johnson EC, Border R, Melroy-Greif WE, de Leeuw CA, Ehringer MA, Keller MC (noviembre de 2017). "No hay evidencia de que los genes candidatos a esquizofrenia estén más asociados a la esquizofrenia que los genes no candidatos". Psiquiatría biológica . 82 (10): 702–708. doi :10.1016/j.biopsych.2017.06.033. PMC 5643230 . PMID 28823710.
- ^ Willi Y, Kristensen TN, Sgrò CM, Weeks AR, Ørsted M, Hoffmann AA (enero de 2022). "La genética de la conservación como herramienta de gestión: los cinco paradigmas mejor respaldados para ayudar a la gestión de especies amenazadas". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 119 (1): e2105076119. Bibcode :2022PNAS..11905076W. doi : 10.1073/pnas.2105076119 . PMC 8740573 . PMID 34930821.
- ^ Turuspekov Y, Baibulatova A, Yermekbayev K, Tokhetova L, Chudinov V, Sereda G, et al. (noviembre de 2017). "GWAS para etapas de crecimiento de plantas y componentes de rendimiento en trigo de primavera (Triticum aestivum L.) cosechado en tres regiones de Kazajstán". BMC Plant Biology . 17 (Supl 1): 190. doi : 10.1186/s12870-017-1131-2 . PMC 5688510 . PMID 29143598.
- ^ Zhao K, Tung CW, Eizenga GC, Wright MH, Ali ML, Price AH, et al. (septiembre de 2011). "El mapeo de asociaciones de todo el genoma revela una rica arquitectura genética de rasgos complejos en Oryza sativa". Nature Communications . 2 (1): 467. Bibcode :2011NatCo...2..467Z. doi :10.1038/ncomms1467. PMC 3195253 . PMID 21915109.
- ^ Bartoli C, Roux F (2017). "Estudios de asociación de todo el genoma en patosistemas vegetales: hacia un enfoque de genómica ecológica". Frontiers in Plant Science . 8 : 763. doi : 10.3389/fpls.2017.00763 . PMC 5441063 . PMID 28588588.
- ^ Abasht B, Lamont SJ (octubre de 2007). "El análisis de asociación de todo el genoma revela que los alelos crípticos son un factor importante en la heterosis para la obesidad en la población de pollos F2". Animal Genetics . 38 (5): 491–498. doi :10.1111/j.1365-2052.2007.01642.x. PMID 17894563.
- ^ Sebastiani P, Solovieff N, Puca A, Hartley SW, Melista E, Andersen S, Dworkis DA, Wilk JB, Myers RH, Steinberg MH, Montano M, Baldwin CT, Perls TT (julio de 2010). "Firmas genéticas de longevidad excepcional en humanos". Science . 2010 (5987). doi :10.1126/science.1190532. PMID 20595579.(Retractado, véase doi :10.1126/science.333.6041.404-a, PMID 21778381, Retraction Watch)

- ^ MacArthur D (8 de julio de 2010). "Se revelan graves fallas en el estudio de los "genes de la longevidad"". Wired . Consultado el 7 de diciembre de 2011 .
- ^ Sebastiani P, Solovieff N, Puca A, Hartley SW, Melista E, Andersen S, Dworkis DA, Wilk JB, Myers RH, Steinberg MH, Montano M, Baldwin CT, Perls TT (julio de 2011). "Retractación". Science . 333 (6041): 404. doi :10.1126/science.333.6041.404-a. PMID 21778381.

- ^ Sebastiani P, Solovieff N, Dewan AT, Walsh KM, Puca A, Hartley SW, Melista E, Andersen S, Dworkis DA, Wilk JB, Myers RH, Steinberg MH, Montano M, Baldwin CT, Hoh J, Perls TT (18 enero de 2012). "Firmas genéticas de longevidad excepcional en humanos". MÁS UNO . 7 (1): e29848. Código Bib : 2012PLoSO...729848S. doi : 10.1371/journal.pone.0029848 . PMC 3261167 . PMID 22279548.
- ^ Tam V, Patel N, Turcotte M, Bossé Y, Paré G, Meyre D (mayo de 2019). "Beneficios y limitaciones de los estudios de asociación de todo el genoma". Nature Reviews Genetics . 20 (8): 467–484. doi :10.1038/s41576-019-0127-1. ISSN 1471-0056. PMID 31068683. S2CID 148570302.
- ^ Rosenberg NA, Huang L, Jewett EM, Szpiech ZA, Jankovic I, Boehnke M (mayo de 2010). "Estudios de asociación de todo el genoma en diversas poblaciones". Nature Reviews Genetics . 11 (5): 356–66. doi :10.1038/nrg2760. PMC 3079573 . PMID 20395969.
- ^ Sham PC, Cherny SS, Purcell S, Hewitt JK (mayo de 2000). "Poder del análisis de ligamiento versus asociación de rasgos cuantitativos, mediante el uso de modelos de componentes de varianza, para datos de hermandad". American Journal of Human Genetics . 66 (5): 1616–30. doi :10.1086/302891. PMC 1378020 . PMID 10762547.
- ^ Borecki IB (2006). "Estudios de asociación y vinculación". Enciclopedia de ciencias de la vida . John Wiley & Sons, Ltd. doi :10.1038/npg.els.0005483. ISBN 9780470015902.
{{cite book}}:|work=ignorado ( ayuda ) - ^ Visscher PM, Goddard ME , Derks EM, Wray NR (mayo de 2012). "Genética psiquiátrica basada en evidencia, también conocida como la falsa dicotomía entre hipótesis de variantes comunes y raras". Psiquiatría molecular . 17 (5): 474–85. doi : 10.1038/mp.2011.65 . PMID 21670730.

- ^ Border R, Athanasiadis G, Buil A, Schork AJ, Cai N, Young AI, Werge T, Flint J, Kendler KS, Sankararaman S, Dahl AW, Zaitlen NA (2022). "El apareamiento selectivo entre rasgos está muy extendido e infla las estimaciones de correlación genética". Science . 378 (6621): 754–761. Bibcode :2022Sci...378..754B. doi :10.1126/science.abo2059. ISSN 0036-8075. PMC 9901291 . PMID 36395242.
Enlaces externos
- Herramientas y bases de datos de interacción genotipo-fenotipo en omicX [ enlace muerto permanente ]
- Métodos estadísticos para el análisis de estudios de asociación de todo el genoma [serie de videoconferencias]
- Estudios de asociación del genoma completo — por el Instituto Nacional de Investigación del Genoma Humano
- GWAS Central: una base de datos central de hallazgos de asociaciones genéticas a nivel de resumen
- Barrett J (18 de julio de 2010). "Cómo leer un estudio de asociación de todo el genoma". Genomes Unzipped.
- Consorcios de estudios de asociación de todo el genoma (GWAS) Archivado el 26 de febrero de 2018 en Wayback Machine . — por Bennett SN, Caporaso, NE, et al.
- PLINK: conjunto de herramientas para el análisis de asociaciones del genoma completo
- Explorador de hilos ENCODE Impacto de la información funcional en la comprensión de la variación. Nature (revista)


