Filovirus
| Filovirus | |
|---|---|
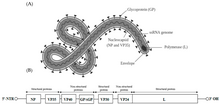 | |
| Estructura y genoma del virus del Ébola | |
 | |
| Micrografía electrónica del virus de Marburgo | |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Virus de la ortiga |
| Filo: | Negarnaviricota |
| Clase: | Monjiviricetes |
| Orden: | Mononegavirus |
| Familia: | Filovirus |
| Géneros | |
| |
Filoviridae ( / ˌfaɪloʊˈvɪrɪdiː / [1] ) es una familia de virus de ARN monocatenario de sentido negativo en el orden Mononegavirales . [ 2 ] Dos miembros de la familia que son comúnmente conocidos son el virus del Ébola y el virus de Marburgo . Ambos virus, y algunos de sus parientes menos conocidos, causan enfermedades graves en humanos y primates no humanos en forma de fiebres hemorrágicas virales . [3]
Todos los filovirus están clasificados por los EE. UU. como agentes selectos , [4] por la Organización Mundial de la Salud como patógenos del Grupo de riesgo 4 (que requieren contención equivalente al Nivel de bioseguridad 4 ), [5] por los Institutos Nacionales de Salud / Instituto Nacional de Alergias y Enfermedades Infecciosas como patógenos prioritarios de categoría A, [6] y por los Centros para el Control y la Prevención de Enfermedades como agentes de bioterrorismo de categoría A , [7] y están enumerados como agentes biológicos para el control de exportaciones por el Grupo de Australia . [8]
Uso del término
La familia Filoviridae es un taxón virológico que fue definido en 1982 [3] y enmendado en 1991, [9] 1998, [10] 2000, [11] 2005, [12] 2010 [13] y 2011. [14] La familia actualmente incluye los seis géneros de virus Cuevavirus , Dianlovirus , Ebolavirus , Marburgvirus , Striavirus y Thamnovirus y está incluida en el orden Mononegavirales . [13] Los miembros de la familia (es decir, las entidades físicas reales) se denominan filovirus o filoviridos. [13] El nombre Filoviridae se deriva del sustantivo latino filum (en alusión a la morfología filamentosa de los filoviriones) y el sufijo taxonómico -viridae (que denota una familia de virus). [3]
Nota
Según las reglas de denominación de taxones establecidas por el Comité Internacional de Taxonomía de Virus (ICTV) , el nombre Filoviridae siempre debe escribirse con mayúscula , cursiva , nunca abreviado y debe ir precedido de la palabra "familia". Los nombres de sus miembros (filovirus o filoviridos) deben escribirse con minúscula, no en cursiva y usarse sin artículos . [13] [14]
Ciclo vital


El ciclo de vida del filovirus comienza con la unión del virión a receptores específicos de la superficie celular , seguida de la fusión de la envoltura del virión con las membranas celulares y la liberación concomitante de la nucleocápside del virus en el citosol . La ARN polimerasa dependiente del ARN viral (RdRp, o ARN replicasa) desviste parcialmente la nucleocápside y transcribe los genes en ARNm de cadena positiva , que luego se traducen en proteínas estructurales y no estructurales . Las RdRp del filovirus se unen a un único promotor ubicado en el extremo 3' del genoma. La transcripción termina después de un gen o continúa hasta el siguiente gen aguas abajo. Esto significa que los genes cercanos al extremo 3' del genoma se transcriben en mayor abundancia, mientras que los que están hacia el extremo 5' tienen menos probabilidades de transcribirse. Por lo tanto, el orden de los genes es una forma simple pero efectiva de regulación transcripcional. La proteína más abundante producida es la nucleoproteína , cuya concentración en la célula determina cuándo la RdRp pasa de la transcripción génica a la replicación genómica. La replicación da como resultado antígenos de cadena positiva de longitud completa que a su vez se transcriben en copias genómicas de la progenie del virus de cadena negativa. Las proteínas estructurales y los genomas recién sintetizados se autoensamblan y se acumulan cerca del interior de la membrana celular . Los viriones se desprenden de la célula y obtienen sus envolturas de la membrana celular de la que brotan. Las partículas de la progenie madura luego infectan otras células para repetir el ciclo. [12]
Criterios de inclusión familiar

Un virus que cumple los criterios para ser miembro del orden Mononegavirales es miembro de la familia Filoviridae si: [13] [14]
- Provoca fiebre hemorrágica viral en ciertos primates.
- Infecta a primates , cerdos o murciélagos en la naturaleza.
- Es necesario adaptarlo a través de pasajes en serie para causar enfermedades en roedores.
- Se replica exclusivamente en el citoplasma de una célula huésped .
- Tiene un genoma de ≈19 kbp de longitud.
- Tiene un genoma de ARN que constituye aproximadamente el 1,1% de la masa del virión.
- Su genoma tiene un peso molecular de ≈4,2 × 106
- Su genoma contiene uno o más genes superpuestos.
- Su genoma contiene siete genes en el orden 3'-UTR - NP - VP35 - VP40 - GP - VP30 - VP24 - L - 5'-UTR
- Su gen VP24 no es homólogo a los genes de otros mononegavirus.
- Su genoma contiene señales de inicio y terminación de la transcripción que no se encuentran en los genomas de otros mononegavirus.
- Forma nucleocápsides con una densidad flotante en CsCl de ≈1,32 g/cm 3
- Forma nucleocápsides con un canal axial central (≈10–15 nm de ancho) rodeado por una capa oscura (≈20 nm de ancho) y una capa helicoidal externa (≈50 nm de ancho) con una estría cruzada (periodicidad de ≈5 nm)
- Expresa una glicoproteína de fusión de clase I que está altamente N- y O - glicosilada y acilada en su cola citoplasmática.
- Expresa una proteína de matriz primaria que no está glicosilada.
- Forma viriones que brotan de la membrana plasmática.
- Forma viriones que son predominantemente filamentosos (en forma de U y de 6) y que tienen un ancho de aproximadamente 80 nm y una longitud de varios cientos de nm y hasta 14 μm.
- Forma viriones que tienen proyecciones superficiales de ≈7 nm de longitud espaciadas ≈10 nm entre sí
- Forma viriones con una masa molecular de ≈3,82 × 108 ; una S 20W de al menos 1,40; y una densidad de flotabilidad en tartrato de potasio de ≈1,14 g/cm 3
- Forma viriones que se neutralizan mal in vivo
Organización familiar
| Nombre del género | Nombre de la especie | Nombre del virus (abreviatura) |
|---|---|---|
| Cuevavirus | Cuevavirus de Lloviu | Virus Llovizna (LLOV) |
| Dianlovirus | Dialovirus de Mengla | Virus de la influenza (MLAV) |
| Virus del Ébola | Virus del ébola de Bombali | Virus Bombali (BOMV) |
| Virus del ébola Bundibugyo | Virus Bundibugyo (BDBV; anteriormente BEBOV) | |
| Virus del ébola en Reston | Virus Reston (RESTV; anteriormente REBOV) | |
| Virus del Ébola en Sudán | Virus de Sudán (SUDV; anteriormente SEBOV) | |
| Virus del ébola en el bosque de Tai | Virus del bosque de Tai (TAFV; anteriormente CIEBOV) | |
| Virus del Ébola en Zaire | Virus del Ébola (EBOV; anteriormente ZEBOV) | |
| Virus de Marburgo | Virus de Marburgo (Marburgvirus) | Virus de Marburgo (MARV) |
| Virus Ravn (RAVV) | ||
| Estriavirus | Estriavirus de Xilang | Virus Xilang (XILV) |
| Tamnovirus | Tamnovirus de Huangjiao | Virus Huangjiao (HUJV) |
Filogenética
Se ha estimado que las tasas de mutación en estos genomas están entre 0,46 × 10 −4 y 8,21 × 10 −4 sustituciones de nucleótidos/sitio/año. [15] Se estimó que el ancestro común más reciente de las variantes secuenciadas del filovirus fue 1971 (1960-1976) para el virus del Ébola, 1970 (1948-1987) para el virus de Reston y 1969 (1956-1976) para el virus de Sudán, y se estimó que el ancestro común más reciente entre las cuatro especies incluidas en el análisis (virus del Ébola, virus del bosque Tai, virus de Sudán y virus de Reston) data de hace 1000-2100 años. [16] El ancestro común más reciente de las especies de Marburgo y Sudán parece haber evolucionado 700 y 850 años antes del presente, respectivamente. Aunque los relojes mutacionales ubicaron el tiempo de divergencia de los filovirus existentes en ~10.000 años antes del presente, la datación de elementos endógenos ortólogos (paleovirus) en los genomas de hámsteres y topillos indicó que los géneros existentes de filoviridos tenían un ancestro común al menos tan antiguo como el Mioceno (hace unos 16–23 millones de años). [17]
El cladograma de Filoviridae es el siguiente: [18] [19]
| Filovirus |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Paleovirología
Se conocen elementos paleovirales de cada uno de los cuatro clados divergentes principales de filovirus. Si bien los elementos ortólogos en los genomas de mamíferos respaldan una edad mínima para los filovirus de decenas de millones de años, la existencia de filovirus y sus elementos en linajes divergentes de peces sugiere que la familia de virus tiene cientos de millones de años. [20] Se han identificado paleovirus que parecen derivar de virus similares a filovirus en los genomas de muchas especies de cuerpo pequeño, incluidos murciélagos , roedores , musarañas , tenrecs , tarseros , marsupiales [21] [22] [23] y peces. [24] Aunque la mayoría de los elementos similares a filovirus parecen ser pseudogenes , los análisis evolutivos y estructurales sugieren que los ortólogos aislados de varias especies del género de murciélagos Myotis y la familia de roedores Spalacidae se han mantenido por selección. [25] [26]
Vacunas
En la actualidad, existen vacunas muy limitadas contra los filovirus conocidos. [27] Una vacuna eficaz contra el virus del Ébola, desarrollada en Canadá, [28] fue aprobada para su uso en 2019 en los EE. UU. y Europa. [29] [30] De manera similar, se están realizando esfuerzos para desarrollar una vacuna contra el virus de Marburgo. [31]
Preocupaciones por las mutaciones y el potencial pandémico
Existe una preocupación acuciante por la posibilidad de que una mutación genética muy leve en un filovirus como el EBOV pudiera provocar un cambio en el sistema de transmisión, pasando de la transmisión directa por fluidos corporales a la transmisión aérea, como se observó en el virus Reston (otro miembro del género Ebolavirus) entre macacos infectados. Un cambio similar en las cepas circulantes actuales del EBOV podría aumentar considerablemente las tasas de infección y enfermedad causadas por el EBOV. Sin embargo, no hay registros de que ninguna cepa del Ébola haya realizado esta transición en humanos. [32]
El Centro Nacional de Análisis y Contramedidas de Biodefensa del Departamento de Seguridad Nacional considera que el riesgo de que en el futuro surja una cepa mutada del virus del Ébola con capacidad de transmisión por aerosoles es una amenaza grave para la seguridad nacional y ha colaborado con los Centros para el Control y la Prevención de Enfermedades (CDC) para diseñar métodos para detectar aerosoles del EBOV. [33]
Referencias
- ^ "Filoviridae". Diccionario Merriam-Webster.com . Merriam-Webster . Consultado el 28 de julio de 2018 .
- ^ Kuhn, JH; Amarasinghe, GK; Basler, CF; Bávari, S; Bukreyev, A; Chandran, K; Báculo, yo; Dolnik, O; Tinte, JM; Formenty, PBH; Griffiths, A; Hewson, R; Kobinger, médico de cabecera ; Leroy, EM; Mühlberger, E; Netesov, SV; Palacios, G; Palyi, B; Pawęska, JT; Smither, SJ; Takada, A; Towner, JS; Wahl, V; Informe ICTV, Consorcio (junio 2019). "Perfil de taxonomía de virus ICTV: Filoviridae". La Revista de Virología General . 100 (6): 911–912. doi : 10.1099/jgv.0.001252 . PMC 7011696 . Número de modelo: PMID31021739.
- ^ abc Kiley MP, Bowen ET, Eddy GA, Isaäcson M, Johnson KM, McCormick JB, Murphy FA, Pattyn SR, Peters D, Prozesky OW, Regnery RL, Simpson DI, Slenczka W, Sureau P, van der Groen G, Webb PA, Wulff H (1982). "Filoviridae: ¿un hogar taxonómico para los virus de Marburg y Ébola?". Intervirología . 18 (1–2): 24–32. doi : 10.1159/000149300 . PMID 7118520.
- ^ Servicio de Inspección Sanitaria de Animales y Plantas de los Estados Unidos (APHIS) y Centros para el Control y la Prevención de Enfermedades de los Estados Unidos (CDC). "Registro Nacional de Agentes Selectos (NSAR)" . Consultado el 16 de octubre de 2011 .
- ^ Departamento de Salud y Servicios Humanos de los Estados Unidos. "Bioseguridad en laboratorios microbiológicos y biomédicos (BMBL), quinta edición" . Consultado el 16 de octubre de 2011 .
- ^ Institutos Nacionales de Salud de Estados Unidos (NIH), Instituto Nacional de Alergias y Enfermedades Infecciosas de Estados Unidos (NIAID). «Biodefense — Patógenos prioritarios de categorías A, B y C del NIAID». Archivado desde el original el 22 de octubre de 2011. Consultado el 16 de octubre de 2011 .
- ^ Centros para el Control y la Prevención de Enfermedades de Estados Unidos (CDC). «Bioterrorism Agents/Diseases». Archivado desde el original el 22 de julio de 2014. Consultado el 16 de octubre de 2011 .
- ^ The Australia Group. «Lista de agentes biológicos para el control de las exportaciones». Archivado desde el original el 6 de agosto de 2011. Consultado el 16 de octubre de 2011 .
- ^ McCormick, JB (1991). "Familia Filoviridae". En Francki, RIB; Fauquet, CM; Knudson, DL; et al. (eds.). Clasificación y nomenclatura de virus: quinto informe del Comité Internacional de Taxonomía de Virus. Suplemento de Archives of Virology . Vol. 2. Viena, Austria: Springer. págs. 247–49. ISBN. 0-387-82286-0.
- ^ Jahrling, PB; Kiley, MP; Klenk, H.-D.; Peters, CJ; Sanchez, A.; Swanepoel, R. (1995). "Familia Filoviridae". En Murphy, FA; Fauquet, CM; Bishop, DHL; Ghabrial, SA; Jarvis, AW; Martelli, GP; Mayo, MA; Summers, MD (eds.). Taxonomía de virus: sexto informe del Comité Internacional de Taxonomía de Virus. Suplemento de Archives of Virology . Vol. 10. Viena, Austria: Springer. págs. 289–92. ISBN. 3-211-82594-0.
- ^ Netesov, SV; Feldmann, H.; Jahrling, PB; Klenk, HD; Sanchez, A. (2000). "Familia Filoviridae". En van Regenmortel, MHV; Fauquet, CM; Bishop, DHL; Carstens, EB; Estes, MK; Lemon, SM; Maniloff, J.; Mayo, MA; McGeoch, DJ; Pringle, CR; Wickner, RB (eds.). Taxonomía de virus: séptimo informe del Comité Internacional de Taxonomía de Virus . San Diego, EE. UU.: Academic Press. págs. 539–48. ISBN 0-12-370200-3.
- ^ ab Feldmann, H.; Geisbert, T. W.; Jahrling, PB; Klenk, H.-D.; Netesov, S. V.; Peters, C. J.; Sanchez, A.; Swanepoel, R.; Volchkov, VE (2005). "Familia Filoviridae". En Fauquet, C. M.; Mayo, MA; Maniloff, J.; Desselberger, U.; Ball, LA (eds.). Taxonomía de virus: octavo informe del Comité Internacional de Taxonomía de Virus . San Diego, EE. UU.: Elsevier/Academic Press. págs. 645–653. ISBN 0-12-370200-3.
- ^ abcde Kuhn JH, Becker S, Ebihara H, Geisbert TW, Johnson KM, Kawaoka Y, Lipkin WI, Negredo AI, Netesov SV, Nichol ST, Palacios G, Peters CJ, Tenorio A, Volchkov VE, Jahrling PB (2010). "Propuesta de taxonomía revisada de la familia Filoviridae: clasificación, nombres de taxones y virus, y abreviaturas de virus". Archivos de Virología . 155 (12): 2083–2103. doi :10.1007/s00705-010-0814-x. PMC 3074192 . PMID 21046175.
- ^ abc Kuhn, JH; Becker, S.; Ebihara, H.; Geisbert, TW; Jahrling, PB; Kawaoka, Y.; Netesov, SV; Nichol, ST; Peters, CJ; Volchkov, VE; Ksiazek, TG (2011). "Familia Filoviridae". En King, Andrew MQ; Adams, Michael J.; Carstens, Eric B.; et al. (eds.). Taxonomía de virus: noveno informe del Comité Internacional de Taxonomía de Virus . Londres, Reino Unido: Elsevier/Academic Press. págs. 665–671. ISBN 978-0-12-384684-6.
- ^ Carroll SA, Towner JS, Sealy TK, McMullan LK, Khristova ML, Burt FJ, Swanepoel R, Rollin PE, Nichol ST (marzo de 2013). "Evolución molecular de los virus de la familia Filoviridae basada en 97 secuencias de genoma completo". J. Virol . 87 (5): 2608–16. doi :10.1128/JVI.03118-12. PMC 3571414 . PMID 23255795.
- ^ Li YH, Chen SP (2014). "Historia evolutiva del virus del Ébola" (PDF) . Epidemiol. Infect . 142 (6): 1138–1145. doi :10.1017/S0950268813002215. PMC 9151191 . PMID 24040779. S2CID 9873900.
- ^ Taylor, DJ; Ballinger, MJ; Zhan, JJ; Hanzly, LE; Bruenn, JA (2014). "Evidencia de que los virus del Ébola y los cuevavirus se han ido diferenciando de los virus de Marburgo desde el Mioceno". PeerJ . 2 : e556. doi : 10.7717/peerj.556 . PMC 4157239 . PMID 25237605.
- ^ Melanie M. Hierweger, Michel C. Koch, Melanie Rupp, Piet Maes, Nicholas Di Paola, Rémy Bruggmann, Jens H. Kuhn, Heike Schmidt-Posthaus y Torsten Seuberlich (22 de noviembre de 2021). "Nuevos filovirus, hantavirus y rabdovirus en peces de agua dulce, Suiza, 2017". Enfermedades Infecciosas Emergentes . 27 (12): 3082–3091. doi : 10.3201/eid2712.210491 . PMC 8632185 . PMID 34808081.
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ) - ^ Biao He, Tingsong Hu, Xiaomin Yan, Fuqiang Zhang, Changchun Tu (7 de agosto de 2023). "Detección y caracterización de un nuevo filovirus de murciélago (virus Dehong, DEHV) en murciélagos frugívoros". bioRxiv . doi : 10.1101/2023.08.07.552227 .
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ) - ^ Taylor, Derek J.; Barnhart, Max H. (2024). "Las transferencias genómicas ayudan a descifrar la antigua evolución de los filovirus y las interacciones con los hospedadores vertebrados". PLOS Pathogens . 20 (9): e1011864. doi : 10.1371/journal.ppat.1011864 . PMC 11398700 . PMID 39226335.
- ^ Taylor DJ, Leach RW, Bruenn J (2010). "Los filovirus son antiguos y están integrados en los genomas de los mamíferos". BMC Evolutionary Biology . 10 (1): 193. Bibcode :2010BMCEE..10..193T. doi : 10.1186/1471-2148-10-193 . PMC 2906475 . PMID 20569424.
- ^ Belyi VA, Levine AJ, Skalka AM (2010). Buchmeier (ed.). "Herencia inesperada: integraciones múltiples de secuencias antiguas de bornavirus y ébolavirus/marburgvirus en genomas de vertebrados". PLOS Pathogens . 6 (7): e1001030. doi : 10.1371/journal.ppat.1001030 . PMC 2912400 . PMID 20686665.
- ^ Katzourakis A, Gifford RJ (2010). "Elementos virales endógenos en genomas animales". PLOS Genetics . 6 (11): e1001191. doi : 10.1371/journal.pgen.1001191 . PMC 2987831 . PMID 21124940.
- ^ Taylor, Derek J.; Barnhart, Max H. (2024). "Las transferencias genómicas ayudan a descifrar la antigua evolución de los filovirus y las interacciones con los hospedadores vertebrados". PLOS Pathogens . 20 (9): e1011864. doi : 10.1371/journal.ppat.1011864 . PMC 11398700 . PMID 39226335.
- ^ Taylor DJ, Dittmar K, Ballinger MJ, Bruenn JA (2011). "Mantenimiento evolutivo de genes similares a filovirus en genomas de murciélagos". BMC Evolutionary Biology . 11 (336): 336. Bibcode :2011BMCEE..11..336T. doi : 10.1186/1471-2148-11-336 . PMC 3229293 . PMID 22093762.
- ^ Taylor, Derek J.; Barnhart, Max H. (2024). "Las transferencias genómicas ayudan a descifrar la antigua evolución de los filovirus y las interacciones con los hospedadores vertebrados". PLOS Pathogens . 20 (9): e1011864. doi : 10.1371/journal.ppat.1011864 . PMC 11398700 . PMID 39226335.
- ^ Peters CJ, LeDuc JW (febrero de 1999). "Introducción al ébola: el virus y la enfermedad". The Journal of Infectious Diseases . 179 (suplemento 1): ix–xvi. doi : 10.1086/514322 . JSTOR 30117592. PMID 9988154.
- ^ Plummer, Francis A.; Jones, Steven M. (30 de octubre de 2017). "La historia de la vacuna canadiense contra el ébola". CMAJ: Revista de la Asociación Médica Canadiense . 189 (43): E1326–E1327. doi :10.1503/cmaj.170704. ISSN 0820-3946. PMC 5662448 . PMID 29084758.
- ^ Investigación, Centro de Evaluación de Productos Biológicos y (2020-01-27). "ERVEBO". FDA .
- ^ CZARSKA-THORLEY, Dagmara (16 de octubre de 2019). «Ervebo». Agencia Europea de Medicamentos . Consultado el 3 de mayo de 2020 .
- ^ Keshwara, Rohan; Hagen, Katie R.; Abreu-Mota, Tiago; Papaneri, Amy B.; Liu, David; Wirblich, Christoph; Johnson, Reed F.; Schnell, Matthias J. (5 de marzo de 2019). "Un virus de la rabia recombinante que expresa la glicoproteína del virus de Marburgo depende de la citotoxicidad celular mediada por anticuerpos para la protección contra la enfermedad del virus de Marburgo en un modelo murino". Revista de Virología . 93 (6). doi :10.1128/JVI.01865-18. ISSN 0022-538X. PMC 6401435 . PMID 30567978.
- ^ Kelland, Kate (19 de septiembre de 2014). "Los científicos consideran remoto el riesgo de transmisión aérea de virus mutantes del ébola". Reuters . Consultado el 10 de octubre de 2014 .
- ^ "Artículo destacado: Nueva tecnología permite detectar el virus del ébola transmitido por el aire". Departamento de Seguridad Nacional . 20 de abril de 2021 . Consultado el 13 de diciembre de 2021 .
Lectura adicional
- Klenk, Hans-Dieter (1999). Virus de Marburgo y Ébola. Temas actuales en microbiología e inmunología . Vol. 235. Berlín, Alemania: Springer-Verlag. ISBN 978-3-540-64729-4.
- Klenk, Hans-Dieter; Feldmann, Heinz (2004). Virus del Ébola y de Marburgo: biología molecular y celular . Wymondham, Norfolk, Reino Unido: Horizon Bioscience. ISBN 978-0-9545232-3-7.
- Kuhn, Jens H. (2008). Filovirus: un compendio de 40 años de estudios epidemiológicos, clínicos y de laboratorio. Suplemento de Archives of Virology . Vol. 20. Viena, Austria: Springer. ISBN 978-3-211-20670-6.
- Ryabchikova, Elena I.; Price, Barbara B. (2004). Virus del Ébola y Marburgo: una visión de la infección mediante microscopía electrónica . Columbus, Ohio, EE. UU.: Battelle Press. ISBN 978-1-57477-131-2.
Enlaces externos
- Informe del ICTV: Filoviridae
- "Filoviridae". Navegador de taxonomía del NCBI . 11266.
- "FILOVIR". Recursos científicos para la investigación sobre filovirus. Archivado desde el original el 2020-07-30 . Consultado el 2014-08-08 .
- Evidencia teórica de que la cepa Zaire del virus del Ébola puede ser dependiente del selenio: ¿un factor en la patogénesis y los brotes virales? Taylor 1995
- ¿Puede la selenita ser un inhibidor definitivo del ébola y otras infecciones virales? Lipinski 2015 Archivado el 19 de noviembre de 2020 en Wayback Machine
- Muchos habitantes de África occidental podrían ser inmunes al virus del Ébola (The New York Times)

