Haplogrupo L0
| Haplogrupo L0 | |
|---|---|
| Posible época de origen | 130 a 200 ka [1] [2] |
| Posible lugar de origen | África meridional o África sudoriental |
| Antepasado | L ( Eva mitocondrial ) |
| Descendientes | Que le jodan, señor |
| Definición de mutaciones | 263!, 1048, 3516A, 5442, 6185, 9042, 9347, 10589, 12007, 12720 [3] |
El haplogrupo L0 es un haplogrupo de ADN mitocondrial (ADNmt) humano .
Origen

L0 es una de las dos ramas del ancestro común más reciente (MRCA) del linaje materno humano compartido. El haplogrupo consta de cinco ramas principales (L0a, L0b, L0d, L0f, L0k). Cuatro de ellas se clasificaron originalmente en subclados L1, L1a, L1d, L1f y L1k.
En 2014, un análisis de ADN antiguo de un esqueleto de un cazador-recolector masculino de 2330 años de antigüedad en el sur de África determinó que el espécimen pertenecía al subclado de ADNmt L0d2c1c. Este haplogrupo materno está hoy más estrechamente asociado con los ju, un subgrupo del pueblo indígena san , lo que indica una continuidad de la población en la región. [4] En 2016, también se descubrió que una momia desecada de la Edad del Hierro tardía de la región de Tuli en el norte de Botsuana pertenecía al haplogrupo L0. [5]
Distribución

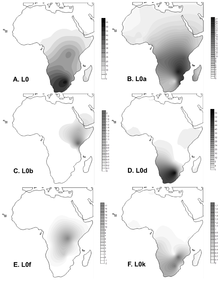
La L0 se encuentra con mayor frecuencia en el África subsahariana . Alcanza su frecuencia más alta en el pueblo Khoisan con un 73% en promedio. [6] Algunas de las frecuencias más altas son: [7] Namibia ( !Xun ) 79%, Sudáfrica ( Khwe /!Xun ) 83% y Botswana ( !Kung ) 100%.
El haplogrupo L0d se encuentra entre los grupos khoisan del sur de África más cerca del lado khoid y (siguiendo a L0k) es más sanid pero está restringido en gran medida a los khoisan en su conjunto. [7] [8] [9] [10] L0d también se encuentra comúnmente en sectores de la población de color de Sudáfrica y las frecuencias varían del 60% [11] al 71%. [10] Esto ilustra la contribución materna masiva de las personas khoisan a sectores de la población de color de Sudáfrica.
El haplogrupo L0k es el segundo haplogrupo más común en los grupos khoisan más cercanos al lado sanid y (después de L0d) es más khoid pero está restringido en gran medida a los khoisan en su conjunto. [7] [8] [9] [10] Aunque el haplogrupo L0d asociado a los khoisan se encontró en altas frecuencias en secciones de la población de color de Sudáfrica, L0k no se observó en dos estudios que involucraron grandes grupos de individuos de color. [10] [11]
El haplogrupo L0f está presente en frecuencias relativamente pequeñas en Tanzania , África Oriental, entre el pueblo Sandawe de Tanzania.
El haplogrupo L0a es más frecuente en las poblaciones del sudeste de África (25% en Mozambique ). [6] Entre los guineanos , tiene una frecuencia de entre el 1% y el 5%, y el grupo Balanta muestra una frecuencia aumentada de alrededor del 11%. El haplogrupo L0a tiene una profundidad de tiempo paleolítico de unos 33.000 años y probablemente llegó a Guinea hace entre 10.000 y 4.000 años. También se ve a menudo en los pigmeos Mbuti y Biaka . L0a se encuentra con una frecuencia de casi el 25% en Hadramawt ( Yemen ). [12]
El haplogrupo L0b se encuentra en Etiopía .
Interacciones entre fármacos y enfermedades
En pacientes a quienes se les administra el medicamento estavudina para tratar el VIH , el haplogrupo L0a2 se asocia con una mayor probabilidad de neuropatía periférica como efecto secundario. [13]
Subclados
Árbol
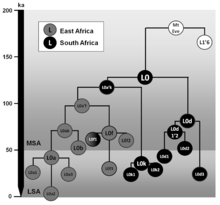
Este árbol filogenético de los subclados del haplogrupo L0 se basa en el artículo de Mannis van Oven y Manfred Kayser Árbol filogenético completo actualizado de la variación global del ADN mitocondrial humano [3] y en investigaciones publicadas posteriormente.
- Ancestro común más reciente (ARM)
- L0
- L0d
- L0d3
- L0d1'2
- L0d1
- L0d1a
- L0d1b
- L0d1c
- L0d1c1
- L0d2
- L0d2a'b
- L0d2a
- L0d2a1
- L0d2b
- L0d2a
- L0d2c
- L0d2c1c [14]
- L0d2a'b
- L0d1
- L0a'b'joder
- Mirar
- Mirar1
- L0k2
- L0a'b'f
- L0f
- L0f1
- L0f2
- L0f2a
- L0f2b
- L0a'b
- L0a
- L0a1
- L0a1a
- L0a1a2
- L0a1b
- L0a1b1
- L0a1b1a
- L0a1b2
- L0a1b1
- L0a1c
- Cargado
- L0a1a
- L0a2
- L0a2a
- L0a2a1
- L0a2a1a
- L0a2a1a1
- L0a2a1a2
- L0a2a1a
- L0a2a2
- L0a2a2a
- L0a2a1
- L0a2b
- L0a2ba
- L0a2c
- L0a2d
- L0a2a
- L0a3
- L0a4
- L0a1
- L0b
- L0a
- L0f
- Mirar
- L0d
- L0
Véase también
- Prueba de ADN genealógica
- Genealogía genética
- Genética mitocondrial humana
- Genética de poblaciones
- Haplogrupos del ADN mitocondrial humano
Referencias
- ^ Estimación puntual de 168,5 ka ( IC del 95 % de 136,3 a 201,1 ka ) según Heinz, Tanja; et al. (2017). "Actualización del árbol de ADN mitocondrial humano africano: relevancia para la genética forense y de poblaciones". Forensic Science International: Genetics . 27 : 156–159. doi :10.1016/j.fsigen.2016.12.016. PMID 28086175.(tabla 2). 150 ka sugerido en: Soares, Pedro; Ermini, Luca; Thomson, Noel; Mormina, Maru; Rito, Teresa; Röhl, Arne; Salas, Antonio; Oppenheimer, Stephen; MacAulay, Vincent (2009). "Corrección para la selección purificadora: un reloj molecular mitocondrial humano mejorado". The American Journal of Human Genetics . 84 (6): 740–59. doi :10.1016/j.ajhg.2009.05.001. PMC 2694979 . PMID 19500773. .
- ^ Estimaciones de edad (ka, IC del 95 % entre corchetes angulares): estimación de edad de todo el ADNmt de ML: 128,2 [IC del 95 %: 107,9-148,9], estimación de edad de todo el ADNmt de ρ: 121,3 [99,2; 143,7], estimación de edad sinónima de ρ (ka): 131,0 [97,8; 164,2]: Rito T, Richards MB, Fernandes V, Alshamali F, Cerny V, Pereira L, Soares P., "Las primeras dispersiones humanas modernas en África", PLoS One 2013 Nov 13; 8(11):e80031. doi: 10.1371/journal.pone.0080031.
- ^ ab Van Oven, Mannis; Kayser, Manfred (2009). "Árbol filogenético completo actualizado de la variación global del ADN mitocondrial humano". Human Mutation . 30 (2): E386–94. doi : 10.1002/humu.20921 . PMID 18853457. S2CID 27566749.
- ^ Alan G. Morris; Anja Heinze; Eva KF Chan; Andrew B. Smith; Vanessa M. Hayes (2014). "Primer genoma humano mitocondrial antiguo de un africano meridional prepastoralista". Genome Biology and Evolution . 6 (10): 2647–53. doi :10.1093/gbe/evu202. PMC 4224329 . PMID 25212860.
- ^ Frank J. Rühli; Maryna Steyn; Morongwa N. Mosothwane; Lena Öhrström; Molebogeng K. Bodiba; Abigail Bouwman (enero-febrero de 2016). «Análisis radiológico y genético de una momia de la Edad del Hierro Tardía del Bloque Tuli, Botsuana» (PDF) . Revista sudafricana de ciencias . 112 (1/2). Archivado desde el original (PDF) el 21 de junio de 2016 . Consultado el 26 de abril de 2016 .
- ^ ab Rosa, Alexandra; Brehm, Antonio; Kivisild, Toomas; Metspalu, Ene; Villems, Richard (2004). "Perfil MtDNA de los guineanos de África occidental: hacia una mejor comprensión de la región de Senegambia". Anales de genética humana . 68 (4): 340–52. doi :10.1046/j.1529-8817.2004.00100.x. hdl : 10400.13/3044 . PMID 15225159. S2CID 15391342.
- ^ abc Tishkoff, SA; Gonder, MK; Henn, BM; Mortensen, H.; Knight, A.; Gignoux, C.; Fernandopulle, N.; Lema, G.; Nyambo, TB (2007). "Historia de las poblaciones de habla chasquida de África inferida a partir de la variación genética del ADNmt y del cromosoma Y". Biología molecular y evolución . 24 (10): 2180–95. doi : 10.1093/molbev/msm155 . PMID 17656633.
- ^ ab Chen, Yu-Sheng; Olckers, Antonel; Schurr, Theodore G.; Kogelnik, Andreas M.; Huoponen, Kirsi; Wallace, Douglas C. (2000). "Variación del ADNmt en los Kung y Khwe sudafricanos y sus relaciones genéticas con otras poblaciones africanas". La Revista Estadounidense de Genética Humana . 66 (4): 1362–83. doi :10.1086/302848. PMC 1288201 . PMID 10739760.
- ^ ab Knight, Alec; Underhill, Peter A.; Mortensen, Holly M.; Zhivotovsky, Lev A.; Lin, Alice A.; Henn, Brenna M.; Louis, Dorothy; Ruhlen, Merritt; Mountain, Joanna L. (2003). "La divergencia del cromosoma Y africano y el ADNmt proporciona información sobre la historia de los lenguajes de clic". Current Biology . 13 (6): 464–73. doi : 10.1016/S0960-9822(03)00130-1 . PMID 12646128. S2CID 52862939.
- ^ abcd Schlebusch, Carina M.; Naidoo, Thijessen; Soodyall, Himla (2009). "Minisecuenciación de SNaPshot para resolver macrohaplogrupos mitocondriales encontrados en África". Electroforesis . 30 (21): 3657–64. doi :10.1002/elps.200900197. PMID 19810027. S2CID 19515426.
- ^ ab Quintana-Murci, Lluis; Harmant, Christine; Quach, Hélène; Balanovsky, Oleg; Zaporozhchenko, Valery; Bormans, Connie; Van Helden, Paul D.; Hoal, Eileen G.; Behar, Doron M. (2010). "Fuerte contribución materna khoisan a la población sudafricana de color: un caso de mezcla con sesgo de género". The American Journal of Human Genetics . 86 (4): 611–20. doi :10.1016/j.ajhg.2010.02.014. PMC 2850426 . PMID 20346436.
- ^ Rídl, Jakub; Edens, Christopher M.; Černý, Viktor (2009). "Estructura del ADN mitocondrial de la población yemení: diferencias regionales e implicaciones para diferentes contribuciones migratorias". La evolución de las poblaciones humanas en Arabia . Paleobiología y paleoantropología de vertebrados. págs. 69–78. doi :10.1007/978-90-481-2719-1_5. ISBN 978-90-481-2718-4.
- ^ Kampira E, Kumwenda J, van Oosterhout JJ, Dandara C. Los subhaplogrupos L0a2 y L2a del ADN mitocondrial modifican la susceptibilidad a la neuropatía periférica en adultos de Malawi que reciben terapia antirretroviral de gran actividad que contiene estavudina., J Acquir Immune Defic Syndr. 15 de agosto de 2013; 63(5):647-52. doi: 10.1097/QAI.0b013e3182968ea5
- ^ Primer genoma humano mitocondrial antiguo de un africano meridional prepastoral
Enlaces externos
- General
- Sitio de ADN mitocondrial de Ian Logan
- El árbol filogenético de Mannis van Oven
- Haplogrupo L0
- Haplogrupo L0 de YFull MTree
- Haplogrupo L0 de MITOMAP
- Rosa, Alexandra; Brehm, Antonio; Kivisild, Toomas; Metspalu, Ene; Villems, Richard (2004). "Perfil de ADNmt de guineanos de África occidental: hacia una mejor comprensión de la región de Senegambia". Anales de genética humana . 68 (4): 340–52. doi :10.1046/j.1529-8817.2004.00100.x. hdl : 10400.13/3044 . PMID 15225159. S2CID 15391342.
Con base en el conocimiento previo de secuencias completas africanas, el clado parafilético L1 se divide en dos unidades monofiléticas L0, que captura los linajes L1a y L1d previamente definidos, y el clado L1 que incluye los clados L1b y L1c…
- Pavesi, A. (2005). "Utilidad del poliomavirus JC en el rastreo del patrón de migraciones humanas que datan de tiempos prehistóricos". Journal of General Virology . 86 (5): 1315–26. doi : 10.1099/vir.0.80650-0 . PMID 15831942.
El primer eje del ADNmt, por otra parte, coloca al haplogrupo ancestral L0 en el extremo izquierdo, ya que dio origen a un único linaje…
- Mishmar, Dan; Ruiz-Pesini, Eduardo; Brandon, Martin; Wallace, Douglas C. (2004). "Secuencias similares al ADN mitocondrial en el núcleo (NUMTs): perspectivas sobre nuestros orígenes africanos y el mecanismo de integración del ADN extraño". Human Mutation . 23 (2): 125–33. doi : 10.1002/humu.10304 . PMID 14722916. S2CID 25109836.
haplogrupo L0 de mtADN, el haplogrupo que habíamos concluido previamente que se encuentra en la base del mtADN humano. árbol basado en análisis filogenético…
- Knight, Alec; Underhill, Peter A.; Mortensen, Holly M.; Zhivotovsky, Lev A.; Lin, Alice A.; Henn, Brenna M.; Louis, Dorothy; Ruhlen, Merritt; Mountain, Joanna L. (2003). "La divergencia del cromosoma Y africano y el ADNmt proporciona información sobre la historia de los lenguajes de clic". Current Biology . 13 (6): 464–73. doi : 10.1016/S0960-9822(03)00130-1 . PMID 12646128. S2CID 52862939.
