EIF4A1
| EIF4A1 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificadores | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Alias | EIF4A1 , DDX2A, EIF-4A, EIF4A, eIF-4A-I, eIF4A-I, factor de iniciación de la traducción eucariótica 4A1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Identificaciones externas | OMIM : 602641; MGI : 95303; HomoloGene : 103998; Tarjetas genéticas : EIF4A1; OMA :EIF4A1 - ortólogos | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidatos | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
El factor de iniciación eucariota 4A-I (también conocido como eIF4A1 o DDX2A) es una proteína citosólica de 46 kDa que, en los seres humanos, está codificada por el gen EIF4A1 , que se encuentra en el cromosoma 17. [5] [6] [7] Es el miembro más frecuente de la familia eIF4A de helicasas de ARN dependientes de ATP , y desempeña un papel fundamental en el inicio de la traducción de proteínas eucariotas dependiente de capuchón como un componente del complejo de iniciación de la traducción eIF4F . [8] eIF4A1 desenrolla la estructura secundaria del ARN dentro del 5'-UTR del ARNm , un paso crítico necesario para el reclutamiento del complejo de preiniciación 43S y, por lo tanto, la traducción de proteínas en eucariotas . [8] Fue caracterizado por primera vez en 1982 por Grifo, et al. , quienes lo purificaron a partir de lisado de reticulocitos de conejo . [9]
Fondo
La regulación de la traducción de las transcripciones de ARNm en proteínas es una de las mejores formas en que una célula puede alterar su respuesta a su entorno, ya que los cambios en la transcripción de genes a menudo tardan considerablemente más en realizarse. La traducción de proteínas se puede dividir en cuatro fases: activación, iniciación, elongación y terminación. De estos pasos, la iniciación es uno sobre el que las células tienen el mayor control. Es el paso limitante de la velocidad de la síntesis de proteínas, controlado por una miríada de proteínas conocidas como factores de iniciación eucariotas o eIF. La abundancia relativa de estos factores o sus actividades individuales relativas brindan a las células eucariotas un amplio control sobre la velocidad de iniciación y, por lo tanto, la síntesis de proteínas. Los eIF están regulados por vías de señalización intracelular bien conocidas, como la vía PI3K/AKT/mTOR , sin embargo, otras capas bioquímicas de regulación, como la complejidad de la estructura secundaria del ARN en el 5'-UTR, se están volviendo evidentes con más investigaciones. [8]
La subfamilia eIF4A en mamíferos está formada por tres parálogos , eIF4A1, eIF4A2 y eIF4A3 . [10] eIF4A1 y eIF4A2 comparten un 90% de similitud de secuencia, y ambas son proteínas citoplasmáticas, mientras que eIF4A3 está localizado en el núcleo y comparte solo un 60% de homología . [10] Históricamente, eIF4A1 y eIF4A2 se consideraban intercambiables, debido a que esto se observó en experimentos in vitro , pero investigaciones posteriores han demostrado que eIF4A1 es más frecuente en células en división, mientras que eIF4A2 es más abundante en células que no se dividen, y además, evidencia más reciente sugiere que podrían tener roles funcionalmente distintos in vivo . [8] [10]
Estructura
eIF4A1 es un miembro de la familia DEAD box de helicasas de ARN. [11] Las helicasas de ARN son enzimas que utilizan la energía liberada de la hidrólisis de ATP para manipular la estructura secundaria del ARN, y la familia DEAD box es la familia más grande de helicasas de ARN. [11] El nombre "DEAD box" se refiere a la secuencia clave de aminoácidos DEAD en el motivo II de la helicasa que participa en la unión del trifosfato de nucleósido (en el caso de eIF4A1, ATP ). Otros motivos conservados , compartidos por todas las proteínas de la familia eIF4A, son los motivos Q, I, Ia, Ib, III, IV, V y VI. Los motivos Ia, Ib, IV y V se unen al ARN, los motivos I, II y III median la actividad de la ATPasa dependiente del ARN , y el motivo VI es necesario tanto para la unión del ARN como para la hidrólisis del ATP. [10]

La familia DEAD box se caracteriza por un núcleo de helicasa estructuralmente muy conservado que consta de dos dominios similares a RecA unidos por una región de bisagra flexible alrededor de la cual la proteína puede abrirse y cerrarse tras la hidrólisis del ATP. [13] [10] [14] La hendidura formada entre estos dos dominios forma el bolsillo de unión del ATP. [11] La molécula de ARN se une en sentido opuesto a este bolsillo de unión, extendiéndose a través de cada uno de los dominios. [11] Este núcleo está flanqueado por dominios auxiliares variables, que les confieren la función única de cada helicasa de ARN en parte al permitir la unión específica a proteínas accesorias. [11]
Función
eIF4A1 es una helicasa de ARN dependiente de ATP, [15] sin embargo, la naturaleza exacta de su dependencia del ATP para su función aún se debate. [10] Aunque después de la unión del ATP, la hidrólisis posterior induce cambios conformacionales en eIF4A1, se ha demostrado que otras helicasas de ARN DEAD-box poseen actividad helicasa en presencia de análogos no hidrolizables del ATP, lo que sugiere que la unión, y no la hidrólisis, es el elemento más importante en la regulación de la actividad. [10]
eIF4A1 es un componente del complejo de iniciación de la traducción eIF4F, junto con eIF4E , la proteína de unión de la tapa 5'-terminal , y eIF4G , la proteína de andamiaje que mantiene unidos a eIF4A y eIF4E. [10] El complejo eIF4F suele ir acompañado de las proteínas accesorias eIF4B y eIF4H , cualquiera de las cuales puede mejorar de forma diferencial la actividad de eIF4A1. Después de que el ARNm se transcribe a partir del ADN y se transloca al citoplasma, y la PABP citosólica se une a la cola de poli(A) del ARNm naciente, su tapa 5' se unirá a eIF4E y la PABP se unirá a eIF4G. [8] A continuación, eIF4A1 desenrollará la estructura secundaria del ARN de 5' a 3' a medida que el PIC 43S se recluta al complejo eIF4F. [8] El PIC 43S escaneará el ARNm desenrollado desde 5' hasta 3' también, hasta que alcance el codón de inicio AUG , momento en el que se reclutará la subunidad ribosomal 60S para comenzar el proceso de elongación. [8]

(B) Desenrollado de la estructura secundaria del ARNm por eIF4A1 y reclutamiento del PIC 43S.
(C) Subunidad ribosómica 40S escaneando el 5'-UTR del transcrito del ARNm en busca de un codón de inicio.
(D) Reclutamiento de la subunidad ribosómica 60S y comienzo de la elongación.
Regulación
La transcripción de eIF4A1 está impulsada por el factor de transcripción MYC . [8] Por sí sola, la actividad helicasa de eIF4A1 es pobre, sin embargo, esta característica impone una restricción práctica en eIF4A1, ya que la actividad helicasa no específica, "no intencionada", en la célula sería perjudicial para la función de ciertas estructuras de ARN endógenas y necesarias. [10] Su eficacia mejora considerablemente en presencia de eIF4B y eIF4H, socios de unión que modulan su actividad. Cuando eIF4B se une a eIF4A1, la actividad helicasa de eIF4A1 aumenta más de 100 veces, pero cuando eIF4H se une en su lugar, el aumento no es tan grande, lo que sugiere que diferentes concentraciones relativas de estas proteínas accesorias pueden conferir un nivel adicional de regulación de la eficiencia de eIF4A1. [10]
Por el contrario, la actividad de eIF4A1 se suprime cuando se une a PDCD4 , un supresor tumoral modulado por mTOR y miR-21 . [8] PCDC4 se localiza típicamente en el núcleo en células sanas, sin embargo, en condiciones cancerígenas, se transloca al núcleo y dos moléculas separadas de eIF4A1 se unirán a él, inhibiendo la capacidad de eIF4A1 de unirse al ARN al bloquear las moléculas en su conformación inactiva, evitando así la unión a eIF4G. [16] [11]
Papel en la enfermedad
Cáncer
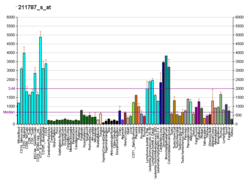
La desregulación de la traducción es un sello distintivo de la transformación maligna de las células cancerosas . Las células cancerosas en tumores en crecimiento se vuelven "adictas" a niveles elevados de traducción de proteínas, y particularmente dependientes de la traducción regulada al alza de ARNm prooncogénicos. Estos ARNm prooncogénicos tienen UTR 5' característicamente más largos con estructuras secundarias más complejas, y la regulación al alza de eIF4A1 se ha implicado en varios cánceres humanos (ver Tabla). [8] [17] [18] Dada la tendencia general de la sobreexpresión de eIF4A1 que impulsa el cáncer, existe interés en desarrollar inhibidores para la enzima. Se han identificado varios compuestos naturales como inhibidores candidatos para el desarrollo, aunque inhiben tanto a eIF4A1 como a eIF4A2 de forma no específica. [8] Estos incluyen hippuristanol , silvestrol y pateamina A, entre otros. [8] El silvestrol, en particular, es un derivado de rocaglato, y esta clase de compuestos podrían ser inhibidores viables de eIF4A. [19]
| Tipo de cáncer | Desregulación/asociación de eIF4A1 |
|---|---|
| Carcinoma hepatocelular | Sobreexpresión [17] |
| Melanoma | Sobreexpresión [17] |
| Carcinoma de pulmón de células no pequeñas (CPCNP) | Expresión asociada a metástasis [8] |
| Cáncer de endometrio | Sobreexpresión en hiperplasia atípica [8] |
| Cáncer de cuello uterino | Sobreexpresión; disminución de la expresión después de la braquiterapia asociada con un mejor resultado [8] |
| Cáncer de mama | Expresión asociada con malos resultados en la enfermedad con receptores de estrógeno negativos [8] |
Infecciones virales
Los virus dependen del secuestro de la maquinaria celular de las células que infectan para crear sus propias proteínas virales y permitirles seguir infectando nuevas células. Su capacidad para manipular eIFs como eIF4A1, por lo tanto, afecta considerablemente su virulencia . Por ejemplo, el citomegalovirus depende de eIF4A para impulsar su síntesis de proteínas. La proteína viral pUL69 estabiliza la formación de eIF4F, a través de la unión a eIF4A, un proceso por el cual se evita que eIF4E se disocie del complejo eIF4F. [14] eIF4E, por lo tanto, ya no puede ser secuestrado por su regulador negativo, 4EBP . [14] Además, el citomegalovirus estimula la síntesis de todos los elementos del complejo eIF4F para impulsar la síntesis de proteínas. [14] Otros virus, como el bracovirus Cotesia plutellae (CpBV), que favorecen la traducción independiente de la caperuza, aprovecharán el eIF4A1 en el contexto inverso, secuestrando el eIF4A1 del complejo eIF4F con socios de unión virales, en este caso una proteína llamada CpBV15β, inhibiendo así la traducción endógena del ARNm dependiente de la caperuza y favoreciendo la traducción de la proteína viral. [14] Los compuestos mencionados en la sección anterior sobre el cáncer, hippuristanol, silvestrol, pateamina A, derivados de rocaglato, etc., también podrían aplicarse como inhibidores virales putativos. [8] [19]
Referencias
- ^ abc GRCh38: Lanzamiento de Ensembl 89: ENSG00000161960 – Ensembl , mayo de 2017
- ^ abc GRCm38: Lanzamiento de Ensembl 89: ENSMUSG00000059796 – Ensembl , mayo de 2017
- ^ "Referencia de PubMed humana:". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ "Referencia de PubMed sobre ratón". Centro Nacional de Información Biotecnológica, Biblioteca Nacional de Medicina de EE. UU .
- ^ Kim NS, Kato T, Abe N, Kato S (abril de 1993). "Secuencia de nucleótidos del ADNc humano que codifica el factor de iniciación eucariota 4AI". Nucleic Acids Research . 21 (8): 2012. doi :10.1093/nar/21.8.2012. PMC 309447 . PMID 8493113.
- ^ Jones E, Quinn CM, See CG, Montgomery DS, Ford MJ, Kölble K, et al. (octubre de 1998). "Los genes del factor de iniciación de elongación humana 4A1 (EIF4A1) y CD68 se asignan al cromosoma 17p13". Genomics . 53 (2): 248–50. doi :10.1006/geno.1998.5515. PMID 9790779.
- ^ "Gen Entrez: factor de iniciación de la traducción eucariota EIF4A1 4A, isoforma 1".
- ^ abcdefghijklmnopq Raza F, Waldron JA, Quesne JL (diciembre de 2015). "Desregulación translacional en el cáncer: isoformas de eIF4A y determinantes de secuencia de la dependencia de eIF4A". Biochemical Society Transactions . 43 (6): 1227–33. doi :10.1042/BST20150163. PMID 26614665.
- ^ Grifo JA, Tahara SM, Leis JP, Morgan MA, Shatkin AJ, Merrick WC (mayo de 1982). "Caracterización del factor de iniciación eucariota 4A, una proteína implicada en la unión dependiente de ATP del ARNm de globina". The Journal of Biological Chemistry . 257 (9): 5246–52. doi : 10.1016/S0021-9258(18)34662-3 . PMID 7068683.
- ^ abcdefghijk Lu WT, Wilczynska A, Smith E, Bushell M (febrero de 2014). "Los diversos roles de la familia eIF4A: eres la compañía que tienes". Biochemical Society Transactions . 42 (1): 166–72. doi :10.1042/BST20130161. PMID 24450646.
- ^ abcdef Linder P, Jankowsky E (julio de 2011). "Del desenrollado al enganche: la familia de helicasas de ARN de caja DEAD". Nature Reviews. Molecular Cell Biology . 12 (8): 505–16. doi :10.1038/nrm3154. PMID 21779027. S2CID 2037710.
- ^ "EIF4A1 - Factor de iniciación eucariota 4A-I - Homo sapiens (humano) - Gen y proteína EIF4A1". www.uniprot.org .
- ^ Sharma D, Jankowsky E (20 de julio de 2014). "La subfamilia Ded1/DDX3 de helicasas de ARN de caja DEAD". Critical Reviews in Biochemistry and Molecular Biology . 49 (4): 343–60. doi :10.3109/10409238.2014.931339. PMID 25039764. S2CID 23470056.
- ^ abcde Montero, Hilda; Pérez-Gil, Gustavo; Sampieri, Clara L. (22 de febrero de 2019). "Factor de iniciación eucariota 4A (eIF4A) durante infecciones virales". Genes de virus . 55 (3): 267–273. doi :10.1007/s11262-019-01641-7. PMC 7088766 . PMID 30796742.
- ^ Shatsky IN, Dmitriev SE, Andreev DE, Terenin IM (1 de marzo de 2014). "Estudios de todo el transcriptoma revelan la diversidad de modos de reclutamiento de ARNm a ribosomas eucariotas". Critical Reviews in Biochemistry and Molecular Biology . 49 (2): 164–77. doi :10.3109/10409238.2014.887051. PMID 24520918. S2CID 207506515.
- ^ "PDCD4 muerte celular programada 4 [Homo sapiens (humano)] - Gen - NCBI". www.ncbi.nlm.nih.gov .
- ^ abc Ali MU, Ur Rahman MS, Jia Z, Jiang C (junio de 2017). "Factores de iniciación de la traducción eucariota y cáncer". Tumour Biology . 39 (6): 1010428317709805. doi : 10.1177/1010428317709805 . PMID 28653885.
- ^ Abdelhaleem M (julio de 2004). "¿Las helicasas de ARN humanas tienen un papel en el cáncer?". Biochimica et Biophysica Acta (BBA) - Reseñas sobre el cáncer . 1704 (1): 37–46. doi :10.1016/j.bbcan.2004.05.001. PMID 15238243.
- ^ ab Pan, Li; Woodard, John L.; Lucas, David M.; Fuchs, James R.; Kinghorn, A. Douglas (2 de mayo de 2014). "Rocaglamida, silvestrol y compuestos bioactivos estructuralmente relacionados de especies de Aglaia". Informes de productos naturales . 31 (7): 924–939. doi :10.1039/c4np00006d. PMC 4091845 . PMID 24788392.
Lectura adicional
- Reddy NS, Roth WW, Bragg PW, Wahba AJ (octubre de 1988). "Aislamiento y mapeo de un gen para el factor de iniciación de la síntesis de proteínas 4A y su expresión durante la diferenciación de células de eritroleucemia murina". Gene . 70 (2): 231–43. doi :10.1016/0378-1119(88)90195-3. PMID 3215517.
- Kukimoto I, Watanabe S, Taniguchi K, Ogata T, Yoshiike K, Kanda T (abril de 1997). "Caracterización del promotor clonado del gen del factor de iniciación humano 4AI". Comunicaciones de investigación bioquímica y biofísica . 233 (3): 844–7. doi :10.1006/bbrc.1997.6555. PMID 9168945.
- Imataka H, Sonenberg N (diciembre de 1997). "El factor de iniciación de la traducción eucariota humano 4G (eIF4G) posee dos sitios de unión separados e independientes para eIF4A". Biología molecular y celular . 17 (12): 6940–7. doi :10.1128/mcb.17.12.6940. PMC 232551 . PMID 9372926.
- Gradi A, Imataka H, Svitkin YV, Rom E, Raught B, Morino S, Sonenberg N (enero de 1998). "Un nuevo factor de iniciación de traducción eucariota humano funcional 4G". Biología Molecular y Celular . 18 (1): 334–42. doi :10.1128/mcb.18.1.334. PMC 121501 . PMID 9418880.
- Craig AW, Haghighat A, Yu AT, Sonenberg N (abril de 1998). "La interacción de la proteína de unión al poliadenilato con el homólogo de eIF4G PAIP mejora la traducción". Nature . 392 (6675): 520–3. Bibcode :1998Natur.392..520C. doi :10.1038/33198. PMID 9548260. S2CID 10891925.
- Henis-Korenblit S, Strumpf NL, Goldstaub D, Kimchi A (enero de 2000). "Una nueva forma de proteína DAP5 se acumula en células apoptóticas como resultado de la escisión de la caspasa y la traducción mediada por el sitio de entrada interno del ribosoma". Biología molecular y celular . 20 (2): 496–506. doi :10.1128/MCB.20.2.496-506.2000. PMC 85113 . PMID 10611228.
- Quinn CM, Wiles AP, El-Shanawany T, Catchpole I, Alnadaf T, Ford MJ, et al. (diciembre de 1999). "El gen del factor de iniciación eucariota humano 4AI (EIF4A1) contiene múltiples elementos reguladores que dirigen la expresión de genes reporteros de alto nivel en líneas celulares de mamíferos". Genomics . 62 (3): 468–76. doi :10.1006/geno.1999.6031. PMID 10644445.
- Cuesta R, Xi Q, Schneider RJ (julio de 2000). "Traducción específica de adenovirus por desplazamiento de la quinasa Mnk1 del complejo de iniciación de capuchón eIF4F". The EMBO Journal . 19 (13): 3465–74. doi :10.1093/emboj/19.13.3465. PMC 313943 . PMID 10880459.
- Mendell JT, Medghalchi SM, Lake RG, Noensie EN, Dietz HC (diciembre de 2000). "Nuevos ortólogos de Upf2p sugieren un vínculo funcional entre la iniciación de la traducción y los complejos de vigilancia sin sentido". Biología molecular y celular . 20 (23): 8944–57. doi :10.1128/MCB.20.23.8944-8957.2000. PMC 86549 . PMID 11073994.
- Li W, Belsham GJ, Proud CG (agosto de 2001). "Los factores de iniciación eucariotas 4A (eIF4A) y 4G (eIF4G) interactúan mutuamente en una proporción de 1:1 in vivo". The Journal of Biological Chemistry . 276 (31): 29111–5. doi : 10.1074/jbc.C100284200 . PMID 11408474.
- Du MX, Johnson RB, Sun XL, Staschke KA, Colacino J, Wang QM (abril de 2002). "Caracterización comparativa de dos helicasas de ARN de caja DEAD en la superfamilia II: factor de iniciación de la traducción humano 4A y proteína no estructural 3 (NS3) helicasa del virus de la hepatitis C". The Biochemical Journal . 363 (Pt 1): 147–55. doi :10.1042/0264-6021:3630147. PMC 1222461 . PMID 11903057.
- Bohnsack MT, Regener K, Schwappach B, Saffrich R, Paraskeva E, Hartmann E, Görlich D (noviembre de 2002). "Exp5 exporta eEF1A a través de ARNt desde el núcleo y se sinergiza con otras vías de transporte para confinar la traducción al citoplasma". The EMBO Journal . 21 (22): 6205–15. doi :10.1093/emboj/cdf613. PMC 137205 . PMID 12426392.
- Yang HS, Cho MH, Zakowicz H, Hegamyer G, Sonenberg N, Colburn NH (mayo de 2004). "Una nueva función de los dominios MA-3 en el supresor de la transformación y la traducción Pdcd4 es esencial para su unión al factor de iniciación de la traducción eucariota 4A". Biología molecular y celular . 24 (9): 3894–906. doi :10.1128/MCB.24.9.3894-3906.2004. PMC 387765 . PMID 15082783.
- Mingot JM, Bohnsack MT, Jäkle U, Görlich D (agosto de 2004). "Exportin 7 define una nueva vía general de exportación nuclear". The EMBO Journal . 23 (16): 3227–36. doi :10.1038/sj.emboj.7600338. PMC 514512 . PMID 15282546.
- Hinton TM, Coldwell MJ, Carpenter GA, Morley SJ, Pain VM (enero de 2007). "Análisis funcional de las actividades de unión individuales de la proteína de andamiaje eIF4G". The Journal of Biological Chemistry . 282 (3): 1695–708. doi : 10.1074/jbc.M602780200 . PMID 17130132.
- Ewing RM, Chu P, Elisma F, Li H, Taylor P, Climie S, et al. (2007). "Mapeo a gran escala de interacciones proteína-proteína humanas mediante espectrometría de masas". Biología de sistemas moleculares . 3 (1): 89. doi :10.1038/msb4100134. PMC 1847948 . PMID 17353931.
Enlaces externos
pUL69 del citomegalovirus (UniProt)
CpBV15β del bracovirus Cotesia plutellae (UniProt)