Virus del mosaico de la coliflor
| Virus del mosaico de la coliflor | |
|---|---|
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Riboviridae |
| Reino: | Pararnavirus |
| Filo: | Arteverviricota |
| Clase: | Revtraviricetes |
| Orden: | Virus ortopédicos |
| Familia: | Caulimovirus |
| Género: | Caulimovirus |
| Especies: | Virus del mosaico de la coliflor |
El virus del mosaico de la coliflor ( CaMV ) es un miembro del género Caulimovirus , uno de los seis géneros de la familia Caulimoviridae , que son pararetrovirus que infectan plantas . [1] Los pararetrovirus se replican a través de la transcripción inversa al igual que los retrovirus , pero las partículas virales contienen ADN en lugar de ARN . [2]
Definición

El virus del mosaico de la coliflor (CaMV) es un miembro de la familia Caulimoviridae . Esta familia está agrupada junto con Belpaoviridae , Metaviridae , Pseudoviridae y Retroviridae (todos los cuales tienen un genoma de ARN replicado a través de un intermediario de ADN) en el orden Ortervirales ; los Hepadnaviridae , a pesar de tener un genoma de ADN replicado a través de un intermediario de ARN (como los Caulimoviridae ), están más distantemente relacionados, y pertenecen al orden separado Blubervirales (ambos órdenes pertenecen a la misma clase, los Revtraviricetes ).
El CaMV infecta principalmente a plantas de la familia Brassicaceae (como la coliflor y el nabo), pero algunas cepas de CaMV (D4 y W260) también pueden infectar especies de Solanaceae de los géneros Datura y Nicotiana . El CaMV induce una variedad de síntomas sistémicos, como mosaico, lesiones necróticas en las superficies de las hojas, retraso del crecimiento y deformación de la estructura general de la planta. Los síntomas que se presentan varían según la cepa viral, el ecotipo del huésped y las condiciones ambientales. [3]
El CaMV se transmite de manera no circulatoria por especies de pulgones como Myzus persicae . [4] Una vez introducidos dentro de una célula huésped vegetal, los viriones migran a la envoltura nuclear de la célula vegetal.
Estructura
La partícula CaMV es un icosaedro con un diámetro de 52 nm construido a partir de 420 subunidades de proteína de la cápside (CP) dispuestas con una triangulación T = 7, que rodea una cavidad central llena de solvente. [5] [6]
El CaMV contiene una molécula de ADN bicatenario circular de aproximadamente 8,0 kilobases, interrumpida por cortes que resultan de las acciones de la ARNasa H durante la transcripción inversa. Estos cortes provienen del Met-ARNt y de dos cebadores de ARN utilizados en la transcripción inversa. Después de ingresar a la célula huésped, estos "cortes" monocatenarios en el ADN viral se reparan, formando una molécula superenrollada que se une a las histonas. Este ADN se transcribe en un ARN 35S de longitud completa, terminalmente redundante y un ARN 19S subgenómico.
Genoma
El promotor del ARN 35S es un promotor constitutivo muy fuerte responsable de la transcripción de todo el genoma de CaMV. Es bien conocido por su uso en la transformación de plantas . Provoca altos niveles de expresión génica en plantas dicotiledóneas. Sin embargo, es menos eficaz en monocotiledóneas, especialmente en cereales. Las diferencias en el comportamiento probablemente se deben a diferencias en la calidad y/o cantidad de factores reguladores. Un estudio reciente ha indicado que el promotor 35S de CaMV también es funcional en algunas células animales, aunque los elementos promotores utilizados son diferentes de los de las plantas. Si bien este promotor tuvo baja actividad en comparación con los promotores animales canónicos, los niveles de productos reporteros fueron significativos. Esta observación sugiere que el promotor 35S puede tener potencial para su uso en animales. [7]
El promotor se denominó promotor 35S de CaMV ("promotor 35S") porque el coeficiente de sedimentación del transcrito viral, cuya expresión está naturalmente impulsada por este promotor, es 35S. Es uno de los promotores constitutivos de uso general más utilizados. Fue descubierto a principios de la década de 1980 por Chua y colaboradores de la Universidad Rockefeller .
El ARN 35S es particularmente complejo y contiene una secuencia líder altamente estructurada de 600 nucleótidos de longitud con seis a ocho marcos de lectura abiertos (ORF) cortos. [8] [9] [10]
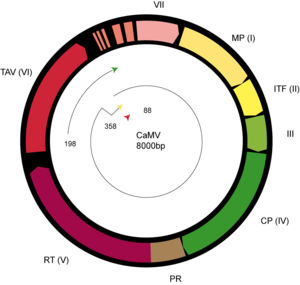
A este líder le siguen siete ORFs más largos y muy bien dispuestos que codifican todas las proteínas virales. El mecanismo de expresión de estas proteínas es único, ya que la proteína ORF VI (codificada por el ARN 19S) controla la reiniciación de la traducción de los principales marcos de lectura abiertos en el ARN policistrónico 35S, un proceso que normalmente solo ocurre en los ARNm bacterianos. La función de TAV depende de su asociación con polisomas y el factor de iniciación eucariota eIF3. [11]
- ORF I – P1: proteína de movimiento ( P03545 )
- ORF II – P2: factor de transmisión pulgón/insecto ( P03548 )
- ORF III – P3: proteína asociada al virión (VAP, P03551 ): proteína estructural, capacidad de unión al ADN
- ORF IV – P4: proteína de la cápside (CP, P03542 )
- ORF V – P5: pro-pol ( P03554 ): proteasa, transcriptasa inversa bifuncional y ARNasa H
- ORF VI – P6: transactivador/viroplasmina ( P03559 ): formación/tráfico de cuerpos de inclusión; posiblemente otras funciones (ver texto)
- ORF VII/VIII – desconocido (no parece ser necesario para la infección, Q83163 , Q83164 )
- Contiene un sitio de unión de ARNt -Met
Además de sus funciones relacionadas con la activación traduccional y la formación de cuerpos de inclusión , se ha demostrado que P6 interactúa con varias otras proteínas CaMV, como P2 y P3, lo que sugiere que también puede contribuir en algún grado al ensamblaje viral y la transmisión mediada por pulgones. Además, se ha demostrado que P6 se une a P7; investigar las interacciones entre los dos puede ayudar a dilucidar la función aún desconocida de P7. [12]
Otra función de P6 implica la modificación del NPR1 (NO EXPRESOR DE PATOGENIA RELACIONADA 1 ) del huésped durante el curso de la infección. NPR1 es un regulador importante de la señalización dependiente del ácido salicílico (SA) y del ácido jasmónico (JA), y está más estrechamente asociado con la comunicación cruzada entre los dos. La modificación de NPR1 sirve para inhibir las respuestas defensivas de las células vegetales al prevenir la señalización dependiente de SA; NPR1 modificado puede desplazarse adecuadamente al núcleo y unirse al promotor PR-1, pero no puede iniciar la transcripción. Debido a que NPR1 activo es necesario para la acumulación de SA, esto conduce a un mayor agotamiento de SA. Mientras que la regulación de la señalización dependiente de SA por NPR1 modificado por P6 se localiza en el núcleo, la regulación de la señalización dependiente de JA es de naturaleza citoplasmática e involucra la vía COI1. A diferencia de la de SA, la señalización dependiente de JA aumenta en presencia de NPR1 modificado. [13]
Replicación

CaMV se replica mediante transcripción inversa:
- Las partículas virales entran en la célula vegetal y no están encapsidadas. En esta etapa, el ADN viral consta de tres fragmentos, uno en la cadena – (α) y dos en la cadena + (β y γ), que están ensamblados de manera imperfecta en un genoma circular con tres huecos o discontinuidades (D1, D2 y D3).
- El ADN viral ingresa al núcleo donde se rellenan las discontinuidades. En este punto, el ADN viral también se asocia con las histonas del huésped , formando un minicromosoma (no se muestra).
- La ARN polimerasa dependiente del ADN del huésped transcribe desde el promotor 35S todo el genoma viral, superando al promotor 35S. (Esto crea dos copias del promotor 35S en el ARN resultante). La transcripción también se inicia en el promotor 19S (no se muestra).
- Los ARN virales pasan al citoplasma del huésped donde se transcriben.
- El extremo 3′ de un ARNt fMet se une a un sitio correspondiente a la discontinuidad 1 (D1) cerca del extremo 5′ del ARN 35S.
- El ARNt fMet prepara la síntesis, por parte de la transcriptasa inversa viral (codificada por ORF V), de una nueva cadena α.
- La ARNasa H elimina el ARN del dúplex ADN-ARN, dejando atrás el ADN.
- Este nuevo ADN se une al promotor 35S en el extremo 3' de la plantilla de ARN y la síntesis de la cadena α de ADN continúa y la ARNasa H continúa degradando el ARN complejado con el ADN.
- Se completa la síntesis de la cadena α. La actividad de la ARNasa H expone regiones ricas en purina en la posición de discontinuidad 3 (D3), lo que prepara la síntesis de la cadena γ de ADN.
- La actividad de la ARNasa H expone las regiones ricas en purina en la posición de la discontinuidad 2 (D2), que prepara la síntesis de la cadena β de ADN. Cuando la nueva cadena γ de ADN alcanza el extremo 5′ de la nueva cadena α, cambia al extremo 5′ de la nueva cadena α, recreando la discontinuidad 1 (D1). Cuando la nueva cadena γ de ADN alcanza el extremo 5′ de la nueva cadena β, desplaza el cebador y parte de la cadena β recién sintetizada, lo que da como resultado la recreación de la discontinuidad 2 (D2). Cuando la nueva cadena β de ADN alcanza el extremo 5′ de la nueva cadena γ, desplaza el cebador y parte de la cadena γ recién sintetizada, lo que da como resultado la recreación de la discontinuidad 3 (D3).
En este punto, el nuevo genoma viral puede empaquetarse en cápsides y liberarse de la célula o puede transportarse mediante proteínas de movimiento a una célula adyacente no infectada. [14]
El promotor del virus del mosaico de la coliflor (CaMV 35S) se utiliza en la mayoría de los cultivos transgénicos para activar genes extraños que se han insertado artificialmente en la planta huésped. Se inserta en las plantas transgénicas en una forma que es diferente de la que se encuentra cuando está presente en sus huéspedes naturales de la especie Brassica . Esto le permite operar en una amplia gama de entornos de organismos huéspedes que de otra manera no serían posibles.
El genoma del CaMV contiene un ADN de doble cadena de aproximadamente 8 kb y produce partículas esféricas. Las infecciones por CaMV son sistémicas, e incluso su ADN es infeccioso cuando se inocula en superficies de plantas desgastadas. El genoma del CaMV tiene 8 genes muy compactos, de los cuales solo dos genes pequeños, los genes II y VII, no son esenciales; como resultado, solo estos dos genes pueden reemplazarse o eliminarse sin una pérdida de infectividad. Además, los genomas modificados del CaMV que exceden el tamaño del genoma natural (8024 pb) incluso por unos pocos cientos de pb no se empaquetan en viriones. Estos dos factores limitan seriamente el tamaño del inserto de ADN clonable en el CaMV. El gen DHFR de la dihidrofolato reductasa bacteriana se ha clonado con éxito en el genoma del CaMV, en lugar del gen II, y se ha expresado con éxito en plantas.
Mecanismos moleculares de la transmisión del CaMV mediada por vectores
El virus se adquiere de un huésped infectado durante la alimentación del pulgón vector. Para que esto ocurra, se forma un complejo transmisible compuesto por viriones y proteína P2 ubicada en los estiletes del vector. El dominio N-terminal de P2 reconoce un receptor proteico ubicado en la punta del estilete y el dominio C-terminal de P2 se une a los viriones decorados con P3. [15]
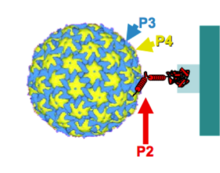
El modo de adquisición por el vector está controlado por la localización tisular e intracelular específica de P2. Esta proteína solo se encuentra en células de la epidermis y el parénquima. Además, en estas células, P2 se localiza en cuerpos de inclusión electrolúcidos virales individuales (ELIB). [16] En las células huésped, las proteínas virales P2 y P3 se producen primero en numerosas fábricas virales (cuerpos de inclusión densos en electrones), y luego se exportan y se co-localizan con microtúbulos, antes de concentrarse en ELIB. CaMV utiliza específicamente los microtúbulos para formar el cuerpo transmisible y así permitir la transmisión del vector. [17] La caracterización molecular completa y el estudio de este virus no se llevaron a cabo más.
Evasión de las defensas de las plantas
El virus del mosaico de la coliflor posee una serie de mecanismos que le permiten contrarrestar las defensas de las células de la planta huésped. Si bien el ARN 35S pregenómico es responsable de la replicación del genoma por la transcriptasa inversa, también contiene una secuencia líder no codificante de 600 pares de bases que sirve como un ARNm importante para la producción de factores involucrados en la contradefensa viral. Varios huéspedes del CaMV poseen pequeños mecanismos de silenciamiento viral basados en ARN que sirven para limitar la infección viral. Los productos de la secuencia de 600 pb antes mencionada son pequeños ARN virales (vsRNA) de 21, 22 y 24 nucleótidos de longitud que sirven como señuelos, uniéndose e inactivando los efectores de la maquinaria de silenciamiento del huésped, como Argonaute 1 ( AGO1 ). Como prueba de principio, la sobreexpresión experimental de estos vsRNA permite una mayor acumulación viral en plantas infectadas. [18]
Preocupaciones sobre el uso del promotor CaMV 35S en plantas transgénicas
A principios de la década de 2010, se han planteado algunas preocupaciones sobre el uso del promotor CaMV 35S para la expresión en plantas transgénicas debido a que existe una superposición de secuencias entre este promotor y las secuencias codificantes de P6. Cincuenta y cuatro eventos transgénicos certificados para su liberación en los EE. UU. contienen hasta 528 pb de ORF VI (que codifica los dominios C-terminales de P6). [19] Como P6 es una proteína multifuncional cuyo rango completo de funciones es desconocido, existe cierta preocupación de que la expresión de uno o más de sus dominios pueda tener consecuencias imprevistas en los organismos transgénicos. Estudios recientes han intentado determinar qué longitud del promotor CaMV 35S tiene la menor probabilidad de producir inadvertidamente dominios P6, mientras que aún conserva la actividad promotora completa. Como se podría esperar, el uso de longitudes de promotor más cortas disminuye la cantidad de dominios P6 incluidos y también disminuye la probabilidad de efectos no deseados. [19]
Referencias
- ^ Pringle, CR. (1999). "Taxonomía de virus--1999. El sistema universal de taxonomía de virus, actualizado para incluir las nuevas propuestas ratificadas por el Comité Internacional de Taxonomía de Virus durante 1998". Arch Virol . 144 (2): 421–9. doi :10.1007/s007050050515. PMC 7086988 . PMID 10470265.
- ^ Rothnie, HM.; Chapdelaine, Y.; Hohn, T. (1994). Pararetrovirus y retrovirus: una revisión comparativa de la estructura viral y las estrategias de expresión génica . Avances en la investigación de virus. Vol. 44. págs. 1–67. doi :10.1016/s0065-3527(08)60327-9. ISBN 9780120398447. Número de identificación personal 7817872.
{{cite book}}:|journal=ignorado ( ayuda ) - ^ Khelifa, M.; Massé, D.; Blanc, S.; Drucker, M. (enero de 2010). "Evaluación del tiempo mínimo de replicación del virus del mosaico de la coliflor en diferentes hospedadores". Virología . 396 (2): 238–45. doi : 10.1016/j.virol.2009.09.032 . PMID 19913268.
- ^ Brault, V.; Uzest, M.; Monsión, B.; Jacquot, E.; Blanc, S. (2010). "Los pulgones como medios de transporte de virus vegetales". Comptes Rendus Biologías . 333 (6–7): 524–38. doi :10.1016/j.crvi.2010.04.001. PMID 20541164.
- ^ Cheng, RH.; Olson, NH.; Baker, TS. (febrero de 1992). "Virus del mosaico de la coliflor: una subunidad 420 (T = 7), estructura multicapa". Virology . 186 (2): 655–68. doi :10.1016/0042-6822(92)90032-k. PMC 4167691 . PMID 1733107.
- ^ Haas, M.; Bureau, M.; Geldreich, A.; Yot, P.; Keller, M. (noviembre de 2002). "Virus del mosaico de la coliflor: todavía en las noticias". Mol Plant Pathol . 3 (6): 419–29. doi : 10.1046/j.1364-3703.2002.00136.x . PMID 20569349.
{{cite journal}}:|last2=tiene nombre genérico ( ayuda ) - ^ Tepfer, M.; Gaubert, S.; Leroux-Coyau, M.; Prince, S.; Houdebine, LM. (2004). "Expresión transitoria en células de mamíferos de transgenes transcritos a partir del promotor 35S del virus del mosaico de la coliflor" (PDF) . Environ Biosafety Res . 3 (2): 91–7. doi : 10.1051/ebr:2004010 . PMID 15612506.
- ^ Fütterer, J.; Gordon, K.; Bonneville, JM.; Sanfaçon, H.; Pisan, B.; Penswick, J.; Hohn, T. (septiembre de 1988). "La secuencia principal del ARN grande del caulimovirus se puede plegar en una gran estructura de tallo-bucle". Nucleic Acids Res . 16 (17): 8377–90. doi :10.1093/nar/16.17.8377. PMC 338565 . PMID 3419922.
- ^ Pooggin, MM.; Hohn, T.; Fütterer, J. (mayo de 1998). "La evolución forzada revela la importancia del marco de lectura abierto corto A y la estructura secundaria en el ARN líder 35S del virus del mosaico de la coliflor". J Virol . 72 (5): 4157–69. doi :10.1128/JVI.72.5.4157-4169.1998. PMC 109645 . PMID 9557705.
- ^ Hemmings-Mieszczak, M.; Steger, G.; Hohn, T. (abril de 1997). "Estructuras alternativas del ARN líder 35 S del virus del mosaico de la coliflor: implicaciones para la expresión y replicación viral". J Mol Biol . 267 (5): 1075–88. doi :10.1006/jmbi.1997.0929. PMID 9150397.
- ^ Park, HS.; Himmelbach, A.; Browning, KS.; Hohn, T.; Ryabova, LA. (septiembre de 2001). "Un factor de reiniciación viral de plantas interactúa con la maquinaria de traducción del huésped". Cell . 106 (6): 723–33. doi : 10.1016/S0092-8674(01)00487-1 . PMID 11572778. S2CID 14384952.
- ^ Lutz, L.; Raikhy, G.; Leisner, SM. (diciembre de 2012). "La proteína del cuerpo de inclusión principal del virus del mosaico de la coliflor interactúa con el factor de transmisión del pulgón, la proteína asociada al virión y el producto del gen VII". Virus Res . 170 (1–2): 150–3. doi :10.1016/j.virusres.2012.08.017. PMC 4215633 . PMID 22982205.
- ^ Love, AJ.; Geri, C.; Laird, J.; Carr, C.; Yun, BW.; Loake, GJ.; Tada, Y.; Sadanandom, A.; Milner, JJ. (2012). "La proteína P6 del virus del mosaico de la coliflor inhibe las respuestas de señalización al ácido salicílico y regula la inmunidad innata". PLOS ONE . 7 (10): e47535. Bibcode :2012PLoSO...747535L. doi : 10.1371/journal.pone.0047535 . PMC 3469532 . PMID 23071821.
- ^ Laliberté, JF.; Sanfaçon, H. (2010). "Remodelación celular durante la infección por virus en plantas". Annu Rev Phytopathol . 48 : 69–91. doi :10.1146/annurev-phyto-073009-114239. PMID 20337516.
- ^ Hoh, F.; Uzest, M.; Drucker, M.; Plisson-Chastang, C.; Bron, P.; Blanc, S.; Dumas, C. (mayo de 2010). "Información estructural sobre los mecanismos moleculares de la transmisión del virus del mosaico de la coliflor por su insecto vector". J Virol . 84 (9): 4706–13. doi :10.1128/JVI.02662-09. PMC 2863735 . PMID 20181714.
- ^ Martinière, A.; Zancarini, A.; Drucker, M. (junio de 2009). "Transmisión del virus del mosaico de la coliflor por pulgones: el papel de la planta huésped". Plant Signal Behav . 4 (6): 548–50. doi :10.4161/psb.4.6.8712. PMC 2688309 . PMID 19816139.
- ^ Martinière, A.; Gargani, D.; Uzest, M.; Lautredou, N.; Blanc, S.; Drucker, M. (abril de 2009). "Un papel de los microtúbulos de las plantas en la formación de cuerpos de inclusión específicos de transmisión del virus del mosaico de la coliflor". Plant J. 58 ( 1): 135–46. doi :10.1111/j.1365-313X.2008.03768.x. PMC 2688309. PMID 19077170 .
- ^ Blevins, T.; Rajeswaran, R.; Aregger, M.; Borah, BK.; Schepetilnikov, M.; Baerlocher, L.; Farinelli, L.; Meins, F.; et al. (julio de 2011). "Producción masiva de ARN pequeños a partir de una región no codificante del virus del mosaico de la coliflor en la defensa de las plantas y la contradefensa viral". Nucleic Acids Res . 39 (12): 5003–14. doi :10.1093/nar/gkr119. PMC 3130284 . PMID 21378120.
- ^ ab Podevin, N.; du Jardin, P. (2012). "Posibles consecuencias de la superposición entre las regiones promotoras de CaMV 35S en los vectores de transformación de plantas utilizados y el gen viral VI en plantas transgénicas". GM Crops Food . 3 (4): 296–300. doi : 10.4161/gmcr.21406 . PMID 22892689.
Enlaces externos
- Virus del mosaico de la coliflor
- "El promotor CaMV 35S". patentlens.net . Archivado desde el original el 7 de enero de 2008.