Barril TIM
| Barril TIM de tipo aldolasa | |
|---|---|
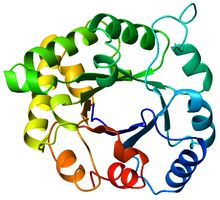 Vista superior de un barril de triosa fosfato isomerasa (TIM) ( PDB : 8TIM ), coloreado de azul ( extremo N ) a rojo (extremo C). | |
| Identificadores | |
| Símbolo | Aldolasa_TIM |
| Clan Pfam | CL0036 |
| ECOD | 2002.1.1 |
| Interprofesional | IPR013785 |
| CATÉTERO | 8tim |
| SCOP2 | 8tim / ALCANCE / SUPFAM |
El barril TIM (triosa-fosfato isomerasa), también conocido como barril alfa/beta , [1] : 252 es un pliegue proteico conservado que consta de ocho hélices alfa (α-hélices) y ocho cadenas beta paralelas (β-cadenas) que se alternan a lo largo de la cadena principal del péptido . [2] La estructura recibe su nombre de la triosa-fosfato isomerasa , una enzima metabólica conservada . [3] Los barriles TIM son omnipresentes, y aproximadamente el 10% de todas las enzimas adoptan este pliegue. [4] Además, cinco de las siete clases de enzimas de la comisión enzimática (EC) incluyen proteínas de barril TIM. [5] [6] El pliegue de barril TIM es evolutivamente antiguo , y muchos de sus miembros poseen poca similitud en la actualidad, [7] en cambio, caen dentro de la zona crepuscular de similitud de secuencia . [8] [9]
El barril beta interno (barril β) en muchos casos está estabilizado por intrincadas redes de puentes salinos . [10] Los bucles en los extremos C-terminales del barril β son responsables de la actividad catalítica [11] [12] mientras que los bucles del extremo N-terminal son importantes para la estabilidad de los barriles TIM. Se pueden insertar inserciones estructurales que van desde bucles extendidos hasta dominios proteicos independientes en lugar de estos bucles o en los extremos N-terminal/C-terminal. Los barriles TIM parecen haber evolucionado a través de eventos de duplicación genética y fusión de dominios de proteínas de medio barril, [13] con una mayoría de barriles TIM originándose a partir de un ancestro común . Esto llevó a que muchos barriles TIM poseyeran simetrías internas. [14] Otros eventos de duplicación genética de este barril TIM ancestral llevaron a enzimas divergentes que poseen la diversidad funcional observada hoy. Los barriles TIM también han sido un objetivo de larga data para los diseñadores de proteínas . Los diseños de barriles TIM exitosos incluyen fusiones de dominios de proteínas existentes y diseños de novo . Los experimentos de fusión de dominios han dado como resultado muchos diseños exitosos, [15] [16] [17] [18] [19] [ 20 ] [21] mientras que los diseños de novo solo dieron resultados exitosos después de 28 años de desarrollo incremental. [22]
Estructura


Topología
El barril TIM recibe su nombre de la enzima triosa-fosfato isomerasa (TIM), que fue la primera proteína que poseía el pliegue para ser cristalizada . [3] Los barriles TIM contienen 200-250 residuos de aminoácidos, [2] plegados en 8 hélices alfa (hélices α) y 8 cadenas beta (cadenas β). Las cadenas β están dispuestas en un barril beta paralelo (barril β), y están rodeadas por las 8 hélices α. La propiedad definitoria de los barriles β TIM es que siempre poseen un número de cizallamiento de 8. [2] El número de cizallamiento se determina eligiendo un residuo x en la cadena β-1, y moviéndose a lo largo del barril β, en una dirección perpendicular a la dirección de las cadenas, hasta que se alcanza el residuo y en la cadena β-1 original. El número de residuos entre las posiciones inicial y final (|y−x|) es el número de cizallamiento. [24] Dado que el número de hebras es igual al número de cizallamiento, las cadenas laterales apuntan alternativamente hacia el poro y el núcleo, lo que da una simetría cuádruple. Las hélices α rodean y encierran por completo el barril β interno. Los bucles cortos suelen conectar las estructuras secundarias α y β, formando una topología de repetición (βα) 8. En algunos casos, se pueden insertar estructuras que van desde bucles extendidos hasta dominios independientes en lugar de estos bucles, o se pueden unir a los terminales N/C. Todas las enzimas de barril TIM poseen sitios catalíticos en el extremo C-terminal del barril β, [25] y las inserciones estructurales presentes cerca de este extremo pueden ayudar en la actividad catalítica.
Regiones del núcleo y de los poros
Los barriles TIM contienen dos regiones enterradas distintas , donde los residuos de aminoácidos están completamente envueltos por sus vecinos y carecen de acceso al solvente. El término "poro" es un nombre inapropiado, ya que no existen canales de solvente dentro de esta región. La región central consta de todos los residuos que constituyen la interfaz α-β y se encuentra fuera del barril β central. La región del poro consta de todos los residuos del barril β interior, que están rodeados y encerrados por la estructura principal del barril β.
Debido a la naturaleza plisada de las cadenas β, los residuos alternos a lo largo de una cadena se dividen casi uniformemente entre el poro (53%) y el núcleo (47%). En el caso de los barriles β, el 95% de los residuos de su núcleo están enterrados. Solo el 11% de sus residuos de núcleo son polares , poseen afinidad por el agua y poseen la capacidad de formar enlaces de hidrógeno o puentes salinos. [10] De manera similar, el 84% de los residuos de los poros de las cadenas β están enterrados. Sin embargo, el 42% de sus residuos de poros son polares. Estos residuos forman intrincadas redes de puentes salinos para compensar su falta de accesibilidad al solvente.
Elementos estabilizadores del cañón TIM

Se cree que los puentes salinos dentro de los poros del barril TIM contribuyen a la estabilidad general del pliegue. Un ejemplo de una gran red de puentes salinos se puede encontrar en la 2-desoxirribosa-5-fosfato aldolasa . Se descubrió que esta red se conservaba en toda la familia de aldolasas de clase I.
La razón exacta de la sobrerrepresentación de residuos polares y puentes salinos dentro del poro sigue sin estar clara. Un estudio propone que mejoran la plegabilidad en lugar de la estabilidad termodinámica de los barriles TIM. Durante el proceso de plegamiento , los residuos del poro interno en las cadenas β estarían expuestos al agua. Los módulos βαβα parcialmente plegados, llamados foldones, se estabilizarían energéticamente mediante residuos de poros polares durante esta etapa de plegamiento.
En otro estudio que involucraba a la proteína barril TIM de la sintetasa de fosfato de indol-3-glicerol de S. solfataricus , se descubrió que un módulo βαβαβ conservado era una plantilla de plegamiento esencial, que guiaba el plegamiento de otras estructuras secundarias. El cierre del barril β solo se produjo al final del proceso de plegamiento. En este caso, sin embargo, los autores atribuyeron la estabilidad del foldón a los aminoácidos alifáticos ramificados (valina, leucina e isoleucina).
Otro elemento estabilizador en los barriles TIM es la pinza de horquilla beta . Los donantes de enlaces de hidrógeno de la cadena lateral en los extremos N de las cadenas β pares a menudo forman enlaces de hidrógeno con los hidrógenos de amida de la cadena principal en las cadenas β impares precedentes. Estas pinzas (o análogos de puentes de cadena lateral hidrofóbicos) se conservan en 3 ortólogos del barril TIM de la indol-3-glicerolfosfato sintasa de los reinos bacteriano y arqueológico, lo que implica que surgieron en su último ancestro común y se han conservado durante más de mil millones de años.
Insertos estructurales

Las regiones N/C-terminal y de bucle de las proteínas de barril TIM son capaces de albergar insertos estructurales que van desde motivos estructurales secundarios simples hasta dominios completos . Estos dominios ayudan en el reconocimiento del sustrato y la actividad catalítica. A continuación se analizan cuatro ejemplos diversos de barriles TIM que contienen motivos y dominios adicionales.
La orotidina 5'-fosfato descarboxilasa de Bacillus subtilis ( PDB : 1DBT ) es una proteína de barril TIM que muestra 4 hélices α en lugar de los bucles βα típicamente presentes en el extremo C del barril β (residuos 35-42, 89-91, 126-133 y 215-219). Una de estas hélices (R215→K219) contiene un residuo de arginina conservado (R215) necesario para interactuar con una fracción de fosfato en la orotidina 5'-monofosfato. No se encontró que las otras hélices albergaran residuos críticos para la actividad catalítica y pueden cumplir funciones estructurales.
La isomerasa bifuncional de biosíntesis de histidina/triptófano (PriA) ( PDB : 2Y85 ) de Mycobacterium tuberculosis posee la capacidad de catalizar dos reacciones: (i) reacción HisA: la conversión de N-[(5-fosforribosil) formimino]-5-aminoimidazol-4-carboxamida ribonucleótido (ProFAR) a N-[(5-fosforibulosil)formimino]-5-aminoimidazol-4-carboxamida ribonucleótido (PRFAR), y (ii) reacción TrpF: N-(5'-fosforribosil)-antranilato (PRA) a 1-(O-carboxifenilamino)-1'-desoxirribosa-5'-fosfato (CdRP). PriA es una enzima de barril TIM que se adapta a ambos sustratos mediante bucles de sitio activo (bucles 1, 5 y 6, bucles βα extendidos en el extremo C-terminal del barril β) que cambian de conformación dependiendo del reactivo presente. El bucle 1 envuelve el sitio activo solo en presencia de ProFAR. El bucle 5 envuelve el sitio activo, adoptando una conformación de lámina β en presencia de CdRP, o una conformación similar a un nudo en presencia de ProFAR. El bucle 6 envuelve el sitio activo para todos los reactivos.
La dihidroorotato deshidrogenasa A (DHODA) ( PDB : 2DOR ) de Lactococcus lactis es un ejemplo de un barril TIM que posee láminas β y bucles extendidos sobre el extremo C-terminal del barril β. La DHODA cataliza la oxidación de dihidroorotato a orotato, que es parte de la vía de síntesis de novo de uridina 5'-monofosfato (UMP). Esta oxidación está mediada por el mononucleótido de flavina (FMN). Aquí, las láminas β y los bucles extendidos encierran el sitio activo formando una cavidad, al mismo tiempo que albergan varios residuos catalíticos.
El barril TIM de la trimetilamina deshidrogenasa de Methylophilus methylotrophus ( PDB : 2TMD ) es un ejemplo de inserción de dominio completo. Aquí, se inserta un dominio de plegamiento de Rossmann en el extremo C-terminal del barril TIM. La trimetilamina deshidrogenasa cataliza la conversión de trimetilamina en formaldehído. Esta reacción requiere tanto un cofactor 6-S-cisteinil flavina mononucleótido (FMN) reducido como un centro de hierro-azufre ([4Fe-4S] + ) reducido. FMN está unido covalentemente dentro de la región C-terminal del barril β. El centro [4Fe-4S] + es demasiado grande para acomodarse dentro del barril TIM y, en cambio, se coloca muy cerca, a 7 Å de distancia, en la interfaz entre el barril TIM y los dominios de plegamiento de Rossmann.
Mecanismos plegables
La conservación del plegamiento en barril de TIM se refleja en la conservación de sus mecanismos de plegamiento cinético y de equilibrio en parálogos bacterianos con linajes filogenéticamente distintos. La desnaturalización química de varias variantes de barril de TIM naturales [27] [28] y 2 diseñadas [28] involucra invariablemente un intermediario de equilibrio altamente poblado. Los intermediarios cinéticos que aparecen después de la dilución a partir de soluciones altamente desnaturalizantes involucran una especie mal plegada temprana que debe desplegarse al menos parcialmente para acceder a la vía de plegamiento productiva. [27] [28] El paso limitante de la velocidad en el plegamiento es el cierre del barril β de 8 hebras, con la forma de barril abierta precedente correspondiente al intermediario de equilibrio. [29] Las simulaciones de dinámica molecular centradas en los nativos recapitulan los resultados experimentales y señalan el camino hacia modelos computacionales comprobables para mecanismos de plegamiento complejos. [30]
Paisajes de aptitud conservados
Las proteínas de barril TIM poseen una plasticidad de secuencia inusualmente alta, formando grandes familias de enzimas ortólogas y parálogas en organismos ampliamente divergentes. Esta plasticidad sugiere un paisaje de secuencia que permite la adaptación de la proteína a una variedad de condiciones ambientales, en gran medida independiente de la historia filogenética, mientras se mantiene la función. Se utilizó un enfoque de escaneo mutacional profundo [31] y un ensayo de competencia [32] para determinar la aptitud de todos los mutantes de aminoácidos posibles en las posiciones en 3 enzimas de barril TIM de indol-3-glicerolfosfato sintasa (IGPS) hipertermófilas para apoyar el crecimiento de un huésped de levadura que carece de IGPS. Aunque las 2 enzimas IGPS bacterianas y 1 arqueal fueron solo 30-40% idénticas en secuencia, sus paisajes de aptitud estaban fuertemente correlacionados: los mismos aminoácidos en las mismas posiciones en las tres proteínas diferentes tenían una aptitud muy similar. La correlación puede considerarse como la conservación del paisaje de aptitud para una enzima de barril TIM a lo largo del tiempo evolutivo.
Regiones de bucle
De los aproximadamente 200 residuos necesarios para formar completamente un barril TIM, alrededor de 160 se consideran estructuralmente equivalentes entre diferentes proteínas que comparten este pliegue. Los residuos restantes se encuentran en las regiones de bucle que unen las hélices y las hebras; los bucles en el extremo C-terminal de las hebras tienden a contener el sitio activo , que es una de las razones por las que este pliegue es tan común: los residuos necesarios para mantener la estructura y los residuos que efectúan la catálisis enzimática son en su mayor parte subconjuntos distintos: [33] Los bucles de enlace pueden, de hecho, ser tan largos que contienen otros dominios proteicos. Recientemente, se ha demostrado que los bucles catalíticos pueden intercambiarse entre diferentes enzimas de barril TIM como unidades semiautónomas de grupos funcionales. [34]
Evolución y orígenes


La teoría predominante para la evolución de barriles TIM implica la duplicación y fusión de genes, comenzando con un medio barril que eventualmente formó un barril TIM completo. Múltiples estudios respaldan la teoría de la evolución divergente a partir de un único ancestro, y se analizan a continuación.
Evolución a partir de un ancestro común
A principios de la década de 1990, se observó que todas las estructuras de barril TIM resueltas en ese momento eran enzimas, lo que indica divergencia de un ancestro común. [11] [12] Además, todos los barriles TIM poseían sitios activos en el extremo C-terminal de los barriles β. sugirió que un sitio de unión de fosfato común, formado por una pequeña hélice α y bucles de barril TIM-7/8, indicaba fuertemente una evolución divergente. [35] Estudios posteriores de estos grupos de fosfato, concluyeron que 12 de las 23 familias de barriles TIM de SCOP divergieron de un ancestro común. [36] De manera similar, hubo indicios de ascendencia común para 17 de las 21 familias de barriles TIM de CATH . [7] Con base en estos informes, se considera plausible que la mayoría de las proteínas de barril TIM evolucionaran a partir de un ancestro común.
Origen por duplicación de genes y fusión de dominios

Muchas proteínas de barril TIM poseen simetría interna de 2, 4 u 8 pliegues, lo que sugiere que los barriles TIM evolucionaron a partir de motivos ancestrales (βα) 4 , (βα) 2 o βα a través de la duplicación de genes y la fusión de dominios . Un buen ejemplo de simetría interna de 2 pliegues se observa en las enzimas ProFAR isomerasa (HisA) e imidazol glicerol fosfato sintasa (HisF) de la vía de biosíntesis de histidina de Thermotoga maritima . [13] Catalizan 2 reacciones sucesivas en la vía, poseen una homología de secuencia del 25% y poseen desviaciones de raíz cuadrada media (RMSD) entre 1,5-2 Å, lo que sugiere divergencia de un ancestro común. Más interesante aún, los bucles en los extremos C-terminales de HisA y HisF mostraron un patrón doblemente repetido, lo que sugiere que su ancestro común también poseía simetría interna doble. Utilizando estas observaciones, se construyó un modelo para la evolución de los barriles TIM. [13] Un medio barril ancestral habría experimentado un evento de duplicación y fusión de genes, lo que dio como resultado una única proteína que contiene dos dominios de medio barril. Se habrían producido adaptaciones estructurales, lo que dio como resultado la fusión de estos dominios para formar un barril β cerrado y formar un barril TIM ancestral. También se habrían producido adaptaciones funcionales, lo que dio como resultado la evolución de una nueva actividad catalítica en el extremo C-terminal del barril β. En este punto, el ancestro común de HisA y HisF habría experimentado un segundo evento de duplicación de genes. La evolución divergente de los genes duplicados del barril TIM ancestral habría dado como resultado la formación de HisA y HisF.
Curiosamente, este modelo evolutivo se ha validado experimentalmente utilizando el diseño racional de proteínas y la evolución dirigida . Höcker et al. primero fusionaron dos mitades C-terminales de HisF, produciendo HisF-CC. Luego, este constructo se estabilizó mediante la inserción de un puente salino interno , produciendo HisF-C*C. [17] Se logró una estabilización y solubilización gradual adicional de HisF-C*C optimizando la interfaz de medio barril, generando HisF-C**C y HisF-C***C, respectivamente. [15] [16] La estructura cristalina de HisF-C***C reveló un barril TIM simétrico doble, validando la posibilidad de fusión de dominios naturales. Además, Höcker creó los primeros barriles TIM quiméricos HisAF y HisFA utilizando medios barriles HisA y HisF. [17] Estos experimentos condujeron a la propuesta de un nuevo medio de diversificación y evolución de las enzimas de barril TIM a través del intercambio de dominios de medio barril (βα)4 entre barriles TIM preexistentes. De acuerdo con esta idea, se estableció una alta actividad catalítica en el constructo HisAF. [18] De manera similar, también se han creado barriles TIM quiméricos de pliegue similar a βα 5 -flavodoxina (CheY)/HisF, [19] [20] y un barril TIM basado en HisF perfectamente simétrico en 2 pliegues [21] [28] .
La existencia de una simetría interna de 4/8 pliegues se sugirió con base en un análisis computacional de secuencias de barriles TIM. [14] Por ejemplo, se sugirió que la aldolasa KDPG de Escherichia coli [37] ( PDB : 1FQ0 ) posee una simetría cuádruple distintiva, con una simetría óctuple discernible. El diseño de un barril TIM simétrico cuádruple [22] confirmó la posibilidad de órdenes superiores de simetría interna en barriles TIM naturales, y se discutirá en detalle en la siguiente sección. Hasta la fecha, no se ha informado de evidencia experimental de la existencia de barriles TIM simétricos óctuple.
De nuevoDiseño de cañón TIM
El pliegue de barril TIM ha sido un objetivo de larga data para los diseñadores de proteínas de novo . Como se describió anteriormente, se han diseñado con éxito numerosos barriles TIM basados en medios barriles naturales preexistentes. En contraste, el diseño de novo de barriles TIM se produjo en pasos incrementales durante un período de 28 años. [38]
La serie Octarellin [39] [40] [41] [42] [43] de proteínas (Octarellin I→VI) fueron los primeros intentos de crear un barril TIM de novo . Como el campo del diseño de proteínas todavía estaba en sus inicios, estos intentos de diseño solo tuvieron un éxito limitado. Aunque mostraron espectros de dicroísmo circular consistentes con las proteínas αβ y algunas características de plegamiento cooperativo, todos los péptidos de la serie Octarellin eran insolubles y tuvieron que ser resolubilizados a partir de cuerpos de inclusión para una caracterización adicional. Curiosamente, Octarellin V.1 [44] mostró un plegamiento similar al de Rossmann en condiciones de cocristal.
La serie de proteínas Symmetrin (Symmetrin-1→4) mostró características biofísicas más favorables. Symmetrin-1 era fácilmente soluble, mostraba espectros de dicroísmo circular consistentes con las proteínas αβ y mostraba excelentes características de desdoblamiento y replegamiento cooperativo. A pesar de estos avances, todas las proteínas de esta familia mostraron características fundidas cuando se analizaron mediante RMN ( resonancia magnética nuclear ), y no se pudo continuar con el trabajo para resolver sus estructuras.
Las proteínas de la serie sTIM [22] representaron el primer diseño exitoso de barril TIM de novo . [45] [38] sTIM-11 ( PDB : 5BVL ) fue diseñado con una simetría interna de 4 pliegues, para reducir la complejidad del diseño computacional utilizando el paquete de software Rosetta. [46] Los primeros principios derivados previamente [47] se utilizaron para delinear topologías y longitudes de estructura secundaria. sTIM-11 demostró ser un diseño altamente termoestable y de plegado cooperativo que adoptó su estructura prevista.
Véase también
Referencias
 Este artículo fue adaptado de la siguiente fuente bajo licencia CC BY 4.0 (2020) (informes de los revisores): Deepesh Nagarajan; Neha Nanajkar (2020). "El pliegue en barril del TIM" (PDF) . WikiJournal of Science . 3 (1): 4. doi : 10.15347/WJS/2020.004 . ISSN 2470-6345. Wikidata Q87400003.
Este artículo fue adaptado de la siguiente fuente bajo licencia CC BY 4.0 (2020) (informes de los revisores): Deepesh Nagarajan; Neha Nanajkar (2020). "El pliegue en barril del TIM" (PDF) . WikiJournal of Science . 3 (1): 4. doi : 10.15347/WJS/2020.004 . ISSN 2470-6345. Wikidata Q87400003.
- ^ Voet D, Voet JG (2011). "Capítulo 8. Estructuras tridimensionales de las proteínas". Bioquímica (4.ª ed.). John Wiley & Sons, Inc. ISBN 978-0470-91745-9.
- ^ abc Wierenga RK (marzo de 2001). "El pliegue en barril TIM: un marco versátil para enzimas eficientes". FEBS Letters . 492 (3): 193–8. Bibcode :2001FEBSL.492..193W. doi : 10.1016/s0014-5793(01)02236-0 . PMID 11257493. S2CID 42044123.
- ^ Jansen R, Gerstein M (marzo de 2000). "Análisis del transcriptoma de la levadura con categorías estructurales y funcionales: caracterización de proteínas altamente expresadas". Nucleic Acids Research . 28 (6): 1481–8. doi : 10.1093/nar/28.6.1481 . PMC 111042 . PMID 10684945.
- ^ Nagano N, Hutchinson EG, Thornton JM (octubre de 1999). "Estructuras de barril en proteínas: identificación y clasificación automáticas, incluyendo un análisis de secuencia de barriles TIM". Protein Science . 8 (10): 2072–84. doi :10.1110/ps.8.10.2072. PMC 2144152 . PMID 10548053.
- ^ Webb EC (1992). Nomenclatura enzimática: Recomendaciones del Comité de Nomenclatura de la Unión Internacional de Bioquímica y Biología Molecular sobre la nomenclatura y clasificación de enzimas . Academic Press. ISBN 978-0-12-227164-9.
- ^ ab Nagano N, Orengo CA, Thornton JM (agosto de 2002). "Un pliegue con muchas funciones: las relaciones evolutivas entre las familias de barriles TIM basadas en sus secuencias, estructuras y funciones". Journal of Molecular Biology . 321 (5): 741–65. doi :10.1016/s0022-2836(02)00649-6. PMID 12206759.
- ^ Livesay DR, La D (mayo de 2005). "Los orígenes evolutivos y la importancia catalítica de las redes electrostáticas conservadas dentro de las proteínas de barril TIM". Protein Science . 14 (5): 1158–70. doi :10.1110/ps.041221105. PMC 2253277 . PMID 15840824.
- ^ Chung SY, Subbiah S (octubre de 1996). "Una explicación estructural de la zona de penumbra de la homología de secuencias de proteínas". Structure . 4 (10): 1123–7. doi : 10.1016/s0969-2126(96)00119-0 . PMID 8939745.
- ^ ab Vijayabaskar MS, Vishveshwara S (2012). "Información sobre la organización de pliegues del barril TIM a partir de redes estructurales basadas en energía de interacción". PLOS Computational Biology . 8 (5): e1002505. Bibcode :2012PLSCB...8E2505V. doi : 10.1371/journal.pcbi.1002505 . PMC 3355060 . PMID 22615547.
- ^ ab Farber GK, Petsko GA (junio de 1990). "La evolución de las enzimas de barril alfa/beta". Tendencias en ciencias bioquímicas . 15 (6): 228–34. doi :10.1016/0968-0004(90)90035-A. PMID 2200166.
- ^ ab Reardon D, Farber GK (abril de 1995). "La estructura y evolución de las proteínas barril alfa/beta". FASEB Journal . 9 (7): 497–503. doi : 10.1096/fasebj.9.7.7737457 . PMID 7737457. S2CID 23208817.
- ^ abcd Lang D, Thoma R, Henn-Sax M, Sterner R, Wilmanns M (septiembre de 2000). "Evidencia estructural de la evolución del andamiaje de barril beta/alfa mediante duplicación y fusión de genes". Science . 289 (5484): 1546–50. Bibcode :2000Sci...289.1546L. doi :10.1126/science.289.5484.1546. PMID 10968789.
- ^ ab Söding J, Remmert M, Biegert A (julio de 2006). "HHrep: detección de repeticiones de proteínas de novo y el origen de los barriles TIM". Nucleic Acids Research . 34 (edición del servidor web): W137-42. doi : 10.1093/nar/gkl130 . PMC 1538828 . PMID 16844977.
- ^ ab Seitz T, Bocola M, Claren J, Sterner R (septiembre de 2007). "Estabilización de una proteína de (βα)8-barril diseñada a partir de medios barriles idénticos". Journal of Molecular Biology . 372 (1): 114–29. doi :10.1016/j.jmb.2007.06.036. PMID 17631894.
- ^ ab Höcker B, Lochner A, Seitz T, Claren J, Sterner R (febrero de 2009). "Estructura cristalina de alta resolución de una proteína de barril (βα)(8) artificial diseñada a partir de semibarriles idénticos". Bioquímica . 48 (6): 1145–7. doi :10.1021/bi802125b. PMID 19166324.
- ^ abc Höcker B, Claren J, Sterner R, Makar AB, McMartin KE, Palese M, Tephly TR (junio de 1975). "Ensayo de formato en fluidos corporales: aplicación en intoxicación por metanol". Medicina bioquímica . 13 (2): 117–26. doi :10.1016/0006-2944(75)90147-7. PMC 534502 . PMID 15539462.
- ^ ab Claren J, Malisi C, Höcker B, Sterner R (marzo de 2009). "Establecimiento de niveles de tipo salvaje de actividad catalítica en estructuras proteicas de barril 8 (beta alfa) naturales y artificiales". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 106 (10): 3704–9. Bibcode :2009PNAS..106.3704C. doi : 10.1073/pnas.0810342106 . PMC 2656144 . PMID 19237570.
- ^ ab Bharat TA, Eisenbeis S, Zeth K, Höcker B (julio de 2008). "Un barril beta alfa construido mediante la combinación de fragmentos de diferentes pliegues". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 105 (29): 9942–7. Bibcode :2008PNAS..105.9942B. doi : 10.1073/pnas.0802202105 . PMC 2481348 . PMID 18632584.
- ^ ab Eisenbeis S, Proffitt W, Coles M, Truffault V, Shanmugaratnam S, Meiler J, Höcker B (marzo de 2012). "Potencial de la recombinación de fragmentos para el diseño racional de proteínas". Revista de la Sociedad Química Americana . 134 (9): 4019–22. doi :10.1021/ja211657k. PMID 22329686.
- ^ ab Fortenberry C, Bowman EA, Proffitt W, Dorr B, Combs S, Harp J, et al. (noviembre de 2011). "Explorando la simetría como una vía para el diseño computacional de dominios proteicos grandes". Revista de la Sociedad Química Estadounidense . 133 (45): 18026–9. doi :10.1021/ja210593m. PMC 3781211. PMID 21978247 .
- ^ abc Huang PS, Feldmeier K, Parmeggiani F, Velasco DA, Höcker B, Baker D (enero de 2016). "Diseño de novo de una proteína barril TIM cuádruple simétrica con precisión a nivel atómico". Nature Chemical Biology . 12 (1): 29–34. doi :10.1038/nchembio.1966. PMC 4684731 . PMID 26595462.
- ^ Nagarajan D, Deka G, Rao M (agosto de 2015). "Diseño de proteínas barril TIM simétricas a partir de los primeros principios". BMC Biochemistry . 16 (1): 18. doi : 10.1186/s12858-015-0047-4 . PMC 4531894 . PMID 26264284.
- ^ Murzin AG, Lesk AM, Chothia C (marzo de 1994). "Principios que determinan la estructura de los barriles de láminas beta en proteínas. I. Un análisis teórico". Journal of Molecular Biology . 236 (5): 1369–81. doi :10.1016/0022-2836(94)90064-7. PMID 8126726.
- ^ Brändén CI (1991). "El barril TIM: el motivo de plegamiento más frecuente en las proteínas". Current Opinion in Structural Biology . 1 (6): 978–983. doi :10.1016/0959-440x(91)90094-a.
- ^ Barber MJ, Neame PJ, Lim LW, White S, Matthews FS (abril de 1992). "Correlación de secuencias de aminoácidos deducidas por rayos X y experimentales de la trimetilamina deshidrogenasa". The Journal of Biological Chemistry . 267 (10): 6611–9. doi : 10.1016/S0021-9258(19)50471-9 . PMID 1551870.
- ^ ab Forsyth WR, Bilsel O, Gu Z, Matthews CR (septiembre de 2007). "Topología y secuencia en el plegamiento de una proteína barril TIM: el análisis global destaca la partición entre intermediarios de plegamiento transitorios fuera de la vía y estables dentro de la vía en el complejo mecanismo de plegamiento de un barril (βα)8 de función desconocida de B. subtilis". Journal of Molecular Biology . 372 (1): 236–53. doi :10.1016/j.jmb.2007.06.018. PMID 17619021.
- ^ abcd Carstensen L, Sperl JM, Bocola M, List F, Schmid FX, Sterner R (agosto de 2012). "Conservación del mecanismo de plegamiento entre las proteínas de barril 8 (βα) primordiales diseñadas y su descendiente moderno". Journal of the American Chemical Society . 134 (30): 12786–91. doi :10.1021/ja304951v. PMID 22758610.
- ^ Gu Z, Rao MK, Forsyth WR, Finke JM, Matthews CR (noviembre de 2007). "Análisis estructural de intermediarios de plegamiento cinético para una proteína barril TIM, la indol-3-glicerol fosfato sintasa, mediante espectrometría de masas de intercambio de hidrógeno y simulación del modelo Gō". Journal of Molecular Biology . 374 (2): 528–46. doi :10.1016/j.jmb.2007.09.024. PMC 2735044 . PMID 17942114.
- ^ Halloran KT, Wang Y, Arora K, Chakravarthy S, Irving TC, Bilsel O, et al. (agosto de 2019). "Frustración y plegamiento de una proteína barril TIM". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 116 (33): 16378–16383. Bibcode :2019PNAS..11616378H. doi : 10.1073/pnas.1900880116 . PMC 6697809 . PMID 31346089.
- ^ Chan YH, Venev SV, Zeldovich KB, Matthews CR (marzo de 2017). "La correlación de los paisajes de aptitud de tres barriles TIM ortólogos se origina a partir de restricciones de secuencia y estructura". Nature Communications . 8 : 14614. Bibcode :2017NatCo...814614C. doi : 10.1038/ncomms14614 . PMC 5343507 . PMID 28262665.
- ^ Hietpas RT, Jensen JD, Bolon DN (mayo de 2011). "Iluminación experimental de un paisaje de fitness". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 108 (19): 7896–901. doi : 10.1073/pnas.1016024108 . PMC 3093508 . PMID 21464309.
- ^ Ochoa-Leyva A, Soberón X, Sánchez F, Argüello M, Montero-Morán G, Saab-Rincón G (abril de 2009). "Diseño de proteínas mediante intercambio catalítico sistemático de bucles en el pliegue (beta/alfa)8". Journal of Molecular Biology . 387 (4): 949–64. doi :10.1016/j.jmb.2009.02.022. PMID 19233201.
- ^ Ochoa-Leyva A, Barona-Gómez F, Saab-Rincón G, Verdel-Aranda K, Sánchez F, Soberón X (agosto de 2011). "Exploración de la adaptabilidad del bucle estructura-función de una enzima de barril (β / α) (8) mediante el intercambio de bucles y la variabilidad de las bisagras". Revista de biología molecular . 411 (1): 143–57. doi :10.1016/j.jmb.2011.05.027. PMID 21635898.
- ^ Brändén CI (1991). "El barril TIM: el motivo de plegamiento más frecuente en las proteínas". Current Opinion in Structural Biology . 1 (6): 978–983. doi :10.1016/0959-440x(91)90094-a.
- ^ Copley RR, Bork P (noviembre de 2000). "Homología entre barriles (βα)(8): implicaciones para la evolución de las vías metabólicas". Journal of Molecular Biology . 303 (4): 627–41. doi : 10.1006/jmbi.2000.4152 . PMID 11054297.
- ^ Wymer N, Buchanan LV, Henderson D, Mehta N, Botting CH, Pocivavsek L, et al. (enero de 2001). "Evolución dirigida de un nuevo sitio catalítico en la 2-ceto-3-desoxi-6-fosfogluconato aldolasa de Escherichia coli". Estructura . 9 (1): 1–9. doi : 10.1016/S0969-2126(00)00555-4 . PMID 11342129.
- ^ ab Borman S (2015). "Los diseñadores de proteínas lanzan un barril". Chemical & Engineering News . Vol. 93, núm. 47. pág. 6.
- ^ Goraj K, Renard A, Martial JA (marzo de 1990). "Síntesis, purificación y caracterización estructural inicial de octarelina, un polipéptido de novo modelado sobre las proteínas de barril alfa/beta". Ingeniería de proteínas . 3 (4): 259–66. doi :10.1093/protein/3.4.259. PMID 2188263.
- ^ Beauregard M, Goraj K, Goffin V, Heremans K, Goormaghtigh E, Ruysschaert JM, Martial JA (octubre de 1991). "Investigación espectroscópica de la estructura en octarelina (una proteína de novo diseñada para adoptar el empaquetamiento en barril alfa/beta)". Ingeniería de proteínas . 4 (7): 745–9. doi :10.1093/protein/4.7.745. PMID 1798699.
- ^ Houbrechts A, Moreau B, Abagyan R, Mainfroid V, Préaux G, Lamproye A, et al. (marzo de 1995). "Octarelinas de segunda generación: dos nuevos polipéptidos de novo (beta/alfa)8 diseñados para investigar la influencia del empaquetamiento de residuos beta en la estabilidad de la estructura de barril alfa/beta". Ingeniería de proteínas . 8 (3): 249–59. doi :10.1093/protein/8.3.249. PMID 7479687.
- ^ Offredi F, Dubail F, Kischel P, Sarinski K, Stern AS, Van de Weerdt C, et al. (enero de 2003). "Diseño de novo de la estructura principal y la secuencia de una proteína barril alfa/beta idealizada: evidencia de una estructura terciaria estable" (PDF) . Journal of Molecular Biology . 325 (1): 163–74. doi :10.1016/S0022-2836(02)01206-8. PMID 12473459.
- ^ Figueroa M, Oliveira N, Lejeune A, Kaufmann KW, Dorr BM, Matagne A, et al. (2013). "Octarelina VI: uso de Rosetta para diseñar una proteína (β/α)8 artificial putativa". PLOS ONE . 8 (8): e71858. Bibcode :2013PLoSO...871858F. doi : 10.1371/journal.pone.0071858 . PMC 3747059 . PMID 23977165.
- ^ Figueroa M, Sleutel M, Vandevenne M, Parvizi G, Attout S, Jacquin O, et al. (julio de 2016). "La estructura inesperada de la proteína diseñada Octarellin V.1 constituye un desafío para las herramientas de predicción de la estructura de proteínas". Journal of Structural Biology . 195 (1): 19–30. doi :10.1016/j.jsb.2016.05.004. hdl :2268/199167. PMID 27181418.
- ^ Nanda V (enero de 2016). "Diseño de proteínas: llegando al fondo del barril de TIM". Nature Chemical Biology . 12 (1): 2–3. doi :10.1038/nchembio.1987. PMID 26678608.
- ^ Kaufmann KW, Lemmon GH, Deluca SL, Sheehan JH, Meiler J (abril de 2010). "Prácticamente útil: lo que la suite de modelado de proteínas Rosetta puede hacer por usted". Bioquímica . 49 (14): 2987–98. doi : 10.1021/bi902153g . PMC 2850155 . PMID 20235548.
- ^ Koga N, Tatsumi-Koga R, Liu G, Xiao R, Acton TB, Montelione GT, Baker D (noviembre de 2012). "Principios para el diseño de estructuras proteínicas ideales". Nature . 491 (7423): 222–7. Bibcode :2012Natur.491..222K. doi :10.1038/nature11600. PMC 3705962 . PMID 23135467.
Enlaces externos
- Lista SCOP de proteínas que adoptan el pliegue de barril TIM Archivado el 8 de julio de 2007 en Wayback Machine.
- Babu MM (1998). "TIM Barrel Analysis". Centro de Biotecnología, Universidad Anna . Archivado desde el original el 15 de junio de 2013. Consultado el 28 de noviembre de 2011 .