Virus Escherichia T4

| Virus Escherichia T4 | |
|---|---|
 | |
| Virus Escherichia T4 ( EM del virión ) | |
| Clasificación de virus | |
| (sin clasificar): | Virus |
| Reino : | Duplodnaviria |
| Reino: | Virus de Heunggong |
| Filo: | Uroviricóta |
| Clase: | Caudoviricetes |
| Familia: | Estrabovirus |
| Género: | Tequatrovirus |
| Especies: | Virus Escherichia T4 |
| Cepas [2] | |
| |
| Sinónimos [3] | |
Fago T4 de enterobacterias | |
El virus Escherichia T4 es una especie de bacteriófago que infecta a la bacteria Escherichia coli . Es un virus de ADN bicatenario de la subfamilia Tevenvirinae de la familia Straboviridae . T4 es capaz de experimentar solo un ciclo de vida lítico y no el ciclo de vida lisogénico . La especie se denominaba anteriormente bacteriófago T-even , un nombre que también engloba, entre otras cepas (o aislados), Enterobacteria fago T2 , Enterobacteria fago T4 y Enterobacteria fago T6 .
Uso en investigación
Los fagos T-even, que datan de la década de 1940 y continúan hasta nuestros días, se consideran los organismos modelo mejor estudiados. Por lo general, se requiere que los organismos modelo sean simples con tan solo cinco genes . Sin embargo, los fagos T-even se encuentran de hecho entre los virus más grandes y de mayor complejidad, en los que la información genética de estos fagos se compone de alrededor de 300 genes . Coincidiendo con su complejidad, se encontró que los virus T-even tienen la base inusual hidroximetilcitosina (HMC) en lugar de la base de ácido nucleico citosina . [4]
Genoma y estructura
El genoma de ADN bicatenario del virus T4 tiene una longitud de aproximadamente 169 kbp [5] y codifica 289 proteínas . El genoma T4 es terminalmente redundante . Tras la replicación del ADN, se forman concatémeros de longitud multigenómica, tal vez mediante un mecanismo de replicación de círculo rodante. [6] Cuando se empaqueta, el concatémero se corta en posiciones no específicas de la misma longitud, lo que da lugar a varios genomas que representan permutaciones circulares del original. [7] El genoma T4 lleva secuencias de intrones similares a las de los eucariotas .
Traducción
La secuencia Shine-Dalgarno GAGG domina en los genes tempranos del virus T4, mientras que la secuencia GGAG es un objetivo para la endonucleasa T4 RegB que inicia la degradación temprana del ARNm. [8]
Estructura de la partícula del virus

El T4 es un virus relativamente grande, de aproximadamente 90 nm de ancho y 200 nm de largo (la mayoría de los virus tienen una longitud de entre 25 y 200 nm). El genoma de ADN se encuentra en una cabeza icosaédrica , también conocida como cápside . [9] La cola del T4 es hueca para que pueda pasar su ácido nucleico a la célula que está infectando después de unirse. Los fagos Myoviridae como el T4 tienen estructuras de cola contráctiles complejas con una gran cantidad de proteínas involucradas en el ensamblaje y la función de la cola. [10] Las fibras de la cola también son importantes para reconocer los receptores de superficie de la célula huésped, por lo que determinan si una bacteria está dentro del rango de hospedaje del virus. [11]
Recientemente se ha descrito con detalle atómico la estructura de la placa base T4 de 6 megadaltons, que comprende 127 cadenas polipeptídicas de 13 proteínas diferentes (productos génicos 5, 5.4, 6, 7, 8, 9, 10, 11, 12, 25, 27, 48 y 53). También se ha creado un modelo atómico de la región proximal del tubo de la cola formada por gp54 y la proteína del tubo principal gp19. La proteína gp29, que se encuentra en forma de cinta métrica, está presente en los complejos placa base-tubo de la cola, pero no se ha podido modelar. [12]
Durante el ensamblaje del virión del bacteriófago (fago) T4 , las proteínas morfogenéticas codificadas por los genes del fago interactúan entre sí en una secuencia característica. Mantener un equilibrio apropiado en las cantidades de cada una de estas proteínas producidas durante la infección viral parece ser crítico para la morfogénesis normal del fago T4. [13] Las proteínas codificadas por el fago T4 que determinan la estructura del virión incluyen componentes estructurales principales, componentes estructurales menores y proteínas no estructurales que catalizan pasos específicos en la secuencia de morfogénesis. [14] La morfogénesis del fago T4 se divide en tres vías independientes: la cabeza, la cola y las fibras largas de la cola, como detallan Yap y Rossman. [15]
Proceso de infección
El virus T4 inicia una infección por Escherichia coli al unirse a las proteínas porinas OmpC y al lipopolisacárido (LPS) en la superficie de las células de E. coli con sus fibras largas de la cola (LTF). [16] [17] Se envía una señal de reconocimiento a través de las LTF a la placa base. Esto desenreda las fibras cortas de la cola (STF) que se unen irreversiblemente a la superficie celular de E. coli . La placa base cambia de conformación y la vaina de la cola se contrae, lo que hace que GP5 en el extremo del tubo de la cola perfore la membrana externa de la célula. [18] El dominio de lisozima de GP5 se activa y degrada la capa periplásmica de peptidoglicano . La parte restante de la membrana se degrada y luego el ADN de la cabeza del virus puede viajar a través del tubo de la cola y entrar en la célula de E. coli . [ cita requerida ]
En 1952, Hershey y Chase [19] aportaron pruebas clave de que el ADN del fago, a diferencia de las proteínas, entra en la célula bacteriana huésped tras la infección y, por tanto, es el material genético del fago. Este hallazgo sugirió que el ADN es, en general, el material genético de diferentes organismos. [ cita requerida ]
Reproducción
El ciclo de vida lítico (desde la entrada en una bacteria hasta su destrucción) dura aproximadamente 30 minutos (a 37 °C). Los bacteriófagos virulentos se multiplican en su huésped bacteriano inmediatamente después de la entrada. Una vez que el número de fagos de la progenie alcanza una cierta cantidad, provocan la lisis o descomposición del huésped, por lo que se liberarían e infectarían nuevas células huésped. [20] El proceso de lisis y liberación del huésped se denomina ciclo lítico . El ciclo lítico es un ciclo de reproducción viral que implica la destrucción de la célula infectada y su membrana. Este ciclo implica que un virus se apodera de la célula huésped y su maquinaria para reproducirse. Por lo tanto, el virus debe pasar por 5 etapas para reproducirse e infectar la célula huésped: [ cita requerida ]
- Adsorción y penetración (inicio inmediato)
- Detención de la expresión del gen del huésped (inicio inmediato)
- Síntesis de enzimas (iniciando después de 5 minutos)
- Replicación del ADN (comienza después de 10 minutos)
- Formación de nuevas partículas virales (a partir de los 12 minutos)
Una vez completado el ciclo de vida, la célula huésped se abre y expulsa los virus recién creados al medio ambiente, destruyéndola. El tamaño de la ráfaga de T4 es de aproximadamente 100 a 150 partículas virales por huésped infectado. [ cita requerida ]
Benzer (1955 – 1959) desarrolló un sistema para estudiar la estructura fina del gen utilizando mutantes del bacteriófago T4 defectuosos en los genes rIIA y rIIB . [21] [22] [23] Las técnicas empleadas fueron pruebas de complementación y cruces para detectar la recombinación , particularmente entre mutaciones por deleción. Estos experimentos genéticos llevaron al hallazgo de un orden lineal único de sitios mutacionales dentro de los genes. Este resultado proporcionó evidencia sólida para la idea clave de que el gen tiene una estructura lineal equivalente a una longitud de ADN con muchos sitios que pueden mutar de forma independiente. [ cita requerida ]
Adsorción y penetración
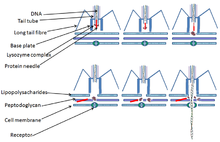
Al igual que todos los demás virus, los fagos T-even no se adhieren aleatoriamente a la superficie de su huésped; en cambio, "buscan" y se unen a receptores , estructuras proteínicas específicas , que se encuentran en la superficie del huésped. Estos receptores varían con el fago; el ácido teicoico , las proteínas de la pared celular y los lipopolisacáridos , los flagelos y los pili pueden servir como receptores a los que se une el fago. Para que el fago T-even infecte a su huésped y comience su ciclo de vida, debe entrar en el primer proceso de infección , la adsorción del fago a la célula bacteriana. La adsorción es un valor característico del par fago-huésped y la adsorción del fago en la superficie de la célula huésped se ilustra como un proceso de 2 etapas: reversible e irreversible. Implica la estructura de la cola del fago que comienza cuando las fibras de la cola del fago ayudan a unir el fago al receptor apropiado de su huésped. Este proceso es reversible. Uno o más de los componentes de la placa base median el proceso irreversible de unión del fago a una bacteria. [ cita requerida ]
La penetración es también un valor característico de la infección fago-huésped que implica la inyección del material genético de los fagos dentro de la bacteria . La penetración del ácido nucleico tiene lugar después de la fase de adsorción irreversible. Los mecanismos que implican la penetración del ácido nucleico de los fagos son específicos para cada fago. Este mecanismo de penetración puede implicar el potencial de membrana electroquímico, las moléculas de ATP , la división enzimática de la capa de peptidoglicano , o los tres factores pueden ser vitales para la penetración del ácido nucleico dentro de la célula bacteriana. Se han realizado estudios sobre el mecanismo de penetración del bacteriófago T2 (fago similar a T4) y se ha demostrado que la cola del fago no penetra dentro de la pared celular bacteriana y la penetración de este fago implica el potencial de membrana electroquímico en la membrana interna. [24] [25]
Replicación y empaquetado
El genoma del virus T4 se sintetiza dentro de la célula huésped mediante replicación por círculo rodante. [6] El tiempo que tarda la replicación del ADN en una célula viva se midió como la tasa de elongación del ADN del virus T4 en E. coli infectada por el virus. [26] Durante el período de aumento exponencial del ADN a 37 °C, la tasa fue de 749 nucleótidos por segundo. La tasa de mutación por par de bases por replicación durante la síntesis de ADN del virus T4 es de 1,7 por 10 −8 , [27] un mecanismo de copia de ADN de alta precisión, con solo 1 error en 300 copias. El virus también codifica mecanismos únicos de reparación del ADN . [28] La cabeza del fago T4 se ensambla vacía alrededor de una proteína de andamiaje, que luego se degrada. En consecuencia, el ADN debe ingresar a la procabeza a través de un poro diminuto, lo que se logra mediante un hexámero de gp17 que interactúa primero con el ADN, que también sirve como motor y nucleasa. Se ha descubierto que el motor de empaquetamiento de ADN T4 carga ADN en las cápsides de los virus a una velocidad de hasta 2000 pares de bases por segundo. La potencia involucrada, si se aumenta en tamaño, sería equivalente a la de un motor de automóvil promedio. [29]
Liberar
El paso final en la reproducción y multiplicación viral está determinado por la liberación de viriones de la célula huésped. La liberación de los viriones ocurre después de la rotura de la membrana plasmática bacteriana. Los virus sin envoltura lisan la célula huésped, lo que se caracteriza por el ataque de proteínas virales al peptidoglicano o membrana. La lisis de las bacterias ocurre cuando las cápsides dentro de la célula liberan la enzima lisozima que rompe la pared celular. Los bacteriófagos liberados infectan otras células y el ciclo de multiplicación viral se repite dentro de esas células. [ cita requerida ]
Reactivación de multiplicidad

La reactivación por multiplicidad (MR) es el proceso por el cual dos o más genomas de virus, cada uno con daño genómico inactivante, pueden interactuar dentro de una célula infectada para formar un genoma de virus viable. Salvador Luria , mientras estudiaba el virus T4 irradiado con UV en 1946, descubrió la MR y propuso que la reactivación observada del virus dañado ocurre por un mecanismo de recombinación. (ver referencias [30] [31] [32] ) Esto precedió a la confirmación del ADN como material genético en 1952 en el virus relacionado T2 por el experimento de Hershey-Chase . [19]
Como recuerda Luria (1984, [33] pág. 97), el descubrimiento de la reactivación de virus irradiados (denominado " reactivación de multiplicidad ") inició inmediatamente una oleada de actividad en el estudio de la reparación de daños por radiación dentro del grupo de fagos tempranos (revisado por Bernstein [28] en 1981). Resultó más tarde que la reparación de virus dañados por ayuda mutua que Luria había descubierto era sólo un caso especial de reparación de ADN. Ahora se sabe que las células de todo tipo, no sólo las bacterias y sus virus, sino todos los organismos estudiados, incluidos los humanos, tienen procesos bioquímicos complejos para reparar daños en el ADN (véase reparación del ADN ). Ahora también se reconoce que los procesos de reparación del ADN desempeñan un papel fundamental en la protección contra el envejecimiento , el cáncer y la infertilidad . [ cita requerida ]
La RM se representa habitualmente mediante "curvas de supervivencia" en las que se representa gráficamente la supervivencia de la capacidad de formación de placas de las células infectadas de forma múltiple (complejos múltiples) frente a la dosis del agente que daña el genoma. A modo de comparación, también se representa gráficamente la supervivencia de la capacidad de formación de placas del virus de las células infectadas individualmente (monocomplejos) frente a la dosis del agente que daña el genoma. La figura superior muestra las curvas de supervivencia de los multicomplejos y monocomplejos del virus T4 con una dosis creciente de luz UV. Dado que la supervivencia se representa gráficamente en una escala logarítmica, resulta claro que la supervivencia de los multicomplejos supera a la de los monocomplejos en factores muy importantes (dependiendo de la dosis). La curva de inactivación UV para los multicomplejos tiene un hombro inicial. Otros agentes que dañan el ADN del virus T4 con hombros en sus curvas de supervivencia de multicomplejos son los rayos X [34] [35] y el etil metano sulfonato (EMS). [28] La presencia de un hombro se ha interpretado como que significa que se utilizan dos procesos de recombinación. [36] La primera repara el ADN con alta eficiencia (en el "hombro"), pero su capacidad se satura a medida que aumenta el daño; la segunda vía funciona en todos los niveles de daño. Los virus T4 sobrevivientes liberados de los multicomplejos no muestran un aumento en la mutación , lo que indica que la RM del virus irradiado con UV es un proceso preciso. [36]
La figura inferior muestra las curvas de supervivencia para la inactivación del virus T4 por el agente que daña el ADN mitomicina C (MMC). En este caso, la curva de supervivencia para multicomplejos no tiene un hombro inicial, lo que sugiere que solo está activo el segundo proceso de reparación recombinacional descrito anteriormente. La eficiencia de la reparación por este proceso está indicada por la observación de que una dosis de MMC que permite la supervivencia de solo 1 en 1000 monocomplejos permite la supervivencia de aproximadamente el 70% de los multicomplejos. También se obtuvieron curvas de supervivencia de multicomplejos similares (sin hombros) para los agentes que dañan el ADN desintegración de P32 , psoraleno más irradiación UV cercana (PUVA), N-metil-N'-nitro-N-nitrosoguanidina (MNNG), metil metanosulfonato (MMS) y ácido nitroso . [28]
Varios de los genes que se encontraron necesarios para la MR en el virus T4 resultaron ser ortólogos de genes esenciales para la recombinación en procariotas , eucariotas y arqueas . Esto incluye, por ejemplo, el gen T4 uvsX [37] que especifica una proteína que tiene homología estructural tridimensional con RecA de Escherichia coli y la proteína homóloga RAD51 en eucariotas y RadA en arqueas . Se ha sugerido que la reparación recombinatoria eficiente y precisa de los daños del ADN durante la MR puede ser análoga al proceso de reparación recombinatoria que ocurre durante la meiosis en eucariotas . [38]
Historia
Los bacteriófagos fueron descubiertos por primera vez por el científico inglés Frederick Twort en 1915 y Félix d'Hérelle en 1917. A finales de la década de 1930, T. L. Rakieten propuso a los investigadores Milislav Demerec y Ugo Fano una mezcla de aguas residuales sin tratar o un lisado de E. coli infectado con aguas residuales sin tratar . Estos dos investigadores aislaron T3, T4, T5 y T6 de E. coli . Además, en 1932, el investigador J. Bronfenbrenner había estudiado y trabajado en el fago T2, en el que se aisló el fago T2 del virus. [39] Este aislamiento se realizó a partir de material fecal en lugar de alcantarillado. En cualquier caso, Max Delbrück estuvo involucrado en el descubrimiento de los fagos T2. Su parte consistió en nombrar los bacteriófagos en Tipo 1 (T1), Tipo 2 (T2), Tipo 3 (T3), etc. [ cita requerida ]
El momento y lugar específicos del aislamiento del virus T4 siguen sin estar claros, aunque es probable que se encontraran en aguas residuales o material fecal. Los virus T4 y similares fueron descritos en un artículo de Thomas F. Anderson , Max Delbrück y Milislav Demerec en noviembre de 1944. [40] En 1943, Salvador Luria y Delbrück demostraron que las mutaciones bacterianas para la resistencia a los fagos surgen en ausencia de selección , en lugar de ser una respuesta a la selección. [33] La sabiduría tradicional entre los bacteriólogos antes de 1943 era que las bacterias no tenían cromosomas ni genes. El experimento de Luria-Delbrück demostró que las bacterias, al igual que otros organismos genéticos modelo establecidos, tienen genes, y que estos pueden mutar espontáneamente para generar mutantes que luego pueden reproducirse para formar linajes clonales. Ese año, también comenzaron a trabajar con Alfred Hershey , otro experimentador de fagos. [41] (Los tres compartirían el Premio Nobel de Fisiología o Medicina de 1969 , "por su trabajo sobre el mecanismo de replicación y la genética de los virus").
El grupo de fagos era una red informal de biólogos centrada en Max Delbrück que llevó a cabo investigación básica principalmente sobre el bacteriófago T4 e hizo numerosas contribuciones seminales a la genética microbiana y los orígenes de la biología molecular a mediados del siglo XX. En 1961, Sydney Brenner , uno de los primeros miembros del grupo de fagos, colaboró con Francis Crick , Leslie Barnett y Richard Watts-Tobin en el Laboratorio Cavendish en Cambridge para realizar experimentos genéticos que demostraron la naturaleza básica del código genético de las proteínas. [42] Estos experimentos, llevados a cabo con mutantes del gen rIIB del fago T4, mostraron que para un gen que codifica una proteína, tres bases secuenciales del ADN del gen especifican cada aminoácido sucesivo de la proteína. Por lo tanto, el código genético es un código de tripletes, donde cada triplete (llamado codón) especifica un aminoácido particular. También obtuvieron evidencia de que los codones no se superponen entre sí en la secuencia de ADN que codifica una proteína, y que dicha secuencia se lee desde un punto de inicio fijo. [ cita requerida ]
Durante 1962-1964, los investigadores del fago T4 brindaron la oportunidad de estudiar la función de prácticamente todos los genes que son esenciales para el crecimiento del fago en condiciones de laboratorio. [43] [44] Estos estudios se vieron facilitados por el descubrimiento de dos clases de mutantes letales condicionales . Una clase de estos mutantes se conoce como mutantes ámbar . [45] Otra clase de mutantes letales condicionales se conoce como mutantes sensibles a la temperatura . [46] Los estudios de estas dos clases de mutantes condujeron a un conocimiento considerable de numerosos problemas biológicos fundamentales. De este modo, se obtuvo conocimiento sobre las funciones e interacciones de las proteínas empleadas en la maquinaria de replicación , reparación y recombinación del ADN , y sobre cómo se ensamblan los virus a partir de componentes de proteínas y ácidos nucleicos ( morfogénesis molecular ). Además, se dilucidó el papel de los codones de terminación de cadena . Un estudio notable utilizó mutantes ámbar defectuosos en el gen que codifica la proteína principal de la cabeza del fago T4. [47] Este experimento proporcionó pruebas sólidas de la "hipótesis de la secuencia", ampliamente aceptada pero aún no demostrada antes de 1964, según la cual la secuencia de aminoácidos de una proteína está determinada por la secuencia de nucleótidos del gen que determina la proteína. Por lo tanto, este estudio demostró la colinealidad del gen con su proteína codificada.
Varios ganadores del Premio Nobel trabajaron con el virus T4 o virus similares a T4, entre ellos Max Delbrück , Salvador Luria , Alfred Hershey , James D. Watson y Francis Crick . Otros científicos importantes que trabajaron con el virus T4 incluyen a Michael Rossmann , Seymour Benzer , Bruce Alberts , Gisela Mosig , [48] Richard Lenski y James Bull .
Véase también
Referencias
- ^ Padilla-Sánchez V (2021). "Modelo estructural del bacteriófago T4". WikiRevista científica . 4 (1): 5. doi : 10.15347/WJS/2021.005 .
- ^ "ICTV 9th Report (2011) Myoviridae". Comité Internacional de Taxonomía de Virus (ICTV) . Archivado desde el original el 26 de diciembre de 2018. Consultado el 26 de diciembre de 2018 .
- ^ "Historia de la taxonomía del ICTV: virus Escherichia T4". Comité Internacional de Taxonomía de Virus (ICTV) . Consultado el 26 de diciembre de 2018.
Caudovirales
>
Myoviridae
>
Tevenvirinae
>
Virus T4
>
Virus Escherichia T4
- ^ Wyatt GR, Cohen SS (diciembre de 1952). "Una nueva base de pirimidina a partir de ácidos nucleicos de bacteriófagos". Nature . 170 (4338): 1072–1073. Bibcode :1952Natur.170.1072W. doi :10.1038/1701072a0. ISSN 1476-4687. PMID 13013321. S2CID 4277592.
- ^ Miller ES, Kutter E, Mosig G, Arisaka F, Kunisawa T, Rüger W (marzo de 2003). "Genoma del bacteriófago T4". Microbiology and Molecular Biology Reviews . 67 (1): 86–156, tabla de contenidos. doi :10.1128/mmbr.67.1.86-156.2003. PMC 150520 . PMID 12626685.
- ^ ab Bernstein H, Bernstein C (julio de 1973). "Concatenaciones circulares y ramificadas como posibles intermediarios en la replicación del ADN del bacteriófago T4". Journal of Molecular Biology . 77 (3): 355–61. doi :10.1016/0022-2836(73)90443-9. PMID 4580243.
- ^ Madigan M, Martinko J, eds. (2006). Brock Biología de los microorganismos (11.ª ed.). Prentice Hall. ISBN 978-0-13-144329-7.
- ^ Malys N (enero de 2012). "Secuencia de Shine-Dalgarno del bacteriófago T4: GAGG prevalece en genes tempranos". Molecular Biology Reports . 39 (1): 33–9. doi :10.1007/s11033-011-0707-4. PMID 21533668. S2CID 17854788.
- ^ Prescott LM, Harley JP, Klein DA (2008). Microbiología (séptima edición). McGraw-Hill. ISBN 978-0-07-126727-4.
- ^ Leiman PG, Arisaka F, van Raaij MJ, Kostyuchenko VA, Aksyuk AA, Kanamaru S, Rossmann MG (diciembre de 2010). "Morfogénesis de la cola T4 y fibras de la cola". Virology Journal . 7 : 355. doi : 10.1186/1743-422X-7-355 . PMC 3004832 . PMID 21129200.
- ^ Ackermann HW, Krisch HM (1997). "Un catálogo de bacteriófagos de tipo T4". Archivos de Virología . 142 (12): 2329–45. doi :10.1007/s007050050246. PMID 9672598. S2CID 39369249.
- ^ Taylor NM, Prokhorov NS, Guerrero-Ferreira RC, Shneider MM, Browning C, Goldie KN, Stahlberg H, Leiman PG (mayo de 2016). "Estructura de la placa base T4 y su función en el desencadenamiento de la contracción de la vaina". Nature . 533 (7603): 346–52. Bibcode :2016Natur.533..346T. doi :10.1038/nature17971. PMID 27193680. S2CID 4399265.
- ^ Floor E (febrero de 1970). "Interacción de genes morfogenéticos del bacteriófago T4". Journal of Molecular Biology . 47 (3): 293–306. doi :10.1016/0022-2836(70)90303-7. PMID 4907266.
- ^ Snustad DP (agosto de 1968). "Interacciones de dominancia en células de Escherichia coli infectadas de forma mixta con bacteriófago T4D de tipo salvaje y mutantes ámbar y sus posibles implicaciones en cuanto al tipo de función del gen-producto: catalítica frente a estequiométrica". Virology . 35 (4): 550–63. doi :10.1016/0042-6822(68)90285-7. PMID 4878023.
- ^ Yap ML, Rossmann MG (2014). "Estructura y función del bacteriófago T4". Future Microbiology . 9 (12): 1319–27. doi :10.2217/fmb.14.91. PMC 4275845 . PMID 25517898.
- ^ Yu F, Mizushima S (agosto de 1982). "Funciones del lipopolisacárido y la proteína de membrana externa OmpC de Escherichia coli K-12 en la función del receptor para el bacteriófago T4". Journal of Bacteriology . 151 (2): 718–22. doi :10.1128/JB.151.2.718-722.1982. PMC 220313 . PMID 7047495.
- ^ Furukawa H, Mizushima S (mayo de 1982). "Funciones de los componentes de la superficie celular de Escherichia coli K-12 en la infección por bacteriófago T4: interacción del núcleo de la cola con los fosfolípidos". Journal of Bacteriology . 150 (2): 916–24. doi :10.1128/JB.150.2.916-924.1982. PMC 216445 . PMID 7040345.
- ^ Maghsoodi A, Chatterjee A, Andricioaei I, Perkins NC (diciembre de 2019). "Cómo funciona la maquinaria de inyección del fago T4, incluidas la energética, las fuerzas y la vía dinámica". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 116 (50): 25097–25105. Bibcode :2019PNAS..11625097M. doi : 10.1073/pnas.1909298116 . PMC 6911207 . PMID 31767752.
- ^ ab HERSHEY AD, CHASE M (mayo de 1952). "Funciones independientes de la proteína viral y el ácido nucleico en el crecimiento del bacteriófago". The Journal of General Physiology . 36 (1): 39–56. doi :10.1085/jgp.36.1.39. PMC 2147348 . PMID 12981234.
- ^ Sherwood L (2011). Microbiología de Prescott (octava edición). McGraw-Hill.
- ^ Benzer S. "Aventuras en la región rII" en Phage and the Origins of Molecular Biology (2007) Editado por John Cairns, Gunther S. Stent y James D. Watson, Laboratorio de Biología Cuantitativa de Cold Spring Harbor, Cold Spring Harbor, Long Island, Nueva York ISBN 978-0879698003
- ^ Benzer S (junio de 1955). "Estructura fina de una región genética en bacteriófagos". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 41 (6): 344–54. Bibcode :1955PNAS...41..344B. doi : 10.1073/pnas.41.6.344 . PMC 528093 . PMID 16589677.
- ^ Benzer S (noviembre de 1959). "Sobre la topología de la estructura fina genética". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 45 (11): 1607–20. Bibcode :1959PNAS...45.1607B. doi : 10.1073/pnas.45.11.1607 . PMC 222769 . PMID 16590553.
- ^ Norkin LC (2010). Virología, biología molecular y patogénesis . Washington: Sociedad Estadounidense de Microbiología. p. 31. ISBN 978-1-55581-453-3.
- ^ Prescott LM, Harley JP, Klein DA (2008). Microbiología (séptima edición). McGraw Hill. pág. 427. ISBN 978-0-07-126727-4.
- ^ McCarthy D, Minner C, Bernstein H, Bernstein C (1976). "Tasas de elongación del ADN y distribuciones de puntos de crecimiento del fago T4 de tipo salvaje y un mutante ámbar con retraso del ADN". J Mol Biol . 106 (4): 963–81. doi :10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ Drake JW (1970) La base molecular de la mutación. Holden-Day, San Francisco ISBN 0816224501 ISBN 978-0816224500
- ^ abcd Bernstein C. "Reparación del ácido desoxirribonucleico en bacteriófagos". Microbiol Rev. 1981 Mar;45(1):72-98. Revisión. PMID 6261109
- ^ Rao VB, Black LW (diciembre de 2010). "Estructura y ensamblaje de la cabeza del bacteriófago T4". Virology Journal . 7 : 356. doi : 10.1186/1743-422X-7-356 . PMC 3012670 . PMID 21129201.
- ^ Luria SE (1947). "Reactivación de bacteriófagos irradiados mediante transferencia de unidades autorreproductoras". Proc. Natl. Sci. USA . 33 (9): 253–64. Bibcode :1947PNAS...33..253L. doi : 10.1073/pnas.33.9.253 . PMC 1079044 . PMID 16588748.
- ^ LURIA SE, DULBECCO R (1948). "Mutaciones letales e inactivación de determinantes genéticos individuales en bacteriófagos". Genética . 33 (6): 618. PMID 18100306.
- ^ Luria SE, Dulbecco R (1949). "Recombinaciones genéticas que conducen a la producción de bacteriófagos activos a partir de partículas de bacteriófagos inactivados por luz ultravioleta". Genética . 34 (2): 93–125. doi :10.1093/genetics/34.2.93. PMC 1209443 . PMID 17247312.
- ^ ab Salvador E. Luria. Una máquina tragamonedas, un tubo de ensayo roto: una autobiografía. Harper & Row, Nueva York: 1984. Pp. 228. ISBN 0-06-015260-5 (Estados Unidos y Canadá)
- ^ WATSON JD (1952). "Las propiedades del bacteriófago inactivado por rayos X". J. Bacteriol . 63 (4): 473–85. doi :10.1128/JB.63.4.473-485.1952. PMC 169298 . PMID 14938320.
- ^ HARM W (1958). "Reactivación de multiplicidad, rescate de marcadores y recombinación genética en el fago T4 después de la inactivación por rayos X". Virología . 5 (2): 337–61. doi :10.1016/0042-6822(58)90027-8. PMID 13544109.
- ^ ab Yarosh DB (1978). "Mutación inducida por UV en el bacteriófago T4". J. Virol . 26 (2): 265–71. doi :10.1128/JVI.26.2.265-271.1978. PMC 354064 . PMID 660716.
- ^ Story RM, Bishop DK, Kleckner N, Steitz TA (1993). "Relación estructural de las proteínas bacterianas RecA con las proteínas de recombinación del bacteriófago T4 y la levadura". Science . 259 (5103): 1892–6. Bibcode :1993Sci...259.1892S. doi :10.1126/science.8456313. PMID 8456313.
- ^ Bernstein C (1979). "¿Por qué los bebés son jóvenes? La meiosis puede prevenir el envejecimiento de la línea germinal". Perspect. Biol. Med . 22 (4): 539–44. doi :10.1353/pbm.1979.0041. PMID 573881. S2CID 38550472.
- ^ Microbiología de Willey J. Prescott (séptima edición). McGraw-Hill.
- ^ Abedon ST (junio de 2000). "El origen turbio de Blancanieves y sus enanos T-even". Genética . 155 (2): 481–6. doi :10.1093/genetics/155.2.481. PMC 1461100 . PMID 10835374.
- ^ Morange, Una historia de la biología molecular , págs. 43-44
- ^ CRICK FH, BARNETT L, BRENNER S, WATTS-TOBIN RJ (diciembre de 1961). "Naturaleza general del código genético de las proteínas". Nature . 192 (4809): 1227–32. Bibcode :1961Natur.192.1227C. doi :10.1038/1921227a0. PMID 13882203. S2CID 4276146.
- ^ Edgar RS Letales condicionales: en Fagos y los orígenes de la biología molecular (2007) Editado por John Cairns, Gunther S. Stent y James D. Watson, Laboratorio de Biología Cuantitativa de Cold Spring Harbor, Cold Spring Harbor, Long Island, Nueva York ISBN 978-0879698003
- ^ Edgar B (octubre de 2004). "El genoma del bacteriófago T4: una excavación arqueológica". Genética . 168 (2): 575–82. doi :10.1093/genetics/168.2.575. PMC 1448817 . PMID 15514035.
- ^ Epstein RH, Bolle A, Steinberg CM, Kellenberger E, Boy de la Tour E, Chevalley R, Edgar RS, Susman M, Denhardt GH, Lielausis A (1963). "Estudios fisiológicos de mutantes letales condicionales del bacteriófago T4D". Simposios de Cold Spring Harbor sobre biología cuantitativa . 28 : 375–394. doi :10.1101/SQB.1963.028.01.053. ISSN 0091-7451.
- ^ Edgar RS, Lielausis I (abril de 1964). "Mutantes sensibles a la temperatura del bacteriófago T4D: su aislamiento y caracterización". Genética . 49 (4): 649–62. doi :10.1093/genetics/49.4.649. PMC 1210603 . PMID 14156925.
- ^ Sarabhai AS, Stretton AO, Brenner S, Bolle A (enero de 1964). "Colinealidad del gen con la cadena polipeptídica". Nature . 201 (4914): 13–7. Bibcode :1964Natur.201...13S. doi :10.1038/201013a0. PMID 14085558. S2CID 10179456.
- ^ Nossal NG, Franklin JL, Kutter E, Drake JW (noviembre de 2004). "Comentarios anecdóticos, históricos y críticos sobre genética. Gisela Mosig". Genética . 168 (3): 1097–104. doi :10.1093/genetics/168.3.1097. PMC 1448779 . PMID 15579671.
Lectura adicional
- Leiman PG, Kanamaru S, Mesyanzhinov VV, Arisaka F., Rossmann MG (2003). "Estructura y morfogénesis del bacteriófago T4". Ciencias de la vida celular y molecular . 60 (11): 2356–2370. doi :10.1007/s00018-003-3072-1. PMC 11138918 . PMID 14625682. S2CID 2228357.
- Karam, J., Petrov, V., Nolan, J., Chin, D., Shatley, C., Krisch, H. y Letarov, A. The T4-like phages genome project. https://web.archive.org/web/20070523215704/http://phage.bioc.tulane.edu/. (El depósito de la secuencia genómica completa de los fagos similares a T4)
- Mosig, G. y F. Eiserling. 2006. Fagos T4 y relacionados: estructura y desarrollo, R. Calendar y ST Abedon (eds.), The Bacteriophages. Oxford University Press, Oxford. (Revisión de la biología del fago T4) ISBN 0-19-514850-9
- Filee J. Tetart F., Suttle CA, Krisch HM (2005). "Bacteriófagos marinos de tipo T4, un componente ubicuo de la materia oscura de la biosfera". Proc. Natl. Sci. USA . 102 (35): 12471–6. Bibcode :2005PNAS..10212471F. doi : 10.1073/pnas.0503404102 . PMC 1194919 . PMID 16116082. (Indicación de prevalencia y fagos similares a T4 en la naturaleza)
- Chibani-Chennoufi S., Canchaya C., Bruttin A., Brussow H. (2004). "Genómica comparativa del fago JS98 de Escherichia coli similar a T4: implicaciones para la evolución de los fagos T4". J. Bacteriol . 186 (24): 8276–86. doi :10.1128/JB.186.24.8276-8286.2004. PMC 532421 . PMID 15576776. (Caracterización de un fago tipo T4)
- Desplats C, Krisch HM (mayo de 2003). "La diversidad y evolución de los bacteriófagos de tipo T4". Res. Microbiol . 154 (4): 259–67. doi : 10.1016/S0923-2508(03)00069-X . PMID 12798230.
- Miller, ES, Kutter E., Mosig G., Arisaka F., Kunisawa T., Ruger W. (2003). "Genoma del bacteriófago T4". Microbiol. Mol. Biol. Rev. 67 ( 1): 86–156. doi : 10.1128/MMBR.67.1.86-156.2003. PMC 150520. PMID 12626685. (Revisión del fago T4, desde la perspectiva de su genoma)
- Desplats C., Dez C., Tetart F., Eleaume H., Krisch HM (2002). "Instantánea del genoma del bacteriófago pseudo-T-even RB49". J. Bacteriol . 184 (10): 2789–2804. doi :10.1128/JB.184.10.2789-2804.2002. PMC 135041 . PMID 11976309. (Descripción general del genoma RB49, un fago similar a T4)
- Malys N, Chang DY, Baumann RG, Xie D, Black LW (2002). "Una biblioteca de presentación de péptidos aleatorizada de SOC y HOC del bacteriófago T4 bipartito: detección y análisis de la interacción entre la terminasa del fago T4 (gp17) y el factor sigma tardío (gp55)". J Mol Biol . 319 (2): 289–304. doi :10.1016/S0022-2836(02)00298-X. PMID 12051907. (Aplicación del fago T4 en biotecnología para estudiar la interacción de proteínas)
- Tétart F., Desplats C., Kutateladze M., Monod C., Ackermann H.-W., Krisch HM (2001). "Filogenia de los principales genes de cabeza y cola de los bacteriófagos de tipo T4 de amplio espectro". J. Bacteriol . 183 (1): 358–366. doi :10.1128/JB.183.1.358-366.2001. PMC 94885 . PMID 11114936. (Indicación de la prevalencia de secuencias de tipo T4 en la naturaleza)
- Abedon ST (2000). "El origen turbio de Blancanieves y sus enanos T-even". Genética . 155 (2): 481–6. doi :10.1093/genetics/155.2.481. PMC 1461100 . PMID 10835374. (Descripción histórica del aislamiento de los fagos similares a T4 T2, T4 y T6)
- Ackermann HW, Krisch HM (1997). "Un catálogo de bacteriófagos de tipo T4". Arch. Virol . 142 (12): 2329–45. doi :10.1007/s007050050246. PMID 9672598. S2CID 39369249. Archivado desde el original el 1 de noviembre de 2001. (Lista casi completa de fagos similares a T4 conocidos en ese momento)
- Monod C, Repoila F, Kutateladze M, Tétart F, Krisch HM (marzo de 1997). "El genoma de los bacteriófagos pseudo-T-even, un grupo diverso que se asemeja al T4". J. Mol. Biol . 267 (2): 237–49. doi :10.1006/jmbi.1996.0867. PMID 9096222. (Descripción general de varios fagos similares a T4 desde la perspectiva de sus genomas)
- Kutter E., Gachechiladze K., Poglazov A., Marusich E., Shneider M., Aronsson P., Napuli A., Porter D., Mesyanzhinov V. (1995). "Evolución de fagos relacionados con T4". Genes de virus . 11 (2–3): 285–297. doi :10.1007/BF01728666. PMID 8828153. S2CID 20529415. (Comparación de los genomas de varios fagos similares a T4)
- Karam, JD et al. 1994. Molecular Biology of Bacteriophage T4. ASM Press, Washington, DC. (La segunda biblia de T4, vaya aquí, así como Mosig y Eiserling, 2006, para comenzar a aprender sobre la biología del fago T4) ISBN 1-55581-064-0
- Eddy, SR 1992. Intrones en los bacteriófagos T-Even. Tesis doctoral. Universidad de Colorado en Boulder. (El capítulo 3 ofrece una descripción general de varios fagos similares a T4, así como el aislamiento de los nuevos fagos similares a T4)
- Surdis, TJ "et al" UC Santa Cruz, noviembre de 1978, "Métodos de unión de bacteriófagos específicos de T4", análisis, descripción general.
- Mathews, CK, EM Kutter, G. Mosig y PB Berget. 1983. Bacteriófago T4. Sociedad Estadounidense de Microbiología, Washington, DC. (La primera biblia sobre T4; no toda la información que aparece aquí está duplicada en Karam et al. , 1994; véase especialmente el capítulo introductorio de Doermann para obtener una descripción histórica de los fagos similares a T4) ISBN 0-914826-56-5
- Russell, RL 1967. Especiación entre los bacteriófagos T-Even. Tesis doctoral. Instituto Tecnológico de California. (Aislamiento de la serie RB de fagos similares a T4)
- Malys N, Nivinskas R (2009). "Disposición no canónica del ARN en fagos pares T4: sitio de unión al ribosoma acomodado en la unión intercistrónica del gen 26-25". Mol Microbiol . 73 (6): 1115–1127. doi :10.1111/j.1365-2958.2009.06840.x. PMID 19708923. S2CID 8187771. (tipo raro de regulación traduccional caracterizado en T4)
- Kay D., Fildes P. (1962). "Bacteriófagos que contienen hidroximetilcitosina y que dependen del triptófano aislados de efluentes urbanos". J. Gen. Microbiol . 27 : 143–6. doi : 10.1099/00221287-27-1-143 . PMID 14454648. (Aislamiento de fagos similares a T4, incluido el del fago Ox2)
Enlaces externos
- Viralzone: virus similares a T4
- Animación del bacteriófago T4 que infecta a E. coli
- Animación del empaquetamiento del ADN del bacteriófago T4
