- Subunidad 40S vista desde el lado expuesto al solvente, identificador PDB 2XZM
- Subunidad 60S vista desde el lado expuesto al solvente, identificadores PDB 4A17, 4A19
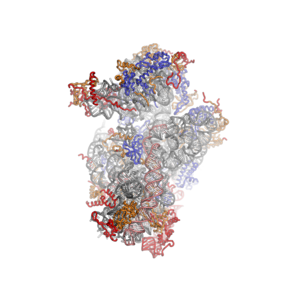
Ribosoma eucariota

Los ribosomas son una máquina molecular grande y compleja que cataliza la síntesis de proteínas , conocida como traducción . El ribosoma selecciona ARN de transferencia aminoacilados (ARNt) basándose en la secuencia de un ARN mensajero (ARNm) que codifica proteínas y une covalentemente los aminoácidos en una cadena polipeptídica . Los ribosomas de todos los organismos comparten un centro catalítico altamente conservado . Sin embargo, los ribosomas de los eucariotas (animales, plantas, hongos y un gran número de organismos unicelulares, todos con núcleo ) son mucho más grandes que los ribosomas procariotas ( bacterias y arqueas ) y están sujetos a vías de regulación y biogénesis más complejas. [1] [2] Los ribosomas eucariotas también se conocen como ribosomas 80S , en referencia a sus coeficientes de sedimentación en unidades de Svedberg , porque sedimentan más rápido que los ribosomas procariotas ( 70S ). Los ribosomas eucariotas tienen dos subunidades desiguales, denominadas subunidad pequeña (40S) y subunidad grande (60S) según sus coeficientes de sedimentación. Ambas subunidades contienen docenas de proteínas ribosómicas dispuestas sobre un andamiaje compuesto de ARN ribosómico (ARNr). La subunidad pequeña controla la complementariedad entre el anticodón del ARNt y el ARNm, mientras que la subunidad grande cataliza la formación de enlaces peptídicos .
Composición
En comparación con sus homólogos procariotas, muchas de las proteínas ribosomales eucariotas se agrandan por inserciones o extensiones al núcleo conservado. Además, se encuentran varias proteínas adicionales en las subunidades pequeñas y grandes de los ribosomas eucariotas, que no tienen homólogos procariotas. La subunidad 40S contiene un ARN ribosómico 18S (abreviado 18S rRNA), que es homólogo al ARN ribosómico 16S procariota . La subunidad 60S contiene un ARN ribosómico 28S que es homólogo al ARN ribosómico 23S procariota . Además, contiene un ARN ribosómico 5.8S que corresponde al extremo 5' del ARN ribosómico 23S, y un ARN ribosómico 5S corto. Tanto 18S como 28S tienen múltiples inserciones en el pliegue del ARN ribosómico central de sus contrapartes procariotas, que se denominan segmentos de expansión. Para obtener una lista detallada de proteínas, incluidos los homólogos de arqueas y bacterias, consulte los artículos separados sobre las subunidades 40S y 60S . Investigaciones recientes sugieren heterogeneidad en la composición ribosómica, es decir, que la estequiometría entre las proteínas ribosómicas centrales en células de levadura de tipo salvaje y células madre embrionarias depende tanto de las condiciones de crecimiento como de la cantidad de ribosomas unidos por ARNm. [3]
| Eucariota [4] | Bacteriano [4] | ||
|---|---|---|---|
| Ribosoma | Coeficiente de sedimentación | 80 S | 70 S |
| Masa molecular | ~3,2×10 6 Da | ~2,0×10 6 Da | |
| Diámetro | ~250–300 Å | ~200 Å | |
| Subunidad grande | Coeficiente de sedimentación | 60 S | 50 S |
| Masa molecular | ~2,0×10 6 Da | ~1,3×10 6 Da | |
| Proteínas | 46 | 33 | |
| ARNr |
|
| |
| Subunidad pequeña | Coeficiente de sedimentación | 40 S | 30 S |
| Masa molecular | ~1,2×10 6 Da | ~0,7×10 6 Da | |
| Proteínas | 33 | 20 | |
| ARNr |
|
|
Determinación de la estructura
Las estructuras iniciales de los ribosomas eucariotas se determinaron mediante microscopía electrónica . Las primeras estructuras 3D se obtuvieron con una resolución de 30-40 Å para ribosomas de levadura [5] y mamíferos. [6] [7] Las estructuras de mayor resolución del ribosoma de levadura mediante criomicroscopía electrónica permitieron la identificación de elementos estructurales de proteínas y ARN. [8] Más recientemente, se obtuvieron estructuras con una resolución subnanómetro para complejos de ribosomas y factores involucrados en la traducción. [9] [10] [11] Después de la determinación de las primeras estructuras de ribosomas bacterianos [12] [13] [14] y arqueales [15] con resolución atómica en la década de 1990, tomó otra década hasta que en 2011, se obtuvieron estructuras de alta resolución de ribosomas eucariotas mediante cristalografía de rayos X , principalmente debido a las dificultades para obtener cristales de suficiente calidad . [16] [17] [18] Se publicó y describió la estructura completa de una estructura ribosomal eucariota 40S en Tetrahymena thermophila , así como mucho sobre la interacción de la subunidad 40S con eIF1 durante la iniciación de la traducción. [16] También se determinó la estructura de la subunidad eucariota 60S de T. thermophila en complejo con eIF6 . [17] La estructura completa del ribosoma eucariota 80S de la levadura Saccharomyces cerevisiae se obtuvo por cristalografía a una resolución de 3,0 A. [18] Estas estructuras revelan la arquitectura precisa de los elementos específicos de eucariotas, su interacción con el núcleo universalmente conservado y todos los puentes específicos de eucariotas entre las dos subunidades ribosomales.
Las coordenadas atómicas (archivos PDB) y los factores de estructura del ribosoma eucariota se han depositado en el Protein Data Bank (PDB) bajo los siguientes códigos de acceso:
| Complejo | Organismo fuente | Resolución | Identificador PDB [19] |
|---|---|---|---|
| Años 80:Stm1 | S. cerevisiae | 3,0 Å |
|
| 40S:eIF1 | T. thermophila | 3,9 Å |
|
| 60S:eIF6 | T. thermophila | 3,5 Å |
|
Arquitectura
Características generales
Algunas características arquitectónicas generales del ribosoma se conservan en todos los reinos: [20] La estructura de la subunidad pequeña se puede subdividir en dos grandes segmentos, la cabeza y el cuerpo. Las características del cuerpo incluyen los pies izquierdo y derecho, el hombro y la plataforma. La cabeza presenta una protuberancia puntiaguda que recuerda al pico de un pájaro. En la característica "vista de corona" de la subunidad grande, los puntos de referencia estructurales incluyen la protuberancia central, el tallo L1 y el tallo P. [21] [22] La mayoría de los elementos proteicos y de ARN específicos de eucariotas se encuentran en los lados expuestos al solvente de las subunidades 40S [16] y 60S [17] . La interfaz de la subunidad, así como las regiones funcionales importantes como el centro de la peptidil transferasa y el sitio de decodificación se conservan en su mayoría, con algunas diferencias observadas en las regiones circundantes. En marcado contraste con las proteínas ribosomales procariotas, que interactúan principalmente con el ARN, los segmentos proteicos específicos de eucariotas participan en una multitud de interacciones proteína-proteína. Las interacciones a larga distancia están mediadas por extensiones helicoidales específicas de eucariotas de proteínas ribosomales, y varias proteínas ribosomales eucariotas se unen para formar láminas beta interproteicas .
El núcleo del ARN ribosómico se representa como un tubo gris, los segmentos de expansión se muestran en rojo. Las proteínas universalmente conservadas se muestran en azul. Estas proteínas tienen homólogos en eucariotas, arqueas y bacterias. Las proteínas compartidas solo entre eucariotas y arqueas se muestran en naranja, y las proteínas específicas de eucariotas se muestran en rojo.
Coevolución del ARNr y las proteínas
La estructura de la subunidad 40S reveló que las proteínas específicas de eucariotas (rpS7, rpS10, rpS12 y RACK1), así como numerosas extensiones de proteínas específicas de eucariotas, se encuentran en el lado expuesto al solvente de la subunidad pequeña. [16] Aquí, participan en la estabilización de los segmentos de expansión de ARNr. Además, el pico de la subunidad 40S se remodela, ya que el ARNr ha sido reemplazado por las proteínas rpS10 y rpS12. [16] Como se observó para la subunidad 40S, todas las proteínas específicas de eucariotas de la subunidad 60S (RPL6, RPL22, RPL27, RPL28, RPL29 y RPL36) y muchas extensiones se encuentran en el lado expuesto al solvente, formando una red intrincada de interacciones con segmentos de expansión de ARN específicos de eucariotas. RPL6, RPL27 y RPL29 median los contactos entre los conjuntos ES7–ES39, ES31–ES20–ES26 y ES9–ES12, respectivamente, y el segmento de expansión estabilizado por RPL28 ES7A. [17]
Proteínas de fusión de ubiquitina
En eucariotas, la proteína de subunidad pequeña RPS27A (o eS31) y la proteína de subunidad grande RPL40 (o eL40) son polipéptidos procesados, que se traducen como proteínas de fusión que llevan dominios de ubiquitina N-terminales . Ambas proteínas se encuentran junto a importantes centros funcionales del ribosoma: los dominios de ubiquitina no escindidos de eS31) y eL40 estarían posicionados en el sitio de decodificación y cerca del sitio de unión del factor de traducción, respectivamente. Estas posiciones sugieren que la escisión proteolítica es un paso esencial en la producción de ribosomas funcionales. [16] [17] De hecho, las mutaciones del enlazador entre el núcleo de eS31 y el dominio de ubiquitina son letales en levaduras. [23]
Sitio activo
Las comparaciones entre las estructuras de los ribosomas bacterianos, arqueológicos y eucariotas revelan un grado muy alto de conservación en la región del sitio activo, también conocido como el centro de la peptidil transferasa (PTC). Ninguno de los elementos proteicos específicos de eucariotas está lo suficientemente cerca como para participar directamente en la catálisis. [17] Sin embargo, RPL29 se proyecta a 18Å del sitio activo en T. thermophila , y las extensiones específicas de eucariotas interconectan varias proteínas en la proximidad del PTC de la subunidad 60S, [17] [21] mientras que las proteínas 50S correspondientes son entidades singulares. [15]
Puentes entre subunidades
Los contactos entre las dos subunidades ribosómicas se conocen como puentes intersubunitarios. En el ribosoma eucariota, los segmentos de expansión 60S y las proteínas realizan contactos adicionales. [24] Específicamente, la extensión C-terminal de la proteína 60S RPL19 interactúa con ES6E del ARNr 40S, y la extensión C-terminal de la proteína 60S RPL24 interactúa con rpS6 40S y la hélice h10 del ARNr. Además, los segmentos de expansión 60S ES31 y ES41 interactúan con rpS3A(S1) y rpS8 de la subunidad 40S, respectivamente, y el péptido básico de 25 aminoácidos RPL41 se ubica en la interfaz de la subunidad en el ribosoma 80S, interactuando con elementos del ARNr de ambas subunidades. [21] [24]
Proteínas ribosómicas con funciones en la señalización
Dos proteínas ribosomales 40S ( RACK1 y RPS6 (o eS6) ) han sido implicadas en la señalización celular: RACK1, descrita por primera vez como el receptor de la proteína quinasa C activada (PKC) , es un componente integral del ribosoma eucariota y está ubicado en la parte posterior de la cabeza. [16] Puede vincular las vías de transducción de señales directamente al ribosoma, aunque también tiene un papel en múltiples procesos de traducción que parecen no estar relacionados (revisados en [25] ). La proteína ribosomal eS6 está ubicada en el pie derecho de la subunidad 40S [16] y se fosforila en respuesta a la señalización de la diana mamífera de la rapamicina (mTOR) . [26]
Aspectos funcionales
Iniciación de la traducción
La síntesis de proteínas se regula principalmente en la etapa de iniciación de la traducción . En eucariotas, la vía de iniciación canónica requiere al menos 12 factores de iniciación de proteínas , algunos de los cuales son en sí mismos grandes complejos. [27] Las estructuras de los complejos 40S:eIF1 [16] y 60S:eIF6 [17] proporcionan los primeros conocimientos detallados sobre las interacciones atómicas entre el ribosoma eucariota y los factores reguladores. eIF1 está involucrado en la selección del codón de inicio, y eIF6 impide estéricamente la unión de subunidades. Sin embargo, la información estructural sobre los factores de iniciación eucariotas y sus interacciones con el ribosoma es limitada y se deriva en gran medida de modelos de homología o análisis de baja resolución. [28] La elucidación de las interacciones entre el ribosoma eucariota y los factores de iniciación a nivel atómico es esencial para una comprensión mecanicista de los procesos reguladores, pero representa un desafío técnico significativo, debido a la dinámica inherente y la flexibilidad de los complejos de iniciación. La primera estructura del complejo de preiniciación de los mamíferos se realizó mediante microscopía crioelectrónica. [29] Pronto se siguieron otras estructuras de complejos de iniciación, impulsadas por mejoras técnicas de crio-EM. [30] [31] Esas estructuras ayudarán a comprender mejor el proceso de iniciación de la traducción en eucariotas.
Funciones reguladoras de las proteínas ribosómicas
Se ha interpretado que evidencia genética reciente sugiere que las proteínas individuales del ribosoma eucariota contribuyen directamente a la regulación de la traducción. [32] [33] [34] Sin embargo, esta interpretación es controvertida y algunos investigadores han propuesto que los cambios genéticos en los genes de las proteínas ribosómicas afectan indirectamente el número total de ribosomas o los procesos de biogénesis de los ribosomas. [35] [36]
Translocación y orientación de proteínas
Para ejercer sus funciones en la célula, las proteínas recién sintetizadas deben ser dirigidas a la ubicación apropiada en la célula, lo que se logra mediante sistemas de orientación y translocación de proteínas . [37] El polipéptido en crecimiento sale del ribosoma a través de un túnel estrecho en la subunidad grande. La región alrededor del túnel de salida de la subunidad 60S es muy similar a las subunidades 50S bacterianas y arqueales. Los elementos adicionales están restringidos al segundo nivel de proteínas alrededor de la salida del túnel, posiblemente por interacciones conservadas con componentes de la maquinaria de translocación. [17] La maquinaria de orientación y translocación es mucho más compleja en eucariotas. [38]
Enfermedades ribosómicas y cáncer
Las ribosomopatías son trastornos humanos congénitos que resultan de defectos en los genes de la proteína ribosomal o del ARNr, u otros genes cuyos productos están implicados en la biogénesis de los ribosomas. [39] Los ejemplos incluyen disqueratosis congénita ligada al cromosoma X (X-DC) , [40] anemia de Diamond-Blackfan , [41] síndrome de Treacher Collins (TCS) [41] [42] y síndrome de Shwachman-Bodian-Diamond (SBDS) . [39] El SBDS es causado por mutaciones en la proteína SBDS que afecta su capacidad para acoplar la hidrólisis de GTP por la GTPasa EFL1 a la liberación de eIF6 de la subunidad 60S. [43]
Oportunidades terapéuticas
El ribosoma es un objetivo farmacológico destacado para los antibacterianos , que interfieren con la traducción en diferentes etapas del ciclo de elongación [44]. La mayoría de los compuestos de traducción clínicamente relevantes son inhibidores de la traducción bacteriana, pero los inhibidores de la traducción eucariota también pueden tener potencial terapéutico para su aplicación en la quimioterapia contra el cáncer o antimicótica. [45] Los inhibidores de la elongación muestran actividad antitumoral "in vivo" e "in vitro". [46] [47] [48] Un inhibidor tóxico de la elongación de la traducción eucariota es el antibiótico glutarimida cicloheximida (CHX), que se ha cocristalizado con la subunidad 60S eucariota [17] y se une en el sitio E ribosómico. La caracterización estructural del ribosoma eucariota [16] [17] [24] puede permitir el uso de métodos basados en la estructura para el diseño de nuevos antibacterianos, en donde las diferencias entre los ribosomas eucariotas y bacterianos se pueden explotar para mejorar la selectividad de los fármacos y, por lo tanto, reducir los efectos adversos .
Mecanismo de formación
Los ribosomas eucariotas se producen y ensamblan en el nucléolo . Las proteínas ribosómicas ingresan al nucléolo y se combinan con las cuatro cadenas de ARNr para crear las dos subunidades ribosómicas (una pequeña y una grande) que formarán el ribosoma completo. Las unidades ribosómicas salen del núcleo a través de los poros nucleares y se unen una vez en el citoplasma con el propósito de sintetizar proteínas.
Referencias
- ^ "Diferencia entre ribosomas 70S y ribosomas 80S, ARN, micromoléculas". www.microbiologyprocedure.com. Archivado desde el original el 5 de septiembre de 2008. Consultado el 6 de agosto de 2009 .
- ^ "Ribosomas 80S, ribosomas eucariotas, ribosomas procariotas, ácidos nucleicos, coeficiente de sedimentación". www.microbiologyprocedure.com. Archivado desde el original el 23 de junio de 2009. Consultado el 6 de agosto de 2009 .
- ^ Slavov, Nikolai; Semrau, Stefan; Airoldi, Edoardo ; Budnik, Bogdan; van Oudenaarden, Alexander (2015). "Estequiometría diferencial entre proteínas ribosomales centrales". Cell Reports . 13 (5): 865–873. doi :10.1016/j.celrep.2015.09.056. ISSN 2211-1247. PMC 4644233 . PMID 26565899.
- ^ Los valores ab se basan en los ribosomas de Tetrahymena thermophila (PDB: 4V8P) y Thermus thermophilus (PDB: 4V5D). El tamaño, el peso y la cantidad exactos de proteínas varían de un organismo a otro.
- ^ Verschoor, A; Warner, JR; Srivastava, S; Grassucci, RA; Frank, J (enero de 1998). "Estructura tridimensional del ribosoma de la levadura". Nucleic Acids Res . 26 (2): 655–661. doi :10.1093/nar/26.2.655. PMC 147289 . PMID 9421530.
- ^ Verschoor, A; Frank, J (agosto de 1990). "Estructura tridimensional del ribosoma citoplasmático de los mamíferos". J Mol Biol . 214 (3): 737–749. doi :10.1016/0022-2836(90)90289-X. PMID 2388265.
- ^ Dube, P; Wieske, M; Stark, H; Schatz, M; Stahl, J; Zemlin, F; Lutsch, G; van Heel, M (marzo de 1998). "El ribosoma de hígado de rata 80S a una resolución de 25 A mediante criomicroscopía electrónica y reconstitución angular". Estructura . 6 (3): 389–399. doi : 10.1016/s0969-2126(98)00040-9 . PMID 9551559.
- ^ Spahn, CM; Beckmann, R; Eswar, N; Penczek, PA; Sali, A; Blobel, G; Frank, J (noviembre de 2001). "Estructura del ribosoma 80S de Saccharomyces cerevisiae: interacciones ARNt-ribosoma y subunidad-subunidad". Cell . 107 (3): 373–386. doi : 10.1016/s0092-8674(01)00539-6 . PMID 11701127.
- ^ Halic, M; Gartmann, M; Schlenker, O; Mielke, T; Pool, MR; Sinning, I; Beckmann, R (mayo de 2006). "El receptor de partículas de reconocimiento de señales expone el sitio de unión del translocón ribosomal". Science . 312 (5774): 745–747. Bibcode :2006Sci...312..745H. doi :10.1126/science.1124864. hdl : 11858/00-001M-0000-0010-842E-9 . PMID 16675701. S2CID 7237420.
- ^ Becker, T; Bhushan, S; Jarasch, A; Armache, JP; Funes, S; Jossinet, F; Gumbart, J; Mielke, T; Berninghausen, O; Schulten, K; Westhof, E; Gilmore, R; Mandon, EC; Beckmann, R (diciembre de 2009). "Estructura de complejos monoméricos de levadura y mamíferos Sec61 que interactúan con el ribosoma traductor". Science . 326 (5958): 1369–1373. Bibcode :2009Sci...326.1369B. doi :10.1126/science.1178535. PMC 2920595 . PMID 19933108.
- ^ Schüler, M; Connell, SR; Lescoute, A; Giesebrecht, J; Dabrowski, M; Schroeer, B; Mielke, T; Penczek, PA; Westhof, E; Spahn, CM (diciembre de 2006). "Estructura del ARN IRES del virus de la parálisis del grillo unido al ribosoma". Nat Struct Mol Biol . 13 (12): 1092–1096. doi :10.1038/nsmb1177. hdl : 11858/00-001M-0000-0010-8321-7 . PMID 17115051. S2CID 8243970.
- ^ Clemons, WM Jr; May, JL; Wimberly, BT; McCutcheon, JP; Capel, MS; Ramakrishnan, V (agosto de 1999). "Estructura de una subunidad ribosomal bacteriana 30S con una resolución de 5,5 A". Nature . 400 (6747): 833–840. Bibcode :1999Natur.400..833C. doi :10.1038/23631. PMID 10476960. S2CID 14808559.
- ^ Cate, JH; Yusupov, MM; Yusupova, GZ; Earnest, TN; Noller, HF (septiembre de 1999). "Estructuras cristalinas de rayos X de complejos funcionales del ribosoma 70S". Science . 285 (5436): 2095–2104. doi :10.1126/science.285.5436.2095. PMID 10497122.
- ^ Yusupov, MM; Yusupova, GZ; Baucom, A; Lieberman, K; Earnest, TN; Cate, JH; Noller, HF (mayo de 2001). "Estructura cristalina del ribosoma a una resolución de 5,5 A". Science . 292 (5518): 883–896. Bibcode :2001Sci...292..883Y. doi : 10.1126/science.1060089 . PMID 11283358. S2CID 39505192.
- ^ ab Ban, N; Nissen, P; Hansen, J; Moore, PB; Steitz, TA (agosto de 2000). "La estructura atómica completa de la subunidad ribosomal grande con una resolución de 2,4 A". Science . 289 (5481): 905–920. Bibcode :2000Sci...289..905B. doi :10.1126/science.289.5481.905. PMID 10937989. S2CID 14056415.
- ^ abcdefghij Rabl, J; Leibundgut, M; Ataide, SF; Haag, A; Ban, N (febrero de 2011). "Estructura cristalina de la subunidad ribosomal eucariota 40S en complejo con el factor de iniciación 1". Science . 331 (6018): 730–736. Bibcode :2011Sci...331..730R. doi :10.1126/science.1198308. hdl : 20.500.11850/153130 . PMID 21205638. S2CID 24771575.
- ^ abcdefghijk Klinge, S; Voigts-Hoffmann, F; Leibundgut, M; Arpagaus, S; Ban, N (noviembre de 2011). "Estructura cristalina de la subunidad ribosomal eucariota 60S en complejo con el factor de iniciación 6". Science . 334 (6058): 941–948. Bibcode :2011Sci...334..941K. doi :10.1126/science.1211204. PMID 22052974. S2CID 206536444.
- ^ ab Ben-Shem A, Garreau de Loubresse N, Melnikov S, Jenner L, Yusupova G, Yusupov M (febrero de 2011). "La estructura del ribosoma eucariota con una resolución de 3,0 Å". Science . 334 (6062): 1524–1529. Bibcode :2011Sci...334.1524B. doi : 10.1126/science.1212642 . PMID 22096102. S2CID 9099683.
- ^ Debido a limitaciones de tamaño, las estructuras de los ribosomas a menudo se dividen en varios archivos de coordenadas
- ^ Melnikov, S; Ben-Shem, A; Garreau; de Loubresse, N; Jenner, L; Yusupova, G; Yusupov, M (junio de 2012). "Un núcleo, dos capas: ribosomas bacterianos y eucariotas". Nat Struct Mol Biol . 19 (6): 560–567. doi :10.1038/nsmb.2313. PMID 22664983. S2CID 6267832.
- ^ abc Klinge, S; Voigts-Hoffmann, F; Leibundgut, M; Ban, N (mayo de 2012). "Estructuras atómicas del ribosoma eucariota". Trends Biochem Sci . 37 (5): 189–198. doi :10.1016/j.tibs.2012.02.007. PMID 22436288.
- ^ Jenner, L; Mélnikov, S; de Loubresse, NG; Ben-Shem, A; Iskakova, M; Urzhumtsev, A; Meskauskas, A; Dinman, J; Yusupova, G; Yusupov, M (diciembre de 2012). "Estructura cristalina del ribosoma de levadura 80S". Estructura de opinión actual Biol . 22 (6): 759–767. doi :10.1016/j.sbi.2012.07.013. PMID 22884264.
- ^ Lacombe, T; García-Gómez, JJ; de la Cruz, J; Roser, D; Hurt, E; Linder, P; Kressler, D (abril de 2009). "La fusión lineal de la ubiquitina con Rps31 y su posterior escisión son necesarias para la producción eficiente y la integridad funcional de las subunidades ribosomales 40S". Mol Microbiol . 72 (1): 69–84. doi :10.1111/j.1365-2958.2009.06622.x. PMID 19210616. S2CID 33924290.
- ^ abc Ben-Shem, A; Garreau; de Loubresse, N; Melnikov, S; Jenner, L; Yusupova, G; Yusupov, M (diciembre de 2011). "La estructura del ribosoma eucariota a una resolución de 3,0 Ã". Science . 334 (6062): 1524–1529. Bibcode :2011Sci...334.1524B. doi : 10.1126/science.1212642 . PMID 22096102. S2CID 9099683.
- ^ Nilsson, J; Sengupta, J; Frank, J; Nissen, P (diciembre de 2004). "Regulación de la traducción eucariota por la proteína RACK1: una plataforma para moléculas de señalización en el ribosoma". EMBO Rep . 5 (12): 1137–1141. doi :10.1038/sj.embor.7400291. PMC 1299186 . PMID 15577927.
- ^ Palm, L; Andersen, J; Rahbek-Nielsen, H; Hansen, TS; Kristiansen, K; Højrup, P (marzo de 1995). "La proteína ribosomal fosforilada S7 en Tetrahymena es homóloga con la S4 de mamíferos y los residuos fosforilados se encuentran en la región C-terminal. Caracterización estructural de proteínas separadas por electroforesis en gel de poliacrilamida bidimensional". J Biol Chem . 270 (11): 6000–6005. doi : 10.1074/jbc.270.11.6000 . PMID 7890730.
- ^ Hinnebusch, AG; Lorsch, JR (octubre de 2012). "El mecanismo de iniciación de la traducción eucariota: nuevos conocimientos y desafíos". Cold Spring Harb Perspect Biol . 4 (10): a011544. doi :10.1101/cshperspect.a011544. PMC 3475172. PMID 22815232 .
- ^ Voigts-Hoffmann, F; Klinge, S; Ban, N (diciembre de 2012). "Información estructural sobre los ribosomas eucariotas y el inicio de la traducción". Curr Opin Struct Biol . 22 (6): 768–777. doi :10.1016/j.sbi.2012.07.010. PMID 22889726.
- ^ Hashem, Y.; Georges, A.; Dhote, V.; Langlois, R.; Liao, HY; Grassucci, RA; Frank, J. (2013). "Estructura del complejo de preiniciación ribosómica 43S de mamíferos unido al factor de exploración DHX29". Celúla . 153 (5): 1108-1119. doi :10.1016/j.cell.2013.04.036. PMC 3730827 . PMID 23706745.
- ^ Hashem, Y., Des Georges, A., Dhote, V., Langlois, R., Liao, HY, Grassucci, RA, ... y Frank, J. (2013). Los sitios de entrada internos a los ribosomas similares al virus de la hepatitis C desplazan a eIF3 para acceder a la subunidad 40S. Nature.
- ^ Fernández, IS; Bai, XC; Hussain, T.; Kelley, AC; Lorsch, JR; Ramakrishnan, V.; Scheres, SH (2013). "Arquitectura molecular de un complejo de iniciación traduccional eucariota". Science . 342 (6160): 1240585. doi :10.1126/science.1240585. PMC 3836175 . PMID 24200810.
- ^ Gilbert, Wendy V. (2011). "¿Especialización funcional de los ribosomas?". Tendencias en ciencias bioquímicas . 36 (3): 127–132. doi :10.1016/j.tibs.2010.12.002. ISSN 0968-0004. PMC 3056915. PMID 21242088 .
- ^ Topisirovic, I; Sonenberg, N (abril de 2011). "Control de la traducción por el ribosoma eucariota". Cell . 145 (3): 333–334. doi : 10.1016/j.cell.2011.04.006 . PMID 21529706.
- ^ Preiss, Thomas (2015). "Todos los ribosomas son creados iguales. ¿En serio?". Tendencias en ciencias bioquímicas . 41 (2): 121–123. doi :10.1016/j.tibs.2015.11.009. ISSN 0968-0004. PMID 26682497.
- ^ Ferretti, Max B.; Karbstein, Katrin (7 de febrero de 2019). "¿Existe realmente la especialización funcional de los ribosomas?". ARN . 25 (5). Cold Spring Harbor Laboratory: 521–538. doi : 10.1261/rna.069823.118 . ISSN 1355-8382. PMC 6467006 . PMID 30733326.
- ^ Farley-Barnes, Katherine I.; Ogawa, Lisa M.; Baserga, Susan J. (2019). "Ribosomopatías: viejos conceptos, nuevas controversias". Tendencias en genética . 35 (10). Elsevier BV: 754–767. doi :10.1016/j.tig.2019.07.004. ISSN 0168-9525. PMC 6852887 . PMID 31376929.
- ^ Boehringer, Daniel; Greber, Basil; Ban, Nenad (2011). "Información mecanicista sobre el procesamiento, plegamiento, orientación e inserción en membrana de proteínas co-traduccionales". Ribosomas . págs. 405–418. doi :10.1007/978-3-7091-0215-2_32. ISBN 978-3-7091-0214-5.
- ^ Bohnsack, Markus T.; Schleiff, Enrico (2010). "La evolución de los sistemas de orientación y translocación de proteínas". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research . 1803 (10): 1115–1130. doi :10.1016/j.bbamcr.2010.06.005. PMID 20600359.
- ^ ab Narla, A; Ebert, BL (abril de 2010). "Ribosomopatías: trastornos humanos de disfunción de los ribosomas". Blood . 115 (16): 3196–3205. doi :10.1182/blood-2009-10-178129. PMC 2858486 . PMID 20194897.
- ^ Stumpf, CR; Ruggero, D (agosto de 2011). "El aparato de traducción canceroso". Curr Opin Genet Dev . 21 (4): 474–483. doi :10.1016/j.gde.2011.03.007. PMC 3481834 . PMID 21543223.
- ^ ab Narla, A; Ebert, BL (octubre de 2011). "Medicina traslacional: ribosomopatías". Blood . 118 (16): 4300–1. doi :10.1182/blood-2011-08-372250. PMID 22021450.
- ^ Dauwerse, JG; Dixon, J; Seland, S; Ruivenkamp, California; van Haeringen, A; Hoefsloot, LH; Peters, DJ; Bóers, AC; Daumer-Haas, C; Maiwald, R; Zweier, C; Kerr, B; Cobo, AM; Toral, JF; Hoogeboom, AJ; Lohmann, DR; Hehr, U; Dixon, MJ; Breuning, MH; Wieczorek, D (enero de 2011). "Las mutaciones en genes que codifican subunidades de las ARN polimerasas I y III provocan el síndrome de Treacher Collins". Nat Genet . 43 (1): 20-22. doi :10.1038/ng.724. PMID 21131976. S2CID 205357102.
- ^ Finch, AJ; Hilcenko, C; Basse, N; Drynan, LF; Goyenechea, B; Menne, TF; González Fernández, A; Simpson, P; D'Santos, CS; Arends, MJ; Donadieu, J; Bellanné-Chantelot, C; Costanzo, M; Boone, C; McKenzie, AN ; Freund, SM; Warren, AJ (mayo de 2011). "El desacoplamiento de la hidrólisis de GTP de la liberación de eIF6 en el ribosoma causa el síndrome de Shwachman-Diamond". Genes & Development . 25 (9): 917–929. doi :10.1101/gad.623011. PMC 3084026 . PMID 21536732.
- ^ Blanchard, SC; Cooperman, BS; Wilson, DN (junio de 2010). "Investigación de la traducción con inhibidores de moléculas pequeñas". Chem. Biol . 17 (6): 633–645. doi :10.1016/j.chembiol.2010.06.003. PMC 2914516. PMID 20609413 .
- ^ Pelletier, J.; Peltz, SW (2007). "Oportunidades terapéuticas en la traducción". Archivo monográfico de Cold Spring Harbor . 48 : 855–895.
- ^ Schneider-; Poetsch, T.; Usui, T.; et al. (2010a). "Mensajes ilegibles y traducciones corruptas". Nature Methods . 6 (3): 189–198. doi :10.1038/nchembio.326. PMID 20154667.
- ^ Schneider; Poetsch, T.; Ju, J.; et al. (2010). "2010b. Inhibición de la elongación de la traducción eucariota por cicloheximida y lactimidomicina". Nat Chem Biol . 6 (3): 209–217. doi :10.1038/nchembio.304. PMC 2831214 . PMID 20118940.
- ^ Dang, Y.; et al. (2011). "Inhibición de la elongación de la traducción eucariota por el producto natural antitumoral Mycalamide B". ARN . 17 (8): 1578–1588. doi :10.1261/rna.2624511. PMC 3153980 . PMID 21693620.
Notas
- "EMDB-1067: complejo ribosomal 80S-eEF2-sordarina de S. cerevisiae - EM Navigator". emnavi.protein.osaka-u.ac.jp. Archivado desde el original el 19 de diciembre de 2012. Consultado el 6 de agosto de 2009 .
- Giavalisco P, Wilson D, Kreitler T, et al. (marzo de 2005). "Alta heterogeneidad dentro de las proteínas ribosomales del ribosoma 80S de Arabidopsis thaliana". Plant Mol. Biol . 57 (4): 577–591. doi :10.1007/s11103-005-0699-3. hdl : 11858/00-001M-0000-0010-86C6-1 . PMID: 15821981. S2CID : 14500573.
- "Ribosomas". www.cs.stedwards.edu. Archivado desde el original el 20 de marzo de 2009. Consultado el 6 de agosto de 2009 .