PubMed
 | |
| Contacto | |
|---|---|
| Centro de investigación | Biblioteca Nacional de Medicina de los Estados Unidos (NLM) |
| Fecha de lanzamiento | Enero de 1996 ( 1996-01 ) |
| Acceso | |
| Sitio web | pubmed.ncbi.nlm.nih.gov |
PubMed es una base de datos gratuita que incluye principalmente la base de datos MEDLINE de referencias y resúmenes sobre ciencias biológicas y temas biomédicos . La Biblioteca Nacional de Medicina de los Estados Unidos (NLM) de los Institutos Nacionales de Salud mantiene la base de datos como parte del sistema Entrez de recuperación de información . [1]
Desde 1971 hasta 1997, el acceso en línea a la base de datos MEDLINE se había realizado principalmente a través de instalaciones institucionales, como bibliotecas universitarias . [2] PubMed, lanzado por primera vez en enero de 1996, marcó el comienzo de la era de la búsqueda privada y gratuita en MEDLINE desde el hogar y la oficina. [3] El sistema PubMed se ofreció gratuitamente al público a partir de junio de 1997. [2]
Contenido
Además de MEDLINE, PubMed proporciona acceso a:
- Referencias más antiguas de la versión impresa del Index Medicus , desde 1951 y antes
- referencias a algunas revistas antes de que fueran indexadas en Index Medicus y MEDLINE, por ejemplo Science , BMJ y Annals of Surgery
- Entradas muy recientes en los registros de un artículo antes de que se indexe con Medical Subject Headings (MeSH) y se agregue a MEDLINE.
- una colección de libros disponibles en texto completo y otros subconjuntos de registros de NLM [4]
- Citas de PMC
- Estantería de libros del NCBI
Muchos registros de PubMed contienen enlaces a artículos de texto completo, algunos de los cuales están disponibles gratuitamente, a menudo en PubMed Central [5] y espejos locales, como Europe PubMed Central . [6]
La información sobre las revistas indexadas en MEDLINE y disponibles a través de PubMed se encuentra en el Catálogo NLM. [7]
A fecha de 23 de mayo de 2023 [actualizar], PubMed tiene más de 35 millones de citas y resúmenes que datan desde 1966, selectivamente hasta el año 1865 y muy selectivamente hasta 1809. A la misma fecha [actualizar], 24,6 millones de registros de PubMed están listados con sus resúmenes, y 26,8 millones de registros tienen enlaces a versiones de texto completo (de los cuales 10,9 millones de artículos están disponibles, en texto completo de forma gratuita). [8] Durante los últimos 10 años (hasta el 31 de diciembre de 2019), se agregaron un promedio de casi un millón de nuevos registros cada año.
En 2016, NLM cambió el sistema de indexación para que los editores puedan corregir directamente errores tipográficos y errores en los artículos indexados en PubMed. [9]
Se ha informado que PubMed incluye algunos artículos publicados en revistas depredadoras. Las políticas de MEDLINE y PubMed para la selección de revistas para su inclusión en bases de datos son ligeramente diferentes. Las debilidades en los criterios y procedimientos para la indexación de revistas en PubMed Central pueden permitir que las publicaciones de revistas depredadoras se filtren a PubMed. [10]
Características
Diseño de sitios web
En octubre de 2009 se lanzó una nueva interfaz de PubMed que fomentaba el uso de fórmulas de búsqueda rápidas, similares a las de Google; también se las ha descrito como búsquedas de "telegrama". [11] De manera predeterminada, los resultados se ordenan por Más reciente, pero esto se puede cambiar a Mejor coincidencia, Fecha de publicación, Primer autor, Último autor, Revista o Título. [12]
El diseño y el dominio del sitio web de PubMed se actualizaron en enero de 2020 y se convirtieron en predeterminados el 15 de mayo de 2020, con las características actualizadas y nuevas. [13] Hubo una reacción crítica de muchos investigadores que utilizan el sitio con frecuencia. [14]
PubMed para dispositivos móviles y de mano
Se puede acceder a PubMed/MEDLINE a través de dispositivos portátiles, utilizando por ejemplo la opción "PICO" (para preguntas clínicas específicas) creada por la NLM. [15] También está disponible la opción "PubMed Mobile", que brinda acceso a una versión simplificada y compatible con dispositivos móviles de PubMed. [16]
Buscar
Búsqueda estándar
Se pueden realizar búsquedas simples en PubMed ingresando aspectos clave de un tema en la ventana de búsqueda de PubMed.
PubMed traduce esta formulación de búsqueda inicial y agrega automáticamente nombres de campos, términos MeSH (encabezados de temas médicos) relevantes, sinónimos, operadores booleanos y "anida" los términos resultantes de manera apropiada, mejorando significativamente la formulación de búsqueda, en particular al combinar rutinariamente (usando el operador OR) palabras de texto y términos MeSH. [ cita requerida ]
Los ejemplos dados en un tutorial de PubMed [17] demuestran cómo funciona este proceso automático:
Causas El sonambulismo se traduce como ("etiología"[Subtítulo] O "etiología"[Todos los campos] O "causas"[Todos los campos] O "causalidad"[Términos MeSH] O "causalidad"[Todos los campos]) Y ("sonambulismo"[Términos MeSH] O "sonambulismo"[Todos los campos] O ("dormir"[Todos los campos] Y "caminar"[Todos los campos]) O "sonambulismo"[Todos los campos])
Asimismo,
Prevención de Ataque Suave Aspirina se traduce como ("infarto de miocardio"[Términos MeSH] O ("miocardio"[Todos los campos] Y "infarto"[Todos los campos]) O "infarto de miocardio"[Todos los campos] O ("corazón"[Todos los campos] Y "ataque"[Todos los campos]) O "ataque cardíaco"[Todos los campos]) Y ("aspirina"[Términos MeSH] O "aspirina"[Todos los campos]) Y ("prevención y control"[Subtítulo] O ("prevención"[Todos los campos] Y "control"[Todos los campos]) O "prevención y control"[Todos los campos] O "prevención"[Todos los campos])
Búsqueda exhaustiva
Para realizar búsquedas óptimas en PubMed, es necesario conocer su componente principal, MEDLINE, y especialmente el vocabulario controlado MeSH (Medical Subject Headings) que se utiliza para indexar los artículos de MEDLINE. También pueden requerir estrategias de búsqueda complejas, uso de nombres de campos (etiquetas), uso adecuado de límites y otras características; los bibliotecarios de referencia y los especialistas en búsquedas ofrecen servicios de búsqueda. [18] [19]
La búsqueda en la ventana de búsqueda de PubMed sólo se recomienda para la búsqueda de temas unívocos o de nuevas intervenciones que aún no tengan un encabezado MeSH creado, así como para la búsqueda de marcas comerciales de medicamentos y nombres propios. También es útil cuando no hay un encabezado adecuado o el descriptor representa un aspecto parcial. La búsqueda mediante el tesauro MeSH es más precisa y dará menos resultados irrelevantes. Además, ahorra el inconveniente de la búsqueda de texto libre en la que hay que tener en cuenta las diferencias ortográficas, singular/plural o abreviadas. Por otro lado, no se encontrarán artículos incorporados más recientemente a la base de datos a los que aún no se les haya asignado descriptores. Por lo tanto, para garantizar una búsqueda exhaustiva, se debe utilizar una combinación de encabezados de lenguaje controlado y términos de texto libre. [20]
Parámetros del artículo de revista
Cuando se indexa un artículo de una revista, se extraen y almacenan numerosos parámetros del artículo como información estructurada. Estos parámetros son: tipo de artículo (términos MeSH, p. ej., "ensayo clínico"), identificadores secundarios (términos MeSH), idioma, país de la revista o historial de publicación (fecha de publicación electrónica, fecha de publicación en la revista impresa).
Tipo de publicación: Consultas clínicas/revisiones sistemáticas
El parámetro de tipo de publicación permite realizar búsquedas por tipo de publicación , incluidos informes de diversos tipos de investigación clínica. [21]
Identificación secundaria
Desde julio de 2005, el proceso de indexación de artículos de MEDLINE extrae identificadores del resumen del artículo y los coloca en un campo llamado Identificador secundario (SI). El campo de identificador secundario se utiliza para almacenar números de acceso a varias bases de datos de secuencias moleculares, expresión genética o compuestos químicos e identificadores de ensayos clínicos. Para los ensayos clínicos, PubMed extrae los identificadores de los ensayos de los dos registros de ensayos más grandes: ClinicalTrials.gov (identificador NCT) y el Registro Internacional Normalizado de Números de Ensayos Controlados Aleatorizados (identificador IRCTN). [22]
Véase también
Se puede marcar una referencia que se considere particularmente relevante y se pueden identificar "artículos relacionados". Si es relevante, se pueden seleccionar varios estudios y se pueden generar artículos relacionados con todos ellos (en PubMed o en cualquiera de las otras bases de datos NCBI Entrez) utilizando la opción "Buscar datos relacionados". A continuación, los artículos relacionados se enumeran en orden de "relación". Para crear estas listas de artículos relacionados, PubMed compara palabras del título y el resumen de cada cita, así como los encabezados MeSH asignados, utilizando un potente algoritmo de ponderación de palabras. [23] Se ha considerado que la función "artículos relacionados" es tan precisa que los autores de un artículo sugirieron que se puede utilizar en lugar de una búsqueda completa. [24]
Asignación a MeSH
PubMed enlaza automáticamente a términos y subtítulos MeSH. Algunos ejemplos serían: "mal aliento" enlaza a (e incluye en la búsqueda) "halitosis", "ataque cardíaco" a "infarto de miocardio", "cáncer de mama" a "neoplasias de mama". Cuando corresponde, estos términos MeSH se "expanden" automáticamente, es decir, incluyen términos más específicos. Términos como "enfermería" se enlazan automáticamente a "Enfermería [MeSH]" o "Enfermería [Subtítulo]". Esta característica se llama Asignación automática de términos y se activa, de forma predeterminada, en la búsqueda de texto libre, pero no en la búsqueda de frases exactas (es decir, encerrando la consulta de búsqueda entre comillas dobles). [25] Esta característica hace que las búsquedas de PubMed sean más sensibles y evita los resultados falsos negativos (perdidos) al compensar la diversidad de la terminología médica. [25]
PubMed no aplica el mapeo automático del término en las siguientes circunstancias: al escribir la frase citada (por ejemplo, "aloinjerto de riñón"), cuando se trunca en el asterisco (por ejemplo, aloinjerto de riñón*) y cuando se busca con etiquetas de campo (por ejemplo, Cáncer [ti]). [20]
Mi NCBI
La función opcional de PubMed "My NCBI" (con registro gratuito) proporciona herramientas para
- guardando búsquedas
- Filtrar resultados de búsqueda
- Configurar actualizaciones automáticas enviadas por correo electrónico
- guardar conjuntos de referencias recuperadas como parte de una búsqueda en PubMed
- configurar formatos de visualización o resaltar términos de búsqueda
y una amplia gama de otras opciones. [26] Se puede acceder al área "My NCBI" desde cualquier computadora con acceso a la web. Una versión anterior de "My NCBI" se llamaba "PubMed Cubby". [27]
Enlace de salida
LinkOut es una herramienta de la NLM para vincular y poner a disposición el texto completo de las publicaciones periódicas locales. [28] Alrededor de 3200 sitios (principalmente instituciones académicas) participan en esta herramienta de la NLM (a marzo de 2010 [actualizar]), desde la Universidad de Aalborg en Dinamarca hasta ZymoGenetics en Seattle. [29] Los usuarios de estas instituciones ven el logotipo de su institución dentro del resultado de búsqueda de PubMed (si la publicación se encuentra en esa institución) y pueden acceder al texto completo. LinkOut se está consolidando con Outside Tool a partir de la actualización principal de la plataforma que se realizará en el verano de 2019. [30]
PubMed Commons
En 2016, PubMed permite a los autores de artículos comentar los artículos indexados por PubMed. Esta función se probó inicialmente en modo piloto (desde 2013) y se hizo permanente en 2016. [31] En febrero de 2018, PubMed Commons se suspendió debido a que "su uso se ha mantenido mínimo". [32] [33]
pregúntele a MEDLINE
askMEDLINE, una herramienta de consulta en lenguaje natural y texto libre para MEDLINE/PubMed, desarrollada por la NLM, también adecuada para dispositivos portátiles. [34]
Identificador de PubMed
Un PMID (identificador de PubMed o identificador único de PubMed) [35] es un valor entero único , que comienza en 1, asignado a cada registro de PubMed. Un PMID no es lo mismo que un PMCID (identificador de PubMed Central), que es el identificador de todos los trabajos publicados en PubMed Central, de libre acceso . [36]
La asignación de un PMID o PMCID a una publicación no le dice al lector nada sobre el tipo o la calidad del contenido. Los PMID se asignan a cartas al editor , opiniones editoriales, columnas de opinión y cualquier otro artículo que el editor elija incluir en la revista, así como a artículos revisados por pares. La existencia del número de identificación tampoco es prueba de que los artículos no hayan sido retractados por fraude, incompetencia o mala conducta. El anuncio sobre cualquier corrección a los artículos originales puede tener asignado un PMID.
Cada número que se introduce en la ventana de búsqueda de PubMed se trata de forma predeterminada como si fuera un PMID. Por lo tanto, cualquier referencia en PubMed se puede localizar mediante el PMID.
Interfaces alternativas
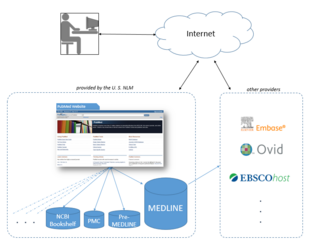
La Biblioteca Nacional de Medicina alquila la información de MEDLINE a varios proveedores privados, como Embase , Ovid , Dialog , EBSCO , Knowledge Finder y muchos otros proveedores comerciales, no comerciales y académicos. [37] Hasta octubre de 2008 [actualizar], se habían emitido más de 500 licencias, más de 200 de ellas a proveedores fuera de los Estados Unidos. Como las licencias para usar los datos de MEDLINE están disponibles de forma gratuita, la NLM proporciona en efecto un campo de pruebas gratuito para una amplia gama [38] de interfaces alternativas y adiciones de terceros a PubMed, una de las pocas bases de datos grandes y curadas profesionalmente que ofrece esta opción.
Lu identifica una muestra de 28 versiones actuales y gratuitas de PubMed basadas en la Web, que no requieren instalación ni registro, que se agrupan en cuatro categorías: [38]
- Clasificación de resultados de búsqueda, por ejemplo: eTBLAST ; MedlineRanker; [39] MiSearch; [40]
- Agrupamiento de resultados por temas, autores, revistas, etc., por ejemplo: Anne O'Tate ; [41] ClusterMed; [42]
- Mejorar la semántica y la visualización, por ejemplo: EBIMed; [43] MedEvi. [44]
- Interfaz de búsqueda mejorada y experiencia de recuperación, por ejemplo, askMEDLINE [45] [46] BabelMeSH; [47] y PubCrawler. [48]
Como la mayoría de estas y otras alternativas se basan esencialmente en datos de PubMed/MEDLINE alquilados bajo licencia de la NLM/PubMed, se ha sugerido el término "derivados de PubMed". [38] Sin la necesidad de almacenar unos 90 GB de conjuntos de datos originales de PubMed, cualquiera puede escribir aplicaciones de PubMed utilizando la interfaz del programa eutils-application como se describe en "The E-utilities In-Depth: Parameters, Syntax and More", de Eric Sayers, PhD. [49] Varios generadores de formatos de citas, que toman números PMID como entrada, son ejemplos de aplicaciones web que hacen uso de la interfaz del programa eutils-application. Las páginas web de muestra incluyen Citation Generator – Mick Schroeder, Pubmed Citation Generator – Ultrasound of the Week, PMID2cite y Cite this for me.
Minería de datos de PubMed
Los métodos alternativos para extraer datos de PubMed utilizan entornos de programación como Matlab , Python o R. En estos casos, las consultas de PubMed se escriben como líneas de código y se pasan a PubMed, y la respuesta se procesa directamente en el entorno de programación. El código se puede automatizar para realizar consultas sistemáticas con diferentes palabras clave como enfermedad, año, órganos, etc.
Además de su papel tradicional como base de datos biomédica, PubMed se ha convertido en un recurso común para el entrenamiento de modelos de lenguaje biomédico . [50] Los avances recientes en este campo incluyen el desarrollo de modelos como PubMedGPT, un modelo de parámetros 2.7B entrenado en datos de PubMed por Stanford CRFM, y BiomedCLIP-PubMedBERT de Microsoft, que utiliza pares de figuras y leyendas de PubMed Central para el procesamiento de visión y lenguaje. Estos modelos demuestran el potencial significativo de los datos de PubMed para mejorar las capacidades de la IA en la investigación médica y las aplicaciones de atención médica. Estos avances subrayan la creciente intersección entre la minería de datos a gran escala y el desarrollo de la IA en el campo biomédico.
Los datos accesibles a través de PubMed se pueden reflejar localmente utilizando una herramienta no oficial como MEDOC. [51]
Millones de registros de PubMed se suman a varios conjuntos de datos abiertos sobre acceso abierto , como Unpaywall . Las bibliotecas utilizan herramientas de análisis de datos como Unpaywall Journals para ayudar con las cancelaciones de grandes acuerdos : las bibliotecas pueden evitar suscripciones a materiales que ya se ofrecen mediante acceso abierto instantáneo a través de archivos abiertos como PubMed Central. [52]
Véase también
- Europa PubMed Central
- RevistaReview.org
- Lista de bases de datos y motores de búsqueda académicos
- Centro de PubMed
- PubMed Central Canadá
Referencias
- ^ "PubMed". Archivado desde el original el 13 de diciembre de 2020. Consultado el 22 de febrero de 2019 .
- ^ ab Lindberg DA (2000). "Acceso a la Biblioteca Nacional de Medicina por Internet" (PDF) . Effective Clinical Practice . 3 (5): 256–60. PMID 11185333. Archivado desde el original (PDF) el 2 de noviembre de 2013.
- ^ "PubMed celebra su décimo aniversario". Boletín técnico . Biblioteca Nacional de Medicina de los Estados Unidos . 5 de octubre de 2006. Archivado desde el original el 23 de abril de 2018 . Consultado el 22 de marzo de 2011 .
- ^ "PubMed: Recuperación de datos de MEDLINE en la World Wide Web". Hoja informativa . Biblioteca Nacional de Medicina de los Estados Unidos. 7 de junio de 2002. Archivado desde el original el 1 de septiembre de 2018 . Consultado el 22 de marzo de 2011 .
- ^ Roberts RJ (enero de 2001). "PubMed Central: El GenBank de la literatura publicada". Actas de la Academia Nacional de Ciencias de los Estados Unidos de América . 98 (2): 381–2. Bibcode :2001PNAS...98..381R. doi : 10.1073/pnas.98.2.381 . PMC 33354 . PMID 11209037.
- ^ McEntyre JR, Ananiadou S, Andrews S, Black WJ, Boulderstone R, Buttery P, et al. (enero de 2011). "UKPMC: un recurso de artículos de texto completo para las ciencias de la vida". Nucleic Acids Research . 39 (número de la base de datos): D58-65. doi :10.1093/nar/gkq1063. PMC 3013671 . PMID 21062818.
- ^ "Catálogo NLM: Revistas referenciadas en las bases de datos del NCBI". NCBI. 2011. Archivado desde el original el 13 de octubre de 2023. Consultado el 8 de septiembre de 2017 .
- ^ "PubMed". PubMed . Archivado desde el original el 6 de enero de 2022 . Consultado el 5 de enero de 2023 .La consulta de búsqueda "1800:2100[dp]" recupera todos los resultados cuya fecha de publicación esté entre 1800 y 2100 inclusive.
- ^ "Mejoras en la producción de MEDLINE/PubMed en marcha". Boletín Técnico de la NLM (411): e1. Julio-agosto de 2016. Archivado desde el original el 29 de marzo de 2023 . Consultado el 29 de julio de 2016 .
- ^ Manca A, Moher D, Cugusi L, Dvir Z, Deriu F (septiembre de 2018). "Cómo las revistas depredadoras se filtran en PubMed". CMAJ . 190 (35): E1042–E1045. doi :10.1503/cmaj.180154. PMC 6148641 . PMID 30181150.
- ^ Clarke J, Wentz R (septiembre de 2000). "El enfoque pragmático es eficaz en la atención sanitaria basada en la evidencia". BMJ . 321 (7260): 566–7. doi :10.1136/bmj.321.7260.566/a. PMC 1118450 . PMID 10968827.
- ^ Fatehi F, Gray LC, Wootton R (enero de 2014). "Cómo mejorar sus búsquedas en PubMed/MEDLINE: 2. Configuración de visualización, consultas de búsqueda complejas y búsqueda de temas". Journal of Telemedicine and Telecare . 20 (1): 44–55. doi :10.1177/1357633X13517067. PMID 24352897. S2CID 43725062.
- ^ Trawick B (21 de enero de 2020). "Un PubMed® nuevo y mejorado". NLM Musings From the Mezzanine . Archivado desde el original el 7 de octubre de 2023. Consultado el 23 de mayo de 2020 .
- ^ Price M (22 de mayo de 2020). «Rediseñaron PubMed, un sitio web muy querido. No ha sido bien recibido». Science . Archivado desde el original el 21 de mayo de 2022 . Consultado el 30 de junio de 2022 .
- ^ "PubMed a través de dispositivos portátiles (PICO)". Boletín técnico . Biblioteca Nacional de Medicina de los Estados Unidos. 2004. Archivado desde el original el 30 de mayo de 2023 . Consultado el 7 de abril de 2016 .
- ^ "PubMed Mobile Beta". Boletín técnico . Biblioteca Nacional de Medicina de los Estados Unidos. 2011. Archivado desde el original el 11 de abril de 2023 . Consultado el 7 de abril de 2016 .
- ^ "Búsqueda de materias sencilla con cuestionario". NCBI. 2010. Archivado desde el original el 11 de mayo de 2020. Consultado el 7 de abril de 2016 .
- ^ Jadad AR, McQuay HJ (julio de 1993). "Búsqueda de la literatura. Sea sistemático en su búsqueda". BMJ . 307 (6895): 66. doi :10.1136/bmj.307.6895.66-a. PMC 1678459 . PMID 8343701.
- ^ Allison JJ, Kiefe CI, Weissman NW, Carter J, Centor RM (primavera de 1999). "El arte y la ciencia de buscar en MEDLINE para responder preguntas clínicas. Encontrar el número correcto de artículos". Revista internacional de evaluación de tecnología en atención sanitaria . 15 (2): 281–96. doi :10.1017/S0266462399015214. PMID 10507188. S2CID 11023273. Archivado desde el original el 19 de febrero de 2022. Consultado el 13 de diciembre de 2019 .
- ^ ab Campos-Asensio C (2018). "Cómo elaborar una estrategia de búsqueda bibliográfica". Enfermería Intensiva (en español). 29 (4): 182–186. doi :10.1016/j.enfi.2018.09.001. PMID 30291015. S2CID 188132546.
- ^ Términos de filtro de consultas clínicas explicados. NCBI. 2010. Archivado desde el original el 29 de noviembre de 2022. Consultado el 8 de septiembre de 2017 .
- ^ Huser V, Cimino JJ (junio de 2013). "Evaluación de la adherencia a la política del Comité Internacional de Editores de Revistas Médicas sobre el registro obligatorio y oportuno de ensayos clínicos". Revista de la Asociación Estadounidense de Informática Médica . 20 (e1): e169-74. doi :10.1136/amiajnl-2012-001501. PMC 3715364 . PMID 23396544.
- ^ "Explicación del cálculo de artículos relacionados". NCBI. Archivado desde el original el 18 de diciembre de 2008. Consultado el 8 de septiembre de 2017 .
- ^ Chang AA, Heskett KM, Davidson TM (febrero de 2006). "Búsqueda de la literatura utilizando encabezamientos de temas médicos frente a palabras de texto con PubMed" (PDF) . The Laryngoscope . 116 (2): 336–40. doi : 10.1097/01.mlg.0000195371.72887.a2 . PMID 16467730. S2CID 42510351. Consultado el 11 de septiembre de 2018 .
- ^ ab Fatehi F, Gray LC, Wootton R (marzo de 2014). "Cómo mejorar sus búsquedas en PubMed/MEDLINE: 3. Búsqueda avanzada, MeSH y My NCBI". Revista de telemedicina y teleasistencia . 20 (2): 102–12. doi :10.1177/1357633X13519036. PMID 24614997. S2CID 9948223.
- ^ "Mi ayuda en el NCBI". Mi NCBI explicado. NCBI. 13 de diciembre de 2010. Archivado desde el original el 26 de julio de 2023. Consultado el 8 de septiembre de 2017 .
- ^ "PubMed Cubby". Boletín técnico . Biblioteca Nacional de Medicina de los Estados Unidos. 2000. Archivado desde el original el 20 de febrero de 2023 . Consultado el 7 de abril de 2016 .
- ^ "Descripción general de LinkOut". NCBI. 2010. Archivado desde el original el 10 de septiembre de 2023. Consultado el 8 de septiembre de 2017 .
- ^ "Participantes de LinkOut 2011". NCBI. 2011. Archivado desde el original el 14 de octubre de 2017. Consultado el 8 de septiembre de 2017 .
- ^ "Ya está en camino una versión actualizada de PubMed". Archivado desde el original el 16 de mayo de 2023. Consultado el 1 de abril de 2019 .
- ^ PubMed Commons Team (17 de diciembre de 2015). «Commenting on PubMed: A Successful Pilot». Archivado desde el original el 25 de octubre de 2017. Consultado el 29 de julio de 2016 .
- ^ "PubMed Commons dejará de funcionar". NCBI Insights . 1 de febrero de 2018. Archivado desde el original el 28 de agosto de 2023 . Consultado el 2 de febrero de 2018 .
- ^ "PubMed cierra su función de comentarios, PubMed Commons". Retraction Watch . 2 de febrero de 2018. Archivado desde el original el 28 de junio de 2022 . Consultado el 2 de febrero de 2018 .
- ^ "askMedline". NCBI. 2005. Archivado desde el original el 17 de julio de 2013. Consultado el 3 de abril de 2011 .
- ^ "Descripciones y etiquetas de campos de búsqueda". Centro Nacional de Información Biotecnológica. Archivado desde el original el 11 de julio de 2013. Consultado el 15 de julio de 2013 .
- ^ Keener M. "PMID vs. PMCID: ¿Cuál es la diferencia?" (PDF) . Universidad de Chicago. Archivado desde el original (PDF) el 6 de julio de 2014. Consultado el 19 de enero de 2014 .
- ^ "Leasing journal citations from PubMed/Medline". NLM. 2011. Archivado desde el original el 9 de julio de 2023. Consultado el 7 de abril de 2016 .
- ^ abc Lu Z (2011). "PubMed y más allá: una encuesta sobre herramientas web para buscar literatura biomédica". Base de datos . 2011 : baq036. doi :10.1093/database/baq036. PMC 3025693 . PMID 21245076.
- ^ Fontaine JF, Barbosa-Silva A, Schaefer M, Huska MR, Muro EM, Andrade-Navarro MA (julio de 2009). "MedlineRanker: clasificación flexible de la literatura biomédica". Nucleic Acids Research . 37 (número del servidor web): W141-6. doi :10.1093/nar/gkp353. PMC 2703945 . PMID 19429696.
- ^ Estados DJ, Ade AS, Wright ZC, Bookvich AV, Athey BD (abril de 2009). "Herramienta de búsqueda adaptativa en pubMed MiSearch". Bioinformática . 25 (7): 974–6. doi :10.1093/bioinformatics/btn033. PMC 2660869 . PMID 18326507.
- ^ Smalheiser NR, Zhou W, Torvik VI (febrero de 2008). "Anne O'Tate: una herramienta para respaldar la síntesis, el análisis detallado y la exploración de los resultados de búsqueda de PubMed impulsados por el usuario". Journal of Biomedical Discovery and Collaboration . 3 : 2. doi : 10.1186/1747-5333-3-2 . PMC 2276193 . PMID 18279519.
- ^ "ClusterMed". Motor de agrupamiento Vivisimo. 2011. Archivado desde el original el 11 de agosto de 2011. Consultado el 3 de julio de 2011 .
- ^ Rebholz-Schuhmann D, Kirsch H, Arregui M, Gaudan S, Riethoven M, Stoehr P (enero de 2007). "EBIMed: análisis de texto para recopilar datos sobre proteínas de Medline". Bioinformática . 23 (2): e237-44. doi : 10.1093/bioinformatics/btl302 . PMID 17237098.
- ^ Kim JJ, Pezik P, Rebholz-Schuhmann D (junio de 2008). "MedEvi: recuperando evidencia textual de relaciones entre conceptos biomédicos de Medline". Bioinformática . 24 (11): 1410–2. doi :10.1093/bioinformatics/btn117. PMC 2387223 . PMID 18400773.
- ^ Fontelo P, Liu F, Ackerman M, Schardt CM, Keitz SA (2006). "askMEDLINE: un informe sobre una experiencia de un año". Actas del Simposio Anual de la AMIA. Simposio de la AMIA . 2006 : 923. PMC 1839379. PMID 17238542 .
- ^ Fontelo P, Liu F, Ackerman M (2005). "MeSH Speller + askMEDLINE: autocompleta los términos MeSH y luego realiza búsquedas en MEDLINE/PubMed mediante consultas de texto libre en lenguaje natural". Actas del Simposio Anual de la AMIA. Simposio de la AMIA . 2005 : 957. PMC 1513542. PMID 16779244 .
- ^ Fontelo P, Liu F, Leon S, Anne A, Ackerman M (2007). "PICO Linguist y BabelMeSH: desarrollo y evaluación parcial de herramientas de búsqueda multilingüe basadas en evidencia para MEDLINE/PubMed". Estudios en Tecnología e Informática de la Salud . 129 (Pt 1): 817–21. PMID 17911830. Archivado desde el original el 18 de octubre de 2023. Consultado el 31 de mayo de 2014 .
- ^ Hokamp K, Wolfe KH (julio de 2004). "PubCrawler: a la par de PubMed y GenBank". Nucleic Acids Research . 32 (edición del servidor web): W16-9. doi :10.1093/nar/gkh453. PMC 441591 . PMID 15215341.
- ^ Eric Sayers, PhD (24 de octubre de 2018). The E-utilities In-Depth: Parameters, Syntax and More [Las utilidades electrónicas en profundidad: parámetros, sintaxis y más]. NCBI. Archivado desde el original el 23 de junio de 2023. Consultado el 8 de septiembre de 2017 .
- ^ Singhal K, Azizi S, Tu T, Mahdavi SS, Wei J, Chung HW, et al. (3 de agosto de 2023). "Los modelos de lenguaje de gran tamaño codifican el conocimiento clínico". Nature . 620 (7972): 172–180. arXiv : 2212.13138 . Bibcode :2023Natur.620..172S. doi :10.1038/s41586-023-06291-2. PMC 10396962 . PMID 37438534.
- ^ MEDOC en GitHub
- ^ Denise Wolfe (7 de abril de 2020). «SUNY negocia un nuevo acuerdo modificado con Elsevier – Libraries News Center University at Buffalo Libraries». library.buffalo.edu . Universidad de Buffalo . Archivado desde el original el 6 de diciembre de 2020 . Consultado el 18 de abril de 2020 .
Enlaces externos
- Sitio web oficial
- Etiquetas de búsqueda y calificadores de campo de PubMed
[[Categoría:Biblioteca Nacional de Medicina de los Estados Unidos|PubMed]

